Note: Descriptions are shown in the official language in which they were submitted.
~ WO 96/01324 2 1 9 4 1 5 ~ /rl ~
Nouveau site interne d'entrée des ribosomes. vecteur le cr~nî~nam et utili~o~ir~n
L~ d~ULiU,UÇ
15 La présente invention concerne un fragment d~ADN isolé d~un ~G~ ls~ et
~Jn~ Glldlll un site interne d'entrée des ribosomes (IRES). Plus particulièrement,
elle concerne des vecteurs d'expression comportant ce fragment d'ADN et
notamment des vecteurs POI~/U;~LI~1n;qUGS permenant l'expression efficace et stable
de plusieurs gènes d'intéret sous la dépendance d'un même promoteur. La présente20 invention trouve une application hlLG~G~..LG dans le domaine des vecteurs de
thérapie génique.
La faisabililé de la thérapie génique appliquée à l'homme n'est plus à démontreret ceci concerne de nombreuses appli-~a~ n~ thérapeutiques comme les maladies
25 génétiques, les maladies hlrGuLiGusG~ et les cancers. De nombreux documents de
l'art antérieur décrivent les moyens de menre en oeuvre une thérapie génique,
notamment par l'intermédiaire de vecteurs viraux. D'une manière générale, les
vecteurs sont obtenus par délétion d'au moins une partie des gènes viraux qui sont
remplacés par les gènes d'intérét th.~dlJGuLique. De tels vecteurs peuvent être
30 propagés dans une lignée de complémentation qui fournit en trans les fonctions
virales délétées pour générer une particule de vecteur viral défective pour la
réplication mais capable d infecter une cellule hote. A ce jour, les vecteurs
rétroviraux sont parmi les plus utilisés mais on peut citer également des vecteurs
issus des adénovirus, virus associés aux adénovirus, poxvirus et virus de l'herpès.
35 Ce type de vecteur, leur organisation et leur mode d'infection sont largement
Wo 96101324 ~ l 9 4 1 5 5 r~l" lv ~ ~
-- 2 --~ = =
décrits dans la littérature accessible à l'homme de l'art.
Il peut être avantageux de disposer de vecteurs de thérapie génique plus
p~,~rv~ a,-l.~ et capables notamment de produire Grriua~c.,l~ plusieurs protéines
5 d'intérêt. Cependant, Ia présence de plusieurs ,.nvllluLGu,.~ au sein du même vecteur
se traduit très souvent par une réduction voire meme une perte de l'expression au
cours du temps. Ceci est du à un phénomène bien connu d'illlGlrGlGlll_G entre les
séquences IJIVUIVIIiUG~. Dans ce contexte, la publication de la demande
,,,t~.,.~li.,,,~l(' W093/03143 propose une solution à ce problème qui consiste à10 mettre en oeuvre un site interne d'entrée des ribosomes (IRES). Elle décrit un
vecteur rétroviral ~ ;q ,~ pour I expression de deux gènes d'intérêt placés sous
le controle du même promoteur. La présence d'un site IRES de IUiUVllldVil u~ entre
ceux-ci permet la production du produit d'expression issu du second gène d'intérêt
par initiation interne de la traduction de l'ARNm dicistronique.
N~rm~lPmf~nî. l'entrée des ribosomes au niveau de l'ARN messager se fait par la
ooiffe située à l'extrémité 5' de l'ensemble des ARNm eucaryotes. Cependant cette
règle universelle connaît des exceptions. L'absence de coiffe chez certains ARNmviraux laissait supposer l'existence de structures alternatives permertant l'entrée
20 des ribo~somes à un site interne de ces ARN, A ce jour, un certain nombre de ces
structures, nommées IRES du fait de leur fonction, ont été identifiées dans la
région 5' non codante des ARNm viraux non coiffés comme celle notamment des
~ ulllavilu:~ tel que le virus de la poliomyélite (Pelletier et al., 1988, Mol. Cell.
Biol., (~, 1103-1112) et l'EMCV (Fnl~rhz~ yvcclldili~ virus (Jang et al., J.
25 Virol., 1988, 62, 2636-2643).
On a maintenant trouvé un nouveau site interne d'entrée des ribosomes dans les
IGO1VI~all:~JV~UIIS murins de type VL30 et montré que ce site améliore la traduction
des séquence~s codantes placées à sa suite.
Le génome des cellules eucar~ otes comprend un certain nombre d'éléments
génétiques cellulaires mobiles, appelés lla,l~luv5u"5, qui ont la capacité de sedéplacer d'un site du génome à un autre choisi au hasard. A l'heure actuelle, onignore leurs fonction et signification biologique. Certains d'entre eux, les
35 léllvllall~ vlli, a,ul~dlab~Glll apparèntes aux~provirus rétroviraux par leur
~ wo 96101324 2 1 9 4 ~ 5 5 1 ~l~r
ui~;c~ Liull el leur mode de l~ uu~;liw~ (par un ill:~,.llléd;dilt: ARN, LLall~ulil"iu..
inverse et inlégratiOn dans le génome cellulaire). Parmi les différents
l~Llul~dn~usul's murins identifiés à ce jour, figurent les éléments VL30. Le
genome murin en comprend de 150 a 200 copies. Ils ont une longueur d'environ
5 6 kb et po.ssèdenl à leurs extrémités des répétitions directes qui rappellent les LTRs
rétroviraux. lls sont défectifs pour la réplication et ne nnnti~nn~nt pas de séquences
codantes (codons d'arrêt de la traduction dans les différentes phases de lecture).
Ces éléments en tant que tels sont décrits dans la littérature et leur séquence
n~ Pntir~ lP coMue (Adams et al., 1988, Mol. Cell. Biol., 8, 2989-2998; Van
10 Beveren, Coffn et Hughes 1984. Restriction analysis of hvO genomes and
re'striction maps of ~u~ lLdlive retroviral proviruses and cellular oncogenes
p.559-1209, ed: Weiss, Teich, Varrnus et Coffin; RNA tumor viruses, 2nd ed.
Cold Spring Harbor Laborator~). Par ailleurs, ils peuvent etre transmis d'une
cellule à l'autre par ~n~p~ tinn en presence d'un virus auxiliaire (helper).
Il n'était pas évident d'identifier une séquence IRES dans les éléments VL30
murins puisque ceux-ci sont dépourvus de séquences codant pour des protéines et
ne presentent pas d'homologie frappante de séquence evec les sites déja décrits
dans la littérature. De plus, par rapport à ces derniers, le site IRES d'un VL3020 murin est particulierement avantageux. En premier lieu, il permet un taux de
réinitiation de la traduction efficace et stable à long terme et, d'autre part et de
manière inattendue, il peut égalemenl, dans le cadre d un vecteur rétroviral,
remplir les fonctions de dillla.i:~dLiUII et d ~ A~ et ceci i~ p ~ 1 de
sa position dans le vecteur. Et enfin, du fait de sa faible homologie avec les
25 séquences rétrovirales, son emploi réduit considérablement le risque de production
de virus competents pour la réplication, propriété avantageuse dans le cadre desvecteurs de thérapie génique destinés à un usage humain.
C'est pourquoi la présente invention a pour objet un fragment d'ADN isolé
30 ~Olllult:llallL un site interne d entrée des ribosomes (IRES) et/ou une séquence
d'..". Allci~ l, caractérisé en ce qu'il est dérivé d'un IGnVIl~ )USUll.
~ Par fragment d'ADN isolé, on entend un fragment d'ADN isolé de son contexte
c'est à dire non as.socié à une autre séquence de l~llull~ Ju~ull autre que celle
35 définie ci-dessous. Le terme 'h~lluLldll~lJùsun~ se rapporte à un élément genétique
WO96/01324 21 94 ~ 5~ P~/r~
-- 4 --
cellulaire mobile qui présente des ~ el i~liques de type rétroviral, notamment par
l'existence de répét;tion.s directes à ses deux extrémités. Par IRES, on désigne une G
séquence capabte de ~ lUU~Jil l'entrée des ribosomes dans une moiécule d'ARN
d'une manière i~ rp~ r. de la coiffe, au niveau d'un site interne de cet ARN.
5 Une séquence d'~l,c,l,~;d~ nl, est une séquence impliquée dans le processus
d~f" ~ n des rétrovirus ou vecteurs rétroviraux en favorisant la dimérisation
de deux copies du génome rétroviral et en permettant 1'.~ ir"\ du dimère
dans les particules virales. Le terme ''dérivé" fait référence à une structure ayant
une origine li:llJllG~ wl mais qui peut avoir subi quelque~s modifir~t;on5, avoir
10 été obtenue par synthèse chimique ou encore d'éléments divers Cul~ -.,-ll des parties de ICIll~lcll:~,UUS~.1n, comme le virus HaMSV.
Selon un mode de réalisation préfére, un fragment d'ADN selon l'invention est
capable d'exercer une fonction IRES et une fonction d'f, - ~ lorsqu'il est
15 introduit dans un vecteur rétroviral approprié.
Dans le cadre de la présente invention, un fragment d'ADN selon l'invention est
isole de l'extrémité 5' d'un .~ u~ll et notamment de la région qui suit
directement la répétition directe située à son extrémité 5' (LTR-like 5') et, en20 particulier, le site de liaison à l'ARN transfert (Pi3S). Il va sans dire qu'ii peut être
isolé par toute technique en usage dans le domaine de l'art, par exemple par
clonage à l'aide de sondes appropriées, par PCR (Polymerase Chain reaction) ou
encore synthèse chimique. Aux funs de la présente invention, il comprend au moins
100 nucleotides de ladite région, a~r~ us~ nl au moins 200 nucléotides, de
25 préférence au moins 300 nucléotides, de manière préférée au moins 400 nucléotides
et, de manière tout à fait préférée, au moins 550 nucléotides, ceci en comptant les
nucléotides hors la répétition directe en 5'. Mais, bien entendu, il peut s'étendre
au delà dans la direction 3' jusqu'à au plus 0,88 à 1,5 kb.
30 Selon un mode de réalisation tout à fait préféré, un fragment d'ADN selon
l'invention, est dérivé d'un élément VL30 de rongeur, de préférence, d'origine
murine et, tout particulièrement, de rat ou de souris. S'agissant de la premièrevariante selon laquelle un fragment d'ADN est isolé d un VL30 de rat, on préfèretout particulièrement mettre en oeuvre un fragment d'ADN ayant une séquence
35 5nh~t:mtiellfmfn~ homologue à la séquence pré~sentée dans l'identificateur de
~ wo 96/01324 2 1 q IJ'r 1 rJ 5 1~ l Ir~S.~ I
séquence SEQ ID NO: 1, ~'(IIIIIII..I~AIII au nucléotide I et se terminant au
nucleotide 590 ou, de manière optionnelle, ~ -~ au nucléotide 176 et se
terminant au nucléotide 590.
5 Selon la deuxième variante (fragment d'ADN isole d'un VL30 de souris), on aura de préférence recours à un fragment d'ADN présentant une séquence
substantiellement homologue à la séquence présentée dans l'iflPntifif ~tPlln de
séquence SEQ ID NO: 2, ~ ~ SA"I au nucléotide 1 et se terminant au
nucléotide 788.
Le terme snhct~ntiPllpmpnt homologue fait référence à un degré d'homologie
supérieur à 70%, aV~nt~gpllcpmpnt supérieur à 80%, de préférence supérieur à
905to et, de manière tout à fait préférée, supérieur a 95%. Ainsi, un fragment
d'ADN selon l'invention peut avoir une séquence légèrement différente d'une des
15 séquences décrites dans les idc.lLiri~Ltu!a de séquence I et 2, à la condition
toutefois que la cllhctih~tion, la délétion ou l'addition d'un ou plusieurs nucléotides
n'affecte pas sa fonction IRES et/ou la fonction d'~".A~ ,-",
D'une manière générale, un fragment d'ADN selon l'invention est destiné à être
20 intégré (dans une orientation qnP~ n~lllp~ dans un vecteur de transfert et
d'expression d'un ou plusieurs gène(s) d'intéret Le choix d'un tel vecteur est large
et les techniques de clonage dans le vecteur retenu sont à la portée de l'homme de
l art Conformement aux buts poursuivis par la présente invention, on peut
envisager un vecteur IJlaa~ ur ou un vecteur dérivé d'un virus animal et, en
25 particulier, d'un poxvirus (canari pox ou virus de la vaccine), adénovirus,
baculovirus, virus de l'herpes, virus associé à un adénovirus ou retrovirus. De tels
vecteurs sont largement décrits dans la littérature En particulier, lorsqu'il s'agit
d'un vecteur adénoviral, celui-ci peut être issu d'un adénovirus humain, de
préférence de t~pe 2 ou 5, animal, de préférence canin ou aviaire, ou encore d'un
30 hybride entre des espèces variées La technologie générale concernant les
adénovirus est divulguée dans Graham et Prevec (1991, Methods in Molecular
Biology, Vol 7, Gene transfer and Expression Protocols; Ed E J. Murray, the
human Press Inc, 109-118).
35 Dans le cadre de la présente invention, un fragment d'ADN selon l'invention est
WO96101324 21 9~1 55 r~llr~~
de préférence positionné en amont d'un gène d'interet pour améliorer la traduction
du produit d'expression pour lequel celui-ci code. Il peut être inclus dans une
cassette d'expression de t~pe monocistronique (pour l'expression d'un gène
d'intérêt placé sous le contrôle d'un promoteur) ou puly~iallull;qut~ (pour
5 I'expression d'au moins deux gènes d intéret placés sous le contrôle d'un mêmepromoteur). Cene dernière peut contenir plusieurs éléments en tandem "site IRES-gène d'intérêt" dont au moins un de~s sites IRES e~st constitué par un fragment
d'ADN selon l'invention. On préfère tout ,Udl Licu~ nlc.ll le mettre en oeuvre dans
une cassette iiu;allul~iu,u~ dans laquelle il peut être inséré soit en amont du premier
I0 gène d'intérêt soit en amont du second, cette dernière variante étant la préférée.
Lorsqu'un vecteur selon l'invention comprend plu.sieurs cassettes d'expression,
oelles-ci peuvent être insérées dans une orientation quelconque les unes par rapport
aux autres: dans une même orientation (promoteur agissant dans une même
15 direction) ou en orientation réverse (prom~oteur agissant dans une orientation
opposée) .
Dans le cas où un vecteur selon l'invention comprend plusieurs fragments d'ADN
selon l'invention, il est préférable qu'ils soient issus de l~llul~llalJusu,.~ d'origines
20 différentes. Selon ce mode de réalisation particulier, on préfère qu'un des
fragments soit dérivé d'un VL30 de rat et présente notamment une séquence
5~1h5t~ntiPIlPment homologue à la SEQ ID NO: l et que l'autre soit dérivé d'un
VL30 de~souris et présente notamment une séquence ~l~h5t~n~iPliPmPnt homologue
à la SEQ ID NO: 2.
Selon un mode de réalisation tout à fait préféré, un vecteur selon l'invention dérive
d'un rétrovirus. On peut citer à titre d exemples, les retrovirus aviaires tels que le
virus de l'~-yllllubldaiuac aviaire (AEV), le virus de la leucémie aviaire (AVL), le
virus du sarcome aviaire (ASV), le virus de la nécrose de la rate (SNV) et le virus
30 du sarcome de Rous (RSV), les retrovirus bovins, les rétrovirus félins, les
rétrovirus murins tels que le virus de la leucemie murine (MuLV), le virus de
Friend (F-MLV) et le virus du sarcome murin (MSV) et les rétrovirus de primate
Bien entendu, d'autres rétrovirus peuvent être mis en oeuvre. Cependant, on
préfère tout particulièrement avoir recours au virus de la leucémie murine de
35 Molone~ (MoMuLV). Les nombreux vecteurs rétroviraux dérivés de ce dernier qui
21 94~ 55
wo 96iOI324 . ~I/r~ . I
sont décrit.s dans la littérature, notamment le vecteur N2 ou un de ses dérivés
peuvent être utilisé.s dans le cadre de la présente invention.
Les vecteurs rétroviraux e"~ ables aux fins de la présente invention sont
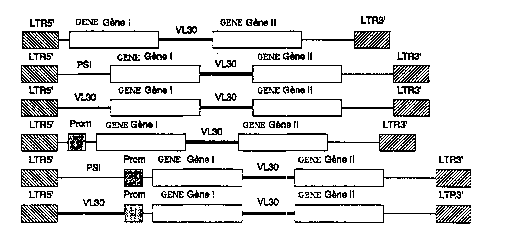
5 ~ dans la Figure I (a, b et c). Bien entendu, ces exemples ne sont pas
limitatifs. Pour plus de clané, les LTRs 5 et 3'rétroviraux sont représentés par une
boite hachuree, les séquences de VL30 murin (hldirr~l.e~ "" souris et/ou rat) par
un trait gras, le promoteur interne par une boîte pointillée, les gènes d'intérêt par
une boite blanche et enfin la région d'~ - (Psi) rétrovirale par un trait
10 fin. Comme cela est illu.stré, le LTR 5' rétroviral peut être utilisé comme
promoteur pour l'expression d'un ou plusieurs gène(s) d'intérêt mais on peut
également avoir recours à un promoteur interne. D'autre pan, un vecteur rétoviral
selon l'invention peut, de facon optionnelle, componer une région d'r~nr~p~iri~rirm
rétrovirale comme la séquence Psi du MoMuLV. Cependant, la présence de cette
15 dernière n'est pDS exigée dans la mesure où un fragment d'ADN selon l'invention
peut également remplir cette fonction et ceci quelle que soit sa position dans le
vecteur rétroviral de l'invention (en amont d'un gène d'intérêt et/ou en aval duLTR 5').
20 Aux fins de la présente invention, un gène d'intérêt en usage dans l'invention peut
être obtenu d'un organisme eucaryote, procaryote ou d'un virus par toute technique
,nnclle. Il est, de préférence, capDble de produire un produit d'expression
ayant un effet thérapeutique et il peut s'agir d'un produit homologue à la cellule
hôte ou, de manière alternative, hétérologue. Le terme produit d'expression désigne
25 une protéine ou un fragment de celle ci. Dans le cadre de la présente invention, un
gène d'intéret peut coder pour un produit (i) intracellulaire (ii) Ill~llblrlllrl;le
présent à la surface de la cellule hote ou (iii) secrété hors de la celiule hote. Il peut
donc ~ le~dle des éléments ~rl.l;li..".,. 1~ appropriés comme, par exemple, une
séquence codant pour un signal de sécretion. Ces signaux sont connus de l'homme
30 de l'an.
G~ L aux buts poursuivis par la présente invention, un gène d'intérêt peutcoder pour une protéine ~:ul~c~JvndallL a tout ou panie d'une protéine native telle
que trouvée dans la nature. Il peut également s'agir d'une protéine r hinnr~riri-,r~ par
35 exemp1e provenant de la fusion de polypeptide d'origines diverses ou d'un mutant
Wo 96/01324 ~ 1 9 ~ i 5 P l/r~
présentant des propriétés biolo=iques améliorées et/ou modifiées. Un tel mutant
peut etre obtenu par des techniques de biologie classiques par sllhctinltinn~ délétion
et/ou addition d'un ou plusieurs résidus acides aminés.
5 On préfère IOut particulièrement mettre en oeuvre un gène d'intérêt Lh~di)t~uli~ut~
codant pour un produit d'expression capable d'inhiber ou retarder l~h~hliccpmpntet/ou le de-eloppement d'une maladie génétique ou acquise. Un vecteur selon
l'invention est particulièrement destiné à la prévention ou au traitement de la
mucoviscidose. de l'hémophilie A ou B, de la myopathie de Duchenne ou de
10 Becker, du cancer, du SIDA et d autres bactéries ou maladies infectieuses dues à
un organisme pathogène: virus, bactérie, parasite ou prion. Les gènes d'intéret
utili.sable.s dans la présente invention, sont ceux qui codent pour les protéines
suivantes:
i
15 - une cytokine et notamment une hlL~ lLlle, un interféron, un facteur de
nécro.se tissulaire et un facteur de croissance et notamment h~ lue
(G-CSF, GM-CSF),
- un facteur ou cofacteur impliqué dans la l-n~enl ~tinn et notamment le
facteur VIII, le facteur IX, le facteur von ~Villebrand, l'~lllill.l-,llll,ille III,
la protéine C, la thrombine et l'hirudine,
- une enzyme et notamment la trypsine, une rihnmlrlP~cP et la B-
g~lzlrtnci~l~ce,
- un inhibiteur d'enzyme tel que l'c~ u~ y,.,sille et les inhibiteurs de
protéases virales
- un produit d'expression d'un gène suicide comme la thymidine kinase du
viru~s HSV (virus de l'herpes) de type 1,
- un activateur ou un inhibiteur de canaux ioniques,
- une protéine dont l'absence, la modification ou la dérégulation de
l'expression est responsable d'une maladie génétique, telle que la protéine
~Iwo96/01324 21 9~1 5~ r~s~ f
Ci~TR, la dyaLlu~ le ou minidy~llu,~ ille~ l'insuline, I'ADA (adénosine
diaminose), la ~luuucéléblua;-laae et la phénylhydlu~yla~>e~
- une protéine capable d'inhiber l'initiation ou la progression de cancers, telie
qu'un produit d'expression de gène~s su~Jle~aeuls de tumeurs (gènes p53,
Rb...), une toxine, un anticorps, une ;Imnu~ lP,
- une protéine capable de stimuler une réponse h~ unildi~e~ et
10 - une protéine capable d'inhiber une infection virale ou son dévelu~
par exemple un épitope antigénique du virus en cause, un anticorps ou un
variant altéré d'une protéine virale susceptible d'entrer en l ~ ;ul~ avec
la protéine virale native.
15 Par ailleurs, un gène d'intérét en usage dans la présente invention, peut également
coder pour un marqueur de sélection permettant de 5PIPrtio~lnPr ou identifier les
cellules hotes ~la"are~lér~a par un vecteur selon l'invention. On peut citer le gène
nco (néomycine) conferrant une résistance à l':~ntiT-iotiq~P G418, le gène dhf~
(dihydrofolate reductase), le gène CAT (Chlu~ l Acethyl Transférase) ou
20 encore le gène~g~tf (xanthine phosphoribosyl).
D'une manière génerale, on aura recours pour I expre~ssion d'un ou des gène(s)
d'intérêt à un promoteur fonctionnel dans la cellule hôte considérée et, de
préférence, une cellule humaine. Le choix du promoteur est très large et à la portée
25 de l'homme du métier. Il peut s'agir d'un promoteur gouvernant naturellement
l'expression d'un gène d'intérêt en usage dans la présente invention ou de tout
autre promoteur (d'origine eucaryote ou viral). Par ailleurs, il peut être de nature
ubiquitaire ou regulable, notamment en réponse à certains signaux cellulaires tissu-
spécifiques ou é~énements-spécifiques. A titre indicatif, il peut être avantageux de
30 mettre en oeuvre un promoteur tissu-spécifique lorsque l'on veut cibler l'expression
du ou des gène(s) d'intéret dans une cellule ou un type cellulaire particulier, par
exemple les Iymphocytes dans le cadre du SIDA, les cellules pulmonaires dans le
cadre de la mlluu~ i.iui iu>e ou les cellules musculaires dans le cadre des
myopathies .
WO96/01324 2 1 9 4 1 5 5 ~I/rl C
- 10 -
A titres d'exemples non limitatifs, on peut citer notamment les l,luluu;~ SV40
(Virus Simian 40), HMG (Hydroxyméthyl-Glutaryl Coenzyme A), TK (Thymidine
kinase), les LTR.s rétroviraux et, en particulier, celui du MoMuLV ou du MSV
lorsqu'on met en oeuvre un vecteur retoviral, le promoteur tardif MPL (Major Late
5 Promoteur) d'adénovirus de type 2 notamment dans le contexte d'un vecteur
adénoviral, les promoteurs 7,5K et H5R notamment destinés à des vecteurs dérivésde poxvirus et surtout du virus de la vaccine, le promoteur PGK (Phosphoglycéro
l;inase), les promoteurs foie-spécifiques des gènes codant pour P~ ,Liily~ le~
le facteur IX, l'albumine et la l~ f~ e~ les l,luuwl~ ul~ des gènes
10 d'immnnnglnb~lin~ qui permettent une expression dans les lY"~ UUYL~ et enfin
les plUII10;~..11~ des gènes codant pour le surfactant ou la protéine CFTR qui
présentent une certaine spécificité pour les tissus pulmonaires.
Par ailleurs, une cassette d'expression pré~sente dans un vecteur selon l'invention
15 peut componer d autres séquences nécessaires à l'expression du ou des gène(s)d'intéret, tant au niveau de la transcription que de la traduction; par exemple des
séquences activatrices de la llr~ ul;~Jliun de type enhancer, des introns, des signaux
de terminaison de la transcription et, comme indiqué LJl&~ "", lh un signal de
sécrétion.
L'invention oouvre également les virus et particules virales obtenus par Llull~r~Liu~
du vecteur ~iral selon l'invention dans une lignée de ~(,",l.l&.,.~ inn adéquate.
Selon le type de vecteur viral utilisé, l'homme du métier connalt les lignées dernmplPmr~ntatinn pouvant etre employées pour générer des particules virales
25 infectieuses ainsi que le procédé à mettre en oeuvre. S'agissant d'un vecteuradénoviral, on peut avoir recours à la lignée 293 (Graham et al., 1977, J. Gen.
Virol., 36, 59-72).
Dans le cadre d'un vecteur rétroviral selon l'invention, on peut envisager
30 d'employer de~s lignées cellulaires éuuLI~ {ues, comme la lignée CRE (Danos et
Mulligan, 1988, Proc. Natl. Acad. Sci. USA, 8~, 6460-6464) ou GP+E-86
(Markosvitz et al., 1988, J. Virol., 62, 1120-1124). Mais on préfère tout
particulièrement mettre en oeuvre une lignée de ~ ,5~ Alinn amphotropique
telle que la lignée PG13 (Miller et al., 1991, J. Virol., 6~, 2220-2224) ou Psi Env-
35 am (Markowitz et al., 1988, T.A.A.P. Vol. Cl, 212-218). Généralement, on
~ WO96101324 2 1 9 4 1 ~ 5 r~l/r~- - .
récupere les particules virales infectieuses dans le surnageant de culture des cellules
de cu~-lpl~."~"laiiun ~ .tf,~ par un vecteur rétroviral selon l'invention.
L'invenlion s'étend également aux cellules eucaryotes CUIIIIU~ I un fragment
5 d'ADN selon l'invention. E11es peuvent etre obtenues par infection par des
particuies virales h~rt:~,Li~u~c~ selon l'invention ou par introduction d'un vecteur
pl~cmiffiquP cu viral soit in UitlO (dans une cellule préievée d'un patient ou d'un
animal) soit directement in ~ Les méthodes pour introduire un vecteur dans une
cellule sont UUlli . a i~ Pc On peut mettre en oeuvre la technique de
10 Ivl~ilJiLdlioll au phosphate de calcium, celie au Di-AE dextrane, I'injection directe
du vecteur ou d'une portion de celui-ci dans une cellule ou encore l'i-.." Al,~"lAli",l
dans des molécules de type liposomes. D'autre part, les vecteurs selon l'invention
peuvent être présents dans la cellule hôte soit sous forme intégrée dans le génome
cellulaire ou sous forme épisomale aussi bien dans le noyau que dans le cytoplasme
15 de la cellule. La cellule selon l'invention est aVAntAep~lcpmpnt une cellule
lliAIIIIII;r~l1 et, de préference, une cellule humaine.
La présente in~ention concerne également l'usage thérapeutique d'un vecteur ou
d'une cellule selon l'invention, pour la préparation d'une ~
20 pha~ .uill~lP destinee au traitement et/ou à la prévention d'une maladie
génétique ou d'une maladie acquise comme le cancer ou une maladie infectieuse.
Cependant, un tel usage n'est pas limité à une application de type therapie génique
somatique. En particulier, un ~ecteur selon l'in-ention peut etre utilisé à d'autres
fins comme la production par voie rerrmhin~-ltP dans des cellules eucaryotes de
25 produits d'expression destinés à être inclus après L L~i;ri~Lùn dans ladite
r~mrr~citii~n ~ ;q~P
L'invention s'adresse egalement à une ~",,,I,..~ili,", ~hAI~ Culll~lr,ll,~lll à
titre d'agent therapeutique ou prophylactique un vecteur ou une cellule selon
30 I'invention, en association a~ec un véhicule acceptable d'un point de vue
~IIAI ",~ I;r~
Une l,umpc~ iull pharmaceutique selon I invention peut etre fabriquée de manièrec~,ll.~,.-lio~ lle~ En particulier, on associe une quantité IhelAI~ lim llrla efficace
35 d'un tel agent a un support, un diluant ou un adjuvant acceptable. Elle peut être
-
Wo 96i01324 2 1 9 4 1 5 5 . ~l/rl~
- 12 -
administrée selon n'importe quelle route d dd~ ,L-dLion et ceci en dose unique ou
répétee après un certain délai d intervalle. La quantité à administrer sera choisie
en fonction de différents critères, en particulier lrusage a titre de traitement ou de
vaccin, la voie d'a.l"~ dLiu", le patient, le type de maladie à traiter et son état
5 d'évolution, la durée du traitement....etc. A titre indicatif, une ~..,..l...~il;..,.
yhc~ Liuue selon l'im~ention comprend entre IOJ et 10'' pfu (unité formant des
plages), d~ n~ .a entre lOi et 101l pfu et, de préférence, entre 106 et 10
pfu de particules virales.
10 Par ailleurs, I'invention concerne une méthode de traitement de maladies
genétiques, cancers et maladie.s infectieuses selon laquelle on administre une
quantité thér~ tiq~ m~nt effcace d'un vecteur ou d'une cellule selon l'inventionà un patient ayant besoin d'un tel traitement. Selon un premier protocole
thérapeutique, on peut les administrer dheut~.,ll~,.lL ill vivo, par exemple par15 injection iulldveillel~e, h~L~du~usuulllhe ou par .~0,v~uli~dLivn dans les poumons. De
manière alternative, on peut adopter un protocole de thérapie génique e~x vivo qui
oonsiste à prélever les cellules d'un patient, cellules souches de la moelle osseuse
oU Iyu~lJllOu~Les du sang l.é,i,.,l.e.ique, à les transfecter avec un vecteur selon
l'invention et à les cultiver i/l VitlO avant de les ,éi,.,Lhl"le, au patient.
L'im~ention concerne également un fragment d'ADN isolé comportant une séquence
d'e.,ual)si.ldLiull (Psi) dérivée d'un virus de la leucémie murine de Moloney
(MoMuLV) à titre de site interne d'entrée des nbosomes (IRES) et sa mise en
oeuvre dans le cadre de vecteurs d expression tels que ceux mentionnés
25 p~ lP,."~ et, en particulier, de vecteurs rétroviraux 11 s'agira d'un vecteurpolycistronique .:ul-,L,,e"d"L, de préférence, deux gènes d'intérêt sous le contrôle
d'un même promoteur et présentant une séquence Psi de MoMuLV entre les deux
gènes Le vecteur peut inclure des gènes d'intérêt ~L~yl~n~ es. soit sous la
forme d'une cassette polycistronique (plusieurs éléments en tandem "IRES-gene
30 d'intérêt" dont au moins un IRES est constitué par une sequence Psi dérivant du
MoMuLV), soit sous la forme d'une cassette ;1ll'~ pourvue de son propre
promoteur. Bien entendu, I'invention couvre également les particules virales et les
oellules eucaryotes comprenant un tel vecteur, leur usage therapeutique ainsi qu'une
l ,l",l~ ,~;li.,ll lJh~l.,l,d~r,uLi Lue. On indique qu'un site IRES selon l'invention est, de
35 préférence, porte par les séquences s'étendant des nucléotides 210 à 1035 du
~ WO96101324 21 9~1 55 r~
génome MoMuLV ( + I "~p~:,cuL~llt le site d'initiation de la ~clls~ ion).
L'invention est illustrée ci-après par référence aux figures suivantes.
5 La Figure ] représente quelques vecteurs rétroviraux utilisables dans le cadre de
l'invention a: de type ~ o.i~LIu~ique~ b: de type dicistronique et c: de type
mixte comprenant des ca.ssettes d'expres.sion mono et dici.~il.,niqu~. Les LTRs 5'
et 3' sont représentés par une boite hachurée, les séquences de VL30 murin (rat
ou souris) par un trait gras, la séquence Psi rétrovirale par un trait fin, le
I0 promoteur interne par une bolte pointillée et enfin les gènes d'intérêt par une bolte
blanche.
La Figure 2 schematise les vecteurs pVL-CT2, pCBTI, pCBT2, pMLV-LacZ+,
pSJE1 et pSJE2.
La Figure 3 schématise les vecteurs pMLV-CBT, pVL-CBT2-Et et pVL-CBT5
(plap représente le gène rhf~Crh-t~c~ alcaline désigné ci-apres PA).
20 EXE~IPLES
Les ~)IL~I UI,~ s ci-après décrites sont réali.sées selon les techniques générales de
génie génétique et de cTonage moléculaire détaillées dans Maniatis et al. (1989,Laboratory Manual, Cold Spring Harbor Laboratory Press, Cold Spring Harbor,
25 NY) ou selon les rer~-mm~n-l~ti-.ns du fabricant lorsqu'on utilise un kit
commercial. Le remplissage des extrémités 5' plvLul,e~ -Lts peut être réalisé à
l'aide du fragment Kleno~v de l'ADN polymérase d'E. coli et la destruction des
extrémités 3' ,ul~JLub~.~llL~a en pré.sence de l'ADN pol)~mérase du phage T4. Les
techniques de PCR sont connues de l'homme de I art et ~ décrites dans
30 PCR Protocols, a guide to methods and rrrlic~ti~ns (Ed: Innis, Gelfand, Sninsky
et White, Academic Press, Inc.).
~ Par ailleurs, la position des séquences du VL30 de rat et de souris est indiquée par
référence à la molécule ARN, la position +I cu.,~pvnd.,..L au premier nucléotide35 de la molécule d'ARN, c'est à dire au site d'initiation de la Llcll~ Li~,n dans la
Wo 96/ot324 2 1 9 4 1 5 5 P~l/r~. , ~
- 14 -
molécule ADN (premier nucléotide de la séquence R). En ce qui concerne les
sequence~s VL30 de souris, les positions ARN 362-575 et 362-1149 ~w-~,onde,.~
Ir ~p~ ellL aux positions ADN 631-844 et 631-1418 de la séquence décrite
dans Adams et al. (]988, Mol. Cell. Biol., ~, 2989-2998)
EXEMPLE 1: Etudes d'expression i/2 ~itrr
I . Constl-ucfiolt des l~ectel~ls d 'ex/7ression
Le vecteur pCB28 est obtenu de la façon suivante:
Le fragment EcoRI-Nhel de pLNPOZ (Adam et al., 1991, J. Virol. 6J, 4985-
4990) est cloné dans le Blue~scr-ipt 11 KS + digére par EcoRI et Spel pour donner
15 pCB25. Le fragment Hindlll-Xbal de pCB25 contenant les séquences codant pour
la néom~cine (néo), le site IRES du poliovirus el le gène B-gal~rt~ci~r~c~ (LacZ)
est cloné dans le vecteur pRc/CMV (Invitrogen) délété des positions 1284 a 3253
et digéré par Hindlll et Xbal. Un fragment portant les séquences virales du virus
F-MLV des positions I a 651 est généré par PCR à l'aide des amorces 6 et 7 ~SEQ
20 ID NO: 3 et 4). On peut utili.ser à titre de matrice une IJlelo~ldLiull d'ADN obtenu
de ce virus par les techniques classiques. Le fragment PCR est digéré par Xhol et
BamHI puis inséré entre les gènes néo et LacZ de pCB27 partiellement digéré par
Xhol et BamHI.
25 Puis on insère entre les gènes néo et LacZ de pCB28 (egalement décrit dans
Perlio~ et Darlix, 1995, J. Virol. 69, 2214-2222) linéarisé par Nhel, une série de
fragments portant des séquence~s VL30 de taille déterrninée, l~al.e.:Li~ L:
- des positions I à 794,
- des positions 205 à 794, et
- des positions 380 à 794.
Les séquences VL30 ont été générees par PCR à partir de la matrice pVL-CG20
(Torrent et a]., 1994, J. Virol. 6,~, 661-667). Il est à la portée de l'homme de l'art
35 de concevoir des amorces ~l-plvlJl;ce5 à partir des données de séquences et
1~ wo 96/013~4 2 l 9 4 1 5 5 t _~/r~ ~
pourvues en outre d'un site Nhel à leurs extrémités (à cet égard les
cligl~n~rl~ot~ c décrits dans les SEQ ID NO: 7 et 8 peuvent convenir pour les
positions 205 à 794). On obtient pVL-D]-7g4, pVL-D205-794 et pVL-D380-794
selon la séquence VL30 insérée.
Dans tou.s ces vecteurs, la cassette dicistronique néo-VL30-LacZ est sous le
contrôle du promoteur de l'ARN po~ eld~e T7 pour l'expression in vitro et du
promoteur précoce du cytomégalovirus (CMV) pour l'e~:pression dans les cellules
eucaryotes. On indique que l'AUG initiateur du gène lacZ est placé dans un
10 contexte favorable à l'initiation de la traduction selon la règle de Kozak (A/G
CCAUGG; Kozal;, 1986, Cell ~, 283-292).
2. Tr~dl~ction ~n ~itr~ des A~Ns dicistl-~niql~cs
15 On génère des ARNs lic;,L-u,,iques par synthèse in vitro à panir de 5 ,ug de
piasmides ~ec~"h;~ a~ pVL linéarisés par Xbal. La réaction de lld~ L;~liclll se
déroule 3 h à 37 C dans 0,1 ml de 40 mM Tris-HCI (pH 7,5), 6 mM MgCI2,
2 mM spermidine, 10 mM diLlliu~ itul, 10 mM NaCl, 0,5 mM de chacun des
rih~n~ cil1l~c L-iluhu,l,]latc en présence de 40 U d'ARN polymérase T7 et 80 U
20 de RNAsine (inhibiteur de RNase). Après un traitement à la DNase (RQ1), les
ARNs sont e~traits au phenol-ul,lu,urulllle et précipités a l'éthanol. Ils sont ensuite
redissous dans de l'eau sterile bidistillée et soumis à une éle~L.ul,:.u.ea~ sur gel
d'agarose pour vénfer leur intégrité (bande unique migrant à la taille attendue de
4,6 à 5 kb selon les plasmides).
Les ARNs dic;aL~u"i~lu~s (10 ~/g d'ARN/ml) sont ensuite traduits in vitro dans un
- système de Iysats de réticulocytes de lapin (système RRL, Proméga) à 50 % de sa
CullCellLldLiull d'origine en présence de 1 mCi [3gS] mt~thirmin~/ml (Amersham)
(temps de reaction Ih à 31 C). Le mélange réactionnel est ~ S en acétate
30 de potassium et en chlorure de potassium à une concentration fnale de 60 mM et
40 mM ~t~ e.8~"-~.,L.
Les tubes sont placés à ]00 C dans 62,5 mM Tris-HCl (pH 6,8), 2 % SDS, 10 %
glycérol, 5 ~o ~3-",~ ,iu3~ ,ol et 0,02 % de bleu de blul--u~ht"ol et les
35 protéines marquées au 358 sont analysées par éle-,L-u,~,h~,,ta~ sur gel de
WO 96/ot324 2 1 9 ~ 1 ~ 5 P~l/r~
- 16 -
polyacrylamide 10 ~o - 0,2 % SDS La quantification des produits de traduction
des gènes néo et LacZ est effectuee par scanning. La protéine néo sert de
d~lu';~aliOll du niveau de traduction et on évalue la quantité de /3 gala,t,.ci,lac,~
synthétisée (PM 1]0 kDa)
Les ARN produits à partir de chacun des vecteurs permettent la synthèse des
produits d'e,~pression des deux gènes (néo et /3 galart--si~laca) ce qui indique qu'ils
sont dici~ J"klu-::, et suggère l'e::istence d'un IRES dans les séquences VL30
mises eh oeuvre, assurant l'initiation de la traduction du second cistron d'où la
10 productjon de /3-gala~t~cl~ace
Lorsqu'on examine la réaction d'une manière ~udllLildli~, le ratio 13gal/néo estsimilaire pour les vecteurs pVL-Dl-794 et pVL-D205-794. Il semble donc que les
séquences 1-205 du VL30 ont peu d'influence sur le ll~f~ -"~ d'initiation de la
15 traduction. De manière illl~r~dnte avec le vecteur pVL-D380-794, le niveau desynthèse de la /3-gala~tl~cirlace est plus élevé. Ces e~l,éliences suggèrent que les
séquences 380 à 794 du VL30 de rat ont la capacité d'initier la traduction d'un
cistron en 3"~ia un mécanisme d'entrée interne des ribosomes.
EXEI~IPLE _: Vecteurs rétrovirau.s comprenant une sequence d'un Vl ~0 de
rat.
l co7Lsrlllct~ dcs I ~CtCIlls dicistl-ol!iqllcs
Le plasmide pCB71 est obtenu de la fa,con suivante:
Un fragment EcoRI-Xbal est isolé du plasmide Clal2-AP et clone entre les mêmes
sites du Bluescript 11 KS + (Stratagène) pour generer pCB70. Ce fragment
comprend le gène de la phosphatase alcaline humaine (PA) décrit dans Part
30 antérieur (~ekete et Cepko, 1993, Mol Cell. Biol., i.7', 2604-2613). L'introduction
de sites de restriction adéquates est à la portée de l'homme du métier.
En parallèle, le gène de la néomS~cine (néo; positions 4 à 844) est amplifié parPCR à partir du vecteur pLNPOZ (Adam et al., 1991, st~pra) et à l'aide des
35 amorces 10 et 11 (SEQ ID NO: 5 et 6)~munies à leurs extrémités de sites de
~ wO g6101L324 2 1 9 4 1 5 ~ r~ I
- 17 -
restriction Sall, Spel et BamHI. Le fragment PCR généré est digéré par Sall et
BamHI et introduit avec le fragment EcoRV-Sall de pCB70 portant le gène de la
pllf~ r alcaline dans le vecteur pLNPOZ digéré par Ball et BamHI.
5 On isole par PCR un fragment d'ADN de 0,59 kb comportant les séquences du
VL30 de rat (positions 205 à 794) et muni à ses extrémités 5' et 3' d'un site Nhel.
On met en oeuvre le piasmide pVL-CG20 (Torrent et al., 1994, s~lpra) à titre de
matrice et les nlignn--rlr-f tiflP~ 12 et 13 reportés dans les SEQ ID NO: 7 et 8. Le
fragment PCR est digéré par Nhel avant d'etre inséré dans le vecteur pCB71 digeré
10 par Spel, pour donner pCBT2. A titre indicatif, on a inclu,s dans l'nli,~ n-lf~",-lr
13 un codon ATG initiateur de la traduction placé dans un contexte favorable de
Kozak (A/GCCATGG) qui permettra ensuite d'introduire une séquence codante
dépourvue de codon initiateur.
15 La ~u~Llu-:Lio~- finale pCBT2 (Figure 2) contient le LTR 5' du MoMuLV, le gène
de la ~ n~ r alcaline humaine, un fragment de 0,59 kb isolé du VL30 de rat
(positions 205 à 794) suivi du gène néom~cine et du LTR 3' du MoMuLV.
La construction du plasmide pCBTI s'effectue de la façon comme suit:
Un fragment d'ADN comportant les séquences VL30 de rat (positions 205 à 379)
est généré par PCR à partir du vecteur pVL-CG20 et des oli~ f~ f~ 8 et 9
(SEQ ID NO: 9 et 10). Après digestion par Nhel, celui-ci est insére dans le
vecteur pCB28 egalement clivé par Nhel, pour donner pCB57. Ce fragment NheI
25 est isolé de pCB57 et introduit dans le vecteur pCB71 digeré par SpeR Comme
prf~cffl-mmf~nt, ce fragment comporte un codon initiateur de la traduction placédans un contexte Kozak. On génère pCBTI (Figure 2) identique à pCBT2 a
l'exception de la longueur du fragment VL30 de rat (0,175 kb au lieu de 0,59 kb).
30 2. Col~trllcti(7ll d~ CCtClll monf7ci.stloniqr~epVL-Cr~
On isole par PCR un fragment d'ADN comportant les séquences d'HaMSV
(position + I à + 794) muni à son extrémité 3' d'un site Ncol. On met en oeuvre
le plasmide pVL-CG20 (Torrent et al, 1994, sllpra) a titre de matrice et les
35 nlignnllflf~oti~f~ 16 et 13 reporté~s dans les SEQ ID NO: 11 et 8 Le fragment PCR
Wo 96/01324 2 1 9 4 1 5 5 . ~l/r~
- 18 -
est digéré par Smal et Ncol avant d'etre inséré dans le vecteur pLNPOZ (Adam
et al., 1991, .sn/71a) digéré par Ncol et partiellement par Smal, pour donner pVL-
CT2 (Figure 2).
5 3. Géncslntioll dc paltic~lle.s ~I;ales infectiell.ses et détcr/nination du taL~
d 'el~r~ ll ct dll talLr d 'erpl es.sioll dcs ~c:nes néo, pllospllatase alcaline
ct LncZ
La lignée de ~ , écotrope GP+E-86 (Markosvitz et al., 1988, J.
10 Virol., 6Z, 1120-1124) et les cellules cibles NIH3T3 (cellules fil)lul/la~Li~lue3 de
souris) disponible.s à l'ATCC, sont cultivées à 37~C en pré~sence de 5% de CO2
dans du milieu DMEM (Dulbecco's Modified Eagle's Medium) ~ ~ '."....' avec
5~o de sérum de veau de nouveau-rlé. La veille de la lLall~feeLiull et de l'infection,
les cellules GP+E-86 et les cellules cibles NIH3T3 sont mises en culture à raison
15 de 5x10; cellules par boite de 10cm et 1,5x105 cellules par puit, le~Je~Li~."~
Les infections virales sont réalisées selon le protocole conventionnel décrit dans la
littérature. La méthode de titration est celle dite du point de dilution limite.
Les vecteurs pCBTI, pCBT2 et pVL-CT2 ainsi que le vecteur de référence pMLV-
20 LacZ (Torrent et al, 1994, .sllpla) sont transfectes en parallèle dans les cellulesGP+E-86 selon la méthode de Chen et Okyama (1987, Mol. Cell. Biol., 7, 2745-
2753). Le jour suis~ant (J+l), le.s cellules sont lavées selon les méthodes de l'art
et on récolte le surnageant de culture à J+2. Différentes dilutions sont utilisées
pour infecter les cellules cibles NIH3T3. Les cellules sont cultivées en milieu
25 sélectif (800 ,tlg/ml de G4]8) 24 heures après la L~all~f~:Liull ou l'infection.
On mesure .sur une aliquote de culture de cellules infectées et transfectées parpMLV-LacZ et pVL-CT2, l expression du gène LacZ après coloration X-Gal.
Cette technique de coloration est decrite dans les ouvrages de base accessibles à
30 I'homme du métier. Il est également possible d'utiliser un kit commercial
(Promega) .
De même, on déterrnine régulièrement au cours du temps, la production de la
phocrh~t~ alcaline dans les cellules GP+E-86 transfectées et dans les cellules
35 NIH3T3 infectées par pCBT1 et pCBT2. Pour ce faire, les cellules sont rincées
WO96/01324 21 9 4 1 ~ P~,l/rh~_
- 19 -
dans du tampon PBS 1 et fixées 5 min à telll,ueldlu,~ ambiante avec une solutioncontenant 2~o de formaldéhyde et 0,25~o de glutaraldéhyde dans du PBS x 1. Les
cellules ont eLsuite rincées deux fois dans du tampon PBS x 1 puis incubées 30 min
à 65~C dans du PBS x 1. Elles sont lavée~s dans du tampon AP (0,1 M Tris-HCl
5 pH 9,5, 0,~ M NaCl, 50 mM MgCl,). Ce tampon est ensuite remplace par la
solution de coloration (contenant 0, I mg/ml de 5-bromo-4-chloro-3-indolyl
phosphate 1, I mg/ml de Nitoblue sel de térazolium 1, Levamisol 1 mM dans du
tampon AP). Ies celluies sont incubées 6 heures à ;.~ példLùle ambiante à l'abri de
la lumière. Les oellules colorées co~ L~u~d~.~L aux cellules pl~ p~ positives.
Le taux d'en~r~ ti~n de chacun des vecteurs est estimé en calculant le rapport
nombre de cellules infectées colorées (PA ou LacZ positives) sur le nombre de
cellules lldu:~le~Lées colorées (PA ou LacZ positives) x 100. Les résultats sontindiqués dans le Tableau I sui~ant.
Tableau I
PLASI IIDESTAUX D'ENCAPSIDATION
pMLV-Lac Z ' 100
pVL-CT2 170
pCBTI 1,8
pCBT2 ~oo
Les données indiquent que le fragment d'ADN de VL30 de rat (positions 205 à
794) comprend un signal d'enud,w~idd~ l au moins aussi efficace que la séquence
30 Psi du MoMuLV. De manière :,UIIJl~lldll;~, la localisation de la séquence VL30 de
rat entre deux gènes affecte peu I efficacité d'~nr~pci~l~tir~n du génome rétroviral
(vecteur pCBT2 comparé à pVL-CT2).
D'autre part, lorsque l'on suit l'expression des gènes PA et néo au cours du temps,
35 la grande majorité des cellules exprimant le gène neo (résistance au G418)
, . , . _ ~ .....
WOg6/01324 21 9 41 ~ r~l~r.
- 20 -
expriment silllulLllle~ .li le gène phosphatase. L'expression des deux gènes eststable au cours du temps, puisque plus de 90% des cellule~s résistantes au G418
sont également pl,.J,~ r positi~es après 40 jours de culture en milieu sélectif~
5 4. Anal~ .~c dcs ARN
Des cellules à confluence sont la~ées (PBS IX) puis incubées 1 heure à 37-C dansun tampon de Iyse (50mM Tris-HCI pH8,8: 0,3M NaCI 0,5% SDS 0,1% ~-
mercapto-éthanol: 100,ug/ml protéinase K). Le Iysat est extrait deux fois au
10 phénol. Les acides nucléiques sont ensuite précipités en pré~sence de 2,5 volumes
d'éthanol froid. Le précipité d ADN est récupéré à l'aide d'une baguette en verre
alors que les ARNs sont préc;pités I heure à -20 C puis centrifuges 30 minutes à10000 rpm a 4 C. L'ARN cellulaire purifié est repris dans 150 ,~d d'eau stérile.
15 Les ARNs cellulaires totaux sont incubes à 65 C pendant 5 min dans un tampon
MOPS (20mM acide morpholinu~,lul,dn~sulfonique, 5mM acétate de sodium, lmM
EDTA, pH7) contenant 6% formaldéhyde, 50% formamide, 30% bleu glycérol.
Les ARNs dénaturés sont dépo.sés sur gel agarose 0,7% en condition denaturante
(MOPS IX, 6% formaldéhyde). Les ARNs sont ensuite transférés sur membrane
20 de nitrocellulose dans un tampon phosphate de sodium 25mM pendant 1 heure 30
min à 800mA. La membrane est exposée sous les UV (252 nm: 0,32 J/cm2) pour
fixer les ARNs. Puis, la membrane est incubée 4 heures à 42 C dans une solution
de ~uléh~ iddliull (50% formamide, IM NaCl, 50mM NaPO4 pH7, 10% Dextran
sulfate, 1% SDS, 250,Llglml ADN de sperme de saumon dénaturé 5 min à 100 C).
25 La membrane est ensuite hybridée pendant 14 heures à 42 C en 50% formamide,
0,8M NaCI, 50mM NaPO4 pH7, 10~o Dextran sulfate et 1% SDS. La sonde
néomycine utilisée à 0,5.109 cpm/~(g est dénaturée 5 min à 100 C et ajoutée à lasolution d'hybridation. Après hybridation, la membrane est lavée dans des bains
successifs: 2 x SSC, 1~" SDS (2 fois 10 min, tel,ll,éldlule ambiante), 2 x SSC,
30 0,1% SDS (30 min, 65 C) et I x SSC, 0,1% SDS (30 min, 65 C). La membrane
est ensuite séchée et exposée à -80 C pendant 72 heures.
La sonde néomycine est ~ u~ e du gène de la néomycine entre les
positions 213 et 596 et correspond au fragment Pstl-Ncol de pLNPOZ (Adam et
35 al, 1991) La sonde a été marquée par extension d amorce avec le kit Nu~ld~ u~"
~ WO96/01324 21 9 4 1 5 5 P~ rl~ _.1 t
- 21 -
kit I (Appligene).
Les ARNs cellulaires ont été extraits de cellule.s GP + E-86 72 heures après
transfection du plasmide pCBT2, ou bien de cellules NIH3T3 infectées avec des
~ 5 virions pC8T2 apres 30 jours de sélection. L'h~ bridation de ces ARNs avec une
sonde ~ ,!5.,~ e du gène néo révèle la présence d'un seul ARN d;~;SIlul~ u~
de taille identique dans les cellules ~Idu.~ U;;..i et infectées. En ~ q.,~...~,I'expression simultanée des gènes ph~$l~ t~ et néomycine dans 90-95~7 des
cellules infectées après 30 jours de sélection est due à la présence d'un seul ARN
10 dicistronique,
5. Vcctc~l/ retr(7vilal dici.stl~7/7iql~e contellal7t dc~.r signal~x d'~nnn/~s;n7nt;n/7.
Cet exemple décrit la COu~iluuLiùll de vecteurs dicistroniques contenant un signal
15 d'encapsidation en position normale (en aval du LTR 5') et le second entre deux
ctstrons, On genere deux vecteurs rétroviraux (I) pVL-CBT2-E~ dans lequel le
signal MoMuLV est placé en aval du LTR 5' et la séquence VL30 de rat (205-794)
entre les gènes codant pour la l,l,;.~ alcaline et néo et (2) pVL-CBT5 dans
lequel leurs positions respectives sont inversées.
pVL-CBT2-E' ré~sulte du clonage du fragment EcoRI isolé de pLNPOZ (Adam et
al,, 1991, sn/Jra) portant les séquences LTR 5 et d Pn~pSi~tion du MoMuLV
dans pCBT2 prP~l~hlpmpnt digére par ce méme enz~me (Figure 3).
25 pVL-CBT5 est obtenu de la fa,con suivante: les séquences uul7~7~ull~ lL au
LTR 5' de MoMuLV suivi des séquences VL30 (positions 205 à 794) sont
amplifiées par PCR a partir de la matrice pVL-CT2. Il est à la portée de l'hommede l'art de déterminer les amorces qui conviennent et de les pourvoir de sites
EcoRI à leurs extrémités S'. Le fragment amplifie est digéré par EcoRI et cloné
30 dans le vecteur pMLV-CTB (voir ci-après) également traité par EcoRI pour donner
pVL-CBT5 (Figure 31.
6. Vectclll r~tl 1l~;ral dici.stl-nni(~ col7t/7~ltallt Ic si~lzal d'rn~n,ns;~7nt;nn de
Mn ~ LV cn pnsitioll ccnrlale.
WO96/01324 21 9 4 ~ ~ 5 1 _llr~
- 22 -
On construit l'équivalent de pCBT2 à la différence que le signal d'rnrar~i~iatirn
de MoMuLV (positions 210 à 1035) remplace celui de VL30 de rat. Il est isolé du
vecteur pLNPOZ par PCR à l'aide d'amorces adéquates comportant à leurs
extrémites un site Nhel. Après digestion Nhel, le fragment amplifié est introduit
5 entre les gènes PA et neo de pMLV-CB71 (Berlioz et Darlix, 1995, J. Virol. 69,2214-2222) digére par Spel. On obtient pMLV-CBT (Figure 3).
7. El~alllatior lles f~aCti~lLc d 'cllcap.cidati~7ll ct If ~7ES dcs I~Cctcllrs phlLV-CBT2-
E~ pVL-CBT7 et plvlLV-CBT
Les titres viraux sont déterminés de manière transitoire ou stable selon la
technologie appliquée ~"~ ",..a par évaluation du nombre de cellules
exprimant la pl~ hc~ alcaline. Les résultats sont indiqués dans le tableau 2
suivant et comparés au vecteur dicistronique pCBT2.
Tableau 2
Vecteur.s Titres viraux (PA' cfu/ml)
Expression tran.sitoire Expression long-terme
pCBT2 I x 10~ I x 10;
pMLV-CBT2,5 x 10~ 2 x 105
pVL-CBT2-E+3 x I OJ 4 x 10;
pVL-CBT52,5 x 10~ 1,8 x 105
25 Ces données indiquent que la presence de deux signaux d',~ p5;.1~ dans un
vecteur rrcf~mhinant n'a pas d'effet (pMLV-CBT comparé a pVL-CBT5) ou un
effet modéré (pCBT2 comparé à pVL-CBT2-E~) sur le titre viral par rapport à un
vecteur n'ayant qu'un signal. On observe également que la position de ces signaux
au sein du génome rétroviral peut influencer leur capacité d'l~nrapsi~lati- r (pVL-
30 CBT2-E comparé à pVL-CBT5) bien qu'à un faible degré. A cet égard, la
r~lmbinaicrn ''signal MoMuLV en position normale et VL30 entre les deux
cistrons" semble plus efficace.
Les cellules GP+E-86 sont transfectées avec chacun des vecteurs rétroviraux et on
35 évalue les niveaux d'expression du gene PA en présence et en absence de
~ wo961013~4 2 1 9 4 ~ 55 P~
-- ~3 --
néom)~cine. Les cellules résistantes à la néom~cine et PA positives sont de~u~bl6~,s
après 15 jours de selection. Les valeurs obtenues sont légèrement plu~s ;IIIIJUI L~l..~
pour les vecteurs dici.stroniques ponant les deux signaux d'Pnr~r~i~lAtinn que pour
leurs homologues n'en ponant qu'un, ce qui suggère que la présence de deux
S séquences d'PnrAr~ inn~ I une d origine rétrovirale et l'autre de VL30
n'influence pas l'expression génique des ci.strons. Par ailleurs, dant tous les cas,
I'expression du gène néo est efficace. Or elle ne peut être obtenue que par
indtiation interne de la traduction des ARNs dicistroniques médiée par les séquences
précédant le cistron néom~ cine, soit VL30 (pCBT2 ou pVL-CBT2-E ' ) ou la région10 d'r~ ;;",l du MoMuLV (pMLV-CBT et pVL-CBT5).
En conclusion, ces e,~lJe,i~.,.es montrent que le.s sequences 5' VL30 et MoMuLV
sont toutes deux capables de promouvoir l'PnrArs;rl~tinn d'un vecteur rétroviral et
l'initiation de la traduction d'un cistron placé en aval.
EXEMPLE 3: Vecteur rétrovjr~l CvlllvlellcllL une séquence d'~ lAl;n
isolee d'un Vl ~Q ~le snuris,
20 1. Ca~L~tl~lCtia/l deS l~e~CtC~
Un fragment d'ADN contenant la séquence VL30 de souris (positions 362 à 1149)
est amplifié par PCR à panir du vecteur pK1403 (Adams et al, 1988, 5~ 7ra) et à
l'aide des amorces nli,~ IPU~ P~ 3 et 4 (SEQ ID NO: 12 et 13). Le fragment
25 généré est digéré: par Ball et Ncol aYant d'être inséré entre les mêmes sites du
vecteur pLNPOZ (Adam et al., 1991, .S~/7la). On obtient le vecteur pSJE2.
Par ailleurs, un fragment d'ADN ~ullll7le~ une séquence plus courte de VL30
de souris (positions 362-575) également obtenu par PCR a panir de la matrice
30 pKT403 et des oligonucleotides 1 et 2 (SEQ ID NO: 14 et 15), est introduit dans
- le vecteur pLNPOZ comme décrit ci-dessus. On obtient pSJEI.
Les vecteurs pSJE1 et pSJE2 uu~ J,e,--,r.--L le LTR 5' du MoMuLV, le fragment
VL30 de souris indiqué, le gène LacZ et le LTR 3' du MoMuLV. A titre de
35 temoin négatif, on construit le vecteur pSJE3 identique aux deux précédents mais
WO 96~01324 2 1 9 2~ 1 ~ 5 P~
- 24 -
dans lequel la séquences VL30 est remplacee par un polylinker. Il est obtenu parintroduclion de l'oligrn~rlPQti~l~ S (SEQ ~1~ NO: 16) entre les sites Ball et Ncol
de pLNPOZ.
5 2. Dr~tc~r7~2ilz(ltil)l2 drl talr.~- d '~'1u'~ s'i~t~7tiOl7
La technologie utilisée est comparable à celle décrite dans l'exemple 1.
Brièvement, les vecteurs pSJEI, 2 et 3 sont transfectes dans des lignées murinesde l~,",pl~ irn, GP+E-86 ou CRIP (Danos et Mulligan, 1988, Proc. Natl.
10 Acad. Sci. USA, ~ , 6460-6464~. Le surnageant de culture est utilisé pour infecter
les oellules cibles murines NIH3T3. On mesure l'expression=du gène LacZ dans lescellules infectées et LIAn~r~L~:e5 après coloration X-Gal. Le titre viral correspond
au rapport nombre de cellules bleues (LacZ + ) infectées sur ~e taux de transfection
multiplié par le volume de surnageant viral utilisé. Le tableau 3 suivant donne une
15 estimation des titres viraux obtenus pour chacun des vecteurs pSJE en fonction de
la lignée de ~ nlllllli~ or de départ. On utilise à titre de référence le vecteur
pMLVLacZ+ (Torrent et al., 1994, ~ ra) C~ L~na,lL la région d'~ Ap~
~ nv~l~Li~ll"elle du MoMuLV.
Tableau 3
PLASMIDES TITRES SUR GP+ ER6 (/ml)TITRES SUR
CRIP (/ml)
En expressionEn expression En cxprcssion
transitoire long terme transitoirc
pSJEI0,1 x 105 0,3 x 105 0,7 x 10~
pSJE2I x 10; 1,3 x 105 6,5 x 104
pSJE3< 0,001 x 10;< 0,001 x 105 .< 0,01 x-lOJ
pMLVlacZ'0,85 x 10;2 x 10; 7 x 10J
, ~ .
Les résultats montrent que la séquence VL30 de souns comprise entre les
nucléotides 362 et 1149 comprend une séquence d f~nrAp~i~lAtirn au moins aussi
30 efficace que celle du MoMuLV. Une séquence VL30 plus courte (positions 362-
575) est encore capable d rnrAp~ Ati~n quoiqu à un taux plus faible. Comme
~ WO96/01324 21 94 1 ~ r~l/r~s- ,
- 25 -
attendu, le vecteur pSJE3, dépour-u de toute région d'~ est incapable
de générer des particules virales.
Par ailleurs, lorsque l'on remplace dans le vecteur pCBT2 la séquence VL30 de rat
5 par une des séquences VL30 de ~souris (soit le fragment 0,78 Icb allant des positions
362 a 1149 soit le fragment 0,2~ I;b allant des positions 362 a 575), on mesure un
taux d'r/~ efficace et une e~:pression correcte du gène de la pl
alcaline.
W O96/01324 2 1 9 ~ 1 ~ 5 P~llrh~ _ C . ~
LISTE DE SEQUENCES
(1) INFORMATION GENERALE:
(i) DE~OSANT:
. NOM: INSERM
RUE 101 rue de Tolbiac
.~ VILLE: Paris cedex 13
. PAY : France
: COD POSTAL: 25654
G TEL.PHONE: (1) 44 23 60 00
IH TEL COPIE: (1) 45 85 68 56 =
(ii) TITRE DE L' INVENTION: Nouveau site interne d~entree des
ribosomes,
vecteur le contenant et utlllsatlon therapeutique.
(iii) NOMBRE DE SEQUENCES: 16
(iv) FORME LI;IB;E PAR ORDINATEUR:
(A) TYP: D- SUPPORT: Tape
(B) ORD NATEUR: IBM PC compatible
(C) SYSTEM- D' EXPLOITATION: PC-DOS/MS-DOS
(D) LOG-CI-L: Pa~entIn P~elease ~1.0, Version #1.25 (OEB)
(2) INFORMATION POUR LA SEQ ID NO: 1:
(i) CARACTER.STIQUES DE LA SEQUENCE:
(~) LON~UEUR: 590 paires de bases
(~) TYP : acide nucléique
(C) NOM RE DE BRINS: simple
(~) CON IGURATION: linéaire
(ii) TYPE DE MOLECULE: ADN (génomigue)
(iii) HYPOTHETIQUE: NON
(iii) ANTI-SENS: NON
(vi) ORIGINE:
(A) ORGANISME: Rat~us
(B) SOUCHE: element VL30 de rat
(xi) DESCRIPTION DE LA SEQUENCE: SEQ ID NO: 1:
GGCAAGCCGG ~C~u~Lll~ L~ LCLL~'I' CCTGTGAACG ATCGATCAAT60
AGGCTCAGAT CTGGGGACTA TCTGGGCGGG CCAGAGAAGG AGCTGACGAG CTCGGACTTC 120
TCCCCCGCAG CCCTGGAAGA CGTTCCAAGG GTGGTTGGAG GAGAGGGAGA TGCGGATCCG 180
TGGCACCTCC ~LC~1111C GGAGGGATCC GCACCCTTGA TGACT5CGTC T=GAATTTTTG I240
GTTTCAGTTT GGTACCGAAG CTGCGCGGCG ~LL~1~1 TACTTGTTTG ACTGTTGGAA 300
~ TCTTTGTGAC CTGACTGTGG TTTTCTGGAC ~ lC TGTTAGTGTC 360
TTTTTGACTT 11~11L~1~ TT~GAATTTG GACTGACGAC TGTGTTTAAA ATCTTAGACC 420
GACGACTGTG TTTGAAATCA TGAAACTGTT '1~CL1~LLC GTCGAAGAGT TTTACTTGGT 480
~ W O96101324 2 1 9 4 1 5 ~ P~llr.
CCCCTTAACG CTTAGTGAGT AAGAAACTTA ATTTTGTAGA U~ Cl~'l'A GTGGCAGTGT 540
GTTGGTTGAT AGCCAAAGTT AATTTTTAAA ACATAGTGTT TTGGGGGTTG 590
(2) INFORMATION POUR LA SEQ ID NO: 2:
(i) CARACTER-STIQUES DE LA SEQUENCE
(~) LON UEUR: 788 paires de bases
(~) TYP : acide nucléique
(C) NOM RE DE BRINS: simple
(D) CON IGURATION: linéaire
(ii) TYPE DE MOLECULE: ADN (génomique)
(iii) HYPOTHETIQUE: NON
(iii) ANTI-SENS: NON
(vi) ORIGINE:
(B) SOUCHE: element VL30 de souris
(xi) DESCRIPTION DE LA SEQUENCE: SEQ ID NO: 2:
GATTCTTTGT l~L~lLLl~ TCTGATGTCT GTGTTCTGAT ~ 11C TGTTTCTAAG 60
TCTGGTGCGA TCGGAGTTTC AGTTTTGCGG ACGCTCAGTG AGACCGCGCT ~A~A~A 120
~1~c~G~1~ GATAAGGATA GACGTGTCCA GGTGTCCACC ~1UUv~1~CC CCTGGGAGAC 180
GTCCCAGGAG GAACAGGGGA GGATCAGGGA u~Cu1~ ACCCCTTTGA AGGCCAAGAG 240
ACCATTTGGG GTTGCGAGAT C~ 1 L-~ AGTCCCACCT CGTGCCCAGT TGCGAGATCG 300
TGGGTTCGAG TCCCACCTCG 1~1111u11u CGAGATCGTG GGTTCGAGTC CCACCTCGCG 360
TCTGGTCACG GGATCGTGGG TTCGAGTCCC ACCTCGTGTT TTGTTGCGAG ATCGTGGGTT 420
CGAGTCCGAC C1~U~ GTCACGGGAT u~ 11.G AGTCCCACCT CGTGCAGAGG 480
GTCTCAATTG ~L~CllA GAGAGGCCAT CTGATTCTTC 1~ lA 540
~ U1~11~11 GTGACTACTG TTTTTCTAAA AATGGGACAA lUl~ l'UUA 600
e1~uc~111c TCTGACTCTG v11.1~1u~ TTGGTAATTT TGTTTGTTTA C~111v1111 660
TGTGAGTCGT GTATGTTGTC TGTTACTATC 11~11111~1 TTGTGGTTTA ~G~ 1 720
~ 1~LLG ~ lml.ll TGTGTTCAGA CTTGGACTGA TGACTGACGA CTGTTTTTAA 780
GTTATGCC 788
(2) INFORMATION POUR LA SEQ ID NO: 3:
(i) CARACTERISTIQUES DE LA SEQUENCE-
(A) LONGUEUR: 30 paires de bases
(B) TYPE: acide nucleique
(C) NOMBRE DE BRINS: simple
(D) CONFIGURATION: linéaire
(ii) TYPE DE MOLECULE: ADN (génomique)
(iii) HYPOTHETIQUE: NON
W 096/01324 2 1 9 ~ 1 5 5 . ~I/r
- 28 -
(iii) ANTI-SENS: NON
(vi) ORIGINE:
(A) ORGANISME: Murine leukemia virus
(B) SOUCHE: souche Friend (ollgQ 6)
(xi) DESCRIPTION DE LA SEQUENCE: SE0 ID NO: 3:
GCTCGAGCTA GCTGCAGCGC CAGTCCTCCG 30
(2) INFORMATION POUR LA sEg ID NO: 4:
(i) CARACT-~.STIQUES DE LA SEQUENCE:
(i) Ln~ruEuR: 26 paires de bases
(~) T ?,: acide nucléique
(r) Nn~ RE DE BRINS: simple
(D) COi.~IGURATION: lineaire
(ii) TYPE DE MOLECULE: ADN (génomique~
(iii) HYPOTHETIQUE: NON
(iii) ANTI-SENS: OUI
(vi) ORIGINE:
(A) ORGANISME: Murine leukemia virus
(B) SOUCHE: souche Friend
(C) INDIVIDUEL ISOLE: oligo 7
(xi) DESCRIPTION DE LA SEQUENCE: SEQ ID NO: 4:
CGGGATCCGC TAGCAAACTT AAGGGG 26
(2) INFORMATION POUR LA SEQ ID NO: 5:
(i) CARACTERIST~QUES DE LA SEQUENCE:
(A) LONGUEUR: 25 paires de bases
(B) TYPE: acide nucléique
(C) NOMBRE DE BRINS: simple
(D) CONFIGURATION: lineaire
(ii) TYPE DE MOLECULE: ADN (génomique)
(iii) HYPOTHETIQUE: NON
(iii) ANTI-SENS: NON
(vi) ORIGINE:
(B) SOUCHE: gene neomycine
(C) INDIVIDUEL ISOLE: oligo 10
(xi) DESCRIPTION DE LA SEQUENCE: SEQ ID NO: 5:
GGGGTCGACA CTAGTGATTG AACAA 25
(2) INFORMATION POUR LA SEQ ID NO: 6:
~i) CARACTERISTIOUES DE LA SEQUENCE:
(A) LONGUEUR: 27 paires de bases
(B) TYPE: acide nucleir~ue
(C) NOMBRE DE BRINS: simple
~ W O96/013Z4 2 1 9 ~ 1 5 ~ F~l/rr~ . ,
-29 -
(D) CONFIGURATION: lineaire
(ii) TYPE DE MOLECULE: ADN (génomique)
(iii) HYPOTHETIQUE: NON
(iii) ANTI-SENS OUI
(vi) ORIGINE:
(B) SOUCHE: gene neomycine
(C) INDIVIDUEL ISOLE: o.ligo 11
(xi) DESCRIPTION DE LA SEQUENCE: SEQ ID NO: 6:
GCTCTAGAGG ATCCGGCAGG TTGGGCG 27
(2) INFORMATION POUR LA SEQ ID NO: 7:
(i) CARACTER-STIQUES DE LA SEQUENCE:
(A) LONtUEUR: 21 paires de bases
(B) TYP-: acide nucléique
(C) NOM RE DE BRINS: simple
~D) CON.IGURATION: linéaire
(ii) TYPE DE MOLECULE: ADN (génomiquej
(iii) HYPOTHETIQUE: NON
(iii) ANTI-SENS: NON
(vi) ORIGINE:
(B) SOUCHE: element VL30 de rat
(C) INDIVIDUEL ISOLE: oligo 12
(xi) DESCRIPTION DE LA SEQUENCE: SEQ ID NO: 7:
GGGCTAGCGG CAAGCCGGCC G 21
(2) INFORMATION POUR LA SEQ ID NO: 8:
(i) CARACTER..STIQUES DE LA sEguENcE:
(A) LON UEUR: 30 palres de bases
(~) TYP,: acide nucléique
(C) NOM RE DE BRINS: simple
(D) CON IGURATION: linéaire
(ii) TYPE:DE MOLECULE: ADN (génomique)
- (iii) HYPOTHETIQUE: NON
(iii) ANTI-SENS: OUI
(vi) ORIGINE:
(B) SOUCHE: element VL30 de rat
(C) INDIVIDUEL ISOLE: oligo 13
(xi) DESCRIPTION DE LA SEQUENCE: SEQ ID NO: ~3:
GGGCTAGCCC CATGGCAACC CCCAAAACAC 30
(2) INFORMATION POUR LA SEQ ID NO: 9:
W 096/01324 2 1 q ~ ~ 5 5 r~llrhv~
- 30 -
(i) CARACTER.STIQUES DE LA SEQUENCE:
(A) LON~UEUR: 21 paires de bases
(B) TYP : acide nucléi4ue
(C) NOM-RE DE BRINS: simple
(D) CON IGURATION: linéaire
(ii) TYPE DE MOLECULE: ADN (génoml~ue)
(iii) HYPOTHETIQUE: NON
~iii) ANTI-SENS: NON
(vi) ORIGINE:
(A) ORGANISME: element VL30 de rat
(C) INDIVIDUEL ISOLE: oligo 8
(xi) DESCRIPTION DE LA SEQUENCE: SEQ ID NO: 9:
GGGrTAr~rGr~ CAAGCCGGCC G . . _ _ _ 21
(2) INFORMATION POUR LA SEQ ID NO: 10:
(i) CARACTE~ISTIQUES DE LA SEQUENCE:
(A) LO~GUEUR: 28 paires de bases
(B) TYPE: acide nucléi4ue
(C) NO~BRE DE BRINS: simple
(D) CO~FIGURATION: linéaire
(ii) TYPE DE MOLECULE: ADN (genomiQue)
(iii) HYPOTHETIQUE: NON
(iii) ANTI-SENS: OUI
(vi) ORIGINE:
(A) ORGANISME: element V130 de rat
(C) INDIVIDUEL ISOLE: oligQ 9
(xi) DESCRIPTION DE LA SEQUENCE: SEQ ID NO: 10:
GGGCTAGCCC CATGGCCGGA TCTCCCTC 28
(2) INFORMATION POUR LA SEQ ID NO: 11:
(i) CARACTERISTIQUES DE LA SEQUENCE:
(A) LONGUEUR: 31 paires de bases
(B) TYPE: acide nucléiQue
(C) NOMBRE DE BRINS: simple ~ ~
(D) CONFIGURATION: lineairç
(ii) TYPE DE MOLECULE: ADN (génomique)
(iii) HYPOTHETIQUE: NON
(iii) ANTI-SENS: NON
(vi) ORIGINE:
(A) ORGANISME: Murine sarcoma Virus
(B) SOUCHE: HaMSV
(C) INDIVIDUEL ISOLE: oligo 16
W0 96/0137A 2 1 9 4 1 5 ~ P~,l /r~5. ~1
-- 31 -
(xi) DESCRIPTION DE LA SEQUENCE: SEQ ID NO: t1:
GCTCGAGCTA GCTGCAGCGC CAGTCCTCCG T 31
(2) INFORMATION POUR LA SEQ ID NO: 12:
(i) CARACTER-STIQUES DE LA SEQUENCE:
(A) LDN'UEUR: 28 paires de bases
(B) TYP.: aclde nucléique
(C) NOM ~RE DE BRINS: simple
(D) CON'IGURATION: lineaire
(ii) TYPE DE MOLECULE: ADN (génomique)
~iii) HYPU~ uU~: NON
(iii) ANTI-SENS: NON
(vi) ORIGINE:
(B) SOUCHE: element VL30 de souris (oligo 3
(xi) DESCRIPTION DE LA SEQUENCE: SEQ ID NO: 12:
CCGAATTCTG GCCAGATTCT TTGTTCTG 28
(2) INFORMATION POUR LA SEQ ID NO: 13:
(i) CARACT_RISTIQUES DE LA SEQUENCE:
(A) LnNGUEUR: 29 paires de bases
(8) T''PE: acide nucléique
(C) N/~MBRE DE BRINS: simple
(D) CONFIGURATION: linéaire
(ii) TYPE DE MOLECULE: ADN (genomique)
(iii~ HYPOTHETIQUE: NON
(iii) ANTI-SENS: OUI
(vi) ORIGINE:
(B) SOUCHE: element VL30 oe souris (oligo 4)
(xi) DESCRIPTION DE LA SEQUENCE: SEQ ID NO: 13:
CCGCTAGCCC CATGGCAACT TAAAAACAG 29
(2) INFORMATION POUR LA SEQ ID NO: 14:
(i) CARACTE~ISTIQUES DE LA SEQUENCE:
(A) LD~GUEUR: 28 paires de bases
(B) TY?E: aciùe nucléique
(C) NO~BRE DE BRINS: simple
(D) CO~FIGURATION: linéaire
(ii) TYPE DE MOLECULE: ADN (génomique)
(iii) HYPOTHETIQUE: NON
(iii) ANTI-SENS: NON
(vi) ORIGINE:
(B) SOUCHE: element VL30 de souris -
W 096/01324 21 9 4 1 5 5 ~ rn~3
(xi) DESCRIPTION DE LA SEQUENCE: SEQ ID NO: 14:
CCGAATTCTG GCCAGATTCT TTGTTCTG 2/3
(2) INFORMATION POUR LA SEQ ID NO: 15:
(i) CARACTER-STIQUES DE LA SEQUENCE:
(~) LO~tUEUR: 27 paires de bases
(~) TY?-: aclde nucléique
(C) NO~lRE DE BRINS: simple ~~
(;) COt.'IGURATION: linéaire
(ii) TYPE DE MOLECULE: ADN (génomique)
(iii) HYPOTHETIQUE: NON
(iii) ANTI-SENS: OUI
(vi) ORIGINE
(B) SOUCHE: element VL30 de souris (oligo 2)- ' ~ ~ ~- - --
(xi) DESCRIPTION DE LA SEQUENCE: SEQ ID NO: 15:
CCGCTAGCCC CATGGCGTCC CTGATCC 27
(2) INFORMATION POUR LA SEQ ID NO: 16:
(i) CARACTER-STIQUES DE LA SEQUENCE:
(A) LONCUEUR: Z4 paires ~de bases .
(B) TYP-.- acide nucléique
(C) NOM RE DE BRINS: simple
(D) CON'IGURATION: linéaire
(ii) TYPE DE MOLECULE: ADN (génomique)
(iii) HYPOTHETIQUE: NON
(iii) ANTI-SENS: NON
(vi) ORIGINE:
(B) SOUCHE: polylinker (oligo 5)
(xi) DESCRIPTION DE LA SEQUENCE: SEQ ID NO: 16:
TGGCCAGCTG AAGCTTGCCA TGGG ~ 24