Note: Descriptions are shown in the official language in which they were submitted.
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
1
METHODE DE BIOSYNTHESE DE LA DIOSMINE ET/OU DE L'HESPERIDINE
DANS UN MICROORGANISME
DOMAINE DE L'INVENTION
La présente invention est relative à une méthode de production de la diosmine
et de
l'hespéridine.
ARRIERE-PLAN TECHNOLOGIQUE
Le Daflon est un mélange de flavonoïdes ayant des effets vaso-toniques et
vasculo-
protecteurs. Ce mélange est principalement composé de Diosmine ¨85 %,
d'Hespéridine
¨8 % et de traces de différentes flavones et de leurs formes oxydées
respectives.
La méthode de production actuelle du médicament est l'extraction acido/basique
à partir
d'écorce de petites oranges, d'un mélange de flavonoïdes contenant
principalement de
l'Hespéridine (94%) et un mélange d'Isonaringine (-3%), de Néoponcirine (-2%)
et
d'Hespérétine (<1%). Ce mélange subit ensuite une oxydation contrôlée par un
procédé
chimique, transformant environ 90 % de l'hespéridine en diosmine, et des
flavonoïdes
minoritaires dans leur forme oxydée.
Ainsi, la production de Daflon est liée à l'approvisionnement en extrait
purifié de
flavonoïdes d'orange, qui peut varier suite à des variations climatiques, des
fluctuations du
cours des monnaies et la difficulté de s'approvisionner sur des dizaines de
sites dans
plusieurs pays (principalement le Mexique, les pays du bassin méditerranéen et
la Chine).
II serait donc précieux de disposer de mode alternatif de production du Daflon
ne
dépendant pas des aléas de l'approvisionnement en extrait purifié de
flavonoïdes d'orange.
Ainsi, il existe un besoin non satisfait de procédé de biosynthèse de la
diosmine et de
l'hespéridine.
RESUME DE L'INVENTION
Les inventeurs ont mis au point une méthode de biosynthèse de la diosmine et
de
l'hespéridine dans un microorganisme recombinant.
Ainsi, la présente invention est relative à un microorganisme recombinant
comprenant :
- une séquence d'acide nucléique hétérologue codant pour une flavanone
7-0-bêta-
D-glucosyltransférase (UGT) capable d'ajouter un glucose en position 7 de
l'hespérétine et/ou la diosmétine ; et
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
2
- une séquence d'acide nucléique hétérologue codant pour une 6"-O-
rhannnosyltransferase (RhaT) capable de transférer un rhannnose en position 6
du
glucose de l'hespérétine-7-0-glucoside et/ou de la diosnnétine-7-0-glucoside;
et
- une séquence d'acide nucléique hétérologue codant pour une UDP-glucose
4,6-
déshydratase/UDP-4-kéto-6-désoxy-D-glucose 3,5-
épinnerase/UDP-4-kéto-L-
rhannnose-réductase (RHM) capable de produire de l'UDP-rhannnose.
De préférence, la flavanone 7-0-bêta-D-glucosyltransférase (UGT) est une
enzyme de Citrus
sinensis, de Citrus clementina, d'Arabidopsis thaliana, de Scutellaria
baicalensis ou d'Homo
sapiens. En particulier, la flavanone 7-0-bêta-D-glucosyltransférase (UGT)
peut être une
enzyme d'Arabidopsis thaliana, de Scutellaria baicalensis ou d'Homo sapiens,
de
préférence d'Arabidopsis thaliana ou de Scutellaria baicalensis. De manière
préférée, la
flavanone 7-0-bêta-D-glucosyltransférase (UGT) est de Citrus sinensis ou de
Scutellaria
baicalensis.
La flavanone 7-0-bêta-D-glucosyltransférase peut être sélectionnée parmi les
enzymes
comprenant une séquence choisie parmi SEQ ID NOs: 113, 115, 91, 93, 95, 97, 99
et 101 et
les polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou
95 %
d'identité de séquence avec une de ces séquences et présentant une activité
flavanone 7-
0-bêta-D-glucosyltransférase, de préférence sélectionnée parmi les enzymes
comprenant
une séquence choisie parmi SEQ ID NOs: 113, 115, 91, 93, 95 et 97 et les
polypeptides
comprenant une séquence ayant au moins 60, 70,80, 85, 90 ou 95% d'identité de
séquence
avec une de ces séquences et présentant une activité flavanone 7-0-bêta-D-
glucosyltransférase. En particulier, elle peut être sélectionnée parmi les
enzymes
comprenant une séquence choisie parmi SEQ ID NOs: 91, 93, 95, 97, 99 et 101 et
les
polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 %
d'identité de séquence avec une de ces séquences et présentant une activité
flavanone 7-
0-bêta-D-glucosyltransférase, de préférence sélectionnée parmi les enzymes
comprenant
une séquence choisie parmi SEQ ID NOs: 91, 93, 95 et 97 et les polypeptides
comprenant
une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence
avec une
de ces séquences et présentant une activité flavanone 7-0-bêta-D-
glucosyltransférase. De
préférence, la flavanone 7-0-bêta-D-glucosyltransférase est sélectionnée parmi
les
enzymes comprenant une séquence choisie parmi SEQ ID NOs: 113 et 95 et les
polypeptides
comprenant une séquence ayant au moins 60, 70,80, 85, 90 ou 95% d'identité de
séquence
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
3
avec une de ces séquences et présentant une activité flavanone 7-0-bêta-D-
glucosyltransférase, de préférence sélectionnée parmi les enzymes comprenant
une
séquence choisie parmi la SEQ ID NOs: 113 et les polypeptides comprenant une
séquence
ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec cette
séquence et
présentant une activité flavanone 7-0-bêta-D-glucosyltransférase
De préférence, la 6"-O-rhamnosyltransferase (RhaT) est une enzyme de plante,
de
préférence du genre Citrus ou de Petunia hybrida, de préférence Citrus
sinensis, Citrus
maxima, ou Citrus clementina, de manière encore plus préférée Citrus sinensis
ou Citrus
clementina. De préférence, la 6"-O-rhannnosyltransferase est sélectionnée
parmi les
enzymes comprenant une séquence choisie parmi SEQ ID NOs: 103, 105 et les
polypeptides
comprenant une séquence ayant au moins 60, 70,80, 85, 90 ou 95% d'identité de
séquence
avec une de ces séquences et présentant une activité 6"-O-
rhannnosyltransferase. De
manière plus particulièrement préférée, la 6"-O-rhannnosyltransferase est
sélectionnée
parmi les enzymes comprenant une séquence choisie parmi la SEQ ID NOs: 103 et
les
polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 %
d'identité de séquence avec cette séquence et présentant une activité 6"-O-
rhannnosyltransferase.
De préférence, l'UDP-glucose 4,6-déshydratase/UDP-4-kéto-6-désoxy-D-glucose
3,5-
épinnerase/UDP-4-kéto-L-rhannnose-réductase (RHM) est une enzyme de plante, de
préférence de Citrus sinensis ou d'Arabidopsis thaliana. De préférence, l'UDP-
glucose 4,6-
déshyd ratase/U DP-4-kéto-6-désoxy-D-glucose 3,5-é
pinnerase/U DP-4-kéto-L-rha nn nase-
réductase est sélectionnée parmi les enzymes comprenant une séquence choisie
parmi SEQ
ID NOs: 107, 109 et 111 et les polypeptides comprenant une séquence ayant au
moins 60,
70, 80, 85, 90 ou 95 % d'identité de séquence avec une de ces séquences et
présentant une
activité UDP-glucose 4,6-déshydratase/UDP-4-kéto-6-désoxy-D-glucose 3,5-
épinnerase/UDP-4-kéto-L-rhannnose-réductase. De manière plus particulièrement
préférée, l'UDP-glucose 4,6-déshydratase/UDP-4-kéto-6-désoxy-D-glucose
3,5-
épinnerase/UDP-4-kéto-L-rhannnose-réductase est sélectionnée parmi les enzymes
comprenant une séquence choisie parmi la SEQ ID NOs: 107 et les polypeptides
comprenant une séquence ayant au moins 60, 70,80, 85, 90 ou 95% d'identité de
séquence
avec cette séquence et présentant une activité UDP-glucose 4,6-
déshydratase/UDP-4-kéto-
6-désoxy-D-glucose 3,5-épinnerase/UDP-4-kéto-L-rhannnose-réductase.
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
4
Dans un mode de réalisation, le microorganisme selon l'invention comprend en
outre :
- une séquence d'acide nucléique hétérologue codant pour une tyrosine
ammonia
lyase (TAL) ; et/ou une séquence d'acide nucléique hétérologue codant pour une
phénylalanine ammonia lyase (PAL) et une séquence d'acide nucléique
hétérologue
codant pour une une cinnamate 4-hydroxylase (C4H) ;
- une séquence d'acide nucléique hétérologue codant pour une 4-coumaroyl-
CoA
ligase (4CL) ;
- une séquence d'acide nucléique hétérologue codant pour une naringénine-
chalcone synthase (CHS) ;
- une séquence d'acide nucléique hétérologue codant pour une chalcone
isomérase
(CHI) ;
- une séquence d'acide nucléique hétérologue codant pour une flavonoïde 3'-
monooxygénase (F3'H) ;
- une séquence d'acide nucléique hétérologue codant pour une 0-méthyl-
transférase (OMT), et
- facultativement, une séquence d'acide nucléique hétérologue codant pour
une 4-
nnéthoxybenzoate 0-dénnéthylase capable de convertir la tyrosine en L-Dopa et
également l'acide p counnarique en acide caféique; ou une séquence d'acide
nucléique hétérologue codant pour une p-counnarate 3-hydroxylase capable de
convertir l'acide p-counnarique en acide caféique.
De préférence, le microorganisme comprend
- une séquence d'acide nucléique hétérologue codant pour une tyrosine ammonia
lyase (TAL) comprenant une séquence choisie parmi SEQ ID NOs: 41 et 39 et les
polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 %
d'identité de séquence avec une de ces séquences et présentant une activité
tyrosine
ammonia lyase, et de préférence une tyrosine ammonia lyase (TAL) comprenant
une
séquence choisie parmi SEQ ID NO: 41 et les polypeptides comprenant une
séquence
ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec cette
séquence
et présentant une activité tyrosine ammonia lyase ; et
- une séquence d'acide nucléique hétérologue codant pour une 4-coumaroyl-CoA
ligase (4CL) comprenant une séquence choisie parmi SEQ ID NOs: 123, 125, 43,
45,
47 et 49 et les polypeptides comprenant une séquence ayant au moins 60, 70,
80, 85,
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
90 ou 95 % d'identité de séquence avec une de ces séquences et présentant une
activité 4-counnarate-CoA ligase, de préférence une 4CL comprenant une
séquence
sélectionnée parmi les SEQ ID NO: 123, 125 et 45 et les polypeptides
comprenant une
séquence ayant au moins 60, 70, 80, 85, 90 ou 95% d'identité de séquence avec
l'une
5 de ces séquences et présentant une activité 4-counnarate-CoA ligase,
et en particulier
une séquence d'acide nucléique hétérologue codant pour une 4-coumaroyl-CoA
ligase (4CL) comprenant une séquence sélectionnée parmi la SEQ ID NO: 45 et
les
polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 %
d'identité de séquence avec cette séquence et présentant une activité 4-
coumarate-
CoA ligase; et
- une séquence d'acide nucléique hétérologue codant pour une chalcone synthase
(CHS) comprenant une séquence choisie parmi SEQ ID NOs: 53, 51, 55 et 57 et
les
polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 %
d'identité de séquence avec une de ces séquences et présentant une activité
chalcone synthase, et de préférence une CHS comprenant une séquence choisie
parmi SEQ ID NO: 53 et les polypeptides comprenant une séquence ayant au moins
60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec cette séquence et
présentant
une activité chalcone synthase; et
- une séquence d'acide nucléique hétérologue codant pour une chalcone
isomérase
(CHI) comprenant une séquence choisie parmi SEQ ID NOs: 61 et 59 et les
polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 %
d'identité de séquence avec une de ces séquences et présentant une activité
chalcone isomérase, et de préférence une CHI comprenant une séquence choisie
parmi la SEQ ID NO: 61 et les polypeptides comprenant une séquence ayant au
moins
60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec cette séquence et
présentant
une activité chalcone isomérase.
De préférence, le microorganisme comprend une séquence d'acide nucléique
hétérologue
codant pour une flavonoïde 3'-monooxygénase (F3'H) de Callistephus chinensis,
Perilla
frutescens var. crispa, Petunia x hybrida, Gerbera hybrida, Citrus sinensis,
Arabidopsis
thaliana, Pilosella officinarum, Osteospermum hybrid cultivar, Phanerochaete
chrysosporium, Citrus clementina ou Streptomyces avermitilis, en particulier
de Callistephus
chinensis, Perilla frutescens var. crispa, Petunia x hybrida, Gerbera hybrida,
Citrus sinensis,
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
6
Arabidopsis thaliana ou Pilosella officinarum, de préférence une enzyme
comprenant une
séquence choisie parmi SEQ ID NOs: 7, 1, 3, 5, 9, 11, 13, 15, 17, 19, 21 et
121 et les
polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 %
d'identité de séquence avec une de ces séquences et présentant l'activité
flavonoïde 3'-
nnonooxygénase, de préférence sélectionnée parmi les enzymes de SEQ ID NOs: 7,
11, 17,
et 121 et les polypeptides ayant au moins 60, 70, 80, 85, 90 ou 95 %
d'identité avec une de
ces séquences et présentant l'activité flavonoïde 3'-nnonooxygénase.
De manière plus particulièrement préférée, le microorganisme comprend une
séquence
d'acide nucléique hétérologue codant pour une flavonoïde 3'-nnonooxygénase
(F3'H)
comprenant une séquence choisie parmi SEQ ID NOs: 7, 17, et 121 et les
polypeptides ayant
au moins 60, 70, 80, 85, 90 ou 95 % d'identité avec une de ces séquences et
présentant
l'activité flavonoïde 3'-nnonooxygénase, de préférence une flavonoïde 3'-
nnonooxygénase
(F3'H) comprenant une séquence choisie parmi SEQ ID NOs: 7 et les polypeptides
ayant au
moins 60, 70, 80, 85, 90 ou 95 % d'identité avec cette séquence et présentant
l'activité
flavonoïde 3'-nnonooxygénase.
De préférence, le microorganisme comprend une séquence d'acide nucléique
hétérologue
codant pour une 0-nnéthyl-transférase (0MT)de Citrus, en particulier Citrus
clementina ou
Citrus sinensis, d'Homo sapiens ou d'Arabidopsis thaliana, de préférence une
enzyme
comprenant une séquence choisie parmi SEQ ID NOs: 119, 117, 87 et 89 et les
polypeptides
comprenant une séquence ayant au moins 60, 70,80, 85, 90 ou 95% d'identité de
séquence
avec une de ces séquences et présentant l'activité 0-nnéthyltransférase. de
manière plus
particulièrement préférée, il comprend une séquence d'acide nucléique
hétérologue
codant pour une 0-nnéthyl-transférase (0MT) comprenant une séquence choisie
parmi SEQ
ID NOs: 119, 117 et 89 et les polypeptides comprenant une séquence ayant au
moins 60,
70, 80, 85, 90 ou 95 % d'identité de séquence avec une de ces séquences et
présentant
l'activité 0-nnéthyltransférase, de préférence une séquence choisie parmi SEQ
ID NOs: 119
et 117 et les polypeptides comprenant une séquence ayant au moins 60, 70, 80,
85, 90 ou
95 % d'identité de séquence avec une de ces séquences et présentant l'activité
0-
nnéthyltransférase.
De préférence, le microorganisme comprend
- une séquence d'acide nucléique hétérologue codant pour une phénylalanine
annnnonia lyase (PAL) , en particulier une PAL comprenant une séquence choisie
parmi SEQ
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
7
ID NOs: 63, 65 et 77, de préférence SEQ ID NO : 65 et 77, et les polypeptides
comprenant
une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence
avec une
de ces séquences et présentant une activité phénylalanine annnnonia lyase, et
de manière
plus particulièrement préférée une phénylalanine annnnonia lyase (PAL)
comprenant une
séquence choisie parmi la SEQ ID NO: 65 et les polypeptides comprenant une
séquence
ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec cette
séquence et
présentant une activité phénylalanine ammonia lyase ; et
- une séquence d'acide nucléique hétérologue codant pour une cinnamate 4-
hydroxylase (C4H), en particulier une C4H comprenant une séquence choisie
parmi SEQ ID
NOs: 67, 69, 79 et les polypeptides comprenant une séquence ayant au moins 60,
70, 80,
85, 90 ou 95 % d'identité de séquence avec une de ces séquences et présentant
une activité
cinnamate 4-hydroxylase ; et de manière tout particulièrement préférée une
cinnamate 4-
hydroxylase (C4H) comprenant une séquence choisie parmi la SEQ ID NO: 79 et
les
polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 %
d'identité de séquence avec cette séquence et présentant une activité
cinnamate 4-
hydroxylase .
Le microorganisme peut comprendre en outre une séquence d'acide nucléique
hétérologue codant pour une flavone synthase (FNS), en particulier une flavone
synthase
capable de produire la lutéoline à partir de l'ériodictyol, de préférence
d'Arabidopsis
thaliana, Petroselinum crispum, Zea mays, Lonicera japonica, Lonicera
macranthoides,
Callistephus chinensis, Apium graveolens, Medicago truncatula, Cuminum
cyminum,
Aethusa cynapium, Angelico archangelica, Conium maculatum, Camellia sinensis,
Cynara
cardunculus var scolymus, Saussurea medusa, Plectranthus barbatus, Scutellaria
baicalensis, Dorcoceras hygrometricum, Antirrhinum malus, Perilla frutescens
var crispa,
Dahlia pinnata ou Erythranthe lewisii. La flavone synthase (FNS) peut être
sélectionnée
parmi les enzymes comprenant une séquence choisie parmi SEQ ID NOs: 33, 35,
37, 127,
129, 131, 133, 135, 137, 139, 141, 143, 145, 147, 149, 151, 153, 155, 157 et
159 et les
polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 %
d'identité de séquence avec une de ces séquences et présentant une activité
flavone
synthase. En particulier la FNS peut être une FNS comprenant une séquence
choisie parmi
SEQ ID NOs: 33, 35, 37 et les polypeptides comprenant une séquence ayant au
moins 60,
70, 80, 85, 90 ou 95 % d'identité de séquence avec une de ces séquences et
présentant une
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
8
activité flavone synthase, et de préférence une flavone synthase (FNS)
comprenant une
séquence choisie parmi SEQ ID NO: 37 et un polypeptide comprenant une séquence
ayant
au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec cette séquence
et
présentant une activité flavone synthase.
II peut également comprendre en outre :
- une séquence d'acide nucléique hétérologue codant pour une cytochrome
P450
réductase (CPR) ; et/ou
- une séquence d'acide nucléique hétérologue ou endogène codant pour une S-
adénosylméthionine synthétase (SAMT).
De préférence, la CPR comprend une séquence choisie parmi SEQ ID NOs: 25, 23,
27, 29 et
31 et les polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85,
90 ou 95
% d'identité de séquence avec une de ces séquences et présentant une activité
cytochrome
P450 réductase, de préférence une séquence choisie parmi SEQ ID NOs: 23, 25 et
29 et les
polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 %
d'identité de séquence avec une de ces séquences et présentant une activité
cytochrome
P450 réductase, et de manière particulière une séquence choisie parmi SEQ ID
NO: 25 et
les polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou
95 %
d'identité de séquence avec cette séquence et présentant une activité
cytochrome P450
réductase.
De préférence, le microorganisme est une levure ou une bactérie, de préférence
une levure
du genre Saccharomyces, en particulier Saccharomyces cerevisiae, ou une
bactérie comme
Escheri chia cou.
La présente invention est également relative à l'utilisation d'un
microorganisme tel que
décrit dans le présent document pour produire de la diosmine et/ou de
l'hespéridine.
En outre, elle est relative à une méthode de production de la diosmine et/ou
de
l'hespéridine comprenant la culture d'un microorganisme tel que décrit dans le
présent
document, et facultativement la récolte de la diosmine et/ou de l'hespéridine.
De préférence, lors de l'utilisation du microorganisme selon l'invention pour
produire de la
diosmine et/ou de l'hespéridine en ce qu'il n'y a pas d'apport de naringénine,
d'apigénine,
d'ériodictyol, de lutéoline, d'hespérétine et/ou de diosmétine dans le milieu
de culture.
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
9
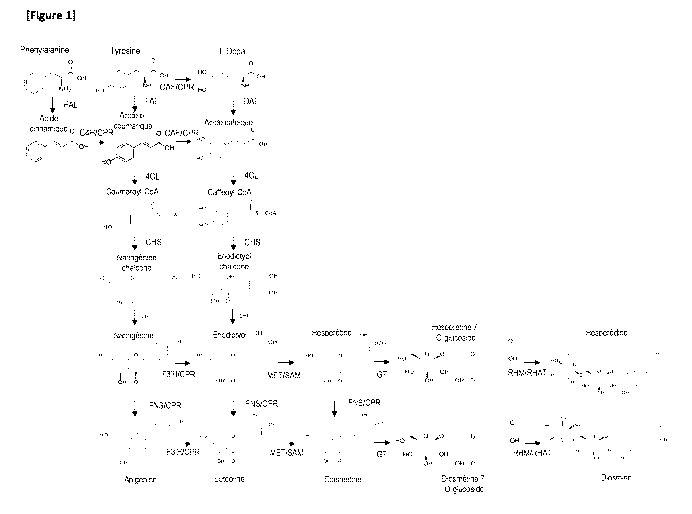
DESCRIPTION DETAILLEE DE L'INVENTION
Les voies de biosynthèse de l'hespéridine dans l'écorce de petites oranges
sont mal
connues. Les inventeurs ont donc exploré plusieurs voies de biosynthèse et ont
réussi à
mettre au point la biosynthèse de la diosmine et de l'hespéridine dans un
microorganisme
recombinant.
Définition
Par microorganisme est entendu un organisme unicellulaire. De préférence,
le
microorganisme est une bactérie ou une levure.
Par microorganisme recombinant est entendu un microorganisme qui ne se
trouve pas
dans la nature et qui contient un génome modifié suite à une insertion,
modification ou
délétion d'un ou plusieurs éléments génétiques hétérologues.
Par acide nucléique recombinant est entendu un acide nucléique qui a été
modifié et
n'existe pas dans un microorganisme naturel. Par exemple, ce terme peut
désigner une
séquence codante ou gène qui est lié de manière opérationnelle à un promoteur
qui n'est
pas le promoteur naturel. Cela peut également désigner une séquence codante
dans
laquelle les introns ont été supprimés pour des gènes comprenant des exons et
des introns.
Par hétérologue est entendu que le gène a été introduit par génie
génétique dans la
cellule. Il peut y être présent sous forme épisomale ou chromosomique.
L'origine du gène
peut être différente de la cellule dans laquelle il est introduit. Cependant,
le gène peut
également provenir de la même espèce que la cellule dans laquelle il est
introduit mais il
est considéré comme hétérologue en raison de son environnement qui n'est pas
naturel.
Par exemple, le gène ou la séquence nucléique est hétérologue car il/elle est
sous le
contrôle d'un promoteur autre que son promoteur naturel, il/elle est
introduit(e) dans un
endroit différent de celui-ci où il/elle est situé(e) naturellement. La
cellule hôte peut
contenir une copie du gène endogène préalablement à l'introduction du gène
hétérologue
ou elle peut ne pas contenir de copie endogène. Par ailleurs, la séquence
d'acide nucléique
peut être hétérologue dans le sens où la séquence codante a été optimisée pour
l'expression dans le microorganisme hôte. De manière préférée, dans le présent
document,
une séquence nucléique hétérologue code pour une protéine qui est hétérologue
à la
cellule hôte, c'est-à-dire qui n'est pas naturellement présente dans la
levure.
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
Tel qu'utilisé ici, le terme natif ou endogène , par rapport au
microorganisme hôte, se
réfère à un élément génétique ou à une protéine naturellement présente dans
ledit
microorganisme.
Le terme gène désigne tout acide nucléique codant pour une protéine. Le
terme gène
5 englobe l'ADN, comme l'ADNc ou l'ADNg, ainsi que l'ARN. Le gène peut
d'abord être
préparé par des techniques recombinantes, enzymatiques et/ou chimiques, et
ensuite
répliqué dans une cellule hôte ou un système in vitro. Le gène comprend
typiquement un
cadre de lecture ouvert codant pour une protéine souhaitée. Le gène peut
contenir des
séquences supplémentaires telles qu'un terminateur de transcription ou un
peptide signal.
10 En raison de la dégénérescence du code génétique, plusieurs acides
nucléiques peuvent
coder un polypeptide particulier. Ainsi, les codons dans la séquence codante
pour un
polypeptide donné peuvent être modifiés de telle sorte que l'expression
optimale dans un
microorganisme particulier soit obtenue, par exemple en utilisant des tables
de traduction
de codon appropriées pour ce microorganisme. Les acides nucléiques peuvent
également
être optimisés selon une teneur en GC préférable pour la levure particulière
et/ou pour
réduire le nombre de séquences répétées. Dans certains modes de réalisation,
les acides
nucléiques hétérologues ont été optimisés par codon pour une expression dans
le
microorganisme concerné. L'optimisation des codons peut être effectuée par des
procédés
de routine connus dans la technique (voir, par exemple, Welch, M., et al.
(2011), Methods
in Enzymology 498: 43-66).
Le terme lié de manière opérationnelle désigne une configuration dans
laquelle une
séquence de contrôle est placée à une position appropriée par rapport à une
séquence
codante, de telle sorte que la séquence de contrôle dirige l'expression de la
séquence
codante.
Le terme séquences de contrôle désigne les séquences d'acide nucléique
nécessaires à
l'expression d'un gène. Les séquences de contrôle peuvent être natives ou
hétérologues.
Des séquences de contrôle bien connues et actuellement utilisées par l'homme
du métier
seront préférées. De telles séquences de contrôle comprennent, mais sans s'y
limiter, un
leader, une séquence de polyadénylation, une séquence de propeptide, un
promoteur, une
séquence de peptide de signal et un terminateur de transcription. De
préférence, les
séquences de contrôle comprennent un promoteur et un terminateur de
transcription.
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
11
Le terme cassette d'expression désigne une construction d'acide nucléique
comprenant
une région codante, c'est-à-dire un gène, et une région de régulation, c'est-à-
dire
comprenant une ou plusieurs séquences de contrôle, liées de manière
opérationnelle. De
préférence, les séquences de contrôle conviennent au microorganisme hôte.
Tel qu'utilisé ici, le terme vecteur d'expression désigne une molécule
d'ADN ou d'ARN
qui comprend une cassette d'expression. De préférence, le vecteur d'expression
est une
molécule d'ADN double brin linéaire ou circulaire. Le vecteur peut comprendre
en outre
une origine de réplication, un marqueur de sélection, etc.
Par pourcentage d'identité entre deux séquences d'acide nucléique ou
d'acides aminés
au sens de la présente invention, on entend désigner un pourcentage de
nucléotides ou de
résidus d'acides aminés identiques entre les deux séquences à comparer, obtenu
après le
meilleur alignement, ce pourcentage étant purement statistique et les
différences entre les
deux séquences étant réparties au hasard et sur toute leur longueur. Le
meilleur
alignement ou alignement optimal est l'alignement pour lequel le pourcentage
d'identité
entre les deux séquences à comparer, comme calculé ci- après, est le plus
élevé. Les
comparaisons de séquences entre deux séquences d'acide nucléique ou d'acides
aminés
sont traditionnellement réalisées en comparant ces séquences après les avoir
alignées de
manière optimale, ladite comparaison étant réalisée par segment ou par fenêtre
de
comparaison pour identifier et comparer les régions locales de similarité de
séquence.
L'alignement aux fins de la détermination du pourcentage d'identité de
séquence d'acides
aminés peut être réalisé de diverses façons qui sont bien connues dans le
domaine, par
exemple en utilisant des logiciels informatiques disponibles sur le Web sur
Internet tels que
http: //blast.ncbi.nInn. Nih.gov/ ou http://www.ebi.ac.uk/Tools/ennboss/).
L'homme du
métier peut déterminer les paramètres appropriés pour mesurer l'alignement, y
compris
tout algorithme nécessaire pour obtenir un alignement maximal sur toute la
longueur des
séquences comparées. Aux fins de la présente invention, les valeurs de
pourcentage
d'identité de séquence d'acides aminés se réfère à des valeurs générées à
l'aide du
programme d'alignement de séquence de paires EMBOSS Needle qui crée un
alignement
global optimal de deux séquences à l'aide de l'algorithme Needlennan-Wunsch,
dans lequel
tous les paramètres de recherche sont définis par défaut Matrice de notation =
BLOSUM62,
Gap ouvert = 10, Gap extension = 0,5, pénalité d'intervalle d'extrémité =
faux, intervalle
d'extrémité ouvert = 10 et écart d'extrémité étendue = 0,5. Dans certains
modes de
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
12
réalisation, tous les pourcentages d'identité mentionnés dans cette demande
peuvent être
fixés à au moins 60%, au moins 70%, au moins 80%, au moins 85%, de préférence
à au
moins 90% d'identité, plus préférablement à au moins 95% d'identité. En
particulier, sont
considérés comme décrits les modes de réalisations dans lesquels tous les
pourcentages
d'identité de séquence des enzymes sont d'au moins 80% ou au moins 85%, de
préférence
d'au moins 90% ou au moins 95% d'identité de séquence.
Dans un mode de réalisation, les polypeptides peuvent présenter 1, 2, 3, 4, 5,
6, 7, 8, 9 ou
additions, substitutions ou délétions par rapport aux séquences décrites dans
les SEQ ID
Nos. En particulier, ces additions, substitutions ou délétions sont
introduites à l'extrémité
10 N-terminale, C-terminale ou aux deux extrémités.
Les polypeptides peuvent éventuellement se présenter sous forme de protéine de
fusion.
Les termes surexpression et augmentation d'expression tels qu'utilisés
ici, sont
utilisés de manière interchangeable et signifient que l'expression d'un gène
ou d'une
enzyme est augmentée par rapport à un microorganisme non modifié, par exemple
un
microorganisme de type sauvage ou ne comprenant pas les modifications
génétiques
décrites ici. Par sauvage est entendu un microorganisme non-modifié
existant dans la
nature. L'expression accrue d'une enzyme est habituellement obtenue en
augmentant
l'expression du gène codant pour ladite enzyme. Dans des modes de réalisation
dans
lesquels le gène ou l'enzyme n'est pas naturellement présent dans le
microorganisme de
l'invention, c'est-à-dire un gène ou une enzyme hétérologue, les termes
surexpression
et expression peuvent être utilisés de manière interchangeable. Pour
augmenter
l'expression d'un gène, l'homme du métier peut utiliser toutes les techniques
connues
telles que l'augmentation du nombre de copies du gène dans le microorganisme,
en
utilisant un promoteur induisant un niveau élevé d'expression du gène, c'est-à-
dire un
promoteur fort, en utilisant des éléments stabilisants l'ARN messager
correspondant ou les
séquences de séquestration du site de liaison de ribosome (RBS) et les
séquences qui les
entourent. En particulier, la surexpression peut être obtenue en augmentant le
nombre de
copies du gène dans le microorganisme. Une ou plusieurs copies du gène peuvent
être
introduites dans le génome par des procédés de recombinaison, connus de
l'homme du
métier, y compris le remplacement des gènes ou l'insertion multicopie (voir
par exemple la
demande de brevet international WO 2015/092013). De préférence, une cassette
d'expression comprenant le gène, de préférence placé sous le contrôle d'un
promoteur
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
13
fort, est intégrée au génome. En variante, le gène peut être porté par un
vecteur
d'expression, de préférence un plasmide, comprenant une cassette d'expression
avec le
gène d'intérêt placé de préférence sous le contrôle d'un promoteur fort. Le
vecteur
d'expression peut être présent dans le microorganisme en une ou plusieurs
copies, selon
la nature de l'origine de la réplication. La surexpression du gène peut
également être
obtenue en utilisant un promoteur induisant un niveau élevé d'expression du
gène. Par
exemple, le promoteur d'un gène endogène peut être remplacé par un promoteur
plus
fort, c'est-à-dire un promoteur induisant un niveau d'expression plus élevé.
Le gène
endogène sous le contrôle d'un promoteur qui n'est pas le promoteur naturel
est qualifié
d'acide nucléique hétérologue. Les promoteurs appropriés pour être utilisés
dans la
présente invention sont connus de l'homme du métier et peuvent être
constitutifs ou
inductibles, et être endogènes ou hétérologues.
Par comprenant est également entendu consistant ou consistant
essentiellement . Par consistant essentiellement est entendu que la
séquence peut
présenter 1, 2, 3, 4, 5, 6, 7, 8, 9 ou 10 additions, substitutions ou
délétions par rapport aux
séquences décrites dans les SEQ ID Nos.
Microorganismes recombinants
Le microorganisme selon la présente invention peut être un microorganisme
eucaryote ou
procaryote.
Dans un premier mode de réalisation, le microorganisme est un eucaryote. De
préférence,
il est une levure de l'ordre des Saccharonnycetales, Sporidiobolales et
Schizosaccharonnycetales. La levure peut par exemple être sélectionnée parmi
Pichia,
Kluyveromyces, Saccharomyces, Schizosaccharomyces, Candida, Lipomyces,
Rhodotorula,
Rhodosporidium, Yarrowia, ou Debaryomyces. Dans un mode de réalisation, la
levure est
choisie parmi Pichia pastoris, Kluyveromyces lactis, Kluyveromyces marxianus,
Saccharomyces cerevisiae, Saccharomyces carlsbergensis, Saccharomyces
diastaticus,
Saccharomyces douglasii, Saccharomyces kluyveri, Saccharomyces norbensis,
Saccharomyces oviformis, Schizosaccharomyces pombe, Candida albicans, Candida
tropicalis, Rhodotorula glutinis, Rhodosporidium toruloides, Yarrowia
lipolytica,
Debaryomyces hansenii et Lipomyces starkeyi. Dans un mode de réalisation
préféré, le
microorganisme est une levure Saccharomyces, de préférence une levure
Saccharomyces
cerevisiae. De manière alternative, le microorganisme peut être un champignon,
de
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
14
préférence un champignon filamenteux. De préférence, il est choisi parmi
Aspergillus,
Trichoderma, Neurospora, Podospora, Endothia, Mucor, Cochiobolus ou
Pyricularia.
Préférentiellement, le champignon est choisi parmi Aspergillus nidulans,
Aspergillus figer,
Aspergillus awomari, Aspergillus oryzae, Aspergillus terre us, Neurospora
crossa,
Trichoderma reesei, et Trichoderma viride.
Dans un deuxième mode de réalisation, le microorganisme est un procaryote. De
préférence, il est une bactérie, notamment choisie parmi les phyla
Acidobacteria,
Actinobacteria, Aquificae, Bacterioide tes,
Chlamydiae, Chlorobi, Chloroflexi,
Chrysiogenetes, Cyanobacteria, Deferribacteres, Deinococcus-Thermus,
Dictyoglomi,
Fibrobacteres, Firmicutes, Fusobacteria, Gemmatimonadetes, Nitrospirae,
Planctomycetes,
Pro teobacteria, Spirochaetes, Thermodesulfobacteria, Thermomicrobia,
Thermotogae, ou
Verrucomicrobia. De manière préférée, la bactérie appartient au genre
Acaryochloris,
Acetobacter, Actinobacillus, Agrobacterium, Alicyclobacillus, Anabaena,
Anacystis,
Anaerobiospirillum, Aquifex, Arthrobacter, Art hrospira,
Azobacter, Bacillus,
Brevibacterium, Burkholderia, Chlorobium, Chromatium, Chlorobaculum,
Clostridium,
Corynebacterium, Cupriavidus, Cyanothece, Enterobacter, Deinococcus, Erwinia,
Escherichia, Geobacter, Gloeobacter, Gluconobacter, Hydrogenobacter,
Klebsiella,
Lactobacillus, Lactococcus, Mannheimia, Mesorhizobium, Methylobacterium,
Microbacterium, Microcystis, Nitrobacter, Nitrosomonas, Nitrospina,
Nitrospira, Nostoc,
Phormidium, Prochlorococcus, Pseudomonas, Ralstonia, Rhizobium, Rhodobacter,
Rhodococcus, Rhodopseudomonas, Rhodospirillum, Salmonella, Scenedesmun,
Serratia,
Shigella, Staphylococcus, Streptomyces, Synechoccus,
Synechocystis,
Thermosynechococcus, Trichodesmium, ou Zymomonas. De manière encore préférée,
la
bactérie est choisie parmi les espèces Agrobacterium tumefaciens,
Anaerobiospirillum
succiniciproducens, Actinobacillus succinogenes, Aquifex aeolicus, Aquifex
pyrophilus,
Bacillus subtilis, Bacillus amyloliquefacines, Brevibacterium ammoniagenes,
Brevibacterium immariophilum, Clostridium pas teurianum, Clostridium
ljungdahlii,
Clostridium acetobutylicum, Clostridium beigerinckii, Corynebacterium
glutamicum,
Cupriavidus necator, Cupriavidus metallidurans, Enterobacter sakazakii,
Escherichia colt
Gluconobacter oxydons, Hydrogenobacter thermophilus, Klebsiella oxytoca,
Lactococcus
lactis, Lactobacillus plan tarum, Mannheimia succiniciproducens, Mesorhizobium
loti,
Pseudomonas aeruginosa, Pseudomonas mevalonii, Pseudomonas pudica, Pseudomonas
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
putida, Pseudomonas fluorescens, Rhizobium etli, Rhodobacter capsulatus,
Rhodobacter
sphaeroides, Rhodospirillum rubrum, Salmonella enterica, Salmonella typhi,
Salmonella
typhimurium, Shigella dysenteriae, Shigella flexneri, Shigella sonnei,
Staphylococcus aureus,
Streptomyces coelicolor, Zymomonas mobilis, Acaryochloris marina, Anabaena
variabilis,
5 Arthrospira platensis, Arthrospira maxima, Chlorobium tepidum, Chlorobaculum
sp.,
Cyanothece sp., Gloeobacter violace us, Microcystis aeruginosa, Nostoc
punctiforme,
Prochlorococcus marin us, Synechococcus elongatus,
Synechocystis sp.,
Thermos ynechococcus elongatus, Trichodesmium erythraeum, et Rhodopseudomonas
palustris. Dans un mode de réalisation préféré, le microorganisme est une
bactérie
10 Escherichia coli, par exemple E. coli BL21, E. coli BL21 (DE3), E. coli
MG1655 ou E. coli W31
10 et leurs dérivés. Dans un mode de réalisation alternatif, le microorganisme
est une
bactérie du genre Streptomyces, en particulier Streptomyces venezuelae.
Les microorganismes peuvent avoir été modifiés pour augmenter la production de
tyrosine
et/ou de phénylalanine, de préférence de la tyrosine. Notamment, les gènes
responsables
15 de la rétro-inhibition de la production de la tyrosine et/ou
phénylalanine, de préférence de
la tyrosine, peuvent être inactivés. De manière alternative ou cumulative, la
voie de la
biosynthèse de la tyrosine et/ou phénylalanine, de préférence de la tyrosine,
peut être
optimisée, notamment en redirigeant le flux de carbone d'autres voies
métaboliques vers
celle de la tyrosine et/ou de la phénylalanine, de préférence de la tyrosine.
Ces
modifications et ces gènes sont bien connus de l'homme du métier (voir US
8,809,028;
Pandey et al, 2016, Biotechnol Adv., 34, 634-662).
Ainsi, dans un mode de réalisation, le microorganisme produit des quantités
importantes
de tyrosine et/ou de phénylalanine, en particulier à partir d'une source
carbonée simple
comme le glucose.
Modifications permettant la production d'hespéridine et/ou de diosmine
Le microorganisme recombinant selon la présente invention a été modifié pour
produire
de l'hespéridine et/ou de la diosnnine. Notamment, pour permettre au
microorganisme de
synthétiser l'hespéridine et/ou la diosnnine à partir de l'hespérétine et/ou
de la diosnnétine
respectivement, le nnicrooganisme a été modifié pour introduire les enzymes
nécessaires à
la glycosylation de l'hespérétine et/ou de la diosnnétine en position 7 et au
transfert d'un
rhannnose en position 6 du glucose de l'hespérétine-7-0-glucoside et/ou la
diosnnétine-7-
0-glucoside.
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
16
Dans un premier mode de réalisation, le microorganisme recombinant est capable
de
produire de l'hespérétine et/ou de la diosnnétine, en particulier il a été
modifié dans ce but.
Dans un mode de réalisation alternatif, l'hespérétine et/ou la diosnnétine
peuvent être
apportées au microorganisme, par exemple par ajout de ces composés dans le
milieu de
culture.
Dans un mode de réalisation particulier, le microorganisme produit
l'hespéridine. La
diosmine peut alors être préparée à partir de l'hespéridine par conversion
chimique,
notamment par oxydation.
Dans un mode de réalisation préféré, le microorganisme produit l'hespéridine
et la
diosnnine.
Ainsi, le microorganisme recombinant comprend :
a. une séquence d'acide nucléique hétérologue codant pour une flavanone 7-
0-bêta-D-glucosyltransférase (UGT) capable d'ajouter un glucose en
position 7 de l'hespérétine et/ou la diosmétine ;
b. une séquence d'acide nucléique hétérologue codant pour une 6-0-
rhamnosyltransferase (RhaT) capable de transférer un rhamnose en position
6 du glucose de l'hespérétine-7-0-glucoside et/ou la diosmétine-7-0-
glucoside; et
c. une séquence d'acide nucléique hétérologue codant pour une UDP-glucose
4,6-déshydratase/UDP-4-kéto-6-désoxy-D-glucose 3,5-épimerase/UDP-4-
kéto-L-rhamnose-réductase (RHM) capable de produire de l'UDP-rhamnose.
Dans un mode de réalisation, la flavanone 7-0-bêta-D-glucosyltransférase
(UGT), la 6"-O-
rhamnosyltransferase (RhaT) et l'UDP-glucose 4,6-déshydratase/UDP-4-kéto-6-
désoxy-D-
glucose 3,5-épimerase/UDP-4-kéto-L-rhamnose-réductase (RHM) sont des enzymes
hétérologues au microorganisme.
UGT : Flavanone 7-0-bêta-glucosyltransférase
L'UGT est une enzyme qui réalise le transfert d'un glucose en position 7 de
l'hespérétine
et/ou de la diosmétine. Le nom de l'UGT est UDP-glucose : flavanone 7-0-bêta-D-
glucosyltransférase ou flavanone 7-0-bêta-D-glucosyltransférase. Elle est
également
appelée par les dénominations suivantes : uridine diphosphoglucose-flavanone 7-
0-
glucosyltransférase, naringénine 7-0-glucosyltransférase, et hespérétine 7-0-
glucosyl-
transférase. Cette enzyme appartient à la classe des EC 2.4.1.185.
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
17
Les inventeurs ont dû identifier et sélectionner des enzymes capables
d'accepter de
l'hespérétine et/ou de la diosmétine comme substrat et d'ajouter un glucose en
position 7
de ces composés. De préférence, l'enzyme est sélectionnée de sorte à présenter
une
préférence pour une glycosylation en position 7 de l'hespérétine et/ou de la
diosmétine.
Dans un mode de réalisation préféré, l'enzyme est spécifique de la position 7
de
l'hespérétine et/ou de la diosmétine.
Le microorganisme comprend donc une séquence d'acide nucléique hétérologue
codant
pour une flavanone 7-0-bêta-D-glucosyltransférase (UGT) capable d'ajouter un
glucose en
position 7 de l'hespérétine et la diosmétine.
Par activité 7-0-bêta-glycosyltransférase est entendue une enzyme UGT
capable de
rajouter un glucose en position 7 d'un flavonoïde. Pour déterminer s'il y a
une activité 7-0-
bêta-glycosyltransférase, un test enzymatique peut être effectué qui consiste
en
l'incubation in vitro de l'enzyme flavanone 7-0-bêta-D-glucosyltransférase en
présence de
NAD(P)H, 02, et d'un flavonoïde, dans des conditions optimales (pH, ions...),
et
l'observation en UPLC-MS et en comparaison au standard attendu de l'apparition
d'un
flavonoïde ayant un glucose supplémentaire en position 7. De préférence, le
flavonoïde est
l'hespérétine ou la diosmétine et le flavonoïde ayant un glucose
supplémentaire en
position 7 est leur forme avec un glucose supplémentaire en position 7, c'est-
à-dire
l'hespérétine 7-0-glucoside et la diosmétine 7-0 glucoside.
Cette enzyme n'est présente que dans des eucaryotes supérieurs, en particulier
dans les
plantes. Par exemple, l'enzyme peut provenir de plantes du genre Citrus, en
particulier
Citrus maxima, Citrus sinensis, Citrus clementina, Citrus mitis et Citrus x
paradisi, Lysium,
en particulier Lysium barbarum, Petunia, en particulier Petunia x hybrida,
Arabidopsis, en
particulier Arabidopsis thaliana, ou Scutellaria, en particulier Scutellaria
baicalensis.
Dans un mode de réalisation, l'UGT est une enzyme d'Arabidopsis thaliana, de
Scutellaria
baicalensis ou d'Homo sapiens. De préférence, l'UGT est une enzyme
d'Arabidopsis
thaliana ou de Scutellaria baicalensis.
Dans un mode de réalisation préféré, l'UGT est une enzyme de Citrus sinensis,
de Citrus
clementina, d'Arabidopsis thaliana, de Scutellaria baicalensis ou d'Homo
sapiens, de
préférence de Citrus sinensis ou de Scutellaria baicalensis.
Dans un mode de réalisation particulier, l'UGT est sélectionnée parmi les
enzymes
comprenant une séquence choisie parmi SEQ ID NOs: 91, 93, 95, 97, 99 et 101 et
les
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
18
polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 %
d'identité de séquence avec une de ces séquences et présentant une activité
flavanone 7-
0-bêta-D-glucosyltransférase, de préférence sélectionnée parmi les enzymes
comprenant
une séquence choisie parmi SEQ ID NOs: 91, 93, 95 et 97 et les polypeptides
comprenant
une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence
avec une
de ces séquences et présentant une activité flavanone 7-0-bêta-D-
glucosyltransférase,
notamment avec l'hespérétine et/ou de la diosmétine comme substrat.
Dans un mode de réalisation préféré, l'UGT est sélectionnée parmi les enzymes
comprenant une séquence choisie parmi SEQ ID NOs: 113, 115, 91, 93, 95, 97, 99
et 101 et
les polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou
95 %
d'identité de séquence avec une de ces séquences et présentant une activité
flavanone 7-
0-bêta-D-glucosyltransférase, de préférence sélectionnée parmi les enzymes
comprenant
une séquence choisie parmi SEQ ID NOs: 113, 115, 91, 93, 95 et 97 et les
polypeptides
comprenant une séquence ayant au moins 60, 70,80, 85, 90 ou 95% d'identité de
séquence
avec une de ces séquences et présentant une activité flavanone 7-0-bêta-D-
glucosyltransférase. De manière plus particulièrement préféré, l'UGT est
sélectionnée
parmi les enzymes comprenant une séquence choisie parmi SEQ ID NOs: 113, 115
et 95 et
les polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou
95 %
d'identité de séquence avec une de ces séquences et présentant une activité
flavanone 7-
O-bêta-D-glucosyltra nsférase
Ainsi, l'UGT peut être d'Arabiclopsis thaliana. Les séquences nucléiques
codant cette
enzyme et séquences protéiques sont décrites dans NCBI sous les numéros de
référence
NM_119576 et NP_567955.1, respectivement, et plus particulièrement dans la SEQ
ID NO:
91. La protéine est également décrite dans UniProtKB/Swiss Prot sous le numéro
de
référence UGT7381.
De manière alternative, l'UGT est de Scutellaria baicalensis. Dans un premier
aspect, les
séquences nucléiques codant une première UGT et séquences protéiques sont
décrites
dans NCBI sous les numéros de référence KU712253 et AMK52071.1,
respectivement, et
plus particulièrement dans la SEQ ID NO : 93. La protéine est décrite dans
UniProtKB/Swiss
Prot sous le numéro de référence A0A140DPB7. Dans un deuxième aspect, les
séquences
nucléiques codant une seconde UGT et séquences protéiques sont décrites dans
NCBI sous
les numéros de référence KU712254 et AMK52072.1, respectivement, et plus
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
19
particulièrement dans la SEQ ID NO : 95. La protéine est décrite dans
UniProtKB/Swiss Prot
sous le numéro de référence A0A140DP68. Dans un troisième aspect, les
séquences
nucléiques codant une troisième UGT et séquences protéiques sont décrites dans
NCBI
sous les numéros de référence KU712255 et AMK52073.1, respectivement, et plus
particulièrement dans les SEQ ID Nos : 97. La protéine est décrite dans
UniProtKB/Swiss
Prot sous le numéro de référence A0A140DP69.
Par ailleurs, l'UGT peut être d'Homo sapiens. Dans un premier aspect, l'UGT
est UGT1A6
(UDP glucuronosyltransferase family 1 member A6). La protéine est décrite dans
UniProtKB/Swiss Prot sous le numéro de référence P19224. La séquence codante
consensus est décrite dans NCBI sous le numéro CCDS2507.1. La séquence de
cette enzyme
est décrite dans la SEQ ID NO : 99. Dans un second aspect, l'UGT est UGT1A7
(UDP
glucuronosyltransferase family 1 member A7). La protéine est décrite dans
UniProtKB/Swiss Prot sous le numéro de référence Q9HAW7. La séquence codante
consensus est décrite dans NCBI sous le numéro CCDS2506.1. la séquence de
cette enzyme
est décrite dans la SEQ ID NO : 101.
L'UGT peut également est de Citrus, en particulier de Citrus sinensis ou
Citrus clementina.
En particulier, l'UGT de Citrus sinensis est décrite dans la SEQ ID NO : 113.
Une séquence
nucléotidique codant pour cette enzyme est décrite dans la SEQ ID NO: 114.
L'UGT de
Citrus clementina est décrite dans la SEQ ID NO: 115. Une séquence
nucléotidique codant
pour cette enzyme est décrite dans la SEQ ID NO: 116.
Selon un mode de réalisation préféré, l'UGT est sélectionnée parmi les enzymes
comprenant une séquence choisie parmi SEQ ID NOs: 113 et 95 et les
polypeptides
comprenant une séquence ayant au moins 60, 70,80, 85, 90 ou 95% d'identité de
séquence
avec une de ces séquences et présentant une activité flavanone 7-0-bêta-D-
glucosyltransférase, de préférence sélectionnée parmi les enzymes comprenant
une
séquence choisie parmi la SEQ ID NOs: 113 et les polypeptides comprenant une
séquence
ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec cette
séquence et
présentant une activité flavanone 7-0-bêta-D-glucosyltransférase.
RhaT: 6-0-rhamnosyltransférase
La RhaT est une enzyme qui réalise le transfert d'un rhamnose en position 6 du
glucose de
l'hespérétine-7-0-glucoside et/ou la diosmétine-7-0-glucoside. La RhaT est une
6-0-
rhamnosyltransferase. Cette enzyme appartient à la classe des EC 2.4.1.653.
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
Les inventeurs ont dû identifier et sélectionner des enzymes capables
d'accepter de
l'hespérétine-7-0-glucoside et/ou la diosnnétine-7-0-glucoside comme substrat
et
d'ajouter un rhamnose en position 6 du glucose de ces composés.
Le microorganisme comprend donc une séquence d'acide nucléique hétérologue
codant
5 pour une 6-0-rhamnosyltransferase (RhaT) capable de transférer un
rhamnose en position
6 du glucose de l'hespérétine-7-0-glucoside et/ou la diosmétine-7-0-glucoside.
Cette
enzyme n'est présente que dans des eucaryotes supérieurs, en particulier dans
les plantes.
Par activité 6-0-rhamnosyltransferase est entendu l'ajout d'un rhamnose en
position 6
du glucose par l'enzyme RhaT. Pour déterminer s'il y a une activité 6-0-
10 rhamnosyltransferase, un test enzymatique peut être effectué qui
consiste en l'incubation
in vitro de l'enzyme 6-0-rhamnosyltransferase en présence de NAD(P)H, 02, et
d'un
flavonoïde, dans des conditions optimales (pH, ions...), et l'observation en
UPLC-MS et en
comparaison au standard attendu de l'apparition d'un flavonoïde où es ajouté
un
rhamnose en position 6 du glucose. De préférence, le flavonoïde est
l'hespérétine 7-0-
15 glucoside ou la diosmétine 7-0-glucoside et le flavonoïde où est ajouté
un rhamnose en
position 6 du glucose sont l'hespéridine et la diosmine.
De préférence, cette enzyme est une enzyme produite par une plante du genre
Citrus ou
de Petunia hybrida, de préférence de l'espèce Citrus sinensis, Citrus maxima,
ou Citrus
clementina. De préférence, l'enzyme est une enzyme provenance de Citrus
sinensis ou
20 Citrus clementina.
Dans un mode de réalisation particulier, la 6-0-rhamnosyltransferase est
sélectionnée
parmi les enzymes comprenant une séquence choisie parmi SEQ ID NOs: 103, 105
et les
polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 %
d'identité de séquence avec une de ces séquences et présentant une activité 6-
0-
rhamnosyltransferase.
Ainsi, la RhaT peut être de Citrus clementina. Elle est décrite dans la base
de données
Genbank de NCBI sous le numéro XM_006420965 pour la séquence nucléique et sous
le
numéro XP_006421028 pour la séquence protéique, et plus particulièrement dans
les SEQ
ID NO : 103. La protéine est décrite dans UniProtKB/Swiss Prot sous le numéro
de référence
V4RJL6.
La RhaT peut également être de Citrus sinensis. Elle est décrite dans la base
de données
Genbank de NCBI sous le numéro DQ119035 pour la séquence nucléique et sous le
numéro
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
21
ABA18631.1 pour la séquence protéique, et plus particulièrement dans les SEQ
ID NO : 105.
La protéine est décrite dans UniProtKB/Swiss Prot sous le numéro de référence
A7ISD3.
Dans un mode de réalisation particulier, la RhaT est de Citrus sinensis et est
une enzyme
comprenant la séquence SEQ ID NO: 105 ou un polypeptide comprenant une
séquence
ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec celle-ci
et présentant
une activité 6"-O-rhannnosyltransferase.
Dans un mode de réalisation préféré, le RhaT est sélectionnée parmi les
enzymes
comprenant une séquence choisie parmi la SEQ ID NO: 103 et les polypeptides
comprenant
une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence
avec cette
séquence et présentant une activité 6"-O-rhamnosyltransferase.
RHM : UDP-glucose 4,6-déshydratase/UDP-4-kéto-6-désoxy-D-glucose 3,5-
épimerase/UDP-
4-kéto-L-rhamnose-réductase
La RHM est une enzyme trifonctionnelle UDP-glucose 4,6-déshydratase/UDP-4-kéto-
6-
désoxy-D-glucose 3,5-épimerase/UDP-4-kéto-L-rhamnose-réductase. Cette enzyme
est
capable de produire de l'UDP-rhamnose à partir de l'UDP-glucose. Cette enzyme
appartient
à la classe des EC 4.2.1.76. L'UDP-rhamnose est nécessaire pour une activité
de la 6-0-
rhamnosyltransferase (RhaT).
Le microorganisme comprend donc une séquence d'acide nucléique hétérologue
codant
pour une UDP-glucose 4,6-déshydratase/UDP-4-kéto-6-désoxy-D-glucose 3,5-
épimerase/UDP-4-kéto-L-rhamnose-réductase (RHM) capable de produire de l'UDP-
rhamnose. Cette enzyme n'est présente que dans des eucaryotes supérieurs, en
particulier
dans les plantes.
Par activité UDP-glucose 4,6-déshydratase/UDP-4-kéto-6-désoxy-D-glucose 3,5-
épimerase/UDP-4-kéto-L-rhamnose-réductase est entendue la transformation de
l'UDP-
glucose en UDP-rhamnose. Pour déterminer s'il y a une activité UDP-glucose 4,6-
déshyd ratase/U DP-4-kéto-6-désoxy-D-glucose 3,5-é
pimerase/U DP-4-kéto-L-rha m nase-
réductase, un test enzymatique peut être effectué qui consiste en l'incubation
in vitro de
l'enzyme UDP-glucose de NAD(P)H, 02, dans des conditions optimales (pH,
ions...), et
l'observation en UPLC-MS et en comparaison au standard attendu de l'apparition
d'un
UDP-rhamnose.
De préférence, cette enzyme est une enzyme produite par une plante du genre
Citrus, en
particulier Citrus sinensis, ou par Arabidopsis thaliana.
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
22
Dans un mode de réalisation particulier, l'UDP-glucose 4,6-déshydratase/UDP-4-
kéto-6-
désoxy-D-glucose 3,5-épinnerase/UDP-4-kéto-L-rhannnose-réductase est
sélectionnée
parmi les enzymes comprenant une séquence choisie parmi SEQ ID NOs: 107, 109,
111 et
les polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou
95 %
d'identité de séquence avec une de ces séquences et présentant une activité
UDP-glucose
4,6-déshydratase/UDP-4-kéto-6-désoxy-D-glucose 3,5-
épinnerase/UDP-4-kéto-L-
rhamnose-réductase.
La RHM peut être de Citrus sinensis. Elle est décrite dans la base de données
Genbank de
NCBI sous le numéro XM_006477756 pour la séquence nucléique et sous le numéro
XP_006477819.1 pour la séquence protéique, et plus particulièrement dans la
SEQ ID NO:
107.
La RHM peut également être d'Arabidopsis thaliana. Dans un premier aspect, la
protéine
est décrite dans la base de données Genbank de NCBI sous le numéro AY081471
pour la
séquence nucléique et sous le numéro AAM10033.1 pour la séquence protéique, et
plus
particulièrement dans la SEQ ID NO : 109. La protéine est décrite dans
UniProtKB/Swiss
Prot sous le numéro de référence Q9SYM5. Dans un deuxième aspect, la protéine
est
décrite dans la base de données Genbank de NCBI sous le numéro AJ565874 pour
la
séquence nucléique et sous le numéro CAD92667.1 pour la séquence protéique, et
plus
particulièrement dans la SEQ ID NO : 111. La protéine est décrite dans
UniProtKB/Swiss
Prot sous le numéro de référence Q9LPG6.
Dans un mode de réalisation particulier, la RHM une enzyme comprenant une
séquence
sélectionnée parmi SEQ ID NOs: 107, 109 et les polypeptides comprenant une
séquence
ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec l'une de
ces
séquences et présentant une activité UDP-glucose 4,6-déshydratase/UDP-4-kéto-6-
désoxy-D-glucose 3,5-épinnerase/UDP-4-kéto-L-rhannnose-réductase.
Dans un mode de réalisation préféré, la RHM est sélectionnée parmi les enzymes
comprenant une séquence choisie parmi la SEQ ID NO: 107 et les polypeptides
comprenant
une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence
avec cette
séquence et présentant une activité UDP-glucose 4,6-déshydratase/UDP-4-kéto-6-
désoxy-
D-glucose 3,5-épinnerase/UDP-4-kéto-L-rhannnose-réductase.
Combinaison d'enzymes
Dans un mode de réalisation particulier, le microorganisme recombinant
comprend :
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
23
- une séquence d'acide nucléique hétérologue codant pour une flavanone
7-0-bêta-
D-glucosyltransférase (UGT) d'Arabidopsis thaliana, de Scutellaria baicalensis
ou
d'Homo sapiens, de préférence d'Arabidopsis thaliana ou de Scutellaria
baicalensis,
de préférence une flavanone 7-0-bêta-D-glucosyltransférase (UGT) sélectionnée
parmi les enzymes comprenant une séquence choisie parmi SEQ ID NOs: 113, 115,
91, 93, 95, 97, 99 et 101 et les polypeptides comprenant une séquence ayant au
moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec une de ces
séquences
et présentant une activité flavanone 7-0-bêta-D-glucosyltransférase ; de
manière
particulière une flavanone 7-0-bêta-D-glucosyltransférase (UGT) comprenant une
séquence choisie parmi SEQ ID NOs: 113, 115, 91, 93, 95 et 97 et les
polypeptides
comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité
de
séquence avec une de ces séquences et présentant une activité flavanone 7-0-
bêta-
D-glucosyltransférase, et de manière tout particulièrement préférée,
comprenant
une séquence choisie parmi SEQ ID NOs: 113, 115 et 95 et les polypeptides
comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité
de
séquence avec une de ces séquences et présentant une activité flavanone 7-0-
bêta-
D-glucosyltransférase, en particulier comprenant une séquence choisie parmi
SEQ
ID NOs: 113 et 95 et les polypeptides comprenant une séquence ayant au moins
60,
70, 80, 85, 90 ou 95 % d'identité de séquence avec une de ces séquences et
présentant une activité flavanone 7-0-bêta-D-glucosyltransférase ;
- une séquence d'acide nucléique hétérologue codant pour une 6-0-
rhannnosyltra nsferase (RhaT) du genre Citrus ou de Petunia hybrida, de
préférence
Citrus sinensis, Citrus maxima, ou Citrus clementina, de manière encore plus
préférée Citrus sinensis ou Citrus clementina, de préférence une 6-0-
rhannnosyltransferase (RhaT) comprenant une séquence choisie parmi SEQ ID NOs:
103, 105 et les polypeptides comprenant une séquence ayant au moins 60, 70,
80,
85, 90 ou 95 % d'identité de séquence avec une de ces séquences et présentant
une
activité 6-0-rhannnosyltransferase, et de préférence une RhaT comprenant une
séquence choisie parmi la SEQ ID NOs: 103 et les polypeptides comprenant une
séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec
cette séquence et présentant une activité 6"-O-rhannnosyltransferase ; et
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
24
- une séquence d'acide nucléique hétérologue codant pour une UDP-glucose 4,6-
déshydratase/UDP-4-kéto-6-désoxy-D-glucose 3,5-
épinnerase/UDP-4-kéto-L-
rhannnose-réductase (RHM) de Citrus sinensis ou d'Arabidopsis thaliana, de
préférence une RHM comprenant une séquence choisie parmi SEQ ID NOs: 107, 109
et 111 et les polypeptides comprenant une séquence ayant au moins 60, 70, 80,
85,
90 ou 95 % d'identité de séquence avec une de ces séquences et présentant une
activité UDP-glucose 4,6-déshydratase/UDP-4-kéto-6-désoxy-D-glucose 3,5-
épinnerase/U DP-4-kéto-L-rhannnose-réductase, en particulier une
RHM
comprenant une séquence sélectionnée parmi SEQ ID NOs: 107, 109 et les
polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 %
d'identité de séquence avec cette séquence et présentant une activité UDP-
glucose
4,6-déshydratase/UDP-4-kéto-6-désoxy-D-glucose 3,5-
épinnerase/UDP-4-kéto-L-
rhannnose-réductase, et de préférence une RHM comprenant une séquence choisie
parmi la SEQ ID NOs: 107 et les polypeptides comprenant une séquence ayant au
moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec cette séquence et
présentant une activité UDP-glucose 4,6-déshydratase/UDP-4-kéto-6-désoxy-D-
glucose 3,5-épinnerase/UDP-4-kéto-L-rhannnose-réductase.
Dans un autre mode de réalisation particulier, le microorganisme recombinant
comprend
une séquence d'acide nucléique hétérologue codant pour une 6-0-
rhannnosyltransferase
(RhaT) et une séquence d'acide nucléique hétérologue codant pour une UDP-
glucose 4,6-
déshydratase/UDP-4-kéto-6-désoxy-D-glucose 3,5-
épinnerase/UDP-4-kéto-L-rhannnose-
réductase (RHM) telles que définies dans le mode de réalisation précédent et
une séquence
d'acide nucléique hétérologue codant pour une flavanone 7-0-bêta-D-
glucosyltransférase
(UGT) d'Arabidopsis thaliana, de Scutellaria baicalensis ou d'Homo sapiens, de
préférence
d'Arabidopsis thaliana ou de Scutellaria baicalensis, de préférence une
flavanone 7-0-bêta-
D-glucosyltransférase (UGT) sélectionnée parmi les enzymes comprenant une
séquence
choisie parmi SEQ ID NOs: 91, 93, 95, 97, 99 et 101 et les polypeptides
comprenant une
séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec
une de ces
séquences et présentant une activité flavanone 7-0-bêta-D-glucosyltransférase
; de
manière particulière une flavanone 7-0-bêta-D-glucosyltransférase (UGT)
comprenant une
séquence choisie parmi SEQ ID NOs: 91, 93, 95 et 97 et les polypeptides
comprenant une
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec
une de ces
séquences et présentant une activité flavanone 7-0-bêta-D-glucosyltransférase.
Dans un mode de réalisation préféré, le microorganisme comprend
- une séquence d'acide nucléique hétérologue codant pour une flavanone 7-0-
5 bêta-D-glucosyltransférase (UGT) sélectionnée parmi les enzymes
comprenant une
séquence choisie parmi SEQ ID NOs: 113, 115 et 95 et les polypeptides
comprenant une
séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec
une de ces
séquences et présentant une activité flavanone 7-0-bêta-D-glucosyltransférase,
de
préférence sélectionnée parmi les enzymes comprenant une séquence choisie
parmi les
10 SEQ ID NOs: 113 et 95 et les polypeptides comprenant une séquence ayant
au moins 60,
70, 80, 85, 90 ou 95 % d'identité de séquence avec l'une de ces séquences et
présentant
une activité flavanone 7-0-bêta-D-glucosyltransférase, et de manière plus
particulièrement
préférée, sélectionnée parmi les enzymes comprenant une séquence choisie parmi
la SEQ
ID NO: 113 et les polypeptides comprenant une séquence ayant au moins 60, 70,
80, 85, 90
15 ou 95 % d'identité de séquence avec cette séquence et présentant une
activité flavanone
7-0-bêta-D-glucosyltransférase; et
- une séquence d'acide nucléique hétérologue codant une 6"-O-
rhamnosyltransferase sélectionnée parmi les enzymes comprenant une séquence
choisie
parmi la SEQ ID NOs: 103 et les polypeptides comprenant une séquence ayant au
moins 60,
20 70, 80, 85, 90 ou 95 % d'identité de séquence avec cette séquence et
présentant une
activité 6"-O-rhamnosyltransferase ; et
- une séquence d'acide nucléique hétérologue codant une UDP-glucose 4,6-
déshyd ratase/U DP-4-kéto-6-désoxy-D-glucose 3,5-
épimerase/U DP-4-kéto-L-rha m nase-
réductase sélectionnée parmi les enzymes comprenant une séquence choisie parmi
la SEQ
25 ID NOs: 107 et les polypeptides comprenant une séquence ayant au moins
60, 70, 80, 85,
90 ou 95 % d'identité de séquence avec cette séquence et présentant une
activité UDP-
glucose 4,6-déshydratase/UDP-4-kéto-6-désoxy-D-glucose 3,5-épimerase/UDP-4-
kéto-L-
rha m nose-réd ucta se.
Dans un mode de réalisation particulier, les enzymes ci-dessus décrites sont
des enzymes
d'eucaryotes supérieurs, de préférence des enzymes de plante. Dans un mode de
réalisation particulier, les enzymes proviennent de plantes du même genre, par
exemple
de même espèce.
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
26
Modifications permettant la production d'hespérétine et/ou de diosmétine
Comme indiqué précédemment, soit l'hespérétine et/ou la diosmétine sont
apportées au
microorganisme, soit le microorganisme est capable de produire l'hespérétine
et/ou la
diosmétine. En particulier, le microorganisme est, ou a été modifié pour être
capable de
produire l'hespérétine et/ou la diosmétine. Les inventeurs ont donc en outre
développé
une voie de biosynthèse permettant au microorganisme de produire l'hespérétine
et/ou la
diosmétine.
De Naringénine/Apigénine à Hespérétine/Diosmétine
L'hespérétine et/ou la diosmétine peuvent être obtenues à partir de la
naringénine et de
l'apigénine.
Plusieurs stratégies de biosynthèse étaient possibles. Pour préparer
l'hespérétine et/ou de
la diosmétine, il est nécessaire de procéder à deux modifications : la
méthylation de
l'hydroxyle en position 4' et l'hydroxylation de la position 3'. Ainsi, pour
augmenter la
spécificité de la méthylation de l'hydroxyle en position 4', il semble logique
de procéder
d'abord à la méthylation du groupe hydroxyle déjà présent avant d'ajouter un
second
groupe hydroxyle en position 3'. Tout au contraire, les inventeurs ont abouti
à la conclusion
qu'il était nécessaire de procéder d'abord à l'hydroxylation puis à la
méthylation, malgré le
risque de problème de spécificité de méthylation dû à l'introduction du
deuxième
hydroxyle.
F3'H : Flayonoïde 3'-monooxygénase
La flavonoïde 3'-monooxygénase (F3'H) est une enzyme qui réalise l'addition
d'un groupe
hydroxyle en position 3' de la naringénine et/ou de l'apigénine. Cette enzyme
appartient à
la classe des EC 1.14.14.82. Elle est également appelée flavone 3'-
hydroxylase.
Les inventeurs ont dû identifier et sélectionner des enzymes capables
d'accepter la
naringénine et/ou l'apigénine comme substrat et d'ajouter un groupe hydroxyle
en
position 3' de ces composés. De préférence, l'enzyme est sélectionnée de sorte
à présenter
une préférence pour une hydroxylation en position 3' de la naringénine et/ou
de
l'apigénine. Dans un mode de réalisation préféré, l'enzyme est spécifique de
la position 3'
de la naringénine et/ou de l'apigénine, en particulier par rapport à la
position 5' afin
d'éviter une double hydroxylation en positions 3' et 5', et de préférence
éviter également
une hydroxylation en position 5'.
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
27
La flavonoïde 3'-nnonooxygénase (F3'H) est une enzyme qui réalise l'addition
d'un groupe
hydroxyle en position 3' de la naringénine et/ou de l'apigénine. Cette enzyme
appartient à
la classe des EC 1.14.14.82. Elle est également appelée flavonoïde 3'-
hydroxylase.
Par activité flavonoïde 3'-nnonooxygénase est entendue la transformation
d'un
flavonoïde en flavonoïde 3' hydroxyle par une enzyme F3'H, CPR dépendante.
Pour
déterminer s'il y a une activité flavonoïde 3'-nnonooxygénase, un test
enzymatique peut
être effectué qui consiste en l'incubation in vitro de l'enzyme flavonoïde 3'
nnonooxygénase
en présence de NAD(P)H, 02, et d'un flavonoïde, dans des conditions optimales
(pH, ions...),
et l'observation en UPLC-MS et en comparaison au standard attendu de
l'apparition d'un
flavonoïde 3'-hydroxylé. De préférence, le flavonoïde est la naringénine ou
l'apigénine et
le flavonoïde 3'-hydroxylé est leur forme 3'-hydroxylé, c'est-à-dire
Eriodictyol ou Lutéoline.
Le microorganisme peut donc comprendre une séquence d'acide nucléique
hétérologue
codant pour une flavonoïde 3'-nnonooxygénase (F3'H) capable d'ajouter un
hydroxyle en
position 3' de la naringénine et/ou de l'apigénine.
Dans un mode de réalisation, la F3'H est une enzyme de plante, notamment de
plantes du
genre Allium, Arabidopsis, Brassica, Callistephus, Columnea, Citrus, Dianthus,
Gentiana,
Gerbera, Glycine, Fragaria, Ipomoea, Malus, Matthiola, Osteospermum, Oryza,
Phanerochaete, Perilla, Petroselinum, Pelargonium, Pilosella, Petunia,
Sinningia, Sorghum,
Torenia, Vitis ou Zea, par exemple de Allium cepa, Arabidopsis thaliana,
Brassica napus,
Columnea hybrida, Callistephus chinensis, Citrus sinensis, Citrus clementina,
Dianthus
caryophyllus, Fragaria vesca, Fragaria x ananassa, Gerbera hybrida, Glycine
max, Gentiana
triflora, Ipomoea nil, Ipomoea purpurea, Ipomoea tricolor, Matthiola incana,
Malus
dom estica, Osteospermum hybrid cultivar, Oryza sativa, Phanerochaete
chrysosporium,
Pilosella officinarum, Petroselinum crispum, Pelargonium x hortorum, Perilla
frutescens var.
crispa, Petunia x hybrida, Sinningia cardinalis, Sorghum bicolor, Torenia sp,
Torenia hybrid
cultivar, Vitis vinifera ou Zea mays. Dans un mode de réalisation plus
spécifique, la F3'H est
une enzyme de plantes du genre Allium, Brassica, Callistephus, Columnea,
Citrus, Dianthus,
Gentiana, Gerbera, Glycine, Fragaria, Ipomoea, Malus, Matthiola, Osteospermum,
Oryza,
Phanerochaete, Perilla, Petroselinum, Pelargonium, Pilosella, Petunia,
Sinningia, Sorghum,
Torenia, Vitis ou Zea, par exemple de Allium cepa, Brassica napus, Columnea
hybrida,
Callistephus chinensis, Citrus sinensis, Citrus clementina, Dianthus
caryophyllus, Fragaria
vesca, Fragaria x ananassa, Gerbera hybrida, Glycine max, Gentiana triflora,
Ipomoea nil,
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
28
lpomoea purpurea, lpomoea tricolor, Matthiola incana, Malus domestica,
Osteospermum
hybrid cultivar, Oryza sativa, Phanerochaete chrysosporium, Pilosella
officinarum,
Petroselinum crispum, Pelargonium x hortorum, Perilla frutescens var. crispa,
Petunia x
hybrida, Sinningia cardinalis, Sorghum bicolor, Torenia sp, Torenia hybrid
cultivar, Vitis
vinifera ou Zea mays.
De préférence, la F3'H est une enzyme de Perilla frutescens var. crispa,
Petunia x hybrida,
Callistephus chinensis, Gerbera hybrida, Citrus clementina, Osteospermum
hybrid cultivar,
Phanerochaete chrysosporium, Streptomyces avermitilis, Citrus sinensis,
Arabidopsis
thaliana ou Pilosella officinarum. En particulier, la F3'H peut être une
enzyme de Perilla
frutescens var. crispa, Petunia x hybrida, Callistephus chinensis, Gerbera
hybrida, Citrus
sinensis et Pilosella officinarum.
Dans un mode de réalisation particulier, la F3'H est sélectionnée parmi les
enzymes
comprenant une séquence choisie parmi SEQ ID NOs: 1, 3, 5, 7, 9, 11, 13, 15,
17, 19, 21 et
121, en particulier parmi SEQ ID NOs: 1, 3, 5, 7, 9, 11, 13, 15, 17, 19 et 21,
et les polypeptides
comprenant une séquence ayant au moins 60, 70,80, 85, 90 ou 95% d'identité de
séquence
avec une de ces séquences et présentant l'activité flavonoïde 3'-
monooxygénase, de
préférence sélectionnée parmi les enzymes comprenant une séquence choisie
parmi SEQ
ID NOs: 1, 5, 7, 11, 17, 19 et 121, en particulier parmi SEQ ID NOs: 1, 5, 7,
11, 17 et 19, et
les polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou
95 %
d'identité avec une de ces séquences et présentant l'activité flavonoïde 3'-
monooxygénase, notamment avec la naringénine et/ou de l'apigénine comme
substrat et
avec une hydroxylation en position 3'. Dans un mode de réalisation
particulier, la F3'H est
une enzyme comprenant une séquence sélectionnée parmi les SEQ ID NOs: 5, 7, 17
et les
polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 %
d'identité avec l'une de ces séquences et présentant l'activité flavonoïde 3'-
monooxygénase.
Dans un mode de réalisation préféré, la F3'H est une enzyme comprenant une
séquence
sélectionnée parmi les SEQ ID NOs: 7, 11, 17 et 121, et les polypeptides
comprenant une
séquence ayant au moins 60, 70, 75, 80, 85, 90 ou 95 % d'identité avec l'une
de ces
séquences et présentant l'activité flavonoïde 3'-monooxygénase. De manière
tout
particulièrement préférée, la F3'H peut être une enzyme comprenant une
séquence
sélectionnée parmi les SEQ ID NOs: 7, 17 et 121 et les polypeptides comprenant
une
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
29
séquence ayant au moins 60, 70, 75, 80, 85, 90 ou 95 %, d'identité avec l'une
de ces
séquences et présentant l'activité flavonoïde 3'-nnonooxygénase
Ainsi, la F3'H peut être de Perilla frutescens var. crispa. Les séquences
nucléiques codant
cette enzyme et séquences protéiques sont décrites dans NCBI sous les numéros
de
référence AB045593.1 et BAB59005.1, respectivement, et plus particulièrement
dans les
SEQ ID Nos : 2 et 1.
La F3'H peut être de Phanerochaete chrysosporium. Les séquences nucléiques
codant cette
enzyme et séquences protéiques sont décrites dans NCBI sous les numéros de
référence
AB597870.1 et BAL05157.1, respectivement, et plus particulièrement dans les
SEQ ID Nos:
4 et 3.
La F3'H peut être de Petunia x hybrida. Les séquences nucléiques codant cette
enzyme et
séquences protéiques sont décrites dans NCBI sous les numéros de référence
AF155332.1
et AAD56282.1, respectivement, et plus particulièrement dans les SEQ ID Nos :
6 et 5.
La F3'H peut être de Callistephus chinensis. Dans un mode de réalisation, les
séquences
nucléiques codant cette enzyme et séquences protéiques sont décrites dans NCBI
sous les
numéros de référence AF313488.1 et AAG49298.1, respectivement, et plus
particulièrement dans les SEQ ID Nos : 8 et 7. Dans un autre mode de
réalisation, les
séquences nucléiques codant cette enzyme et séquences protéiques sont décrites
dans
NCBI sous les numéros de référence AF313489.1 et AAG49299.1, respectivement,
et plus
particulièrement dans les SEQ ID Nos: 10 et 9.
La F3'H peut être de Gerbera hybrida. Les séquences nucléiques codant cette
enzyme et
séquences protéiques sont décrites dans NCBI sous les numéros de référence
DQ218417.1
et ABA64468.1, respectivement, et plus particulièrement dans les SEQ ID Nos :
12 et 11.
La F3'H peut être de Osteospermum hybrid cultivar. Les séquences nucléiques
codant cette
enzyme et séquences protéiques sont décrites dans NCBI sous les numéros de
référence
DQ250711.1 et ABB29899.1, respectivement, et plus particulièrement dans les
SEQ ID Nos:
14 et 13.
La F3'H peut être de Citrus clementina. Les séquences nucléiques codant cette
enzyme et
séquences protéiques sont décrites dans NCBI sous les numéros de référence
XM_006440673.1 et XP_006440736.1, respectivement, et plus particulièrement
dans les
SEQ ID Nos: 16 et 15.
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
La F3'H peut être de Citrus sinensis. Les séquences nucléiques codant cette
enzyme et
séquences protéiques sont décrites dans NCBI sous les numéros de référence
XM_006477592.2 et XP_006477655.1, respectivement, et plus particulièrement
dans les
SEQ ID Nos: 18 et 17.
5 La F3'H peut être de Pilosella officinarum. Les séquences nucléiques
codant cette enzyme
et séquences protéiques sont décrites dans NCBI sous les numéros de référence
DQ319866.2 et ABC47161.1, respectivement, et plus particulièrement dans les
SEQ ID Nos:
20 et 19.
La F3'H peut être de Streptomyces ayermitilis. Les séquences nucléiques codant
cette
10 enzyme et séquences protéiques sont décrites dans NCBI sous les numéros
de référence
SAV_4539 et WP_010985964.1, respectivement, et plus particulièrement dans les
SEQ ID
Nos : 22 et 21.
La F3'H peut être d'Arabidopsis thaliana. Une séquence nucléique codant cette
enzyme et
la séquence protéique sont décrites dans NCBI sous les numéros de référence
15 NM_120881.2 et NP_196416.1, respectivement et plus particulièrement dans
les SEQ ID
NOs: 122 et 121.
De préférence, la F3'H est une enzyme comprenant une séquence sélectionnée
parmi les
SEQ ID NOs: 7, 11, 17 et 121, et les polypeptides comprenant une séquence
ayant au moins
75%, au moins 80%, au moins 85%, au moins 90% ou au moins 95% d'identité avec
l'une de
20 ces séquences et présentant l'activité flavonoïde 3'-monooxygénase. De
manière tout
particulièrement préférée, la F3'H est une enzyme comprenant une séquence
sélectionnée
parmi les SEQ ID NOs: 7, 17 et 121, et les polypeptides comprenant une
séquence ayant au
moins 75%, au moins 80%, au moins 85%, au moins 90% ou au moins 95% d'identité
avec
l'une de ces séquences et présentant l'activité flavonoïde 3'-monooxygénase.
25 Selon un mode de réalisation préféré, la F3'H est une enzyme comprenant
une séquence
choisie parmi SEQ ID NO: 7 et les polypeptides comprenant une séquence ayant
au moins
75 %, au moins 80 %, au moins 85 %, au moins 90 % ou au moins 95 % d'identité
de
séquence avec la séquence SEQ ID NO: 7 et présentant l'activité flavonoïde 3'-
monooxygénase.
30 Selon un autre mode de réalisation particulier, la F3'H est une enzyme
comprenant une
séquence choisie parmi SEQ ID NO: 17 et les polypeptides comprenant une
séquence ayant
au moins 75 %, au moins 80 %, au moins 85 %, au moins 90 % ou au moins 95 %
d'identité
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
31
de séquence avec la séquence SEQ ID NO: 17, et présentant l'activité
flavonoïde 3'-
nnonooxygénase.
Selon un autre mode de réalisation particulier, la F3'H est une enzyme
comprenant une
séquence choisie parmi SEQ ID NO: 121 et les polypeptides comprenant une
séquence
ayant au moins 70%, au moins 75 %, au moins 80%, au moins 85 %, au moins 90%
ou au
moins 95 % d'identité de séquence avec la séquence SEQ ID NO: 121, et
présentant
l'activité flavonoïde 3'-monooxygénase.
Selon un autre mode de réalisation particulier, la F3'H est une enzyme
comprenant une
séquence choisie parmi SEQ ID NO: 11 et les polypeptides comprenant une
séquence ayant
au moins 75 %, au moins 80 %, au moins 85 %, au moins 90 % ou au moins 95 %
d'identité
de séquence avec la séquence SEQ ID NO: 11, et présentant l'activité
flavonoïde 3'-
monooxygénase.
CPR : cytochrome P450 réductase
La flavonoïde 3'-monooxygénase (F3'H) nécessite la présence de NADPH pour
réaliser
l'addition du groupe hydroxyle.
Ainsi, dans un mode de réalisation préféré, le microorganisme comprend un
acide
nucléique hétérologue codant pour une cytochrome P450 réductase, une NADPH-
cytochrome P450 réductase. Cette enzyme appartient à la classe des EC 1.6.2.4.
La cytochrome P450 réductase provient d'un eucaryote, notamment d'une levure,
par
exemple du genre Saccharomycetales, ou d'une plante, par exemple une plante du
genre
Arabidopsis, Ammi, Avicennia, Camellia, Camp totheca, Catharanthus, Citrus,
Glycine,
Helianthus, Lotus, Mesembryanthemum, Phaseolus, Physcomitrella, Pinus,
Populus, Ruta,
Saccharum, Solanum, Vigna, Vitis ou Zea.
Dans un mode de réalisation préféré, la cytochrome P450 réductase provient
d'un
eucaryote, par exemple de levure, en particulier de Saccharomyces cerevisiae,
ou de plante,
par exemple de Catharanthus roseus ou d'Arabidopsis thaliana.
Dans un mode de réalisation particulier, la cytochrome P450 réductase est
sélectionnée
parmi les enzymes comprenant une séquence choisie parmi SEQ ID NOs: 23, 25,
27, 29 et
31 et les polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85,
90 ou 95
% d'identité de séquence avec une de ces séquences et présentant une activité
cytochrome
P450 réductase, de préférence parmi les enzymes comprenant une séquence
choisie parmi
SEQ ID NOs: 23, 25, 29 et 31 et les polypeptides comprenant une séquence ayant
au moins
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
32
60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec une de ces séquences et
présentant
une activité cytochrome P450 réductase. Dans un mode de réalisation très
particulier, la
cytochrome P450 réductase peut être sélectionnée parmi les enzymes comprenant
une
séquence choisie parmi SEQ ID NO: 25 et les polypeptides comprenant une
séquence ayant
au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec cette séquence
et
présentant une activité cytochrome P450 réductase.
Par exemple, la cytochrome P450 réductase peut être de Catharanthus roseus.
Elle est
décrite dans la base de données Genbank de NCBI sous le numéro X69791.1 pour
la
séquence nucléique et sous le numéro CAA49446.1 pour la séquence protéique, et
plus
particulièrement dans les SEQ ID Nos: 24 et 23, respectivement. La protéine
est décrite
dans UniProtKB/Swiss Prot sous le numéro de référence Q05001.
La cytochrome P450 réductase peut être de Saccharomyces cerevisiae. Elle est
décrite dans
la base de données Genbank de NCBI sous le numéro NM_001179172.1 pour la
séquence
nucléique et sous le numéro NP_011908.1 pour la séquence protéique, et plus
particulièrement dans les SEQ ID Nos: 26 et 25, respectivement. La protéine
est décrite
dans UniProtKB/Swiss Prot sous le numéro de référence P16603.
La cytochrome P450 réductase peut être chimérique. Elle est décrite dans
l'article Aigrain
et al (2009, EM BO reports, 10, 742-747. La séquence nucléique codant cette
enzyme et la
séquence protéique sont décrites dans les SEQ ID Nos: 28 et 27,
respectivement.
Par ailleurs, la cytochrome P450 réductase peut être d'Arabidopsis thaliana.
Lorsque la
cytochrome P450 provient d'Arabidopsis thaliana, on peut la nommer l'ATR. Elle
est décrite
dans la base de données Genbank de NCBI sous le numéro NM_118585.4 pour la
séquence
nucléique et sous le numéro NP_194183.1 pour la séquence protéique, et plus
particulièrement dans les SEQ ID Nos : 30 et 29, respectivement. La protéine
est décrite
dans UniProtKB/Swiss Prot sous le numéro de référence Q9SB48.
En outre, la cytochrome P450 réductase peut être d'Arabidopsis thaliana et
être décrite
dans la base de données Genbank de NCBI sous le numéro NM_179141.2 pour la
séquence
nucléique et sous le numéro NP_849472.2 pour la séquence protéique, et plus
particulièrement dans les SEQ ID Nos : 32 et 31, respectivement. La protéine
est décrite
dans UniProtKB/Swiss Prot sous le numéro de référence Q9SUM3.
Dans un mode de réalisation, une nouvelle copie d'une séquence codante pour
CPR comme
définie ci-dessus est introduite dans la levure. Dans un autre mode de
réalisation, lorsque
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
33
la levure est Saccharomyces cerevisiae et que la CPR provient de la même
levure, le
promoteur du gène endogène codant pour CPR est remplacé par un promoteur fort.
Ainsi,
l'expression de la CPR est augmentée par rapport à la levure sauvage ; la CPR
est donc
surexprinnée dans la levure modifiée.
Dans un mode de réalisation particulier, la F3'H et la CPR proviennent de la
même origine,
la même espèce.
OMT : 0-méthyltransférase
Les 0-méthyltransférases (OMT) sont une très large famille d'enzymes
présentant des
cibles difficiles à cerner. Les inventeurs ont dû identifier et sélectionner
des O-
capables de méthyler l'ériodictyol et/ou la lutéoline en position 4'
(position para).
De préférence, l'enzyme a été sélectionnée de sorte à présenter une préférence
pour une
méthylation en position 4' de l'ériodictyol et/ou la lutéoline. Dans un mode
de réalisation
préféré, l'enzyme est spécifique de la position 4' de l'ériodictyol et/ou la
lutéoline. Par
spécifique, est entendu que le groupe méthyle introduit par l'enzyme sur
l'ériodictyol et/ou
la lutéoline se trouve en position 4' dans 60% des cas, le reste étant
introduit en position
3', de préférence dans 70% des cas, et de manière encore préférée dans 80% des
cas.
Par activité 4'0-méthyltransférase est entendue la transformation d'un 4'-
hydroxyflavonoïde en un 4'-méthoxyflavonoïde par une enzyme 4'-0-
méthyltransférase.
Pour déterminer s'il y a une activité 4'0-méthyltransférase, un test
enzymatique peut être
effectué qui consiste en l'incubation in vitro d'un mélange composé de
l'enzyme 4'-0-
méthyltransférase, d'un 4'-hydroxyflavonoïde, de S-adenosyl-L-methionine, dans
les
conditions optimales (pH, température, ions...). Après un certain temps
d'incubation,
l'apparition du 4'-méthoxyflavonoïde est observée en UPLC-MS en comparaison au
standard attendu.
Dans notre cas, le 4'-hydroxyflavonoïde est l'ériodictyol ou la lutéoline et
seront
transformés respectivement en leur forme 4'-méthoxyflavonoïde c'est-à-dire en
hespérétine ou en diosnnétine.
Le microorganisme peut donc comprendre une séquence d'acide nucléique
hétérologue
codant pour une 0-méthyltransférase capable de méthyler l'ériodictyol et/ou la
lutéoline
en position 4'.
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
34
Cette enzyme n'est présente que dans des eucaryotes supérieurs, en particulier
dans les
plantes.
Dans un mode de réalisation, la 0-nnéthyltransférase (OMT) est une enzyme
d'Arabidopsis
thaliana. Dans un autre mode de réalisation, la 0-nnéthyltransférase (OMT)
provient d'un
eucaryote supérieur, de préférence d'un mammifère. En particulier, la 0-
nnéthyltransférase (OMT) est d'origine humaine (Homo sapiens).
Dans un mode de réalisation particulier, l'OMT est sélectionnée parmi les
enzymes
comprenant une séquence choisie parmi SEQ ID NOs: 87 et 89 et les polypeptides
comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité
avec une
de ces séquences et présentant l'activité 0-méthyltransférase, notamment avec
l'ériodictyol et/ou la lutéoline comme substrat et une méthylation en position
4'.
Dans un mode de réalisation, l'OMT est sélectionnée parmi l'enzyme comprenant
une
séquence choisie parmi SEQ ID NO 89 et les polypeptides comprenant une
séquence ayant
au moins 60, 70, 80, 85, 90 ou 95 % d'identité avec cette séquence et
présentant l'activité
0-méthyltransférase.
Dans un autre mode de réalisation, l'OMT est sélectionnée parmi l'enzyme
comprenant
une séquence choisie parmi SEQ ID NO 87 et les polypeptides comprenant une
séquence
ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité avec cette séquence et
présentant
l'activité 0-méthyltransférase.
Ainsi, l'OMT peut être d'Arabidopsis thaliana. Les séquences nucléiques codant
cette
enzyme et séquences protéiques sont décrites dans NCBI sous les numéros de
référence
NM_118755.4 et NP_567739.1, respectivement. La protéine est également décrite
dans
UniProtKB/Swiss Prot sous le numéro de référence Q9C5D7, et plus
particulièrement dans
la SEQ ID No :87.
De manière alternative, l'OMT est d'Homo sapiens. Les séquences nucléiques
codant cette
enzyme et séquences protéiques sont décrites dans NCBI sous les numéros de
référence
NM_007310.2 et NP_009294.1, respectivement. La protéine est également décrite
dans
UniProtKB/Swiss Prot sous le numéro de référence P21964, et plus
particulièrement dans
la SEQ ID No :89.
L'OMT d'Homo sapiens présente l'avantage d'accepter l'ériodictyol et la
lutéoline comme
substrat pour la méthylation alors que l'OMT d'Arabidopsis thaliana présente
une forte
préférence pour l'ériodictyol. A contrario, si la synthèse d'hespérétine est à
favoriser par
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
rapport à celle de diosnnétine, l'OMT d'Arabidopsis thaliana pourrait
présenter un
avantage.
Dans un mode de réalisation préféré, l'OMT est une OMT de Citrus, en
particulier Citrus
clementina ou Citrus sinensis. Dans un mode de réalisation particulièrement
préféré, l'OMT
5 est sélectionnée parmi une enzyme comprenant une séquence choisie parmi
SEQ ID NO
117 et 119 et les polypeptides comprenant une séquence ayant au moins 60, 70,
80, 85, 90
ou 95 % d'identité avec l'une de ces séquences et présentant l'activité 0-
méthyl-
transférase.
De préférence, l'OMT est sélectionnée parmi une enzyme comprenant une séquence
10 choisie parmi SEQ ID NO 117 et les polypeptides comprenant une séquence
ayant au moins
60, 70, 80, 85, 90 ou 95 % d'identité avec cette séquence et présentant
l'activité 0-méthyl-
transférase.
De manière alternative, l'OMT est sélectionnée parmi une enzyme comprenant une
séquence choisie parmi SEQ ID NO 119 et les polypeptides comprenant une
séquence ayant
15 au moins 60, 70, 80, 85, 90 ou 95 % d'identité avec cette séquence et
présentant l'activité
0-méthyl-transférase.
Les OMT de Citrus et d'Arabidopsis thaliana décrites ci-dessus présentent
l'avantage de
méthyler spécifiquement l'ériodictyol en position 4'.
Lors de la conception du microorganisme, les inventeurs ont constaté que cette
étape de
20 méthylation constituait une des étapes limitantes. De manière
surprenante, malgré la
présence du cofacteur S-adénosyl-L-méthionine dans le microorganisme, en
particulier la
levure, l'ajout d'une enzyme augmentant la synthèse de ce cofacteur permettait
de lever
l'aspect limitant de cette étape. Ainsi, dans un mode de réalisation préféré,
le
microorganisme comprend en outre une séquence hétérologue ou endogène codant
pour
25 une enzyme synthétisant la S-adénosyl-L-méthionine, une S-
adénosylméthionine
synthétase (SAMT). Cette enzyme appartient à la classe des EC 2.5.1.6.
Dans un mode de réalisation, le microorganisme comprend une séquence d'acide
nucléique hétérologue codant pour une 0-méthyl-transférase (OMT), en
particulier
capable de méthyler l'ériodictyol et/ou la lutéoline en position 4' et une
séquence d'acide
30 nucléique hétérologue ou endogène codant pour une S-adénosylméthionine
synthétase
(SAMT).
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
36
Dans un mode de réalisation, la SAMT proviennent d'une levure, en particulier
de
Saccharomyces cerevisiae, tout particulièrement lorsque le microorganisme est
une levure.
Dans un mode de réalisation particulier, la S-adénosylnnéthionine synthétase
est une
enzyme comprenant une séquence choisie parmi SEQ ID NO: 81 et un polypeptide
comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité
avec cette
séquence et présentant l'activité S-adénosylméthionine synthétase.
Par exemple, la S-adénosylméthionine synthétase peut être de Saccharomyces
cerevisiae.
Elle est décrite dans la base de données Genbank de NCBI sous le numéro
NM_001180810.3 pour la séquence nucléique et sous le numéro NP_010790.3 pour
la
séquence protéique. La protéine est décrite dans UniProtKB/Swiss Prot sous le
numéro de
référence P19358.
Dans un mode de réalisation, une nouvelle copie d'une séquence codante pour
SAMT
comme définie ci-dessus est introduite dans le microorganisme. Dans un autre
mode de
réalisation, lorsque le microorganisme est Saccharomyces cerevisiae, le
promoteur du gène
endogène codant pour SAMT est remplacé par un promoteur fort. Ainsi,
l'expression de la
SAMT est augmentée par rapport au microorganisme sauvage ; la SAMT est donc
surexprimée dans le microorganisme modifié.
Ainsi, dans un mode de réalisation préféré, le microorganisme comprend donc
une
séquence d'acide nucléique hétérologue codant pour une 0-nnéthyltransférase
capable de
méthyler l'ériodictyol et/ou la lutéoline en position 4' et une séquence
hétérologue ou
endogène codant pour une S-adénosylméthionine synthétase (SAMT) capable de
produire
la S-adénosyl-L-méthionine.
FNS : Flavone synthase
La diosmétine peut être produite à partir de la lutéoline. Elle peut également
être obtenue
à partir de l'ériodictyol, soit en le transformant en lutéoline puis en
préparant la diosmétine
à partir de la lutéoline, soit en le transformant en hespérétine puis en
préparant la
diosmétine à partir de l'hespérétine. L'enzyme capable de transformer
l'ériodictyol en
lutéoline et/ou l'hespérétine en diosmétine est une flavone synthase (FNS).
Dans un mode
de réalisation particulier, la flavone synthase est capable également de
transformer
l'ériodictyol en lutéoline.
Ainsi, le microorganisme peut donc comprendre une séquence d'acide nucléique
hétérologue codant pour une flavone synthase, en particulier une flavone
synthase capable
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
37
de produire la lutéoline à partir de l'ériodictyol et/ou la diosnnétine à
partir de
I' hespérétine.
Par activité flavone synthase est entendue la transformation d'une
flavanone en
flavone par une enzyme FNSI (CPR indépendante) ou une FNSII (CPR dépendante).
Pour déterminer s'il y a une activité flavone synthase, un test enzymatique
peut être
effectué qui consiste en l'incubation in vitro dans le cas de la FNSI d'un
mélange composé
de l'enzyme flavone synthase (FNSI), d'une flavanone, de 2-oxoglutarate, d'Oz,
dans les
conditions optimales (pH, température, ions...) et dans le cas de la FNSII
d'un mélange
composé de l'enzyme FNSII, d'une flavanone, de NAD(P)H, d'02, dans les
conditions
optimales (pH, température, ions...). Après un certain temps d'incubation,
l'apparition de
la flavone correspondante à la flavanone est observée en UPLC-MS en
comparaison au
standard attendu. De préférence, la flavanone est l'ériodictyol ou
l'hespérétine et seront
transformés respectivement en leur forme flavone c'est-à-dire en lutéoline ou
diosnnétine.
Ainsi, dans un mode de réalisation particulier, le microorganisme comprend une
séquence
d'acide nucléique hétérologue codant pour une 0-nnéthyl-transférase (OMT), en
particulier
capable de nnéthyler l'ériodictyol et/ou la lutéoline en position 4' ; et une
séquence d'acide
nucléique hétérologue codant pour une flavone synthase, en particulier une
flavone
synthase capable de produire la lutéoline à partir de l'ériodictyol et/ou la
diosnnétine à
partir de l'hespérétine.
De préférence, la flavone synthase est une enzyme provenant de plante, par
exemple du
genre Aethusa, Angelica, Antirrhinum, Apium, Arabidopsis, Callistephus,
Camellia, Conium,
Cuminum, Cynara, Dahlia, Dorcoceras, Erythranthe, Lonicera, Medicago, Oryza,
Perilla,
Petroselinum, Plectranthus, Populus, Saussurea, Scutellaria ou Zea, en
particulier du genre
Arabidopsis, Lonicera, Medicago, Oryza, Petroselinum, Populus ou Zea notamment
d'Arabidopsis thaliana, Lonicera japonica, Lonicera macranthoides, Medicago
truncatula,
Oryza sativa, Petroselinum crispum, Populus deltoides, Zea mays, Callistephus
chinensis,
Apium graveolens, Cuminum cyminum, Aethusa cynapium, Angelico archangelica,
Conium
maculatum, Camellia sinensis, Cynara cardunculus var scolymus, Saussurea
medusa,
Plectranthus barbatus, Scutellaria baicalensis, Dorcoceras hygrometricum,
Antirrhinum
malus, Perilla frutescens var crispa, Dahlia pinnata ou Erythranthe lewisii,
en particulier
d'Arabidopsis thaliana, Lonicera japonica, Lonicera macranthoides, Medicago
truncatula,
Oryza sativa, Petroselinum crispum, Populus deltoides, ou Zea mays, de
préférence de
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
38
Petroselinum crispum, ou du genre Lonicera comme par exemple Lonicera japonica
et
Lonicera macranthoides.
Dans un mode de réalisation particulier, la flavone synthase (FNS) est
sélectionnée parmi
les enzymes comprenant une séquence choisie parmi SEQ ID NOs: 33, 35, 37, 127,
129, 131,
133, 135, 137, 139, 141, 143, 145, 147, 149, 151, 153, 155, 157 et 159 et les
polypeptides
comprenant une séquence ayant au moins 60, 70,80, 85, 90 ou 95% d'identité de
séquence
avec une de ces séquences et présentant une activité flavone synthase. En
particulier, la
flavone synthase (FNS) est sélectionnée parmi les enzymes comprenant une
séquence
choisie parmi SEQ ID NOs: 33, 35, 37, et les polypeptides comprenant une
séquence ayant
au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec une de ces
séquences et
présentant une activité flavone synthase. De préférence, la FNS est
sélectionnée parmi les
enzymes comprenant une séquence choisie parmi SEQ ID NOs: 37 et les
polypeptides
comprenant une séquence ayant au moins 60, 70,80, 85, 90 ou 95% d'identité de
séquence
avec cette séquence et présentant une activité flavone synthase.
II existe deux sortes de flavone synthase (FNS) : la flavone synthase 1 (FNSI)
et la flavone
synthase 2 (FNSII). A partir d'une flavanone et de 2-oxoglutarate, la FNSI est
capable de
produire la flavone correspondante. L'enzyme FNSI appartient à la classe des
EC 1.14.11.22.
La FNSII appartient au groupe de P450 et nécessite la présence d'une
cytochrome P450
réductase. L'enzyme FNSII appartient à la classe des EC 1.14.13.
Dans un mode de réalisation, la FNS est une flavone synthase de type I. Dans
un autre mode
de réalisation, la FNS est une flavone synthase de type II. Dans un mode de
réalisation
supplémentaire, le microorganisme comprend une flavone synthase de type I et
une
flavone synthase de type II.
Dans un mode de réalisation préféré, le microorganisme comprend une séquence
d'acide
nucléique hétérologue codant pour une flavone synthase de type I (FNSI).
L'avantage de la
FNSI est qu'elle fonctionne sans cytochrome P450 réductase.
La FNSI peut être une flavone synthase de plante comme Petroselinum crispum,
Oryza
sativa, Populus deltoides, Medicago truncatula, Apium graveolens, Cuminum
cyminum,
Aethusa cynapium, Angelico archangelica, ou Conium maculatum, en particulier
de
Petroselinum crispum, Oryza sativa, Populus deltoides, Medicago truncatula, de
préférence
de Petroselinum crispum.
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
39
La FNSI peut être une enzyme comprenant une séquence choisie parmi les SEQ ID
NOs: 37,
127, 137, 141, 143 et 145 et un polypeptide comprenant une séquence ayant au
moins 60,
70, 80, 85, 90 ou 95 % d'identité de séquence avec l'un de ces séquences et
présentant une
activité flavone synthase. Dans un aspect particulier,la FNSI peut être une
enzyme
comprenant une séquence choisie parmi SEQ ID NO: 37 et un polypeptide
comprenant une
séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec
cette
séquence et présentant une activité flavone synthase.
Par exemple, la FNSI peut être de Petroselinum crispum. Elle est décrite dans
la base de
données Genbank de NCBI sous le numéro AY817680.1 pour la séquence nucléique
et sous
le numéro AAX21541.1 pour la séquence protéique. La protéine est décrite dans
UniProtKB/Swiss Prot sous le numéro de référence Q7XZQ8. Les séquences en
acides
aminés et en acides nucléiques sont décrites dans les SEQ ID Nos : 37 et 38,
respectivement.
La FNSI peut également être d'Angelico archangelica. Elle est décrite dans la
base de
données Genbank de NCBI sous le numéro DQ683352.1 pour la séquence nucléique
et sous
le numéro ABG78793.1 pour la séquence protéique. Les séquences en acides
aminés et en
acides nucléiques sont décrites dans les SEQ ID Nos : 127 et 128,
respectivement.
La FNSI peut également être d'Apium graveolens. Elle est décrite dans la base
de données
Genbank de NCBI sous le numéro AY817676.1 pour la séquence nucléique et sous
le
numéro AAX21537.1 pour la séquence protéique. Les séquences en acides aminés
et en
acides nucléiques sont décrites dans les SEQ ID Nos: 137 et 138,
respectivement.
La FNSI peut également être de Cuminum cyminum. Elle est décrite dans la base
de données
Genbank de NCBI sous le numéro DQ683349.1 pour la séquence nucléique et sous
le
numéro ABG78790.1 pour la séquence protéique. Les séquences en acides aminés
et en
acides nucléiques sont décrites dans les SEQ ID Nos : 141 et 142,
respectivement.
La FNSI peut également être d'Aethusa cynapium. Elle est décrite dans la base
de données
Genbank de NCBI sous le numéro DQ683350.1 pour la séquence nucléique et sous
le
numéro DQ683350.1 pour la séquence protéique. Les séquences en acides aminés
et en
acides nucléiques sont décrites dans les SEQ ID Nos : 143 et 144,
respectivement.
La FNSI peut également être de Conium maculatum. Elle est décrite dans la base
de
données Genbank de NCBI sous le numéro DQ683354.1 pour la séquence nucléique
et sous
le numéro ABG78795.1 pour la séquence protéique. Les séquences en acides
aminés et en
acides nucléiques sont décrites dans les SEQ ID Nos : 145 et 146,
respectivement.
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
Dans un autre mode de réalisation, le microorganisme comprend une séquence
d'acide
nucléique hétérologue codant pour une flavone synthase de type II (FNSII).
La FNSII peut être une flavone synthase de plante, par exemple d'Arabidopsis
thaliana, Zea
5 mays, du genre Lonicera comme par exemple Lonicera japonica et Lonicera
macranthoides,
Callistephus chinensis, Medicago truncatula, Camellia sinensis, Cynara
cardunculus var
scolym us, Saussurea medusa, Plectranthus barbatus, Scutellaria baicalensis,
Dorcoceras
hygrometricum, Antirrhinum majus, Perilla frutescens var crispa, Dahlia
pinnata ou
Erythranthe lewisii, en particulier une flavone synthase d'Arabidopsis
thaliana, Zea mays
10 ou du genre Lonicera comme par exemple Lonicera japonica et Lonicera
macranthoides.
Dans un mode de réalisation particulier, la flavone synthase (FNSII) est
sélectionnée parmi
les enzymes comprenant une séquence choisie parmi SEQ ID NOs: 33, 35, 129,
131, 133,
135, 139, 147, 149, 151, 153, 155, 157 et 159 et les polypeptides comprenant
une séquence
ayant au moins 60,70, 80, 85, 90 ou 95% d'identité de séquence avec une de ces
séquences
15 et présentant une activité flavone synthase, de préférence parmi les
enzymes comprenant
une séquence choisie parmi SEQ ID NOs: 33 et 35 et les polypeptides comprenant
une
séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec
une de ces
séquences et présentant une activité flavone synthase.
Dans un mode de réalisation, la flavone synthase FNS est une FNSII provenant
de Lonicera
20 japonica. Dans ce mode de réalisation, l'enzyme peut être une enzyme
décrite dans la base
de données Genbank de NCBI sous le numéro KU127576.1 pour la séquence
nucléique et
sous le numéro AMQ91109.1 pour la séquence protéique, et plus particulièrement
dans les
SEQ ID Nos : 34 et 33, respectivement.
Dans un autre mode de réalisation, la flavone synthase FNS est une FNSII
provenant de
25 Lonicera macranthoides. Les séquences nucléiques codant cette enzyme et
séquences
protéiques sont décrites dans NCBI sous les numéros de référence KU127580.1 et
AMQ91113.1, respectivement, et plus particulièrement dans les SEQ ID Nos: 36
et 35,
respectivement.
Dans un autre mode de réalisation, la flavone synthase FNS est une FNSII
provenant de
30 Cynara cardunculus var scolymus. Les séquences nucléiques codant cette
enzyme et
séquences protéiques sont décrites dans NCBI sous les numéros de référence
JN825735.1
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
41
et AFG31000.1, respectivement, et plus particulièrement dans les SEQ ID Nos:
130 et 129,
respectivement.
Dans un autre mode de réalisation, la flavone synthase FNS est une FNSII
provenant de
Perilla frutescens var crispa. Les séquences nucléiques codant cette enzyme et
séquences
protéiques sont décrites dans NCBI sous les numéros de référence AB045592.1 et
BAB59004.1, respectivement, et plus particulièrement dans les SEQ ID Nos: 132
et 131,
respectivement.
Dans un autre mode de réalisation, la flavone synthase FNS est une FNSII
provenant de
Dahlia pinnata. Les séquences nucléiques codant cette enzyme et séquences
protéiques
sont décrites dans NCBI sous les numéros de référence AB769842.1 et
BAM72335.1,
respectivement, et plus particulièrement dans les SEQ ID Nos: 134 et 133,
respectivement.
Dans un autre mode de réalisation, la flavone synthase FNS est une FNSII
provenant de
Callistephus chinensis. Les séquences nucléiques codant cette enzyme et
séquences
protéiques sont décrites dans NCBI sous les numéros de référence AF188612.1 et
AAF04115.1, respectivement, et plus particulièrement dans les SEQ ID Nos : 136
et 135,
respectivement.
Dans un autre mode de réalisation, la flavone synthase FNS est une FNSII
provenant de
Medicago truncatula. Les séquences nucléiques codant cette enzyme et séquences
protéiques sont décrites dans NCBI sous les numéros de référence DQ354373.1 et
ABC86159.1, respectivement, et plus particulièrement dans les SEQ ID Nos: 140
et 139,
respectivement.
Dans un autre mode de réalisation, la flavone synthase FNS est une FNSII
provenant de
Camellia sinensis. Les séquences nucléiques codant cette enzyme et séquences
protéiques
sont décrites dans NCBI sous les numéros de référence FJ169499.1 et
ACH99109.1,
respectivement, et plus particulièrement dans les SEQ ID Nos: 148 et 147,
respectivement.
Dans un autre mode de réalisation, la flavone synthase FNS est une FNSII
provenant de
Saussurea medusa. Les séquences nucléiques codant cette enzyme et séquences
protéiques sont décrites dans NCBI sous les numéros de référence KF170286.1 et
AGV40781.1, respectivement, et plus particulièrement dans les SEQ ID Nos: 150
et 149,
respectivement.
Dans un autre mode de réalisation, la flavone synthase FNS est une FNSII
provenant de
Plectranthus barbatus. Les séquences nucléiques codant cette enzyme et
séquences
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
42
protéiques sont décrites dans NCBI sous les numéros de référence KF606861.1 et
AHJ89438.1, respectivement, et plus particulièrement dans les SEQ ID Nos: 152
et 151,
respectivement.
Dans un autre mode de réalisation, la flavone synthase FNS est une FNSII
provenant de
Scutellaria baicalensis. Les séquences nucléiques codant cette enzyme et
séquences
protéiques sont décrites dans NCBI sous les numéros de référence KT963454.1 et
AMW91729.1, respectivement, et plus particulièrement dans les SEQ ID Nos: 154
et 153,
respectivement.
Dans un autre mode de réalisation, la flavone synthase FNS est une FNSII
provenant de
Dorcoceras hygrometricum. Les séquences nucléiques codant cette enzyme et
séquences
protéiques sont décrites dans NCBI sous les numéros de référence KV013332.1 et
KZV23934.1, respectivement, et plus particulièrement dans les SEQ ID Nos: 156
et 155,
respectivement.
Dans un autre mode de réalisation, la flavone synthase FNS est une FNSII
provenant de
Antirrhinum majus. Les séquences nucléiques codant cette enzyme et séquences
protéiques sont décrites dans NCBI sous les numéros de référence AB028151.1 et
BAA84071.1, respectivement, et plus particulièrement dans les SEQ ID Nos: 158
et 157,
respectivement.
Dans un autre mode de réalisation, la flavone synthase FNS est une FNSII
provenant de
Erythranthe lewisii. Les séquences nucléiques codant cette enzyme et séquences
protéiques sont décrites dans NCBI sous les numéros de référence KX710102.1 et
A0R81894.1, respectivement, et plus particulièrement dans les SEQ ID Nos: 160
et 159,
respectivement.
Dans un mode de réalisation particulier, le microorganisme comprend une
séquence
d'acide nucléique hétérologue codant pour une flavone synthase de type II
(FNSII) et une
flavone synthase de type I, par exemple une séquence choisie parmi SEQ ID NOs:
33, 35,
129, 131, 133, 135, 139, 147, 149, 151, 153, 155, 157 et 159 et les
polypeptides comprenant
une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence
avec une
de ces séquences et présentant une activité flavone synthase et une enzyme
comprenant
une séquence choisie parmi SEQ ID NOs: 37, 127, 137, 141, 143 et 145 et un
polypeptide
comprenant une séquence ayant au moins 60, 70,80, 85, 90 ou 95% d'identité de
séquence
avec cette séquence et présentant une activité flavone synthase, de préférence
une
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
43
séquence choisie parmi SEQ ID NOs: 33 et 35 et les polypeptides comprenant une
séquence
ayant au moins 60,70, 80, 85, 90 ou 95% d'identité de séquence avec une de ces
séquences
et présentant une activité flavone synthase et une enzyme comprenant une
séquence
choisie parmi SEQ ID NO: 37 et un polypeptide comprenant une séquence ayant au
moins
60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec cette séquence et
présentant une
activité flavone synthase.
Les FNS de type II, FNSII, nécessite la présence d'une cytochrome P450
réductase (CPR). Si
le microorganisme ne comprend pas de cytochrome P450 réductase, il sera donc
nécessaire
d'introduire une cytochrome P450 réductase hétérologue. Si le microorganisme
en
comprend déjà une, il est possible d'envisager soit la surexpression d'une
cytochrome P450
réductase endogène (par exemple en remplaçant le promoteur par un promoteur
fort ou
en ajoutant une ou plusieurs copies de la séquence codante) ou d'en introduire
également
une cytochrome P450 réductase hétérologue.
Dans un mode de réalisation particulier, la FNS de type II et la CPR
proviennent de la même
origine, la même espèce.
Combinaison d'enzymes
Ainsi, outre les enzymes nécessaires à la biosynthèse de l'hespéridine et/ou
la diosmine à
partir de l'hespéritine et/ou la diosmétine, respectivement, telles que
décrites
précédemment, le microorganisme comprend de préférence les enzymes permettant
la
production de l'hespéritine et/ou la diosnnétine à partir de la naringénine
et/ou de
l'apigénine.
Dans un premier mode de réalisation particulier, le microorganisme recombinant
comprend :
- une
séquence d'acide nucléique hétérologue codant pour une flavanone 7-0-bêta-
D-glucosyltransférase (UGT) capable d'ajouter un glucose en position 7 de
l'hespérétine et/ou la diosmétine ; de préférence une flavanone 7-0-bêta-D-
glucosyltransférase (UGT) d'Arabidopsis thaliana, de Citrus sinensis, de
Citrus
clementina, de Scutellaria baicalensis ou d'Homo sapiens, de préférence de
Citrus
sinensis ou de Scutellaria baicalensis ; de préférence une flavanone 7-0-bêta-
D-
glucosyltransférase (UGT) sélectionnée parmi les enzymes comprenant une
séquence choisie parmi SEQ ID NOs: 113, 115, 91, 93, 95, 97, 99 et 101 et les
polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 %
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
44
d'identité de séquence avec une de ces séquences et présentant une activité
flavanone 7-0-bêta-D-glucosyltransférase ; de manière particulière une
flavanone
7-0-bêta-D-glucosyltransférase (UGT) comprenant une séquence choisie parmi SEQ
ID NOs: 113, 115, 91, 93, 95 et 97 et les polypeptides comprenant une séquence
ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec une de
ces
séquences et présentant une activité flavanone 7-0-bêta-D-glucosyltransférase
;
- une séquence d'acide nucléique hétérologue codant pour une 6-0-
rhamnosyltransferase (RhaT) capable de transférer un rhamnose en position 6 du
glucose de l'hespérétine-7-0-glucoside et/ou la diosmétine-7-0-glucoside; de
préférence une RhaT du genre Citrus ou de Petunia hybrida, de préférence
Citrus
sinensis, Citrus maxima, ou Citrus clementina, de manière encore plus préférée
Citrus sinensis ou Citrus clementina, de préférence une 6-0-
rhamnosyltransferase
(RhaT) comprenant une séquence choisie parmi SEQ ID NOs: 103, 105 et les
polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 %
d'identité de séquence avec une de ces séquences et présentant une activité 6-
0-
rhamnosyltransferase ;
- une séquence d'acide nucléique hétérologue codant pour une UDP-glucose 4,6-
déshydratase/UDP-4-kéto-6-désoxy-D-glucose 3,5-
épimerase/UDP-4-kéto-L-
rhamnose-réductase (RHM) capable de produire de l'UDP-rhamnose ; de
préférence une RHM de Citrus sinensis ou d'Arabidopsisthaliana, de préférence
une
RHM comprenant une séquence choisie parmi SEQ ID NOs: 107, 109 et 111 et les
polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 %
d'identité de séquence avec une de ces séquences et présentant une activité
UDP-
glucose 4,6-déshydratase/UDP-4-kéto-6-désoxy-D-glucose 3,5-épimerase/UDP-4-
kéto-L-rhamnose-réductase, en particulier une RHM comprenant une séquence
sélectionnée parmi SEQ ID NOs: 107, 109 et les polypeptides comprenant une
séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec
cette séquence et présentant une activité UDP-glucose 4,6-déshydratase/UDP-4-
kéto-6-désoxy-D-glucose 3,5-épimerase/UDP-4-kéto-L-rhamnose-réductase ;
- une séquence d'acide nucléique hétérologue codant pour une flavonoïde 3'-
monooxygénase (F3'H) capable d'hydroxyler en position 3' la naringénine et/ou
l'apigénine ; en particulier capable d'hydroxyler en position 3' la
naringénine et/ou
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
l'apigénine ; de préférence de Perilla frutescens var. crispa, Petunia x
hybrida,
Callistephus chinensis, Gerbera hybrida, Citrus sinensis, Citrus clementina,
Osteospermum hybrid cultivar, Phanerochaete chrysosporium, Streptomyces
avermitilis ou Pilosella officinarum, en particulier de Perilla frutescens
var. crispa,
5 Petunia x hybrida, Callistephus chinensis, Gerbera hybrida, Citrus
sinensis ou
Pilosella officinarum, de préférence une FIH comprenant une séquence choisie
parmi SEQ ID NOs: 1, 3, 5, 7, 9, 11, 13, 15, 17, 19, 21 et 121 et les
polypeptides
comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité
de
séquence avec une de ces séquences et présentant une activité flavonoïde 3'-
10 nnonooxygénase, de préférence sélectionnée parmi les enzymes
comprenant une
séquence choisie parmi SEQ ID NOs: 1, 5, 7, 11, 17, 19 et 121 et les
polypeptides
comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité
de
séquence avec une de ces séquences et présentant une activité flavonoïde 3'-
nnonooxygénase, en particulier une F3'H comprenant une séquence sélectionnée
15 parmi les SEQ ID NOs: 7, 11, 17 et 121 et les polypeptides
comprenant une séquence
ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec l'une de
ces
séquences et présentant une activité flavonoïde 3'-nnonooxygénase ;
- facultativement, une séquence d'acide nucléique hétérologue codant pour une
cytochrome P450 réductase (CPR) ; de préférence une CPR de Saccharomyces
20 cerevisiae, ou de plante, par exemple de Catharanthus roseus ou
d'Arabidopsis
thaliana ; de préférence une CPR comprenant une séquence choisie parmi SEQ ID
NOs: 23, 25, 27, 29 et 31 et les polypeptides comprenant une séquence ayant au
moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec une de ces
séquences
et présentant une activité cytochrome P450 réductase, de préférence parmi les
25 enzymes comprenant une séquence choisie parmi SEQ ID NOs: 23, 25, 29
et 31 et
les polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou
95
% d'identité de séquence avec une de ces séquences et présentant une activité
cytochrome P450 réductase;
- une séquence d'acide nucléique hétérologue codant pour une 0-
nnéthyltransférase
30 (OMT) capable de nnéthyler l'ériodictyol et/ou la lutéoline en
position 4'; de
préférence une OMT d'Arabidopsis thaliana ou d'Homo sapiens, de préférence une
OMT comprenant une séquence choisie parmi SEQ ID NOs: 117, 119, 87 et 89 et
les
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
46
polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 %
d'identité de séquence avec une de ces séquences et présentant une activité 0-
nnéthyltransférase, notamment avec l'ériodictyol et/ou la lutéoline comme
substrat
et une nnéthylation en position 4', de préférence sélectionnée parmi l'enzyme
comprenant une séquence choisie parmi SEQ ID NO: 117, 119 et 89 et les
polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 %
d'identité de séquence avec l'une de ces séquences et présentant une activité
0-
nnéthyltra nsférase ; et
- facultativement, une séquence d'acide nucléique hétérologue codant pour une
flavone synthase (FNS) capable de produire une flavone à partir d'une
flavanone,
en particulier capable de transformer la naringénine en apigénine, et/ou
l'ériodictyol en lutéoline, de préférence de transformer l'ériodictyol en
lutéoline;
de préférence une FNS d'Arabidopsis thaliana, Lonicera japonica, Lonicera
macranthoides, Medicago truncatula, Oryza sativa, Petroselinum crispum,
Populus
deltoides, Zea mays, Callistephus chinensis, Apium graveolens, Cuminum
cyminum,
Aethusa cynapium, Angelico archangelica, Conium maculatum, Camellia sinensis,
Cynara cardunculus var scolymus, Saussurea medusa, Plectranthus barbatus,
Scutellaria baicalensis, Dorcoceras hygrometricum, Antirrhinum malus, Perilla
frutescens var crispa, Dahlia pinnata ou Erythranthe lewisii, en particulier
d'Arabidopsis thaliana, Lonicera japonica, Lonicera macranthoides, Medicago
truncatula, Oryza sativa, Petroselinum crispum, Populus deltoides ou Zea mays,
de
préférence de Lonicera japonica, Lonicera macranthoides, et Petroselinum
crispum;
de préférence une FNS comprenant une séquence choisie parmi SEQ ID NOs: 33,
35, 37, 127, 129, 131, 133, 135, 137, 139, 141, 143, 145, 147, 149, 151, 153,
155,
157 et 159 et les polypeptides comprenant une séquence ayant au moins 60, 70,
80, 85, 90 ou 95 % d'identité de séquence avec une de ces séquences et
présentant
une activité flavone synthase, de préférence parmi les enzymes comprenant une
séquence choisie parmi SEQ ID NOs: 33, 35 et 37 et les polypeptides comprenant
une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence
avec une de ces séquences et présentant une activité flavone synthase, de
préférence une flavone synthase (FNS) comprenant une séquence choisie parmi
SEQ ID NO: 37 et un polypeptide comprenant une séquence ayant au moins 60, 70,
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
47
80, 85, 90 ou 95 % d'identité de séquence avec cette séquence et présentant
une
activité flavone synthase.
Dans un autre mode de réalisation particulier, le microorganisme recombinant
comprend :
- une séquence d'acide nucléique hétérologue codant pour une flavanone 7-0-
bêta-
D-glucosyltransférase (UGT) capable d'ajouter un glucose en position 7 de
l'hespérétine et/ou la diosmétine ; de préférence une flavanone 7-0-bêta-D-
glucosyltransférase (UGT) d'Arabidopsis thaliana, de Scutellaria baicalensis
ou
d'Homo sapiens, de préférence d'Arabidopsis thaliana ou de Scutellaria
baicalensis ; de préférence une flavanone 7-0-bêta-D-glucosyltransférase (UGT)
sélectionnée parmi les enzymes comprenant une séquence choisie parmi SEQ ID
NOs: 91, 93, 95, 97, 99 et 101 et les polypeptides comprenant une séquence
ayant
au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec une de ces
séquences et présentant une activité flavanone 7-0-bêta-D-glucosyltransférase
; de
manière particulière une flavanone 7-0-bêta-D-glucosyltransférase (UGT)
comprenant une séquence choisie parmi SEQ ID NOs: 91, 93, 95 et 97 et les
polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 %
d'identité de séquence avec une de ces séquences et présentant une activité
flavanone 7-0-bêta-D-glucosyltransférase ;
- une séquence d'acide nucléique hétérologue codant pour une 6-0-
rhamnosyltransferase (RhaT) capable de transférer un rhamnose en position 6 du
glucose de l'hespérétine-7-0-glucoside et/ou la diosmétine-7-0-glucoside; de
préférence une RhaT du genre Citrus ou de Petunia hybrida, de préférence
Citrus
sinensis, Citrus maxima, ou Citrus clementina, de manière encore plus préférée
Citrus sinensis ou Citrus clementina, de préférence une 6-0-
rhamnosyltransferase
(RhaT) comprenant une séquence choisie parmi SEQ ID NOs: 103, 105 et les
polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 %
d'identité de séquence avec une de ces séquences et présentant une activité 6-
0-
rhamnosyltransferase ;
- une séquence d'acide nucléique hétérologue codant pour une UDP-glucose
4,6-
déshydratase/UDP-4-kéto-6-désoxy-D-glucose 3,5-
épimerase/UDP-4-kéto-L-
rhamnose-réductase (RHM) capable de produire de l'UDP-rhamnose ; de
préférence une RHM de Citrus sinensis ou d'Arabidopsis thaliana, de préférence
une
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
48
RHM comprenant une séquence choisie parmi SEQ ID NOs: 107, 109 et 111 et les
polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 %
d'identité de séquence avec une de ces séquences et présentant une activité
UDP-
glucose 4,6-déshydratase/UDP-4-kéto-6-désoxy-D-glucose 3,5-épinnerase/UDP-4-
kéto-L-rhannnose-réductase, en particulier une RHM comprenant une séquence
sélectionnée parmi SEQ ID NOs: 107, 109 et les polypeptides comprenant une
séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec
cette séquence et présentant une activité UDP-glucose 4,6-déshydratase/UDP-4-
kéto-6-désoxy-D-glucose 3,5-épinnerase/UDP-4-kéto-L-rhannnose-réductase ;
- une séquence d'acide nucléique hétérologue codant pour une flavonoïde 3'-
nnonooxygénase (F3'H) capable d'hydroxyler en position 3' la naringénine et/ou
l'apigénine ; en particulier capable d'hydroxyler en position 3' la
naringénine et/ou
l'apigénine ; de préférence de Perilla frutescens var. crispa, Petunia x
hybrida,
Callistephus chinensis, Gerbera hybrida, Citrus sinensis, et Pilosella
officinarum, de
préférence une FIH comprenant une séquence choisie parmi SEQ ID NOs: 1, 3, 5,
7, 9, 11, 13, 15, 17, 19 et 21 et les polypeptides comprenant une séquence
ayant au
moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec une de ces
séquences
et présentant une activité flavonoïde 3'-nnonooxygénase, de préférence
sélectionnée parmi les enzymes comprenant une séquence choisie parmi SEQ ID
NOs: 1, 5, 7, 11, 17 et 19 et les polypeptides comprenant une séquence ayant
au
moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec une de ces
séquences
et présentant une activité flavonoïde 3'-nnonooxygénase, en particulier une
F3'H
comprenant une séquence sélectionnée parmi les SEQ ID NOs: 5, 7, 17 et les
polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 %
d'identité de séquence avec cette séquence et présentant une activité
flavonoïde
3'-nnonooxygénase ;
- facultativement, une séquence d'acide nucléique hétérologue codant pour une
cytochrome P450 réductase (CPR) ; de préférence une CPR de Saccharomyces
cerevisiae, ou de plante, par exemple de Catharanthus roseus ou d'Arabidopsis
thaliana ; de préférence une CPR comprenant une séquence choisie parmi SEQ ID
NOs: 23, 25, 27, 29 et 31 et les polypeptides comprenant une séquence ayant au
moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec une de ces
séquences
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
49
et présentant une activité cytochrome P450 réductase, de préférence parmi les
enzymes comprenant une séquence choisie parmi SEQ ID NOs: 23, 25, 29 et 31 et
les polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou
95
% d'identité de séquence avec une de ces séquences et présentant une activité
cytochrome P450 réductase ;
- une séquence d'acide nucléique hétérologue codant pour une 0-
nnéthyltransférase
(OMT) capable de nnéthyler l'ériodictyol et/ou la lutéoline en position 4'; de
préférence une OMT d'Arabidopsis thallana ou d'Homo sapiens, de préférence une
OMT comprenant une séquence choisie parmi SEQ ID NOs: 87 et 89 et les
polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 %
d'identité de séquence avec une de ces séquences et présentant une activité 0-
nnéthyltransférase, notamment avec l'ériodictyol et/ou la lutéoline comme
substrat
et une nnéthylation en position 4', de préférence sélectionnée parmi l'enzyme
comprenant une séquence SEQ ID NO 89 et les polypeptides comprenant une
séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec
cette séquence et présentant une activité 0-nnéthyltransférase ; et
- facultativement, une séquence d'acide nucléique hétérologue codant pour une
flavone synthase (FNS) capable de produire une flavone à partir d'une
flavanone,
en particulier capable de transformer la naringénine en apigénine, et/ou
l'ériodictyol en lutéoline, de préférence de transformer l'ériodictyol en
lutéoline;
de préférence une FNS d'Arabidopsis thaliana, Lonicera japonica, Lonicera
macranthoides, Medicago truncatula, Oryza sativa, Petroselinum crispum,
Populus
deltoides, ou Zea mays, de préférence de Lonicera japonica, Lonicera
macranthoides, et Petroselinum crispum; de préférence une FNS comprenant une
séquence choisie parmi SEQ ID NOs: 33, 35, 37 et les polypeptides comprenant
une
séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec
une de ces séquences et présentant une activité flavone synthase, de
préférence
une flavone synthase (FNS) comprenant une séquence choisie parmi SEQ ID NO: 37
et un polypeptide comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou
95 % d'identité de séquence avec cette séquence et présentant une activité
flavone
synthase.
Dans un mode de réalisation préféré, le microorganisme comprend
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
- une séquence d'acide nucléique hétérologue codant pour une flavanone 7-0-
bêta-D-glucosyltransférase sélectionnée parmi les enzymes comprenant une
séquence
choisie parmi SEQ ID NOs: 113, 115 et 95 et les polypeptides comprenant une
séquence
ayant au moins 60,70, 80, 85, 90 ou 95% d'identité de séquence avec une de ces
séquences
5 et présentant une activité flavanone 7-0-bêta-D-glucosyltransférase, de
préférence
sélectionnée parmi les enzymes comprenant une séquence choisie parmi SEQ ID
NOs: 113
et 95 et les polypeptides comprenant une séquence ayant au moins 60, 70, 80,
85, 90 ou
95 % d'identité de séquence avec une de ces séquences et présentant une
activité
flavanone 7-0-bêta-D-glucosyltransférase, et de manière tout particulièrement
préférée
10 sélectionnée parmi les enzymes comprenant une séquence choisie parmi la
SEQ ID NO: 113
et les polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90
ou 95 %
d'identité de séquence avec cette séquence et présentant une activité
flavanone 7-0-bêta-
D-glucosyltransférase ; et
- une séquence d'acide nucléique hétérologue codant pour une 6-0-
15 rhannnosyltransferase sélectionnée parmi les enzymes comprenant une
séquence choisie
parmi la SEQ ID NO: 103 et les polypeptides comprenant une séquence ayant au
moins 60,
70, 80, 85, 90 ou 95 % d'identité de séquence avec cette séquence et
présentant une
activité 6"-O-rhannnosyltransferase ; et
- une séquence d'acide nucléique hétérologue codant pour une l'UDP-glucose
4,6-
20 déshydratase/UDP-4-kéto-6-désoxy-D-glucose 3,5-
épinnerase/U DP-4-kéto-L-rha nn nase-
réductase sélectionnée parmi les enzymes comprenant une séquence choisie parmi
la SEQ
ID NO: 107 et les polypeptides comprenant une séquence ayant au moins 60, 70,
80, 85, 90
ou 95 % d'identité de séquence avec cette séquence et présentant une activité
UDP-
glucose 4,6-déshydratase/UDP-4-kéto-6-désoxy-D-glucose 3,5-épimerase/UDP-4-
kéto-L-
25 rhamnose-réductase ; et
-une séquence d'acide nucléique hétérologue codant pour une flavonoïde 3'-
nnonooxygénase (F3'H) sélectionnée parmi les enzymes comprenant une séquence
choisie
parmi SEQ ID NOs: 7, 11, 17 et 121 et les polypeptides comprenant une séquence
ayant au
moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec l'une de ces
séquences et
30 présentant une activité flavonoïde 3'-nnonooxygénase, de préférence une
flavonoïde 3'-
nnonooxygénase (F3'H) comprenant une séquence choisie parmi SEQ ID NOs: 7, 17,
et 121
et les polypeptides ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité avec
une de ces
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
51
séquences et présentant l'activité flavonoïde 3'-nnonooxygénase, et de manière
tout
particulièrement préférée une flavonoïde 3'-nnonooxygénase (F3'H) comprenant
une
séquence choisie parmi la SEQ ID NO: 7 et les polypeptides ayant au moins 60,
70, 80, 85,
90 ou 95 % d'identité avec cette séquence et présentant l'activité flavonoïde
3'-
nnonooxygénase ;
-facultativement, une séquence d'acide nucléique hétérologue codant pour une
cytochrome P450 réductase (CPR) comprenant une séquence choisie parmi SEQ ID
NOs:
23, 25, 29 et 31 et les polypeptides comprenant une séquence ayant au moins
60, 70, 80,
85, 90 ou 95 % d'identité de séquence avec une de ces séquences et présentant
une activité
cytochrome P450 réductase, de préférence parmi les enzymes comprenant une
séquence
choisie parmi SEQ ID NOs: 23, 25 et 29 et les polypeptides comprenant une
séquence ayant
au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec une de ces
séquences et
présentant une activité cytochrome P450 réductase, et de manière tout
particulièrement
préférée parmi les enzymes comprenant une séquence choisie parmi la SEQ ID NO:
25 et
les polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou
95 %
d'identité de séquence avec cette séquence et présentant une activité
cytochrome P450
réductase ; et
-une séquence d'acide nucléique hétérologue codant pour une 0-
nnéthyltransférase (OMT) capable de nnéthyler l'ériodictyol et/ou la lutéoline
en position 4'
et comprenant une séquence choisie parmi SEQ ID NOs: 117, 119 et 89 et les
polypeptides
comprenant une séquence ayant au moins 60, 70,80, 85, 90 ou 95% d'identité de
séquence
avec une de ces séquences et présentant une activité 0-nnéthyltransférase,
notamment
avec l'ériodictyol et/ou la lutéoline comme substrat et une nnéthylation en
position 4', de
préférence sélectionnée parmi l'enzyme comprenant une séquence choisie parmi
SEQ ID
NOs: 117 et 119 et les polypeptides comprenant une séquence ayant au moins 60,
70, 80,
85, 90 ou 95 % d'identité de séquence avec l'une de ces séquences et
présentant une
activité 0-méthyltransférase ; et
-facultativement, une séquence d'acide nucléique hétérologue codant pour une
flavone synthase (FNS) capable de produire une flavone à partir d'une
flavanone, en
particulier capable de transformer la naringénine en apigénine, et/ou
l'ériodictyol en
lutéoline, de préférence de transformer l'ériodictyol en lutéoline et
comprenant une
séquence choisie parmi SEQ ID NO: 37 et un polypeptide comprenant une séquence
ayant
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
52
au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec cette séquence
et
présentant une activité flavone synthase.
De préférence, le microorganisme comprend chacune de ces séquences d'acide
nucléique
hétérologues.
Dans un autre mode de réalisation particulier, le microorganisme recombinant
comprend :
- une séquence d'acide nucléique hétérologue codant pour une flavanone 7-0-
bêta-
D-glucosyltransférase (UGT) capable d'ajouter un glucose en position 7 de
l'hespérétine et/ou la diosnnétine ;
- une séquence d'acide nucléique hétérologue codant pour une 6"-O-
rhannnosyltransferase (RhaT) capable de transférer un rhannnose en position 6
du
glucose de l'hespérétine-7-0-glucoside et/ou la diosnnétine-7-0-glucoside;
- une séquence d'acide nucléique hétérologue codant pour une UDP-glucose
4,6-
déshydratase/UDP-4-kéto-6-désoxy-D-glucose 3,5-
épinnerase/UDP-4-kéto-L-
rhannnose-réductase (RHM) capable de produire de l'UDP-rhannnose ;
- une séquence d'acide nucléique hétérologue codant pour une flavonoïde 3'-
nnonooxygénase (F3'H) capable d'hydroxyler en position 3' la naringénine et/ou
l'apigénine ;
- une séquence d'acide nucléique hétérologue codant pour une cytochrome
P450
réductase ;
- une séquence d'acide nucléique hétérologue codant pour une 0-
nnéthyltransférase
capable de nnéthyler l'ériodictyol et/ou la lutéoline en position 4'; et
- une séquence d'acide nucléique hétérologue codant pour une flavone
synthase
(FNS) capable de produire une flavone à partir d'une flavanone, en particulier
capable de transformer la naringénine en apigénine, l'ériodictyol en lutéoline
et/ou
l'hespérétine en diosnnétine, de préférence de transformer l'ériodictyol en
lutéoline.
Dans un autre mode de réalisation particulier, le microorganisme recombinant
comprend
en outre une séquence d'acide nucléique hétérologue ou endogène codant pour
une S-
adénosylnnéthionine synthétase (SAMT), en particulier de Saccharomyces
cerevisiae, par
exemple une SAMT comprenant une séquence choisie parmi SEQ ID NO: 81 et un
polypeptide comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 %
d'identité
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
53
de séquence avec cette séquence et présentant une activité S-
adénosylnnéthionine
synthétase.
Chaque enzyme peut être choisie parmi les enzymes décrites ci-dessus.
Jusqu'à Naringénine et Apigénine
Différentes voies de biosynthèses de naringénine et de l'apigénine sont
connues dans les
plantes, en particulier à partir du glucose, de la tyrosine ou de la
phénylalanine. Des
microorganismes, notamment E. cou i et Saccharomyces cerevisiae, ont été
modifiés pour
produire de la naringénine et/ou et de l'apigénine (Hwang El, et al. 2003.
Appl Environ
Microbiol. 2003, 69(5):2699-2706; Jiang H1, et al. 2005. Appl Environ
Microbiol. 2005,
71(6):2962-9; Pandey et al, 2016, Biotechnol Adv., 34, 634-662).
Par exemple, la voie de biosynthèse de la naringénine et de l'apigénine peut
être celle
décrite dans la Figure 1.
Dans un premier mode de réalisation, le microorganisme comprend les enzymes
nécessaires pour la synthèse de la naringénine et/ou de l'apigénine à partir
de la tyrosine.
Dans un deuxième mode de réalisation, le microorganisme comprend les enzymes
nécessaires pour la synthèse de la naringénine et/ou de l'apigénine à partir
de la
phénylalanine.
Dans un troisième mode de réalisation, le microorganisme comprend les enzymes
nécessaires pour la synthèse de la naringénine et/ou de l'apigénine à partir
de la tyrosine
et de la phénylalanine.
TAL : Tyrosine ammonia lyase
La TAL est une tyrosine ammonia lyase. Cette enzyme est capable de produire de
l'acide p-
coumarique à partir de la tyrosine. Cette enzyme appartient à la classe des EC
4.3.1.23.
Par activité phénylalanine ammonia lyase est entendue la transformation de
la
Phénylalanine en acide trans-cinnamique par une enzyme Phénylalanine ammonia
lyase.
Pour déterminer s'il y a une activité phénylalanine ammonia lyase, un test
enzymatique
peut être effectué qui consiste en l'incubation in vitro d'un mélange composé
de l'enzyme
phénylalanine ammonia lyase et de phénylalanine dans les conditions optimales
(pH,
température, ions...). Après un certain temps d'incubation, l'apparition
d'acide trans-
cinnamique est observée en UPLC-MS en comparaison au standard attendu.
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
54
Une tyrosine ammonia lyase (TAL) peut présenter en outre une activité
phénylalanine
ammonia lyase (PAL) telle que définie ci-dessus et/ou une activité
dihydroxyphénylalanine
ammonia-lyase (DAL).
Le microorganisme peut donc comprendre une séquence d'acide nucléique
hétérologue
codant pour une tyrosine ammonia lyase.
De préférence, cette enzyme est une enzyme produite par une bactérie du genre
Rhodobacter ou une bactérie du genre Flavobacteriaceae. Dans un mode de
réalisation
particulier, cette enzyme est produite par une bactérie Rhodobacter
capsulatus, ou
Rhodobacter sphaeroides. Dans un autre mode de réalisation particulier, cette
enzyme est
produite par une bactérie Flavobacterium johnsoniae. Dans un autre mode de
réalisation,
cette enzyme est une enzyme produite par une levure, en particulier une levure
du genre
Rhodotorula, par exemple Rhodotorula glutinis. D'autres organismes produisent
également
une telle enzyme, par exemple Camellia sinensis, Fragaria x ananassa,
Ralstonia
metallidurans, ou Zea mays.
Dans un mode de réalisation particulier, la tyrosine ammonia lyase est
sélectionnée parmi
les enzymes comprenant une séquence choisie parmi SEQ ID NOs: 39 et 41 et les
polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 %
d'identité de séquence avec une de ces séquences et présentant une activité
tyrosine
ammonia lyase.
Dans un mode de réalisation, la TAL est de Flavobacterium johnsoniae. Elle est
décrite dans
la base de données Genbank de NCBI sous le numéro KR095306.1 pour la séquence
nucléique et sous le numéro AKE50827.1 pour la séquence protéique, et plus
particulièrement dans les SEQ ID Nos :40 et 39.
Dans un mode de réalisation particulier préféré, la TAL est de Rhodotorula
glutinis. Elle est
décrite dans la base de données Genbank de NCBI sous le numéro KF765779.1 pour
la
séquence nucléique et sous le numéro AGZ04575.1 pour la séquence protéique, et
plus
particulièrement dans les SEQ ID Nos : 42 et 41, respectivement.
Dans un mode de réalisation particulièrement préféré, la tyrosine ammonia
lyase est
sélectionnée parmi l'enzyme comprenant une séquence choisie parmi SEQ ID NO:
41 et les
polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 %
d'identité de séquence avec cette séquence et présentant une activité tyrosine
ammonia
lyase.
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
4CL : 4-Coumarate-CoA ligase
La 4CL est une 4-counnarate-CoA ligase. Cette enzyme est capable de produire
du 4-
counnaroyl-CoA à partir de l'acide p-counnarique et de Coenzyme A et de
produire du
caffeoyl-CoA à partir de l'acide caféique et de Coenzyme A. Cette enzyme
appartient à la
5 classe des EC 6.2.1.12.
Par activité 4-coumarate-CoA ligase est entendue la transformation de
l'acide p-
coumarique en p-coumaroyl-CoA ou de l'acide caféique en caffeoyl-CoA par une
enzyme 4-
coumarate CoA ligase. Pour déterminer s'il y a une activité 4-coumarate-CoA
ligase, un test
enzymatique peut être effectué qui consiste en l'incubation in vitro d'un
mélange composé
10 de l'enzyme 4-coumarate-CoA ligase, d'acide p-coumarique ou d'acide
caféique, d'ATP et
de CoA dans les conditions optimales (pH, température, ions...). Après un
certain temps
d'incubation, l'apparition du p-coumaroyl-CoA ou du caffeoyl-CoA est observée
au
spectrophotomètre UV avec une longueur d'onde respectivement de 333 nm et de
346 nm
en comparaison au standard attendu.
15 Le microorganisme peut donc comprendre une séquence d'acide nucléique
hétérologue
codant pour une 4-coumarate-CoA ligase.
De préférence, cette enzyme est une enzyme produite par une plante, par
exemple Abies,
Arabidopsis, Agastache, Amorpha, Brassica, Citrus, Cathaya, Cedrus, Crocus,
Larix, Festuca,
Glycine, Jugions, Keteleeria, Lithospermum, Lolium, Lotus, Lycopersicon,
Malus, Medicago,
20 Mesembryanthemum, Nicotiana, Nothotsuga, Oryza, Phaseolus, Pelargonium,
Petroselinum, Physcomitrella, Picea, Prunus, Pseudolarix, Pseudotsuga, Rosa,
Rubus, Ryza,
Saccharum, Suaeda, Pin us, Populus, Solanum, Thellungiella, Triticum, Tsuga,
Vitis ou Zea.
De manière alternative, cette enzyme est une enzyme produite par un
microorganisme, par
exemple Aspergillus, Mycosphaerella, Mycobacterium, Neisseria, Neurospora,
25 Streptomyces, Rhodobacter ou Yarrowia.
Dans une mode de réalisation préféré, cette enzyme est une enzyme produite par
une
plante, de préférence Arabidopsis thaliana, Citrus clementina ou Petroselinum
crispum, en
particulier Arabidopsis thaliana ou Petroselinum crispum, ou par une bactérie,
de
préférence du genre Streptomyces, en particulier Streptomyces clavuligerus.
30 Dans un mode de réalisation particulier, la 4-coumarate-CoA ligase est
sélectionnée parmi
les enzymes comprenant une séquence choisie parmi SEQ ID NOs: 43, 45, 47, 49,
123 et
125 et les polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85,
90 ou 95
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
56
% d'identité de séquence avec une de ces séquences et présentant une activité
4-
counnarate-CoA ligase.
Dans un autre mode de réalisation particulier, la 4-counnarate-CoA ligase est
une enzyme
comprenant une séquence sélectionnée parmi les SEQ ID NOs: 43, 45, 47 et 49,
de
préférence SEQ ID NOs: 45 et 49, et les polypeptides comprenant une séquence
ayant au
moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec une de ces
séquences et
présentant une activité 4-coumarate-CoA ligase.
Dans un premier mode de réalisation particulier, la 4CL est d'Arabidopsis
thaliana. Elle est
décrite dans la base de données Genbank de NCBI sous le numéro AY099747.1 pour
la
séquence nucléique et sous le numéro AAM20598.1 pour la séquence protéique, et
plus
particulièrement dans les SEQ ID Nos : 44 et 43, respectivement.
Dans un deuxième mode de réalisation particulier, la 4CL est de Petroselinum
crispum. Elle
est décrite dans la base de données Genbank de NCBI sous le numéro X13324.1 ou
X13325.1 pour la séquence nucléique et sous le numéro CAA31696.1 ou CAA31697.1
pour
la séquence protéique, respectivement. Les protéines sont décrites dans
UniProtKB/Swiss
Prot sous le numéro de référence P14912 et P14913, respectivement, et plus
particulièrement dans les SEQ ID Nos : 46 et 45, et 48 et 47, respectivement.
De préférence,
la 4CL est de Petroselinum crispum et est décrite dans la base de données
Genbank de NCBI
sous le numéro X13324.1 pour la séquence nucléique et sous le numéro
CAA31696.1 pour
la séquence protéique, et dans UniProtKB/Swiss Prot sous le numéro de
référence P14912,
et plus particulièrement dans les SEQ ID Nos :46 et 45, respectivement.
Dans un troisième mode de réalisation particulier, la 4CL est de Streptomyces
clayuligerus.
Elle est décrite dans la base de données Genbank de NCBI sous le numéro
CP016559.1 pour
la séquence nucléique et sous le numéro ANW18832.1 pour la séquence protéique,
et plus
particulièrement dans les SEQ ID Nos : 50 et 49, respectivement.
Dans un quatrième mode de réalisation particulier, la 4CL est d'Arabidopsis
thaliana. Une
séquence nucléotidique ainsi que la séquence protéique de cette enzyme sont
décrites
respectivement dans les SEQ ID NOs : 124 et 123.
Dans un cinquième mode de réalisation particulier, la 4CL est de Citrus
clementina et une
séquence nucléotidique ainsi que la séquence protéique de cette enzyme sont
décrites
respectivement dans les SEQ ID NOs : 126 et 125.
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
57
Dans un mode de réalisation préféré, la 4CL est une enzyme comprenant une
séquence
sélectionnée parmi les SEQ ID NOs: 45, 123 et 125, de préférence SEQ ID NOs:
123 et 45 et
les polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou
95 %
d'identité de séquence avec une de ces séquences et présentant une activité 4-
counnarate-
CoA ligase. De manière tout particulièrement préférée, la 4CL est une enzyme
comprenant
une séquence sélectionnée parmi la SEQ ID NO: 45 et les polypeptides
comprenant une
séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec
cette
séquence et présentant une activité 4-counnarate-CoA ligase.
CHS : Chalcone synthase
La CHS est une chalcone synthase. Cette enzyme est capable de produire de la
naringénine-
chalcone à partir du 4-counnaroyl-CoA et de nnalonyl-CoA et de produire de
l'ériodictyol-
chalcone à partir de caffeoyl-CoA et de nnalonyl-CoA. Cette enzyme appartient
à la classe
des EC 2.3.1.74.
Par activité chalcone synthase est entendue la transformation de p-
counnaroyl-CoA et
de nnalonyl-CoA a en naringénine chalcone ou de caffeoyl-CoA et de nnalonyl-
CoA¨en
ériodictyol chalcone par une enzyme chalcone synthase. Pour déterminer s'il y
a une
activité chalcone synthase, un test enzymatique peut être effectué qui
consiste en
l'incubation in vitro d'un mélange composé de l'enzyme Chalcone synthase, de
counnaroyl-
CoA ou de caffeoyl-CoA et de nnalonyl-CoA dans les conditions optimales (pH,
température,
ions...). Après un certain temps d'incubation, l'apparition respectivement de
la Naringénine
chalcone ou de l'ériodictyol chalcone est observée en HPLC-MS en comparaison
au
standard attendu.
Le microorganisme comprend donc une séquence d'acide nucléique hétérologue
codant
pour une Cha Icone synthase.
Cette enzyme peut être une enzyme produite par une plante, notamment du genre
Arabidopsis, Avena, Cosmos, Citrus, Daucus, Fagopyrum, Freesia, Glycine,
Glycyrrhiza,
Hum ulus, Hypericum, Hordeum, Jugions, Medicago, Phaseolus, Physcomitrella,
Plagiochasma, Petroselinum, Pueraria, Rubus, Secale, Scutellaria, Silene,
Sinapis, Spin acia,
Stellaria, Triticum, Tulipa, Verbena, Vitis, ou Xanthisma, par exemple
Arabidopsis thaliana,
Avena sativa, Cosmos sulphureus, Citrus sinensis, Daucus carota, Fagopyrum
esculentum,
Freesia hybrid cultivar, Glycine max, Glycyrrhiza echinata, Hum ulus lupulus,
Hypericum
androsaemum, Hordeum vulgare, Jugions sp., Medicago sativa, Phaseolus
vulgaris,
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
58
Physcomitrella patens, Plagiochasma appendiculatum, Petroselinum crispum,
Pueraria
montana var. lobata, Rubus idaeus, Secale cereale, Scutellaria baicalensis,
Silene sp.,
Sinapis alba, Spinacia oleracea, Stellaria longipes, Triticum aestivum, Tulipa
hybrid cultivar,
Verbena sp., Vitis vimfera, ou Xanthisma gracile.
De préférence, cette enzyme est une enzyme produite par une plante, par
exemple du
genre Citrus, en particuliers Citrus sinensis, ou de Hordeum vulgare ou par
une bactérie, de
préférence du genre Streptomyces, en particulier Streptomyces clavuligerus.
Dans un mode de réalisation particulier, la chalcone synthase est sélectionnée
parmi les
enzymes comprenant une séquence choisie parmi SEQ ID NOs: 51, 53, 55 et 57 et
les
polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 %
d'identité de séquence avec une de ces séquences et présentant une activité
chalcone
synthase, de préférence parmi les enzymes comprenant une séquence choisie
parmi SEQ
ID NOs: 53 et 55 et les polypeptides comprenant une séquence ayant au moins
60, 70, 80,
85, 90 ou 95 % d'identité de séquence avec une de ces séquences et présentant
une activité
chalcone synthase.
Dans un mode de réalisation particulièrement préféré, la chalcone synthase est
une
enzyme comprenant une séquence choisie parmi SEQ ID NO: 53 et les polypeptides
comprenant une séquence ayant au moins 60, 70,80, 85, 90 ou 95% d'identité de
séquence
avec cette séquence et présentant une activité chalcone synthase.
Dans un premier mode de réalisation particulier, la CHS est de Hordeum
vulgare. Elle est
décrite dans la base de données Genbank de NCBI sous le numéro Y09233.1 pour
la
séquence nucléique et sous le numéro CAA70435.1 pour la séquence protéique, et
plus
particulièrement dans les SEQ ID Nos : 52 et 51, respectivement. La protéine
est décrite
dans UniProtKB/Swiss Prot sous le numéro de référence Q96562.
Dans un deuxième mode de réalisation particulier, la CHS est de Citrus
sinensis. Elle est
décrite dans la base de données Genbank de NCBI sous le numéro AB009351.1 pour
la
séquence nucléique et sous le numéro BAA81664.1 pour la séquence protéique, et
plus
particulièrement dans les SEQ ID Nos : 54 et 53, respectivement.
Dans un troisième mode de réalisation particulier, la CHS est de Citrus
sinensis. Elle est
décrite dans la base de données Genbank de NCBI sous le numéro XM_006489733.1
pour
la séquence nucléique et sous le numéro XP_006489796.1 pour la séquence
protéique, et
plus particulièrement dans les SEQ ID Nos : 56 et 55, respectivement.
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
59
Dans un quatrième mode de réalisation particulier, la CHS est de Streptomyces
clavuligerus.
Elle est décrite dans la base de données Genbank de NCBI sous le numéro
CP016559.1 pour
la séquence nucléique et sous le numéro ANW16917.1 pour la séquence protéique,
et plus
particulièrement dans les SEQ ID Nos : 58 et 57, respectivement.
La réaction catalysée par la chalcone synthase nécessitant la présence de
malonyl-CoA, le
microorganisme peut être modifié pour augmenter la synthèse de nnalonyl-CoA.
CHI : Chalcone isomérase
La CHI est une chalcone isomérase. Elle est capable de produire la naringénine
à partir de
la Naringénine chalcone et de produire de l'eriodictyol à partir d'ériodictyol
chalcone. Cette
enzyme appartient à la classe des EC 5.5.1.6.
Par activité Chalcone isomérase est entendue la transformation de
naringénine
chalcone ou d'ériodictyol chalcone en naringénine ou ériodictyol par une
enzyme chalcone
isomérase. Pour déterminer s'il y a une activité Chalcone isomérase, un test
enzymatique
peut être effectué qui consiste en l'incubation in vitro d'un mélange composé
de l'enzyme
Chalcone isomérase, de naringénine chalcone ou d'ériodictyol chalcone dans les
conditions
optimales (pH, température, ions...). Après un certain temps d'incubation,
l'apparition
respectivement de la Naringénine ou de l'ériodictyol est observée en HPLC-MS
en
comparaison au standard attendu.
Le microorganisme comprend donc une séquence d'acide nucléique hétérologue
codant
pour une chalcone isomérase.
Cette enzyme peut provenir d'une plante, notamment du genre Arabidopsis,
Ginkgo,
Oncidium, Perilla, Citrus ou Tri gonella, par exemple Arabidopsis thaliana,
Ginkgo biloba,
Oncidium Gower Ramsey, Perilla frutescens, Citrus Sinensis ou Trigonella
foenum-graecum.
De préférence, cette enzyme est une enzyme produite par une plante, par
exemple
Arabidopsis thaliana ou par une bactérie, de préférence du genre Streptomyces,
en
particulier Streptomyces clavuligerus.
Dans un mode de réalisation particulier, la chalcone isomérase est
sélectionnée parmi les
enzymes comprenant une séquence choisie parmi SEQ ID NOs: 59 et 61 et les
polypeptides
comprenant une séquence ayant au moins 60, 70,80, 85, 90 ou 95% d'identité de
séquence
avec une de ces séquences et présentant une activité chalcone isomérase.
Dans un mode de réalisation préféré, la chalcone isomérase est sélectionnée
parmi les
enzymes comprenant une séquence choisie parmi SEQ ID NO: 61 et les
polypeptides
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
comprenant une séquence ayant au moins 60, 70,80, 85, 90 ou 95% d'identité de
séquence
avec cette séquence et présentant une activité chalcone isonnérase.
Dans un premier mode de réalisation particulier, la CHI est de Streptomyces
clavuligerus.
Elle est décrite dans la base de données Genbank de NCBI sous le numéro
CP016559.1 pour
5 la séquence nucléique et sous le numéro ANW16918.1 pour la séquence
protéique, et plus
particulièrement dans les SEQ ID Nos : 60 et 59, respectivement.
Dans un deuxième mode de réalisation particulier, la CHI est d'Arabidopsis
thaliana. Elle
est décrite dans la base de données Genbank de NCBI sous le numéro NM_115370.4
pour
la séquence nucléique et sous le numéro NP_191072.1pour la séquence protéique,
et plus
10 particulièrement dans les SEQ ID Nos : 62 et 61, respectivement.
FNS: Flavone synthase
L'apigénine peut être préparée à partir de la naringénine à l'aide d'une
flavone synthase
(FNS). Elle est capable de produire de l'apigénine à partir de la naringénine.
Le microorganisme peut donc comprendre une séquence d'acide nucléique
hétérologue
15 codant pour une flavone synthase, en particulier capable de produire de
l'apigénine à partir
de la naringénine et/ou une séquence d'acide nucléique hétérologue codant pour
une
flavone synthase, en particulier capable de produire de la lutéoline à partir
d'ériodictyol,
et/ou une séquence d'acide nucléique hétérologue codant pour une flavone
synthase, en
particulier capable de produire la diosmétine à partir de l'hespérétine.
20 La FNS peut être choisie parmi celles décrites précédemment.
A partir de la phénylalanine
Alternativement ou en complément, le microorganisme peut également comprendre
les
enzymes nécessaires pour la synthèse de l'acide p-coumarique à partir de la
phénylalanine.
Dans ce contexte, le microorganisme peut comprendre en outre une séquence
d'acide
25 nucléique hétérologue codant pour une phénylalanine ammonia lyase (PAL)
et une
séquence d'acide nucléique hétérologue codant pour une cinnamate 4-hydroxylase
(C4H).
La PAL appartient à la classe des EC 4.3.1.24. Elle est capable de produire de
l'acide
cinnamique à partir de la phénylalanine.
Par activité phénylalanine ammonia lyase est entendue la transformation de
la
30 Phénylalanine en acide trans-cinnamique par une enzyme Phénylalanine
ammonia lyase.
Pour déterminer s'il y a une activité phénylalanine ammonia lyase, un test
enzymatique
peut être effectué qui consiste en l'incubation in vitro d'un mélange composé
de l'enzyme
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
61
phénylalanine annnnonia lyase et de phénylalanine dans les conditions
optimales (pH,
température, ions...). Après un certain temps d'incubation, l'apparition
d'acide trans-
cinnamique est observée en UPLC-MS en comparaison au standard attendu.
Plusieurs enzymes ont déjà été décrites dans l'art antérieur. De préférence,
l'enzyme
provient d'une plante, par exemple une plante du genre Arabidopsis, Agastache,
Ananas,
Asparagus, Brassica, Bromheadia, Bambusa, Beta, Betula, Citrus, Cucumis,
Camellia,
Capsicum, Cassia, Catharanthus, Cicer, Citrullus, Coffea, Cucurbita, Cynodon,
Daucus,
Dendrobium, Dianthus, Digitalis, Dioscorea, Eucalyptus, Gallus, Ginkgo,
Glycine, Hordeum,
Helianthus, lpomoea, Lactuca, Lithospermum, Lotus, Lycopersicon, Medicago,
Malus,
Manihot, Medicago, Mesembryanthemum, Nicotiana, Olea, Oryza, Phaseolus, Pin
us,
Populus, Pisum, Persea, Petroselinum, Phalaenopsis, Phyllostachys,
Physcomitrella, Picea,
Pyrus, Prunus, Quercus, Raphanus, Rehmannia, Rubus, Solanum, Sorghum,
Sphenostylis,
Stellaria, Stylosanthes, Triticum, Trifolium, Vaccinium, Vigna, Vitis, Zea, ou
Zinnia. Par
exemple, on peut citer celles d'Arabidopsis thaliana ou de Petroselinum
crispum. Dans un
mode de réalisation préféré, la PAL est de Citrus sinensis.
En outre, la phénylalanine annnnonia lyase (PAL) peut présenter en outre une
activité
tyrosine annnnonia lyase (TAL) et/ou une activité dihydroxyphénylalanine
annnnonia-lyase
(DAL) telle que décrite ci-dessous.
Dans un mode de réalisation particulier, la phénylalanine annnnonia lyase
(PAL) est
sélectionnée parmi les enzymes comprenant une séquence choisie parmi SEQ ID
NOs: 63,
65, 77 et les polypeptides comprenant une séquence ayant au moins 60, 70, 80,
85, 90 ou
95 % d'identité de séquence avec une de ces séquences et présentant une
activité
phénylalanine annnnonia lyase.
Dans un mode de réalisation préféré, la PAL est sélectionnée parmi les enzymes
comprenant une séquence choisie parmi SEQ ID NOs: 65 et 77 et les polypeptides
comprenant une séquence ayant au moins 60, 70,80, 85, 90 ou 95% d'identité de
séquence
avec une de ces séquences et présentant une activité phénylalanine annnnonia
lyase. De
manière tout particulièrement préférée, la PAL est sélectionnée parmi les
enzymes
comprenant une séquence choisie parmi SEQ ID NO: 65 et les polypeptides
comprenant
une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence
avec cette
séquences et présentant une activité phénylalanine annnnonia lyase.
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
62
Dans un mode de réalisation particulier, la PAL de Citrus sinensis est décrite
dans la base
de données Genbank de NCBI sous le numéro XM_006481431.2 pour la séquence
nucléique et sous le numéro XP_006481494.1 pour la séquence protéique, et plus
particulièrement dans les SEQ ID Nos : 64 et 63, respectivement.
Dans un autre mode de réalisation particulier, la PAL de Citrus sinensis est
décrite dans la
base de données Genbank de NCBI sous le numéro XM_006488000.2 pour la séquence
nucléique et sous le numéro XP_006488063.1 pour la séquence protéique, et plus
particulièrement dans les SEQ ID Nos : 66 et 65, respectivement.
Dans un autre mode de réalisation particulier, la PAL d'Arabidopsis thaliana
est décrite dans
la base de données Genbank de NCBI sous le numéro NM_115186.4 pour la séquence
nucléique et sous le numéro NP_190894.1 pour la séquence protéique, et plus
particulièrement dans les SEQ ID Nos : 78 et 77, respectivement.
Éventuellement, si la biosynthèse à partir de la tyrosine et de la
phénylalanine est
envisagée, la PAL et la TAL peuvent être remplacées ou complétées par une
phénylalanine/tyrosine annnnonia lyase (PTAL). La PTAL appartient à la classe
des EC
4.3.1.25.
La C4H appartient à la classe des EC 1.14.13.11. Elle est capable de produire
de l'acide p-
counnarique à partir de l'acide cinnamique.
Par activité trans-cinnamate 4-nnonooxygénase est entendue la
transformation de
l'acide trans-cinnamique en acide p-counnarique par une enzyme trans-
cinnannate 4-
nnonooxygénase (CPR dépendante). Pour déterminer s'il y a une activité trans-
cinnannate
4-nnonooxygénase, un test enzymatique peut être effectué qui consiste en
l'incubation in
vitro d'un mélange composé de l'enzyme trans-cinnannate 4-nnonooxygénase,
d'acide
cinnamique, de NADPH, d'H+ et d'Oz dans les conditions optimales (pH,
température,
ions...). Après un certain temps d'incubation, l'apparition de 4-
hydroxycinnannate (acide p-
counnarique) est observée en UPLC-MS en comparaison au standard attendu.
Plusieurs enzymes ont déjà été décrites dans l'art antérieur. De préférence,
l'enzyme
provient d'une plante, par exemple une plante du genre Arabidopsis, Ammi,
Avicennia,
Camellia, Camptotheca, Catharanthus, Citrus, Glycine, Helianthus, Lotus,
Mesembryanthemum, Physcomitreila, Phaseolus, Pin us, Populus, Ruta, Saccharum,
Solanum, Vitis, Vigna ou Zea. Dans un mode de réalisation préféré, la
cinnannate 4-
hydroxylase (C4H) est de Citrus sinensis ou d'Arabidopsis thaliana.
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
63
Dans un mode de réalisation particulier, la cinnannate 4-hydroxylase (C4H) est
sélectionnée
parmi les enzymes comprenant une séquence choisie parmi SEQ ID NOs: 67, 69, 79
et les
polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 %
d'identité de séquence avec une de ces séquences et présentant une activité
cinnannate 4-
hydroxylase.
Dans un mode de réalisation préféré, la C4H est sélectionnée parmi les enzymes
comprenant une séquence choisie parmi SEQ ID NO: 79 et les polypeptides
comprenant
une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence
avec cette
séquence et présentant une activité cinnamate 4-hydroxylase.
Dans un mode de réalisation particulier, la C4H de Citrus sinensis est décrite
dans la base
de données Genbank de NCBI sous le numéro NM_001288840.1 pour la séquence
nucléique et sous le numéro NP_001275769.1 pour la séquence protéique, et plus
particulièrement dans les SEQ ID Nos : 68 et 67, respectivement.
Dans un autre mode de réalisation particulier, la C4H de Citrus sinensis est
décrite dans la
base de données Genbank de NCBI sous le numéro NM_001288895.1 pour la séquence
nucléique et sous le numéro NP_001275824.1 pour la séquence protéique, et plus
particulièrement dans les SEQ ID Nos : 70 et 69, respectivement.
Dans un autre mode de réalisation particulier, la C4H d'Arabidopsis thaliana
est décrite
dans la base de données Genbank de NCBI sous le numéro NM_128601.3 pour la
séquence
nucléique et sous le numéro NP_180607.1 pour la séquence protéique, et plus
particulièrement dans les SEQ ID Nos : 80 et 79, respectivement.
En passant par l'acide caféique
Dans un mode de réalisation supplémentaire, la biosynthèse de l'ériodictyol
peut
également comprendre la synthèse de la L-DOPA (3,4-dihydroxy-L-phenylalanine)
à partir
de la tyrosine puis de l'acide caféique à partir de la L-DOPA (3,4-dihydroxy-L-
phenylalanine). Pour cela, les enzymes suivantes sont nécessaires. Pour
convertir la
tyrosine en L-DOPA (3,4-dihydroxy-L-phenylalanine), deux sous-unités sont
nécessaires,
HpaB et HpaC.
La HpaB est une sous-unité 4-hydroxyphénylacetate 3-monooxygénase oxygénase
(HpaB).
De préférence, cette enzyme est une enzyme produite par une bactérie, de
préférence
Escheri chia cou.
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
64
Dans un mode de réalisation particulier, la 4-hydroxyphénylacetate 3-
nnonooxygénase
oxygénase (HpaB) est une enzyme comprenant une séquence choisie parmi SEQ ID
NO: 83
et les polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90
ou 95 %
d'identité de séquence avec cette séquence et présentant une activité 4-
hydroxyphénylacetate 3-monooxygénase.
Dans un mode de réalisation particulier, la HpaB est de Escheri chia cou. Elle
est décrite dans
la base de données Genbank de NCBI sous le numéro CAQ34705.1 pour la séquence
protéique, et plus particulièrement dans la séquence SEQ ID No: 83. Une
séquence
nucléique codant pour cette enzyme est décrite dans la séquence SEQ ID No :
84. La
protéine est décrite dans UniProtKB/Swiss Prot sous le numéro de référence
A0A14ONG21.
La HpaC est une sous-unité 4-hydroxyphénylacetate 3-monooxygénase réductase.
Le
microorganisme peut donc comprendre une séquence d'acide nucléique hétérologue
codant pour une sous-unité 4-hydroxyphénylacetate 3-monooxygénase réductase
(HpaC).
Par activité p-coumarate 3 hydroxylase est entendue la transformation de
l'acide p-
coumarique en acide caféique et/ou de la L-tyrosine en L-Dopa en utilisant un
complexe
enzymatique composé de HpaB (4-hydroxyphenylacetate 3-hydroxylase oxydase) et
de
HpaC (4-hydroxyphenylacetate 3-hydroxylase réductase). Pour déterminer s'il y
a une
activité p-coumarate 3 hydroxylase, un test enzymatique peut être effectué qui
consiste en
l'incubation in vitro d'un mélange composé des enzymes HpaB, HpaC, d'acide p-
coumarique ou de L-Tyrosine, de FAD et de NADH dans les conditions optimales
(pH,
température, ions...). Après un certain temps d'incubation, l'apparition de
l'acide caféique
ou de la L-Dopa est observée en HPLC-MS en comparaison au standard attendu.
De préférence, cette enzyme est une enzyme produite par une bactérie, de
préférence
Escheri chia coll.
Dans un mode de réalisation particulier, la 4-hydroxyphénylacetate 3-
monooxygénase
réductase (HpaC) est une enzyme comprenant une séquence choisie parmi SEQ ID
NO: 85
et les polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90
ou 95 %
d'identité de séquence avec cette séquence et présentant une activité 4-
hydroxyphénylacetate 3-monooxygénase.
Dans un mode de réalisation particulier, la HpaC est de Escheri chia cou. Elle
est décrite dans
la base de données Genbank de NCBI sous le numéro CAQ34704.1 pour la séquence
protéique, et plus particulièrement dans la séquence SEQ ID No: 85. Une
séquence
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
nucléique codant pour cette enzyme est décrite dans la séquence SEQ ID No :
86. La
protéine est décrite dans UniProtKB/Swiss Prot sous le numéro de référence
A0A14ONG67.
Ensemble, la HpaB et la HpaC sont capables de produire de la L-DOPA (3,4-
dihydroxy-L-
phenylalanine) à partir de la tyrosine.
5 Ainsi, le microorganisme peut donc comprendre une séquence d'acide
nucléique
hétérologue codant pour une 4-hydroxyphénylacetate 3-monooxygénase oxygénase
(HpaB) et une séquence d'acide nucléique hétérologue codant pour 4-
hydroxyphénylacetate 3-monooxygénase réductase (HpaC).
Par ailleurs, cette voie nécessite également la présence d'une enzyme capable
de
10 synthétiser l'acide caféique à partir de la L-DOPA (3,4-dihydroxy-L-
phenylalanine), une
dihydroxyphénylalanine ammonia-lyase (DAL). Cette enzyme appartient à la
classe des EC
4.3.1.11.
Par activité dihydroxyphénylalanine ammonia lyase est entendue la
transformation de
la L-Dopa en acide trans-caféique par une enzyme dihydroxyphénylalanine
ammonia lyase.
15 Pour déterminer s'il y a une activité dihydroxyphénylalanine ammonia
lyase, un test
enzymatique peut être effectué qui consiste en l'incubation in vitro d'un
mélange composé
de l'enzyme dihydroxyphénylalanine ammonia lyase et de L-dopa (levodopa) dans
les
conditions optimales (pH, température, ions...). Après un certain temps
d'incubation,
l'apparition d'acide trans-caféique est observée en UPLC-MS en comparaison au
standard
20 attendu.
En outre, la dihydroxyphénylalanine ammonia-lyase (DAL) peut présenter en
outre une
activité tyrosine ammonia lyase (TAL) et/ou une activité phénylalanine ammonia
lyase
(PAL).
Le microorganisme peut ainsi comprendre une séquence d'acide nucléique
hétérologue
25 codant pour une sous-unité 4-hydroxyphénylacetate 3-monooxygénase
oxygénase (HpaB),
une séquence d'acide nucléique hétérologue codant pour une sous-unité 4-
hydroxyphénylacetate 3-monooxygénase réductase (HpaC) et une séquence d'acide
nucléique hétérologue codant pour une dihydroxyphénylalanine ammonia-lyase
(DAL).
De manière alternative à l'utilisation des HpaB et HpaC ou en combinaison avec
celles-ci, il
30 est possible d'utiliser une enzyme permettant de convertir la tyrosine
en L-Dopa et une
enzyme permettant de convertir l'acide p-coumarique en acide caféique.
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
66
Il s'agit respectivement d'une 4-nnéthoxybenzoate 0-dénnéthylase, également
appelée 4-
nnéthoxybenzoate nnonooxygénase (0-dénnéthylant) qui possède l'activité L-
tyrosine
hydroxylase appartenant à la classe des EC 1.14.99.15 et d'une p-counnarate-3-
hydroxylase
possédant l'activité p-counnarate 3 hydroxylase appartenant à la classe des EC
1.14.13.
Ces différentes enzymes font toutes deux partie de la famille des cytochromes
P450 (CYP).
Par activité L-tyrosine hydroxylase est entendue la transformation de
l'acide p-
counnarique en acide caféique et/ou de la L-tyrosine en L-Dopa en utilisant
une enzyme p-
counnarate 3 hydroxylase (CPR dépendante). Pour déterminer s'il y a une
activité L-tyrosine
hydroxylase, un test enzymatique peut être effectué qui consiste en
l'incubation in vitro
d'un mélange composé de l'enzyme p-counnarate 3 hydroxylase, d'acide p-
counnarique ou
de L-Tyrosine, les co-facteurs nécessaires dans les conditions optimales (pH,
température,
ions...). Après un certain temps d'incubation, l'apparition de l'acide
caféique ou de la L-
Dopa est observée en HPLC-MS en comparaison au standard attendu.
Par activité p-counnarate 3 hydroxylase est entendue la transformation de
l'acide p-
counnarique en acide caféique et/ou de la L-tyrosine en L-Dopa en utilisant
une enzyme p-
counnarate 3 hydroxylase (CPR dépendante). Pour déterminer s'il y a une
activité p-
counnarate 3 hydroxylase, un test enzymatique peut être effectué qui consiste
en
l'incubation in vitro d'un mélange composé de l'enzyme p-counnarate 3
hydroxylase,
d'acide p-counnarique ou de L-Tyrosine, dans les conditions optimales (pH,
température,
ions...). Après un certain temps d'incubation, l'apparition de l'acide
caféique ou de la L-
Dopa est observée en HPLC-MS en comparaison au standard attendu.
Le microorganisme recombinant peut donc comprendre une séquence d'acide
nucléique
hétérologue codant pour une 4-nnéthoxybenzoate 0-déméthylase (CYP) capable de
convertir la tyrosine en L-Dopa et également l'acide p counnarique en acide
caféique.
Dans un mode de réalisation, la 4-nnéthoxybenzoate 0-dénnéthylase est une
enzyme de
bactérie, notamment de Rhodopseudomonas palustris, Pseudomonas putida, ou
Escherichia cou, de plante, notamment de Beta vulgaris, de mammifère,
notamment
d'Oryctolagus cuniculus, ou de champignon, notamment de Rhodotorula glutinis.
Dans un
mode de réalisation particulier, la 4-nnéthoxybenzoate 0-dénnéthylase est une
enzyme de
Rhodopseudomonas palustris, de Saccharothrix espanaensis, ou de Beta vulgaris.
Dans un mode de réalisation particulier, la 4-nnéthoxybenzoate 0-dénnéthylase
est
sélectionnée parmi les enzymes comprenant une séquence choisie parmi SEQ ID
NOs: 73
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
67
et 75 et les polypeptides comprenant une séquence ayant au moins 60, 70, 80,
85, 90 ou
95 % d'identité de séquence avec une de ces séquences et présentant l'activité
L-tyrosine
hydrolase.
La 4-nnéthoxybenzoate 0-dénnéthylase peut également être de Beta vulgaris. Les
séquences nucléiques codant cette enzyme et séquences protéiques sont décrites
dans
dans les SEQ ID Nos: 74 et 73, respectivement. La protéine est décrite dans
UniProtKB/Swiss Prot sous le numéro de référence PODKI2.
En outre, la 4-nnéthoxybenzoate 0-déméthylase peut être de Saccharothrix
espanaensis.
Les séquences nucléiques codant cette enzyme et séquences protéiques sont
décrites dans
NCBI sous les numéros de référence NC_005296.1 et WP_011157377.1,
respectivement, et
plus particulièrement dans les SEQ ID Nos : 76 et 75. La protéine est décrite
dans
UniProtKB/Swiss Prot sous le numéro de référence Q6N8N2.
Dans un mode de réalisation, le microorganisme peut comprendre une séquence
d'acide
nucléique hétérologue codant pour une 4-nnéthoxybenzoate 0-dénnéthylase et une
séquence d'acide nucléique hétérologue codant pour une dihydroxyphénylalanine
annnnonia-lyase (DAL).
Le microorganisme recombinant peut donc comprendre une séquence d'acide
nucléique
hétérologue codant pour une coumarate 3-hydroxylase (Counn3H) capable de
convertir
l'acide p-counnarique en acide caféique.
Dans un mode de réalisation, la coumarate 3-hydroxylase est une enzyme de
bactérie,
notamment de Saccharothrix.
Dans un mode de réalisation particulier, la 4-méthoxybenzoate 0-déméthylase
est
sélectionnée l'enzyme comprenant une séquence choisie parmi la SEQ ID NO: 71
et les
polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 %
d'identité de séquence avec une de ces séquences et présentant l'activité
coumarate 3-
hydroxylase.
La séquence nucléique codant cette enzyme et séquence protéique est décrite
dans NCBI
sous les numéros de référence DQ357071.1 et ABC88666.1, respectivement, et
plus
particulièrement dans les SEQ ID Nos : 72 et 71.
Dans un mode de réalisation, le microorganisme peut comprendre une séquence
d'acide
nucléique hétérologue codant pour une coumarate 3-hydroxylase et une séquence
d'acide
nucléique hétérologue codant pour une dihydroxyphénylalanine ammonia-lyase
(DAL).
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
68
Combinaison d'enzymes supplémentaire
Ainsi, outre les enzymes nécessaires à la biosynthèse de l'hespéridine et/ou
la diosnnine à
partir de la naringénine et/ou de l'apigénine telles que décrites
précédemment, le
microorganisme comprend de préférence les enzymes permettant la production de
la
naringénine et/ou de l'apigénine à partir de la tyrosine et/ou de la
phénylalanine, de
préférence à partir de la tyrosine.
Ainsi, selon des modes de réalisation particuliers, le microorganisme comprend
- une séquence d'acide nucléique hétérologue codant pour une flayanone 7-0-
bêta-D-
glucosyltransférase (UGT) capable d'ajouter un glucose en position 7 de
l'hespérétine et/ou
la diosmétine,
- une séquence d'acide nucléique hétérologue codant pour une 6"-O-
rhamnosyltransferase
(RhaT) capable de transférer un rhamnose en position 6 du glucose de
l'hespérétine-7-0-
glucoside et/ou la diosmétine-7-0-glucoside,
- une séquence d'acide nucléique hétérologue codant pour une UDP-glucose
4,6-
déshydratase/UDP-4-kéto-6-désoxy-D-glucose 3,5-épimerase/UDP-4-kéto-L-rhamnose-
réductase (RHM) capable de produire de l'UDP-rhamnose,
- une séquence d'acide nucléique hétérologue codant pour une 0-
méthyltransférase
(OMT) capable de méthyler l'ériodictyol et/ou la lutéoline en position 4',
- une séquence d'acide nucléique hétérologue codant pour une enzyme F3'H,
- et optionnellement une séquence d'acide nucléique hétérologue codant pour
une enzyme
FNS et une séquence d'acide nucléique hétérologue codant pour une enzyme CPR,
et comprend en outre
- une séquence d'acide nucléique hétérologue codant pour une tyrosine
ammonia lyase
(TAL), une séquence d'acide nucléique hétérologue codant pour une 4-coumaroyl-
CoA
ligase (4CL), une séquence d'acide nucléique hétérologue codant pour une
chalcone
synthase (CHS) et une séquence d'acide nucléique hétérologue codant pour une
chalcone
isomérase (CHI).
Dans une mode de réalisation, le microorganisme comprend
-une séquence d'acide nucléique hétérologue codant pour une 0-
méthyltransférase (OMT) capable de méthyler l'ériodictyol et/ou la lutéoline
en position
4'; de préférence une OMT de Citrus clementina, de Citrus sinensis,
d'Arabidopsis thaliana
ou d'Homo sapiens, de préférence une OMT comprenant une séquence choisie parmi
SEQ
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
69
ID NOs: 117, 119, 87 et 89 et les polypeptides comprenant une séquence ayant
au moins
60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec une de ces séquences et
présentant
une activité 0-nnéthyltransférase, notamment avec l'ériodictyol et/ou la
lutéoline comme
substrat et une nnéthylation en position 4', de préférence une OMT comprenant
une
séquence choisie parmi SEQ ID NOs: 117 et 119 et les polypeptides comprenant
une
séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec
une de ces
séquences et présentant une activité 0-nnéthyltransférase, et manière tout
particulièrement préférée une OMT comprenant une séquence choisie parmi SEQ ID
NO:
117 et les polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85,
90 ou 95
% d'identité de séquence avec cette séquence et présentant une activité 0-
nnéthyltra nsférase; et
- une séquence d'acide nucléique hétérologue codant pour une flavonoïde 3'-
nnonooxygénase (F3'H) capable d'hydroxyler en position 3' la naringénine et/ou
l'apigénine
et comprenant une séquence sélectionnée parmi les SEQ ID NOs: 7, 11, 17 et
121, et les
polypeptides comprenant une séquence ayant au moins 75%, au moins 80%, au
moins 85%,
au moins 90% ou au moins 95% d'identité avec l'une de ces séquences et
présentant
l'activité flavonoïde 3'-nnonooxygénase, de préférence une enzyme comprenant
une
séquence sélectionnée parmi les SEQ ID NOs: 7, 17 et 121, et les polypeptides
comprenant
une séquence ayant au moins 75%, au moins 80%, au moins 85%, au moins 90% ou
au
moins 95% d'identité avec l'une de ces séquences et présentant l'activité
flavonoïde 3'-
nnonooxygénase, et de manière tout particulièrement préférée une enzyme
comprenant
une séquence sélectionnée parmi la SEQ ID NO: 7, et les polypeptides
comprenant une
séquence ayant au moins 75%, au moins 80%, au moins 85%, au moins 90% ou au
moins
95% d'identité avec cette séquence et présentant l'activité flavonoïde 3'-
nnonooxygénase ;
et
-facultativement, une séquence d'acide nucléique hétérologue codant pour une
cytochrome P450 réductase (CPR) et comprenant une séquence choisie parmi SEQ
ID NOs:
23, 25, 27, 29 et 31 et les polypeptides comprenant une séquence ayant au
moins 60, 70,
80, 85, 90 ou 95 % d'identité de séquence avec une de ces séquences et
présentant une
activité cytochrome P450 réductase, de préférence parmi les enzymes comprenant
une
séquence choisie parmi SEQ ID NOs: 23, 25 et 29 et les polypeptides comprenant
une
séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec
une de ces
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
séquences et présentant une activité cytochrome P450 réductase, et de manière
particulière parmi les enzymes comprenant une séquence choisie parmi SEQ ID
NO: 25 et
les polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou
95 %
d'identité de séquence avec cette séquence et présentant une activité
cytochrome P450
5 réductase ; et
-facultativement, une séquence d'acide nucléique hétérologue codant pour une
flavone synthase (FNS) et comprenant une séquence choisie parmi SEQ ID NOs:
33, 35, 37,
127, 129, 131, 133, 135, 137, 139, 141, 143, 145, 147, 149, 151, 153, 155, 157
et 159 et les
polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 %
10 d'identité de séquence avec une de ces séquences et présentant une
activité flavone
synthase, en particulier pour une flavone synthase (FNS) et comprenant une
séquence
choisie parmi SEQ ID NOs: 33, 35, 37 et les polypeptides comprenant une
séquence ayant
au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec une de ces
séquences et
présentant une activité flavone synthase, de préférence une flavone synthase
(FNS)
15 comprenant une séquence choisie parmi SEQ ID NO: 37 et un polypeptide
comprenant une
séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec
cette
séquence et présentant une activité flavone synthase ; et
-une séquence d'acide nucléique hétérologue codant pour une tyrosine ammonia
lyase (TAL) de Rhodotorula glutinis ou Flavobacterium johnsoniae ; en
particulier une TAL
20 comprenant une séquence choisie parmi SEQ ID NOs: 41 et 39 et les
polypeptides
comprenant une séquence ayant au moins 60, 70,80, 85, 90 ou 95% d'identité de
séquence
avec une de ces séquences et présentant une activité tyrosine ammonia lyase ;
de
préférence une TAL comprenant une séquence choisie parmi SEQ ID NO: 41 et les
polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 %
25 d'identité de séquence avec cette séquence et présentant une activité
tyrosine ammonia
lyase ; et
-une séquence d'acide nucléique hétérologue codant pour une 4-coumaroyl-CoA
ligase (4CL) d'Arabidopsis thaliana, Citrus clementina, Petroselinum crispum
ou
Streptomyces clavuligerus ; en particulier une 4CL comprenant une séquence
choisie parmi
30 SEQ ID NOs: 123, 125, 45, 43, 47 et 49 et les polypeptides comprenant
une séquence ayant
au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec une de ces
séquences et
présentant une activité 4-coumarate-CoA ligase ; de préférence une 4CL
comprenant une
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
71
séquence sélectionnée parmi les SEQ ID NOs: 123, 125 et 45 et les polypeptides
comprenant une séquence ayant au moins 60, 70,80, 85, 90 ou 95% d'identité de
séquence
avec une de ces séquences et présentant une activité 4-counnarate-CoA ligase ;
de manière
tout particulièrement préférée une 4CL comprenant une séquence sélectionnée
parmi la
SEQ ID NO: 45 et les polypeptides comprenant une séquence ayant au moins 60,
70, 80, 85,
90 ou 95 % d'identité de séquence avec cette séquence et présentant une
activité 4-
counnarate-CoA ligase, et
-une séquence d'acide nucléique hétérologue codant pour une chalcone synthase
(CHS) de Citrus sinensis, de Hordeum vulgare ou de Streptomyces clavuligerus,
en
particulier une CHS comprenant une séquence choisie parmi SEQ ID NOs: 53, 51,
55 et 57
et les polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90
ou 95 %
d'identité de séquence avec une de ces séquences et présentant une activité
chalcone
synthase, de préférence une CHS comprenant une séquence choisie parmi SEQ ID
NO: 53
et les polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90
ou 95 %
d'identité de séquence avec cette séquence et présentant une activité chalcone
synthase;
et
-une séquence d'acide nucléique hétérologue codant pour une chalcone
isonnérase
(CHI) d'Arabidopsis thaliana ou Streptomyces clavuligerus, en particulier une
CHI
comprenant une séquence choisie parmi SEQ ID NOs: 61 et 59 et les polypeptides
comprenant une séquence ayant au moins 60, 70,80, 85, 90 ou 95% d'identité de
séquence
avec une de ces séquences et présentant une activité chalcone isonnérase ; de
préférence
une CHI comprenant une séquence choisie parmi la SEQ ID NO: 61 et les
polypeptides
comprenant une séquence ayant au moins 60, 70,80, 85, 90 ou 95% d'identité de
séquence
avec cette séquence et présentant une activité cha Icone isonnérase .
De préférence, dans ce mode de réalisation, le microorganisme comprend l'une
des
combinaisons d'enzymes UGT, RhaT et RHM décrites plus haut, en particulier
- la flavanone 7-0-bêta-D-glucosyltransférase sélectionnée parmi les enzymes
comprenant
une séquence choisie parmi SEQ ID NOs: 113 et 95 et les polypeptides
comprenant une
séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec
une de ces
séquences et présentant une activité flavanone 7-0-bêta-D-glucosyltransférase,
de
préférence sélectionnée parmi les enzymes comprenant une séquence choisie
parmi la SEQ
ID NO: 113 et les polypeptides comprenant une séquence ayant au moins 60, 70,
80, 85, 90
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
72
ou 95 % d'identité de séquence avec cette séquence et présentant une activité
flavanone
7-0-bêta-D-glucosyltransférase ; et
- la 6"-O-rhannnosyltransferase sélectionnée parmi les enzymes comprenant
une
séquence choisie parmi la SEQ ID NO: 103 et les polypeptides comprenant une
séquence
ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec cette
séquence et
présentant une activité 6"-O-rhannnosyltransferase ; et
- l'UDP-glucose 4,6-
déshydratase/UDP-4-kéto-6-désoxy-D-glucose 3,5-
épinnerase/UDP-4-kéto-L-rhannnose-réductase sélectionnée parmi les enzymes
comprenant une séquence choisie parmi la SEQ ID NO: 107 et les polypeptides
comprenant
une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence
avec cette
séquence et présentant une activité UDP-glucose 4,6-déshydratase/UDP-4-kéto-6-
désoxy-
D-glucose 3,5-épinnerase/UDP-4-kéto-L-rhannnose-réductase.
De préférence, dans ce mode de réalisation, le microorganisme comprend en
outre
- une séquence d'acide nucléique hétérologue codant pour une phénylalanine
annnnonia lyase (PAL), en particulier une PAL comprenant une séquence choisie
parmi SEQ
ID NOs: 63, 65 et 77, de préférence SEQ ID NO : 65 et 77, et les polypeptides
comprenant
une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence
avec une
de ces séquences et présentant une activité phénylalanine annnnonia lyase, et
de manière
plus particulièrement préférée une phénylalanine annnnonia lyase (PAL)
comprenant une
séquence choisie parmi la SEQ ID NO: 65 et les polypeptides comprenant une
séquence
ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec cette
séquence et
présentant une activité phénylalanine annnnonia lyase ; et
- une séquence d'acide nucléique hétérologue codant pour une cinnannate 4-
hydroxylase (C4H), en particulier une C4H comprenant une séquence choisie
parmi SEQ ID
NOs: 67, 69, 79 et les polypeptides comprenant une séquence ayant au moins 60,
70, 80,
85, 90 ou 95 % d'identité de séquence avec une de ces séquences et présentant
une activité
cinnannate 4-hydroxylase ; et de manière tout particulièrement préférée une
cinnannate 4-
hydroxylase (C4H) comprenant une séquence choisie parmi la SEQ ID NO: 79 et
les
polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 %
d'identité de séquence avec cette séquence et présentant une activité
cinnamate 4-
hydroxylase.
Dans un autre mode de réalisation, le microorganisme comprend :
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
73
-une séquence d'acide nucléique hétérologue codant pour une 0-
nnéthyltransférase (OMT) comprenant une séquence choisie parmi SEQ ID NOs: 117
et 119
et les polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90
ou 95 %
d'identité de séquence avec une de ces séquences et présentant une activité 0-
nnéthyltransférase, et manière tout particulièrement préférée une OMT
comprenant une
séquence choisie parmi SEQ ID NO: 117 et les polypeptides comprenant une
séquence
ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec cette
séquence et
présentant une activité 0-nnéthyltransférase; et
- une séquence d'acide nucléique hétérologue codant pour une flavonoïde 3'-
monooxygénase (F3'H) capable d'hydroxyler en position 3' la naringénine et/ou
l'apigénine
et comprenant une séquence sélectionnée parmi les SEQ ID NOs: 7, 17 et 121, et
les
polypeptides comprenant une séquence ayant au moins 75%, au moins 80%, au
moins 85%,
au moins 90% ou au moins 95% d'identité avec l'une de ces séquences et
présentant
l'activité flavonoïde 3'-monooxygénase, de préférence une enzyme comprenant
une
séquence sélectionnée parmi la SEQ ID NO: 7, et les polypeptides comprenant
une
séquence ayant au moins 75%, au moins 80%, au moins 85%, au moins 90% ou au
moins
95% d'identité avec cette séquence et présentant l'activité flavonoïde 3'-
monooxygénase ;
et
-facultativement, une séquence d'acide nucléique hétérologue codant pour une
cytochrome P450 réductase (CPR) et comprenant une séquence choisie parmi SEQ
ID NOs:
23, 25 et 29 et les polypeptides comprenant une séquence ayant au moins 60,
70, 80, 85,
90 ou 95 % d'identité de séquence avec une de ces séquences et présentant une
activité
cytochrome P450 réductase, de préférence choisie parmi les enzymes comprenant
une
séquence choisie parmi SEQ ID NO: 25 et les polypeptides comprenant une
séquence ayant
au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec cette séquence
et
présentant une activité cytochrome P450 réductase; et
-facultativement, une séquence d'acide nucléique hétérologue codant pour une
flavone synthase (FNS) et comprenant une séquence choisie parmi SEQ ID NO: 37
et un
polypeptide comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 %
d'identité
de séquence avec cette séquence et présentant une activité flavone synthase ;
et
- une séquence d'acide nucléique hétérologue codant pour une tyrosine
ammonia
lyase (TAL) comprenant une séquence choisie parmi SEQ ID NO: 41 et les
polypeptides
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
74
comprenant une séquence ayant au moins 60, 70,80, 85, 90 ou 95% d'identité de
séquence
avec cette séquence et présentant une activité tyrosine annnnonia lyase ;
- une séquence d'acide nucléique hétérologue codant pour une 4-counnaroyl-
CoA
ligase (4CL) comprenant une séquence sélectionnée parmi les SEQ ID NO: 123,
125 et 45 et
les polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou
95 %
d'identité de séquence avec ces séquences et présentant une activité 4-
coumarate-CoA
ligase de manière tout particulièrement préférée une 4CL comprenant une
séquence
sélectionnée parmi la SEQ ID NO: 45 et les polypeptides comprenant une
séquence ayant
au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec cette séquence
et
présentant une activité 4-cou ma rate-CoA ligase; et
- une séquence d'acide nucléique hétérologue codant pour une chalcone
synthase
(CHS) comprenant une séquence choisi parmi SEQ ID NO: 53 et les polypeptides
comprenant une séquence ayant au moins 60, 70,80, 85, 90 ou 95% d'identité de
séquence
avec cette séquence et présentant une activité cha Icone synthase; et
- une séquence d'acide nucléique hétérologue codant pour une chalcone
isomérase (CHI) comprenant une séquence choisie parmi la SEQ ID NO: 61 et les
polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 %
d'identité de séquence avec cette séquence et présentant une activité chalcone
isomérase .
De préférence, dans ce mode de réalisation, le microorganisme comprend l'une
des
combinaisons d'enzymes UGT, RhaT et RHM décrites plus haut, en particulier
- la flavanone 7-0-bêta-D-glucosyltransférase sélectionnée parmi les
enzymes
comprenant une séquence choisie parmi SEQ ID NOs: 113 et 95 et les
polypeptides
comprenant une séquence ayant au moins 60, 70,80, 85, 90 ou 95% d'identité de
séquence
avec une de ces séquences et présentant une activité flavanone 7-0-bêta-D-
glucosyltransférase, de préférence sélectionnée parmi les enzymes comprenant
une
séquence choisie parmi la SEQ ID NO: 113 et les polypeptides comprenant une
séquence
ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec cette
séquence et
présentant une activité flavanone 7-0-bêta-D-glucosyltransférase ; et
- la 6"-O-rhamnosyltransferase sélectionnée parmi les enzymes comprenant une
séquence choisie parmi la SEQ ID NO: 103 et les polypeptides comprenant une
séquence
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec cette
séquence et
présentant une activité 6"-O-rhannnosyltransferase ; et
- l'UDP-glucose 4,6-déshydratase/UDP-4-kéto-6-désoxy-D-glucose
3,5-
épinnerase/UDP-4-kéto-L-rhannnose-réductase sélectionnée parmi les enzymes
5 comprenant une séquence choisie parmi la SEQ ID NO: 107 et les
polypeptides comprenant
une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence
avec cette
séquence et présentant une activité UDP-glucose 4,6-déshydratase/UDP-4-kéto-6-
désoxy-
D-glucose 3,5-épinnerase/UDP-4-kéto-L-rhannnose-réductase.
De préférence, dans ce mode de réalisation, le microorganisme comprend en
outre une
10 séquence d'acide nucléique hétérologue codant pour une phénylalanine
annnnonia lyase
(PAL) et une séquence d'acide nucléique hétérologue codant pour une cinnannate
4-
hydroxylase (C4H) comme décrites dans le mode de réalisation précédent.
Facultativement, dans ces différents modes de réalisation, le microorganisme
comprend
en outre une séquence d'acide nucléique hétérologue ou endogène codant pour
une S-
15 adénosylnnéthionine synthétase (SAMT); en particulier de
Saccharomyces cerevisiae, par
exemple une SAMT comprenant une séquence choisie parmi SEQ ID NO: 81 et un
polypeptide comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 %
d'identité
de séquence avec cette séquence et présentant une activité S-
adénosylnnéthionine
synthétase.
20 Dans un autre mode de réalisation particulier, le microorganisme
comprend :
- une séquence d'acide nucléique hétérologue codant pour une flavanone
7-0-bêta-
D-glucosyltransférase (UGT) d'Arabidopsis thaliana, de Scutellaria baicalensis
ou
d'Homo sapiens, de préférence d'Arabidopsis thaliana ou de Scutellaria
baicalensis,
de préférence une flavanone 7-0-bêta-D-glucosyltransférase (UGT) sélectionnée
25 parmi les enzymes comprenant une séquence choisie parmi SEQ ID NOs:
91, 93, 95,
97, 99 et 101 et les polypeptides comprenant une séquence ayant au moins 60,
70,
80, 85, 90 ou 95 % d'identité de séquence avec une de ces séquences et
présentant
une activité flavanone 7-0-bêta-D-glucosyltransférase ; de manière
particulière une
flavanone 7-0-bêta-D-glucosyltransférase (UGT) comprenant une séquence choisie
30 parmi SEQ ID NOs: 91, 93, 95 et 97 et les polypeptides comprenant
une séquence
ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec une de
ces
séquences et présentant une activité flava none 7-0-bêta-D-
glucosyltransférase, et
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
76
de préférence une UGT comprenant une séquence choisie parmi la SEQ ID NO: 113
et les polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90
ou
95 % d'identité de séquence avec cette séquence et présentant une activité
flavanone 7-0-bêta-D-glucosyltransférase;
- une séquence d'acide nucléique hétérologue codant pour une 6"-O-
rhannnosyltransferase (RhaT) du genre Citrus ou de Petunia hybrida, de
préférence
Citrus sinensis, Citrus maxima, ou Citrus clementina, de manière encore plus
préférée Citrus sinensis ou Citrus clementina, de préférence une 6"-O-
rhannnosyltransferase (RhaT) comprenant une séquence choisie parmi SEQ ID NOs:
103, 105 et les polypeptides comprenant une séquence ayant au moins 60, 70,
80,
85, 90 ou 95 % d'identité de séquence avec une de ces séquences et présentant
une
activité 6"-O-rhannnosyltransferase, et de préférence une RhaT comprenant une
séquence choisie parmi la SEQ ID NO: 103 et les polypeptides comprenant une
séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec
cette séquence et présentant une activité 6"-O-rhannnosyltransferase; et
- une séquence d'acide nucléique hétérologue codant pour une UDP-glucose 4,6-
déshyd ratase/U DP-4-kéto-6-désoxy-D-gl ucose 3,5-
épinnerase/UDP-4-kéto-L-
rhannnose-réductase (RHM) de Citrus sinensis ou d'Arabidopsis thaliana, de
préférence une RHM comprenant une séquence choisie parmi SEQ ID NOs: 107, 109
et 111 et les polypeptides comprenant une séquence ayant au moins 60, 70, 80,
85,
90 ou 95 % d'identité de séquence avec une de ces séquences et présentant une
activité UDP-glucose 4,6-déshydratase/UDP-4-kéto-6-désoxy-D-glucose 3,5-
épinnerase/U DP-4-kéto-L-rhannnose-réductase, en particulier une
RHM
comprenant une séquence sélectionnée parmi SEQ ID NOs: 107, 109 et les
polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 %
d'identité de séquence avec cette séquence et présentant une activité UDP-
glucose
4,6-déshydratase/UDP-4-kéto-6-désoxy-D-glucose 3,5-
épinnerase/UDP-4-kéto-L-
rhannnose-réductase, et de préférence une RHM comprenant une séquence choisie
parmi la SEQ ID NO: 107 et les polypeptides comprenant une séquence ayant au
moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec cette séquence et
présentant une activité UDP-glucose 4,6-déshydratase/UDP-4-kéto-6-désoxy-D-
glucose 3,5-épinnerase/UDP-4-kéto-L-rhannnose-réductase;
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
77
- une séquence d'acide nucléique hétérologue codant pour une flavonoïde 3'-
nnonooxygénase (F3'H) capable d'hydroxyler en position 3' la naringénine et/ou
l'apigénine ; de préférence de Perilla frutescens var. crispa, Petunia x
hybrida,
Callistephus chinensis, Gerbera hybrida, Citrus sinensis, Citrus clementina,
Osteospermum hybrid cultivar, Phanerochaete chrysosporium, Streptomyces
avermitilis ou Pilosella officinarum, en particulier Perilla frutescens var.
crispa,
Petunia x hybrida, Callistephus chinensis, Gerbera hybrida, Citrus sinensis ou
Pilosella officinarum, de préférence une F3'H comprenant une séquence choisie
parmi SEQ ID NOs: 1, 3, 5, 7, 9, 11, 13, 15, 17, 19 et 21 et les polypeptides
comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité
de
séquence avec une de ces séquences et présentant une activité flavonoïde 3'-
nnonooxygénase, de préférence sélectionnée parmi les enzymes comprenant une
séquence choisie parmi SEQ ID NOs: 1, 5, 7, 11, 17 et 19 et les polypeptides
comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité
de
séquence avec une de ces séquences et présentant une activité flavonoïde 3'-
nnonooxygénase, en particulier une F3'H comprenant une séquence sélectionnée
parmi les SEQ ID NOs: 5, 7, 17 et les polypeptides comprenant une séquence
ayant
au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec cette séquence
et
présentant une activité flavonoïde 3'-monooxygénase, de manière plus
particulièrement préférée une F3'H comprenant une séquence choisie parmi SEQ
ID NO: 7 et les polypeptides comprenant une séquence ayant au moins 60, 70,
80,
85, 90 ou 95 % d'identité de séquence avec la séquence SEQ ID NO: 7 et
présentant
l'activité flavonoïde 3'-nnonooxygénase ;
- facultativement, une séquence d'acide nucléique hétérologue codant pour une
cytochrome P450 réductase (CPR) ; de préférence une CPR de Saccharomyces
cerevisiae, ou de plante, par exemple de Catharanthus roseus ou d'Arabidopsis
thaliana ; de préférence une CPR comprenant une séquence choisie parmi SEQ ID
NOs: 23, 25, 27, 29 et 31 et les polypeptides comprenant une séquence ayant au
moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec une de ces
séquences
et présentant une activité cytochrome P450 réductase, de préférence parmi les
enzymes comprenant une séquence choisie parmi SEQ ID NOs: 23, 25 et 29 et les
polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 %
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
78
d'identité de séquence avec une de ces séquences et présentant une activité
cytochrome P450 réductase, en particulier une CPR comprenant une séquence
choisie parmi SEQ ID NO: 25 et les polypeptides comprenant une séquence ayant
au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec cette
séquences et
présentant une activité cytochrome P450 réductase ;
- une séquence d'acide nucléique hétérologue codant pour une 0-
nnéthyltransférase
(OMT) capable de nnéthyler l'ériodictyol et/ou la lutéoline en position 4'; de
préférence une OMT d'Arabidopsis thaliana ou d'Homo sapiens, de préférence une
OMT comprenant une séquence choisie parmi SEQ ID NOs: 87 et 89 et les
polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 %
d'identité de séquence avec une de ces séquences et présentant une activité 0-
nnéthyltransférase, notamment avec l'ériodictyol et/ou la lutéoline comme
substrat
et une nnéthylation en position 4', de préférence sélectionnée parmi l'enzyme
comprenant une séquence choisie parmi SEQ ID NO 89 et les polypeptides
comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité
de
séquence avec cette séquence et présentant une activité 0-nnéthyltransférase ;
- facultativement, une séquence d'acide nucléique hétérologue ou endogène
codant
pour une S-adénosylnnéthionine synthétase (SAMT) ; en particulier de
Saccharomyces cerevisiae, par exemple une SAMT comprenant une séquence
choisie parmi SEQ ID NO: 81 et un polypeptide comprenant une séquence ayant au
moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec cette séquence et
présentant une activité S-adénosylnnéthionine synthétase ; et
- facultativement, une séquence d'acide nucléique hétérologue codant pour
une
flavone synthase (FNS) capable de transformer la naringénine en apigénine,
l'ériodictyol en lutéoline et/ou l'hespérétine en diosnnétine, de préférence
de
transformer l'ériodictyol en lutéoline; de préférence une FNS d'Arabidopsis
thaliana, Lonicera japonica, Lonicera macranthoides, Medicago truncatula,
Oryza
sativa, Petroselinum crispum, Pop ulus deltoides, Zea mays, Callistephus
chinensis,
Apium graveolens, Medicago truncatula, Cuminum cyminum, Aethusa cynapium,
Angelico archangelica, Conium maculatum, Camellia sinensis, Cynara cardunculus
var scolymus, Saussurea medusa, Plectranthus barbatus, Scutellaria
baicalensis,
Dorcoceras hygrometricum, Antirrhinum majus, Perilla frutescens var crispa,
Dahlia
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
79
pinnata ou Erythranthe lewisii, en particulier d'Arabidopsis thaliana,
Lonicera
japonica, Lonicera macranthoides, Medicago truncatula, Oryza sativa,
Petroselinum
crispum, Populus deltoides ou Zea mays, de préférence de Lonicera japonica,
Lonicera macranthoides, et Petroselinum crispum; de préférence une FNS
comprenant une séquence choisie parmi SEQ ID NOs: 33, 35, 37, 127, 129, 131,
133,
135, 137, 139, 141, 143, 145, 147, 149, 151, 153, 155, 157 et 159 et les
polypeptides
comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité
de
séquence avec une de ces séquences et présentant une activité flavone
synthase,
de préférence parmi les enzymes comprenant une séquence choisie parmi SEQ ID
NOs: 33, 35 et 37 et les polypeptides comprenant une séquence ayant au moins
60,
70, 80, 85, 90 ou 95 % d'identité de séquence avec une de ces séquences et
présentant une activité flavone synthase, de préférence une flavone synthase
(FNS)
comprenant une séquence choisie parmi SEQ ID NO: 37 et un polypeptide
comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité
de
séquence avec cette séquence et présentant une activité flavone synthase;
- une séquence d'acide nucléique hétérologue codant pour une tyrosine
annnnonia
lyase (TAL) capable de produire de l'acide p-counnarique à partir de la
tyrosine; de
préférence de Rhodotorula glutinis ou Flavobacterium johnsoniae ; en
particulier
une TAL comprenant une séquence choisie parmi SEQ ID NOs: 39 et 41 et les
polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 %
d'identité de séquence avec une de ces séquences et présentant une activité
tyrosine annnnonia lyase ; de préférence une TAL comprenant une séquence
choisie
parmi SEQ ID NO: 41 et les polypeptides comprenant une séquence ayant au moins
60, 70, 80, 85, 90 ou 95% d'identité de séquence avec cette séquence et
présentant
une activité tyrosine annnnonia lyase;
- une séquence d'acide nucléique hétérologue codant pour une 4-counnarate-CoA
ligase (4CL) capable de produire de le coumaryl-CoA à partir de l'acide p-
counnarique et de Coenzyme A; de préférence d'Arabidopsis thaliana,
Petroselinum
crispum ou Streptomyces clavuligerus ; une 4CL comprenant une séquence choisie
parmi SEQ ID NOs: 43, 45, 47 et 49 et les polypeptides comprenant une séquence
ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec une de
ces
séquences et présentant une activité 4-counnarate-CoA ligase, de préférence
une
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
4CL comprenant une séquence sélectionnée parmi la SEQ ID NO: 45 et les
polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 %
d'identité de séquence avec cette séquence et présentant une activité 4-
counnarate-CoA ligase ;
5 - une
séquence d'acide nucléique hétérologue codant pour une chalcone synthase
(CHS) capable de produire de la naringénine-chalcone à partir du 4-coumaroyl-
CoA
et de malonyl-CoA ; de préférence de Citrus sinensis, de Hordeum vulgare ou de
Streptomyces clavuligerus, en particulier une CHS comprenant une séquence
choisie parmi SEQ ID NOs: 51, 53, 55 et 57 et les polypeptides comprenant une
10 séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité de
séquence avec
une de ces séquences et présentant une activité chalcone synthase, de
préférence
parmi les enzymes comprenant une séquence choisie parmi SEQ ID NOs: 53 et 55
et les polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90
ou
95 % d'identité de séquence avec une de ces séquences et présentant une
activité
15 chalcone synthase, et de manière plus particulièrement préférée une
CHS
comprenant une séquence choisie parmi SEQ ID NO: 53 et les polypeptides
comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité
de
séquence avec cette séquences et présentant une activité chalcone synthase ;
- une
séquence d'acide nucléique hétérologue codant pour une chalcone isomérase
20 (CHI)
capable de produire la naringénine à partir de la naringénine chalcone; de
préférence d'Arabidopsis thaliana ou Streptomyces clavuligerus, en particulier
une
CHI comprenant une séquence choisie parmi SEQ ID NOs: 59 et 61 et les
polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 %
d'identité de séquence avec une de ces séquences et présentant une activité
25 chalcone isomérase, et de manière plus particulièrement préférée une
CHI
comprenant une séquence choisie parmi la SEQ ID NO: 61 et les polypeptides
comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité
de
séquence avec cette séquence et présentant une activité chalcone isomérase .
Dans un autre mode de réalisation particulier, le microorganisme comprend les
séquences
30 d'acides nucléiques hétérologues codant pour les enzymes UGT, RhaT,
RHM, F3'H, OMT,
4CL, CHS, CHI, et facultativement pour les enzymes CPR, FNS et SAMT, telles
que décrites
dans le mode de réalisation précédent et comprend en outre :
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
81
- une séquence d'acide nucléique hétérologue codant pour une phénylalanine
annnnonia lyase (PAL), en particulier une PAL comprenant une séquence choisie
parmi SEQ ID NOs: 63, 65, 77 et les polypeptides comprenant une séquence ayant
au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec une de ces
séquences et présentant une activité phénylalanine annnnonia lyase, en
particulier
une PAL comprenant une séquence choisie parmi SEQ ID NO: 65 et les
polypeptides
comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité
de
séquence avec cette séquences et présentant une activité phénylalanine
annnnonia
lyase, et une séquence d'acide nucléique hétérologue codant pour une
cinnannate
4-hydroxylase (C4H), en particulier une C4H comprenant une séquence choisie
parmi SEQ ID NOs: 67, 69, 79 et les polypeptides comprenant une séquence ayant
au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec une de ces
séquences et présentant une activité cinnannate 4-hydroxylase, capables de
produire de l'acide p-counnarique à partir de la phénylalanine, en particulier
une
C4H comprenant une séquence choisie parmi SEQ ID NO: 79 et les polypeptides
comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité
de
séquence avec cette séquence et présentant une activité cinnannate 4-
hydroxylase.
Dans un troisième mode de réalisation particulier, le microorganisme comprend
les
séquences d'acides nucléiques hétérologues codant pour les enzymes UGT, RhaT,
RHM,
F3'H, OMT, 4CL, CHS, CHI, et facultativement pour les enzymes CPR, FNS et
SAMT, telles
que décrites dans le mode de réalisation précédent et comprend en outre :
- une séquence d'acide nucléique hétérologue codant pour une
dihydroxyphénylalanine annnnonia-lyase (DAL) capable de produire de l'acide
caféique à partir de la L-DOPA (3,4-dihydroxy-L-phenylalanine);
- une séquence d'acide nucléique hétérologue codant pour une sous-unité 4-
hydroxyphénylacetate 3-nnonooxygénase oxygénase (HpaB), de préférence
comprenant une séquence choisie parmi SEQ ID NO: 83 et les polypeptides
comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité
de
séquence avec celle-ci et présentant une activité 4-hydroxyphénylacetate 3-
nnonooxygénase oxygénase, et une séquence d'acide nucléique hétérologue codant
pour une sous-unité 4-hydroxyphénylacetate 3-nnonooxygénase réductase (HpaC),
de préférence comprenant une séquence choisie parmi SEQ ID NO: 85 et les
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
82
polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 %
d'identité de séquence avec celle-ci et présentant une activité 4-
hydroxyphénylacetate 3-nnonooxygénase réductase; ou une séquence d'acide
nucléique hétérologue codant pour une 4-nnéthoxybenzoate 0-déméthylase
capable de convertir la tyrosine en L-Dopa et également l'acide p counnarique
en
acide caféique, de préférence comprenant une séquence choisie parmi SEQ ID
NOs:
73 et 75 et les polypeptides comprenant une séquence ayant au moins 60, 70,
80,
85, 90 ou 95 % d'identité de séquence avec une de ces séquences et présentant
une
activité L-tyrosine hydroxylase; ou une séquence d'acide nucléique hétérologue
codant pour une p-coumarate 3-hydroxylase capable de convertir l'acide p-
coumarique en acide caféique, de préférence comprenant une séquence choisie
parmi SEQ ID No : 71 et les polypeptides comprenant une séquence ayant au
moins
60, 70, 80, 85, 90 ou 95 % d'identité d'une séquence avec une de ces séquences
et
présentant une activité p-coumarate 3 hydroxylase.
Dans un autre mode de réalisation particulier, le microorganisme comprend les
séquences
d'acides nucléiques hétérologues codant pour les enzymes UGT, RhaT, RHM, F3'H,
OMT,
4CL, CHS, CHI, et facultativement pour les enzymes CPR, FNS et SAMT, telles
que décrites
dans le mode de réalisation précédent et comprend en outre :
- une séquence d'acide nucléique hétérologue codant pour une tyrosine ammonia
lyase (TAL) capable de produire de l'acide p-coumarique à partir de la
tyrosine; de
préférence de Rhodotorula glutinis ou Flavobacterium johnsoniae ; en
particulier
une TAL comprenant une séquence choisie parmi SEQ ID NOs: 39 et 41 et les
polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 %
d'identité de séquence avec une de ces séquences et présentant une activité
tyrosine ammonia lyase ; de préférence une TAL comprenant une séquence choisie
parmi SEQ ID NO: 41 et les polypeptides comprenant une séquence ayant au moins
60, 70, 80, 85, 90 ou 95% d'identité de séquence avec cette séquence et
présentant
une activité tyrosine ammonia lyase ;
- facultativement, une séquence d'acide nucléique hétérologue codant pour une
phénylalanine ammonia lyase (PAL), en particulier une PAL comprenant une
séquence choisie parmi SEQ ID NOs: 63, 65, 77 et les polypeptides comprenant
une
séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
83
une de ces séquences et présentant une activité phénylalanine annnnonia lyase,
de
préférence une PAL comprenant une séquence choisie parmi la SEQ ID NO: 65 et
les
polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 %
d'identité de séquence avec cette séquence et présentant une activité
phénylalanine annnnonia lyase ; et une séquence d'acide nucléique hétérologue
codant pour une cinnannate 4-hydroxylase (C4H), en particulier une C4H
comprenant une séquence choisie parmi SEQ ID NOs: 67, 69, 79 et les
polypeptides
comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité
de
séquence avec une de ces séquences et présentant une activité cinnannate 4-
hydroxylase, capables de produire de l'acide p-counnarique à partir de la
phénylalanine, de préférence une C4H comprenant une séquence choisie parmi la
SEQ ID NO: 79 et les polypeptides comprenant une séquence ayant au moins 60,
70, 80, 85, 90 ou 95 % d'identité de séquence avec cette séquence et
présentant
une activité cinnannate 4-hydroxylase;
- facultativement, une séquence d'acide nucléique hétérologue codant pour une
sous-unité 4-hydroxyphénylacetate 3-nnonooxygénase oxygénase (HpaB), de
préférence comprenant une séquence choisie parmi SEQ ID NO: 83 et les
polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 %
d'identité de séquence avec celle-ci et présentant une activité 4-
hydroxyphénylacetate 3-nnonooxygénase oxygénase, et une séquence d'acide
nucléique hétérologue codant pour une sous-unité 4-hydroxyphénylacetate 3-
nnonooxygénase réductase (HpaC), de préférence comprenant une séquence
choisie parmi SEQ ID NO: 85 et les polypeptides comprenant une séquence ayant
au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec celle-ci et
présentant une activité 4-hydroxyphénylacetate 3-nnonooxygénase réductase; ou
une séquence d'acide nucléique hétérologue codant pour une 4-nnéthoxybenzoate
0-dénnéthylase capable de convertir la tyrosine en L-Dopa et également l'acide
p
counnarique en acide caféique, de préférence comprenant une séquence choisie
parmi SEQ ID NOs: 73 et 75 et les polypeptides comprenant une séquence ayant
au
moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec une de ces
séquences
et présentant une activité L-tyrosine hydrolase; ou une séquence d'acide
nucléique
hétérologue codant pour une p-counnarate 3-hydroxylase capable de convertir
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
84
l'acide p-counnarique en acide caféique, de préférence comprenant une séquence
choisie parmi SEQ ID No: 71 et les polypeptides comprenant une séquence ayant
au moins 60, 70, 80, 85, 90 ou 95 % d'identité d'une séquence avec une de ces
séquences et présentant une activité p-counnarate 3 hydroxylase ;
-
facultativement, une séquence d'acide nucléique hétérologue codant pour une
dihydroxyphénylalanine annnnonia-lyase (DAL) capable de produire de l'acide
caféique à partir de la L-DOPA (3,4-dihydroxy-L-phenylalanine).
Dans un autre mode de réalisation particulier, le microorganisme comprend :
- une séquence d'acide nucléique hétérologue codant pour une flavanone 7-0-
bêta-D-
glucosyltransférase (UGT) sélectionnée parmi les enzymes comprenant une
séquence
choisie parmi la SEQ ID NO: 113 et les polypeptides comprenant une séquence
ayant au
moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec cette séquence et
présentant
une activité flavanone 7-0-bêta-D-glucosyltransférase ;
- une séquence d'acide nucléique hétérologue codant pour une 6"-O-
rhannnosyltransferase
(RhaT) comprenant une séquence choisie parmi la SEQ ID NO: 103 et les
polypeptides
comprenant une séquence ayant au moins 60, 70,80, 85, 90 ou 95% d'identité de
séquence
avec cette séquence et présentant une activité 6"-O-rhamnosyltransferase; et
- une séquence d'acide nucléique hétérologue codant pour une UDP-glucose 4,6-
déshydratase/UDP-4-kéto-6-désoxy-D-glucose 3,5-épimerase/UDP-4-kéto-L-rhamnose-
réductase (RHM) comprenant une séquence choisie parmi la SEQ ID NO: 107 et les
polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 %
d'identité de séquence avec cette séquence et présentant une activité UDP-
glucose 4,6-
déshyd ratase/U DP-4-kéto-6-désoxy-D-glucose 3,5-
épinnerase/U DP-4-kéto-L-rhann nase-
réductase ;
- une séquence d'acide nucléique hétérologue codant pour une phénylalanine
annnnonia
lyase (PAL) comprenant une séquence choisie parmi la SEQ ID NO: 65 et les
polypeptides
comprenant une séquence ayant au moins 60, 70,80, 85, 90 ou 95% d'identité de
séquence
avec cette séquence et présentant une activité phénylalanine annnnonia lyase ;
- une séquence d'acide nucléique hétérologue codant pour une cinnannate 4-
hydroxylase
(C4H) comprenant une séquence choisie parmi la SEQ ID NO: 79 et les
polypeptides
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
comprenant une séquence ayant au moins 60, 70,80, 85, 90 ou 95% d'identité de
séquence
avec cette séquence et présentant une activité cinnannate 4-hydroxylase ;
- une séquence d'acide nucléique hétérologue codant pour une tyrosine
annnnonia lyase
(TAL) comprenant une séquence choisie parmi SEQ ID NO: 41 et les polypeptides
5 comprenant une séquence ayant au moins 60, 70,80, 85, 90 ou 95%
d'identité de séquence
avec cette séquence et présentant une activité tyrosine annnnonia lyase ;
- une séquence d'acide nucléique hétérologue codant pour une 4-counnaroyl-
CoA ligase
(4CL) comprenant une séquence sélectionnée parmi la SEQ ID NO: 45 ou 123 et
les
polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 %
10 d'identité de séquence avec l'une de ces séquences et présentant une
activité 4-
counnarate-CoA ligase, de préférence une séquence sélectionnée parmi la SEQ ID
NO: 45 et
les polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou
95 %
d'identité de séquence avec cette séquence et présentant une activité 4-
counnarate-CoA
ligase;
15 - une séquence d'acide nucléique hétérologue codant pour une chalcone
synthase (CHS)
comprenant une séquence choisie parmi SEQ ID NO: 53 et les polypeptides
comprenant
une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence
avec cette
séquence et présentant une activité chalcone synthase;
- une séquence d'acide nucléique hétérologue codant pour une chalcone
isonnérase (CHI)
20 comprenant une séquence choisie parmi la SEQ ID NO: 61 et les
polypeptides comprenant
une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence
avec cette
séquence et présentant une activité chalcone isonnérase ;
- une séquence d'acide nucléique hétérologue codant pour une flavonoïde 3'-
nnonooxygénase (F3'H) comprenant une séquence choisie parmi SEQ ID NO: 7 et
les
25 polypeptides comprenant une séquence a ayant au moins 60, 70, 80, 85, 90
ou 95 %
d'identité de séquence avec la séquence SEQ ID NO: 7 et présentant l'activité
flavonoïde
3'-monooxygénase ;
- une séquence d'acide nucléique hétérologue codant pour une flavone
synthase (FNS)
comprenant une séquence choisie parmi SEQ ID NO: 37 et un polypeptide
comprenant une
30 séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité de
séquence avec cette
séquence et présentant une activité flavone synthase ; et
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
86
- une séquence d'acide nucléique hétérologue codant pour une cytochrome P450
réductase (CPR) comprenant une séquence choisie parmi SEQ ID NO: 25 et les
polypeptides
comprenant une séquence ayant au moins 60, 70,80, 85, 90 ou 95% d'identité de
séquence
avec une de ces séquences et présentant une activité cytochrome P450 réductase
; et
5- une séquence d'acide nucléique hétérologue codant pour une 0-
méthyltransférase (OMT)
capable de méthyler l'ériodictyol et/ou la lutéoline en position 4' et
comprenant une
séquence choisie parmi SEQ ID NOs: 117 et 119 et les polypeptides comprenant
une
séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec
une de ces
séquences et présentant une activité 0-méthyltransférase ,et de préférence une
OMT
comprenant une séquence choisie parmi SEQ ID NO: 117 et les polypeptides
comprenant
une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence
avec cette
séquence et présentant une activité 0-méthyltransférase.
De préférence, le microorganisme comprend en outre une séquence d'acide
nucléique
hétérologue ou endogène codant pour une S-adénosylméthionine synthétase
(SAMT), en
particulier une SAMT comprenant une séquence choisie parmi SEQ ID NO: 81 et un
polypeptide comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 %
d'identité
de séquence avec cette séquence et présentant une activité S-
adénosylméthionine
synthétase.
L'origine des enzymes ou d'un ensemble d'enzymes pourra être choisie de sorte
à ce que
leur origine soit la même ou soit proche. Par exemple, les enzymes ou
l'ensemble
d'enzymes peuvent être issues de bactéries, par exemple de bactéries du même
genre ou
de la même espèce. Dans un autre exemple, les enzymes ou l'ensemble d'enzymes
peuvent
être issues de plantes, par exemple de plantes du même genre ou de la même
espèce. En
effet, ces origines communes permettent que les enzymes fonctionnent entre
elles de
manière optimale.
Dans un mode de réalisation, les microorganismes comprennent une voie
métabolique de
biosynthèse de la tyrosine. Notamment, les microorganismes peuvent avoir été
modifiées
pour avoir une production augmentée de tyrosine par rapport à une souche
sauvage.
Notamment, les microorganismes peuvent avoir été modifiées pour que le flux de
carbone
soit redirigé vers la biosynthèse de tyrosine. En outre, les microorganismes
peuvent avoir
été modifiées pour réduire ou supprimer les rétro-inhibitions de la
biosynthèse de tyrosine.
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
87
Dans un autre mode de réalisation, les microorganismes comprennent une voie
métabolique de biosynthèse de la phénylalanine. Notamment, les microorganismes
peuvent avoir été modifiées pour avoir une production augmentée de
phénylalanine par
rapport à une souche sauvage. Notamment, les microorganismes peuvent avoir été
modifiées pour que le flux de carbone soit redirigé vers la biosynthèse de
phénylalanine.
En outre, les microorganismes peuvent avoir été modifiées pour réduire ou
supprimer les
rétro-inhibitions de la biosynthèse de phénylalanine.
Dans un autre mode de réalisation, les microorganismes comprennent une voie
métabolique de biosynthèse de la phénylalanine et de la tyrosine. Notamment,
les
microorganismes peuvent avoir été modifiées pour avoir une production
augmentée de
phénylalanine et de tyrosine par rapport à une souche sauvage. Notamment, les
microorganismes peuvent avoir été modifiées pour que le flux de carbone soit
redirigé vers
la biosynthèse de phénylalanine et de tyrosine. En outre, les microorganismes
peuvent
avoir été modifiées pour réduire ou supprimer les rétro-inhibitions de la
biosynthèse de
phénylalanine et de tyrosine.
Acide nucléique recombinant et Cassette d'expression
Chaque séquence nucléique codant pour une enzyme telle que décrite
précédemment est
comprise dans une cassette d'expression. De préférence, les séquences
nucléiques
codantes ont été optimisées pour l'expression dans le microorganisme hôte. La
séquence
nucléique codante est liée de manière opérationnelle aux éléments nécessaires
à
l'expression du gène, notamment à la transcription et traduction. Ces éléments
sont choisis
de sorte à être fonctionnels dans le microorganisme recombinant hôte. Ces
éléments
peuvent comporter par exemple des promoteurs de transcription, des activateurs
de
transcription, des séquences terminatrices, des codons d'initiation et de
terminaison. Les
méthodes pour sélectionner ces éléments en fonction de la cellule hôte dans
laquelle
l'expression est souhaitée, sont bien connues de l'homme du métier.
De préférence, le promoteur est un promoteur fort. Le promoteur peut
éventuellement
être inductible.
Par exemple, si le microorganisme est procaryote, le promoteur peut être
sélectionné
parmi les promoteurs suivants : Lad, LacZ, pLacT, ptac, pARA, pBAD, les
promoteurs d'ARN
polymérase de bactériophage T3 or T7, le promoteur de la polyhèdrine, le
promoteur PR
ou PL du phage lambda. Dans un mode de réalisation particulier, le promoteur
est pLac. Si
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
88
le microorganisme est eucaryote et en particulier une levure, le promoteur
peut être
sélectionné parmi les promoteurs suivants : le promoteur pTDH3, le promoteur
pTEF1, le
promoteur pTEF2, le promoteur pCCW12, le promoteur pH HF2, le promoteur pHTB2
et le
promoteur pRPL18B. Des exemples de promoteurs inductibles utilisables chez la
levure
sont les promoteurs tet0-2, GAL10, GAL10-CYC1, PH05.
Toutes ou une partie des cassettes d'expression comprenant les séquences
nucléiques
codant pour les enzymes telles que décrites ou une combinaison de certaines de
celles-ci
peuvent être comprises dans un vecteur d'expression commun ou dans différents
vecteurs
d'expression.
la présente invention est donc relative à un vecteur comprenant deux séquences
d'acide
nucléique choisies parmi une séquence d'acide nucléique hétérologue codant
pour une
flavanone 7-0-bêta-D-glucosyltransférase (UGT), une séquence d'acide nucléique
hétérologue codant pour une 6"-O-rhannnosyltransferase (RhaT) et une séquence
d'acide
nucléique hétérologue codant pour une UDP-glucose 4,6-déshydratase/UDP-4-kéto-
6-
désoxy-D-glucose 3,5-épinnerase/UDP-4-kéto-L-rhannnose-réductase (RHM), de
préférence
un vecteur comprend ces trois séquences.
En particulier, la présente invention est donc relative à un vecteur
comprenant deux
séquences d'acide nucléique choisies parmi
(i) une
séquence d'acide nucléique hétérologue codant pour une flavanone 74)-
bêta-D-glucosyltransférase (UGT) d'Arabidopsis thaliana, de Scutellaria
baicalensis ou d'Homo sapiens, de préférence d'Arabidopsis thaliana ou de
Scutellaria baicalensis, de préférence une flavanone 7-0-bêta-D-
glucosyltransférase (UGT) sélectionnée parmi les enzymes comprenant une
séquence choisie parmi SEQ ID NOs: 113, 115, 91, 93, 95, 97, 99 et 101 et les
polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95
% d'identité de séquence avec une de ces séquences et présentant une activité
flavanone 7-0-bêta-D-glucosyltransférase ; de manière particulière une
flavanone 7-0-bêta-D-glucosyltransférase (UGT) comprenant une séquence
choisie parmi SEQ ID NOs: 113, 115, 91, 93, 95 et 97 ou parmi SEQ ID Nos : 113
et 95 et les polypeptides comprenant une séquence ayant au moins 60, 70, 80,
85, 90 ou 95 % d'identité de séquence avec une de ces séquences et présentant
une activité flavanone 7-0-bêta-D-glucosyltransférase, de préférence
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
89
sélectionnée parmi les enzymes comprenant une séquence choisie parmi la SEQ
ID NO: 113 et les polypeptides comprenant une séquence ayant au moins 60,
70, 80, 85, 90 ou 95 % d'identité de séquence avec cette séquence et
présentant
une activité flava none 7-0-bêta-D-glucosyltransférase;
(ii) une séquence d'acide nucléique hétérologue codant pour une 6"-O-
rhannnosyltransferase (RhaT) du genre Citrus ou de Petunia hybrida, de
préférence Citrus sinensis, Citrus maxima, ou Citrus clementina, de manière
encore plus préférée Citrus sinensis ou Citrus clementina, de préférence une
6"-
0-rhannnosyltransferase (RhaT) comprenant une séquence choisie parmi SEQ ID
NOs: 103, 105 et les polypeptides comprenant une séquence ayant au moins 60,
70, 80, 85, 90 ou 95 % d'identité de séquence avec une de ces séquences et
présentant une activité 6"-O-rhannnosyltransferase, et de préférence une RhaT
comprenant une séquence choisie parmi la SEQ ID NO: 103 et les polypeptides
comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité
de séquence avec cette séquence et présentant une activité 6"-O-
rhannnosyltransferase; et
(iii) une séquence d'acide nucléique hétérologue codant pour une UDP-
glucose 4,6-
dés hyd ratase/U DP-4-kéto-6-désoxy-D-gl ucose 3,5-
épi nnerase/U DP-4-kéto-L-
rha nnnose-réductase (RHM) de Citrus sinensis ou d'Arabidopsis thaliana, de
préférence une RHM comprenant une séquence choisie parmi SEQ ID NOs: 107,
109 et 111 et les polypeptides comprenant une séquence ayant au moins 60,
70, 80, 85, 90 ou 95 % d'identité de séquence avec une de ces séquences et
présentant une activité UDP-glucose 4,6-déshyd ratase/UDP-4-kéto-6-désoxy-D-
glucose 3,5-épinnerase/UDP-4-kéto-L-rhannnose-réductase, en particulier une
RHM comprenant une séquence sélectionnée parmi SEQ ID NOs: 107, 109 et les
polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95
% d'identité de séquence avec cette séquence et présentant une activité UDP-
glucose 4,6-déshydratase/UDP-4-kéto-6-désoxy-D-glucose 3,5-épinnerase/UDP-
4-kéto-L-rhannnose-réductase, et de préférence un RHM comprenant une
séquence choisie parmi la SEQ ID NO: 107 et les polypeptides comprenant une
séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
cette séquence et présentant une activité UDP-glucose 4,6-déshydratase/UDP-
4-kéto-6-désoxy-D-glucose 3,5-épinnerase/UDP-4-kéto-L-rhannnose-réductase.
En particulier, le vecteur peut comprendre deux séquences d'acide nucléique
choisies
parmi
5 (i) une séquence d'acide nucléique hétérologue codant pour une
flavanone 7-0-
bêta-D-glucosyltra nsférase (UGT) d'Arabidopsis thaliana, de Scutellaria
baicalensis ou d'Homo sapiens, de préférence d'Arabidopsis thaliana ou de
Scutellaria baicalensis, de préférence une flavanone 7-0-bêta-D-
glucosyltransférase (UGT) sélectionnée parmi les enzymes comprenant une
10 séquence choisie parmi SEQ ID NOs: 91, 93, 95, 97, 99 et 101 et
les polypeptides
comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité
de séquence avec une de ces séquences et présentant une activité flavanone 7-
0-bêta-D-glucosyltransférase ; de manière particulière une flavanone 7-0-bêta-
D-glucosyltransférase (UGT) comprenant une séquence choisie parmi SEQ ID
15 NOs: 91, 93, 95 et 97 et les polypeptides comprenant une séquence
ayant au
moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec une de ces
séquences et présentant une activité flavanone 7-0-bêta-D-glucosyltransférase;
(ii) une séquence d'acide nucléique hétérologue codant pour une 6"-O-
rhannnosyltransferase (RhaT) du genre Citrus ou de Petunia hybrida, de
20 préférence Citrus sinensis, Citrus maxima, ou Citrus clementina,
de manière
encore plus préférée Citrus sinensis ou Citrus clementina, de préférence une
6"-
0-rhannnosyltransferase (RhaT) comprenant une séquence choisie parmi SEQ ID
NOs: 103, 105 et les polypeptides comprenant une séquence ayant au moins 60,
70, 80, 85, 90 ou 95 % d'identité de séquence avec une de ces séquences et
25 présentant une activité 6"-O-rhannnosyltransferase; et
(iii) une séquence d'acide nucléique hétérologue codant pour une UDP-
glucose 4,6-
déshyd ratase/U DP-4-kéto-6-désoxy-D-glucose 3,5-
épinnerase/UDP-4-kéto-L-
rhannnose-réductase (RHM) de Citrus sinensis ou d'Arabidopsis thaliana, de
préférence une RHM comprenant une séquence choisie parmi SEQ ID NOs: 107,
30 109 et 111 et les polypeptides comprenant une séquence ayant au
moins 60,
70, 80, 85, 90 ou 95 % d'identité de séquence avec une de ces séquences et
présentant une activité UDP-glucose 4,6-déshydratase/UDP-4-kéto-6-désoxy-D-
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
91
glucose 3,5-épirnerase/UDP-4-kéto-L-rharnnose-réductase, en particulier une
RHM comprenant une séquence sélectionnée parmi SEQ ID NOs: 107, 109 et les
polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95
% d'identité de séquence avec cette séquence et présentant une activité UDP-
glucose 4,6-déshydratase/UDP-4-kéto-6-désoxy-D-glucose 3,5-épimerase/UDP-
4-kéto-L-rhamnose-réductase.
De préférence, le vecteur comprend deux séquences d'acide nucléique choisies
parmi
(i) une séquence d'acide nucléique hétérologue codant pour une flavanone 7-
0-
bêta-D-glucosyltransférase (UGT) sélectionnée parmi les enzymes comprenant
une séquence choisie parmi SEQ ID NOs: 113 et 95 et les polypeptides
comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité
de séquence avec une de ces séquences et présentant une activité flavanone 7-
0-bêta-D-glucosyltransférase ; de préférence une flavanone 7-0-bêta-D-
glucosyltransférase (UGT) comprenant une séquence choisie parmi SEQ ID NO:
113 et les polypeptides comprenant une séquence ayant au moins 60, 70, 80,
85, 90 ou 95 % d'identité de séquence avec cette séquence et présentant une
activité flavanone 7-0-bêta-D-glucosyltransférase;
(ii) une séquence d'acide nucléique hétérologue codant pour une 6"-O-
rhamnosyltransferase (RhaT) comprenant une séquence choisie parmi SEQ ID
NO: 103 et les polypeptides comprenant une séquence ayant au moins 60, 70,
80, 85, 90 ou 95 % d'identité de séquence avec cette séquence et présentant
une activité 6"-O-rhamnosyltransferase; et
(iii) une séquence d'acide nucléique hétérologue codant pour une UDP-
glucose 4,6-
déshydratase/UDP-4-kéto-6-désoxy-D-glucose 3,5-
épimerase/UDP-4-kéto-L-
rhamnose-réductase (RHM) comprenant une séquence choisie parmi SEQ ID
NO: 107 et les polypeptides comprenant une séquence ayant au moins 60, 70,
80, 85, 90 ou 95 % d'identité de séquence avec cette séquence et présentant
une activité UDP-glucose 4,6-déshydratase/UDP-4-kéto-6-désoxy-D-glucose
3,5-épimerase/UDP-4-kéto-L-rhamnose-réductase.
Dans un mode de réalisation préféré, le microorganisme comprend les trois.
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
92
Les séquences nucléiques codantes et les séquences des enzymes sont telles que
décrites
ci-dessus. Par comprendre une séquence d'acide nucléique est également entendu
comprendre une cassette d'expression comprenant la séquence d'acide nucléique.
Facultativement, le vecteur peut comprendre en outre une ou plusieurs
séquences d'acide
nucléique choisies parmi : une séquence d'acide nucléique codant pour une 0-
méthyltransférase (OMT), une séquence d'acide nucléique codant pour une F3'H,
une
séquence d'acide nucléique codant pour une CPR, une séquence d'acide nucléique
codant
pour une FNS, une séquence d'acide nucléique codant pour une SAMT, une
séquence
d'acide nucléique codant pour une TAL, une séquence d'acide nucléique codant
pour une
4CL, une séquence d'acide nucléique codant pour une CHS, une séquence d'acide
nucléique
codant pour une CHI, une séquence d'acide nucléique codant pour une PAL, une
séquence
d'acide nucléique codant pour une C4H, une séquence d'acide nucléique codant
pour une
HpaB, et une séquence d'acide nucléique codant pour une DAL, chacune de ces
enzymes
étant telles que définies ci-dessus, ainsi que leurs combinaisons.
De préférence, le vecteur comprend en outre une ou plusieurs séquences
choisies parmi
une séquence d'acide nucléique codant pour une OMT, une séquence d'acide
nucléique
codant pour une F3'H, une séquence d'acide nucléique codant pour une CPR, une
séquence
d'acide nucléique codant pour une FNS, une séquence d'acide nucléique codant
pour une
TAL, une séquence d'acide nucléique codant pour une 4CL, une séquence d'acide
nucléique
codant pour une CHS, une séquence d'acide nucléique codant pour une CHI, une
séquence
d'acide nucléique codant pour une PAL et une séquence d'acide nucléique codant
pour une
C4 H.
En particulier, le vecteur peut comprendre en outre une ou plusieurs séquences
d'acide
nucléique choisies parmi :
- une séquence d'acide nucléique hétérologue codant pour une flavonoïde 3'-
monooxygénase (F3'H) capable d'hydroxyler en position 3' la naringénine et/ou
l'apigénine ; de préférence de Perilla frutescens var. crispa, Petunia x
hybrida,
Callistephus chinensis, Gerbera hybrida, Citrus sinensis, Arabidopsis
thaliana, Citrus
clementina, Osteospermum hybrid cultivar, Phanerochaete chrysosporium,
Streptomyces avermitilis ou Pilosella officinarum, en particulier Perilla
frutescens
var. crispa, Petunia x hybrida, Callistephus chinensis, Gerbera hybrida,
Citrus
sinensis ou Pilosella officinarum de préférence une F3'H comprenant une
séquence
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
93
choisie parmi SEQ ID NOs: 1, 3, 5, 7, 9, 11, 13, 15, 17, 19, 21 et 121 et les
polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 %
d'identité de séquence avec une de ces séquences et présentant une activité
flavonoïde 3'-nnonooxygénase, de préférence sélectionnée parmi les enzymes
comprenant une séquence choisie parmi SEQ ID NOs: 1, 5, 7, 11, 17, 19 et 121
et les
polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 %
d'identité de séquence avec une de ces séquences et présentant une activité
flavonoïde 3'-monooxygénase, en particulier une F3'H comprenant une séquence
sélectionnée parmi les SEQ ID NOs: 5, 7, 17 et 121 et les polypeptides
comprenant
une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence
avec cette séquence et présentant une activité flavonoïde 3'-monooxygénase, et
de
préférence une enzyme comprenant une séquence sélectionnée parmi la SEQ ID
NO: 7, et les polypeptides comprenant une séquence ayant au moins 75%, au
moins
80%, au moins 85%, au moins 90% ou au moins 95% d'identité avec cette séquence
et présentant l'activité flavonoïde 3'-monooxygénase ;
- une séquence d'acide nucléique hétérologue codant pour une cytochrome P450
réductase (CPR) ; de préférence une CPR de Saccharomyces cerevisiae, ou de
plante,
par exemple de Catharanthus roseus ou d'Arabidopsis thaliana ; de préférence
une
CPR comprenant une séquence choisie parmi SEQ ID NOs: 23, 25, 27, 29 et 31 et
les
polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 %
d'identité de séquence avec une de ces séquences et présentant une activité
cytochrome P450 réductase, de préférence parmi les enzymes comprenant une
séquence choisie parmi SEQ ID NOs: 23, 25 et 29 et les polypeptides comprenant
une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence
avec une de ces séquences et présentant une activité cytochrome P450
réductase,
et de préférence une CPR comprenant une séquence choisie parmi SEQ ID NO: 25
et les polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90
ou
95 % d'identité de séquence avec cette séquence et présentant une activité
cytochrome P450 réductase;
- une séquence d'acide nucléique hétérologue codant pour une 0-
méthyltransférase
(OMT) capable de méthyler l'ériodictyol et/ou la lutéoline en position 4'; de
préférence une OMT d'Arabidopsis thaliana ou d'Homo sapiens, de préférence une
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
94
OMT comprenant une séquence choisie parmi SEQ ID NOs: 117, 119, 87 et 89 et
les
polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 %
d'identité de séquence avec une de ces séquences et présentant une activité 0-
nnéthyltransférase, notamment avec l'ériodictyol et/ou la lutéoline comme
substrat
et une méthylation en position 4', de préférence sélectionnée parmi l'enzyme
comprenant une séquence choisie parmi SEQ ID NO : 117, 119 et 89 et les
polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 %
d'identité de séquence avec cette séquence et présentant une activité 0-
nnéthyltransférase ;
- une séquence d'acide nucléique hétérologue codant pour une flavone synthase
(FNS) capable de transformer la naringénine en apigénine, l'ériodictyol en
lutéoline
et/ou l'hespérétine en diosnnétine, de préférence de transformer l'ériodictyol
en
lutéoline; de préférence une FNS d'Arabidopsis thaliana, Lonicera japonica,
Lonicera macranthoides, Medicago truncatula, Oryza sativa, Petroselinum
crispum,
Populus deltoides, Zea mays, Callistephus chinensis, Apium graveolens,
Medicago
truncatula, Cuminum cyminum, Aethusa cynapium, Angelica archangelica, Conium
maculatum, Camellia sinensis, Cynara cardunculus var scolymus, Saussurea
medusa, Plectranthus barbatus, Scutellaria baicalensis, Dorcoceras
hygrometricum,
Antirrhinum malus, Perilla frutescens var crispa, Dahlia pinnata ou
Erythranthe
lewisii, en particulier d'Arabidopsis thaliana, Lonicera japonica, Lonicera
macranthoides, Medicago truncatula, Oryza sativa, Petroselinum crispum,
Populus
deltoides ou Zea mays, de préférence de Lonicera japonica, Lonicera
macranthoides, et Petroselinum crispum; de préférence une FNS comprenant une
séquence choisie parmi SEQ ID NOs: 33, 35, 37, 127, 129, 131, 133, 135, 137,
139,
141, 143, 145, 147, 149, 151, 153, 155, 157 et 159 et les polypeptides
comprenant
une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence
avec une de ces séquences et présentant une activité flavone synthase, de
préférence parmi les enzymes comprenant une séquence choisie parmi SEQ ID NOs:
33, 35 et 37 et les polypeptides comprenant une séquence ayant au moins 60,
70,
80, 85, 90 ou 95 % d'identité de séquence avec une de ces séquences et
présentant
une activité flavone synthase, de préférence une flavone synthase (FNS)
comprenant une séquence choisie parmi SEQ ID NO: 37 et un polypeptide
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité
de
séquence avec cette séquence et présentant une activité flavone synthase ;
- une séquence d'acide nucléique hétérologue ou endogène codant pour une S-
adénosylnnéthionine synthétase (SAMT) ; en particulier de Saccharomyces
5 cerevisiae, par exemple une SAMT comprenant une séquence choisie
parmi SEQ ID
NO: 81 et un polypeptide comprenant une séquence ayant au moins 60, 70, 80,
85,
90 ou 95 % d'identité de séquence avec cette séquence et présentant une
activité
S-adénosylnnéthionine synthétase ;
- une séquence d'acide nucléique hétérologue codant pour une tyrosine
annnnonia
10 lyase (TAL) capable de produire de l'acide p-counnarique à partir de
la tyrosine; de
préférence de Rhodotorula glutinis ou Flavobacterium johnsoniae ; en
particulier
une TAL comprenant une séquence choisie parmi SEQ ID NOs: 39 et 41 et les
polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 %
d'identité de séquence avec une de ces séquences et présentant une activité
15 tyrosine annmonia lyase ; de préférence une TAL comprenant une
séquence choisie
parmi SEQ ID NO: 41 et les polypeptides comprenant une séquence ayant au moins
60, 70, 80, 85, 90 ou 95% d'identité de séquence avec cette séquence et
présentant
une activité tyrosine annnnonia lyase ;
- une séquence d'acide nucléique hétérologue codant pour une 4-counnarate-
CoA
20 ligase (4CL) capable de produire de le counnaryl-CoA à partir de
l'acide p-
counnarique et de Coenzyme A ; de préférence d'Arabidopsis thaliana, Citrus
clementina, Petroselinum crispum ou Streptomyces clavuligerus; une 4CL
comprenant une séquence choisie parmi SEQ ID NOs: 43, 45, 47, 49, 123 et 125
et
les polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou
95
25 % d'identité de séquence avec une de ces séquences et présentant une
activité 4-
counnarate-CoA ligase, de préférence, une 4CL comprenant une séquence
sélectionnée parmi les SEQ ID NOs: 123, 125 et 45 et les polypeptides
comprenant
une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence
avec une de ces séquences et présentant une activité 4-counnarate-CoA ligase,
et
30 en particulier une 4CL comprenant une séquence sélectionnée parmi la
SEQ ID NO:
45 et les polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85,
90
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
96
ou 95 % d'identité de séquence avec cette séquence et présentant une activité
4-
counnarate-CoA ligase ;
- une séquence d'acide nucléique hétérologue codant pour une chalcone
synthase
(CHS) capable de produire de la naringénine-chalcone à partir du 4-counnaroyl-
CoA
et de malonyl-CoA ; de préférence de Citrus sinensis, de Hordeum vulgare ou de
Streptomyces clavuligerus, en particulier une CHS comprenant une séquence
choisie parmi SEQ ID NOs: 51, 53, 55 et 57 et les polypeptides comprenant une
séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec
une de ces séquences et présentant une activité chalcone synthase, de
préférence
parmi les enzymes comprenant une séquence choisie parmi SEQ ID NOs: 53 et 55
et les polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90
ou
95 % d'identité de séquence avec une de ces séquences et présentant une
activité
chalcone synthase, et de préférence une CHS comprenant une séquence choisie
parmi SEQ ID NO: 53 et les polypeptides comprenant une séquence ayant au moins
60, 70, 80, 85, 90 ou 95% d'identité de séquence avec cette séquence et
présentant
une activité chalcone synthase ;
- une séquence d'acide nucléique hétérologue codant pour une chalcone
isonnérase
(CHI) capable de produire la naringénine à partir de la naringénine chalcone;
de
préférence d'Arabidopsis thaliana ou Streptomyces clavuligerus, en particulier
une
CHI comprenant une séquence choisie parmi SEQ ID NOs: 59 et 61 et les
polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 %
d'identité de séquence avec une de ces séquences et présentant une activité
chalcone isomérase, et de préférence une CHI comprenant une séquence choisie
parmi la SEQ ID NO: 61 et les polypeptides comprenant une séquence ayant au
moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec cette séquence et
présentant une activité chalcone isonnérase ;
- une séquence d'acide nucléique hétérologue codant pour une phénylalanine
amnnonia lyase (PAL), en particulier une PAL comprenant une séquence choisie
parmi SEQ ID NOs: 63, 65, 77 et les polypeptides comprenant une séquence ayant
au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec une de ces
séquences et présentant une activité phénylalanine annmonia lyase, de
préférence
une PAL comprenant une séquence choisie parmi la SEQ ID NO: 65 et les
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
97
polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 %
d'identité de séquence avec cette séquence et présentant une activité
phénylalanine annnnonia lyase ; et
- une séquence d'acide nucléique hétérologue codant pour une cinnannate 4-
hydroxylase (C4H), en particulier une C4H comprenant une séquence choisie
parmi
SEQ ID NOs: 67, 69, 79 et les polypeptides comprenant une séquence ayant au
moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec une de ces
séquences
et présentant une activité cinnannate 4-hydroxylase, capables de produire de
l'acide
p-counnarique à partir de la phénylalanine, et de préférence une C4H
comprenant
une séquence choisie parmi la SEQ ID NO: 79 et les polypeptides comprenant une
séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec
cette séquence et présentant une activité cinnannate 4-hydroxylase ;
- une séquence d'acide nucléique codant pour une sous-unité 4-
hydroxyphénylacetate 3-nnonooxygénase oxygénase (H paB), de préférence
comprenant une séquence choisie parmi SEQ ID NO: 83 et les polypeptides
comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité
de
séquence avec celle-ci et présentant une activité 4-hydroxyphénylacetate 3-
nnonooxygénase oxygénase, et une séquence d'acide nucléique codant pour une
sous-unité 4-hydroxyphénylacetate 3-nnonooxygénase réductase (HpaC), de
préférence comprenant une séquence choisie parmi SEQ ID NO: 85 et les
polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 %
d'identité de séquence avec celle-ci et présentant une activité 4-
hydroxyphénylacetate 3-nnonooxygénase réductase; ou une séquence d'acide
nucléique codant pour une 4-nnéthoxybenzoate 0-dénnéthylase capable de
convertir la tyrosine en L-Dopa et également l'acide p counnarique en acide
caféique, de préférence comprenant une séquence choisie parmi SEQ ID NOs: 73
et
75 et les polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85,
90
ou 95 % d'identité de séquence avec une de ces séquences et présentant une
activité L-tyrosine hydrolase; ou une séquence d'acide nucléique hétérologue
codant pour une p-counnarate 3-hydroxylase capable de convertir l'acide p-
counnarique en acide caféique, de préférence comprenant une séquence SEQ ID
Nos : 71 et les polypeptides comprenant une séquence ayant au moins 60, 70,
80,
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
98
85, 90 ou 95 % d'identité d'une séquence avec une de ces séquences et
présentant
une activité p-counnarate 3 hydroxylase, et
- une séquence d'acide nucléique codant pour une dihydroxyphénylalanine
annnnonia-lyase (DAL).
En particulier, le vecteur peut comprendre en outre une ou plusieurs séquences
d'acide
nucléique choisies parmi :
- une séquence d'acide nucléique hétérologue codant pour une flavonoïde 3'-
monooxygénase (F3'H) capable d'hydroxyler en position 3 la naringénine et/ou
l'apigénine ; de préférence de Perilla frutescens var. crispa, Petunia x
hybrida,
Callistephus chinensis, Gerbera hybrida, Citrus sinensis, et Pilosella
officinarum, de
préférence une F3'H comprenant une séquence choisie parmi SEQ ID NOs: 1, 3, 5,
7, 9, 11, 13, 15, 17, 19 et 21 et les polypeptides comprenant une séquence
ayant au
moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec une de ces
séquences
et présentant une activité flavonoïde 3'-monooxygénase, de préférence
sélectionnée parmi les enzymes comprenant une séquence choisie parmi SEQ ID
NOs: 1, 5, 7, 11, 17 et 19 et les polypeptides comprenant une séquence ayant
au
moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec une de ces
séquences
et présentant une activité flavonoïde 3'-monooxygénase, en particulier une
F3'H
comprenant une séquence sélectionnée parmi les SEQ ID NOs: 5, 7, 17 et les
polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 %
d'identité de séquence avec cette séquence et présentant une activité
flavonoïde
3'-monooxygénase ;
- une séquence d'acide nucléique hétérologue codant pour une cytochrome
P450
réductase (CPR) ; de préférence une CPR de Saccharomyces cerevisiae, ou de
plante,
par exemple de Catharanthus roseus ou d'Arabidopsis thaliana ; de préférence
une
CPR comprenant une séquence choisie parmi SEQ ID NOs: 23, 25, 27, 29 et 31 et
les
polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 %
d'identité de séquence avec une de ces séquences et présentant une activité
cytochrome P450 réductase, de préférence parmi les enzymes comprenant une
séquence choisie parmi SEQ ID NOs: 23, 25 et 29 et les polypeptides comprenant
une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence
avec une de ces séquences et présentant une activité cytochrome P450
réductase;
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
99
- une séquence d'acide nucléique hétérologue codant pour une 0-
nnéthyltransférase
(OMT) capable de nnéthyler l'ériodictyol et/ou la lutéoline en position 4'; de
préférence une OMT d'Arabidopsis thaliana ou d'Homo sapiens, de préférence une
OMT comprenant une séquence choisie parmi SEQ ID NOs: 87 et 89 et les
polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 %
d'identité de séquence avec une de ces séquences et présentant une activité 0-
nnéthyltransférase, notamment avec l'ériodictyol et/ou la lutéoline comme
substrat
et une nnéthylation en position 4', de préférence sélectionnée parmi l'enzyme
comprenant une séquence SEQ ID NO 89 et les polypeptides comprenant une
séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec
cette séquence et présentant une activité 0-nnéthyltransférase ;
- une séquence d'acide nucléique hétérologue codant pour une flavone
synthase
(FNS) capable de transformer la naringénine en apigénine, l'ériodictyol en
lutéoline
et/ou l'hespérétine en diosnnétine, de préférence de transformer l'ériodictyol
en
lutéoline; de préférence une FNS d'Arabidopsis thaliana, Lonicera japonica,
Lonicera macranthoides, Medicago truncatula, Oryza sativa, Petrosehnum
crispum,
Populus deltoides, ou Zea mays, de préférence de Lonicera japonica, Lonicera
macranthoides, et Petroselinum crispum; de préférence une FNS comprenant une
séquence choisie parmi SEQ ID NOs: 33, 35, 37 et les polypeptides comprenant
une
séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec
une de ces séquences et présentant une activité flavone synthase, de
préférence
parmi les enzymes comprenant une séquence choisie parmi SEQ ID NOs: 33, 35 et
37 et les polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85,
90
ou 95 % d'identité de séquence avec une de ces séquences et présentant une
activité flavone synthase, de préférence une flavone synthase (FNS) comprenant
une séquence choisie parmi SEQ ID NO: 37 et un polypeptide comprenant une
séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec
cette séquence et présentant une activité flavone synthase ;
- une séquence d'acide nucléique hétérologue ou endogène codant pour une S-
adénosylnnéthionine synthétase (SAMT) ; en particulier de Saccharomyces
cerevisiae, par exemple une SAMT comprenant une séquence choisie parmi SEQ ID
NO: 81 et un polypeptide comprenant une séquence ayant au moins 60, 70, 80,
85,
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
100
90 ou 95 % d'identité de séquence avec cette séquence et présentant une
activité
S-adénosylnnéthionine synthétase ;
- une séquence d'acide nucléique hétérologue codant pour une tyrosine
annnnonia
lyase (TAL) capable de produire de l'acide p-counnarique à partir de la
tyrosine; de
préférence de Rhodotorula giutinis ou Flavobacterium johnsoniae ; en
particulier
une TAL comprenant une séquence choisie parmi SEQ ID NOs: 39 et 41 et les
polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 %
d'identité de séquence avec une de ces séquences et présentant une activité
tyrosine annnnonia lyase ; de préférence une TAL comprenant une séquence
choisie
parmi SEQ ID NO: 41 et les polypeptides comprenant une séquence ayant au moins
60, 70, 80, 85, 90 ou 95% d'identité de séquence avec cette séquence et
présentant
une activité tyrosine annnnonia lyase ;
- une séquence d'acide nucléique hétérologue codant pour une 4-counnarate-
CoA
ligase (4CL) capable de produire de le counnaryl-CoA à partir de l'acide p-
counnarique et de Coenzyme A; de préférence d'Arabidopsis thaliana,
Petroselinum
crispum ou Streptomyces clavuligerus ; une 4CL comprenant une séquence choisie
parmi SEQ ID NOs: 43, 45, 47 et 49 et les polypeptides comprenant une séquence
ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec une de
ces
séquences et présentant une activité 4-counnarate-CoA ligase ;
- une séquence d'acide nucléique hétérologue codant pour une chalcone synthase
(CHS) capable de produire de la naringénine-chalcone à partir du 4-counnaroyl-
CoA
et de malonyl-CoA ; de préférence de Citrus sinensis, de Hordeum vulgare ou de
Streptomyces clavuligerus, en particulier une CHS comprenant une séquence
choisie parmi SEQ ID NOs: 51, 53, 55 et 57 et les polypeptides comprenant une
séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec
une de ces séquences et présentant une activité chalcone synthase, de
préférence
parmi les enzymes comprenant une séquence choisie parmi SEQ ID NOs: 53 et 55
et les polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90
ou
95 % d'identité de séquence avec une de ces séquences et présentant une
activité
chalcone synthase;
- une séquence d'acide nucléique hétérologue codant pour une chalcone
isonnérase
(CHI) capable de produire la naringénine à partir de la naringénine chalcone;
de
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
101
préférence d'Arabidopsis thaliana ou Streptomyces clavuligerus, en particulier
une
CHI comprenant une séquence choisie parmi SEQ ID NOs: 59 et 61 et les
polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 %
d'identité de séquence avec une de ces séquences et présentant une activité
chalcone isonnérase ;
- une séquence d'acide nucléique hétérologue codant pour une phénylalanine
amnnonia lyase (PAL), en particulier une PAL comprenant une séquence choisie
parmi SEQ ID NOs: 63, 65, 77 et les polypeptides comprenant une séquence ayant
au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec une de ces
séquences et présentant une activité phénylalanine annmonia lyase ; et
- une séquence d'acide nucléique hétérologue codant pour une cinnannate 4-
hydroxylase (C4H), en particulier une C4H comprenant une séquence choisie
parmi
SEQ ID NOs: 67, 69, 79 et les polypeptides comprenant une séquence ayant au
moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec une de ces
séquences
et présentant une activité cinnannate 4-hydroxylase, capables de produire de
l'acide
p-counnarique à partir de la phénylalanine ;
- une séquence d'acide nucléique codant pour une sous-unité 4-
hydroxyphénylacetate 3-nnonooxygénase oxygénase (HpaB), de préférence
comprenant une séquence choisie parmi SEQ ID NO: 83 et les polypeptides
comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité
de
séquence avec celle-ci et présentant une activité 4-hydroxyphénylacetate 3-
nnonooxygénase oxygénase, et une séquence d'acide nucléique codant pour une
sous-unité 4-hydroxyphénylacetate 3-nnonooxygénase réductase (HpaC), de
préférence comprenant une séquence choisie parmi SEQ ID NO: 85 et les
polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 %
d'identité de séquence avec celle-ci et présentant une activité 4-
hydroxyphénylacetate 3-nnonooxygénase réductase; ou une séquence d'acide
nucléique codant pour une 4-nnéthoxybenzoate 0-dénnéthylase capable de
convertir la tyrosine en L-Dopa et également l'acide p counnarique en acide
caféique, de préférence comprenant une séquence choisie parmi SEQ ID NOs: 73
et
75 et les polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85,
90
ou 95 % d'identité de séquence avec une de ces séquences et présentant une
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
102
activité L-tyrosine hydrolase; ou une séquence d'acide nucléique hétérologue
codant pour une p-counnarate 3-hydroxylase capable de convertir l'acide p-
counnarique en acide caféique, de préférence comprenant une séquence SEQ ID
Nos : 71 et les polypeptides comprenant une séquence ayant au moins 60, 70,
80,
85, 90 ou 95 % d'identité d'une séquence avec une de ces séquences et
présentant
une activité p-coumarate 3 hydroxylase, et
- une séquence d'acide nucléique codant pour une dihydroxyphénylalanine
ammonia-lyase (DAL).
De préférence, le vecteur peut comprendre en outre une ou plusieurs séquences
d'acide
nucléique choisies parmi :
- une séquence d'acide nucléique hétérologue codant pour une flavonoïde 3'-
monooxygénase (F3'H) comprenant une séquence choisie parmi SEQ ID NOs: 7, 11,
17, et 121 et les polypeptides comprenant une séquence ayant au moins 60, 70,
80,
85, 90 ou 95 % d'identité de séquence avec une de ces séquences et présentant
une
activité flavonoïde 3'-monooxygénase, de préférence sélectionnée parmi les
enzymes comprenant une séquence choisie parmi SEQ ID NOs: 7, 17 et 121 et les
polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 %
d'identité de séquence avec une de ces séquences et présentant une activité
flavonoïde 3'-monooxygénase, et de manière tout particulièrement préférée une
F3'H comprenant une séquence sélectionnée parmi SEQ ID NO: 7 et les
polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 %
d'identité de séquence avec cette séquence et présentant une activité
flavonoïde
3'-monooxygénase ;
- une séquence d'acide nucléique hétérologue codant pour une cytochrome
P450
réductase (CPR) comprenant une séquence choisie parmi SEQ ID NOs: 23, 25, 27,
29
et 31 et les polypeptides comprenant une séquence ayant au moins 60, 70, 80,
85,
90 ou 95 % d'identité de séquence avec une de ces séquences et présentant une
activité cytochrome P450 réductase, de préférence parmi les enzymes comprenant
une séquence choisie parmi SEQ ID NOs: 23, 25 et 29 et les polypeptides
comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité
de
séquence avec une de ces séquences et présentant une activité cytochrome P450
réductase, et de manière tout particulièrement préférée, parmi les enzymes
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
103
comprenant une séquence choisie parmi SEQ ID NO: 25 et les polypeptides
comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité
de
séquence avec cette séquence et présentant une activité cytochrome P450
réductase ;
- une séquence
d'acide nucléique hétérologue codant pour une 0-nnéthyltransférase
(OMT) capable de nnéthyler l'ériodictyol et/ou la lutéoline en position 4' et
comprenant une séquence choisie parmi SEQ ID NOs: 117 et 119 et les
polypeptides
comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité
de
séquence avec une de ces séquences et présentant une activité 0-
nnéthyltransférase, notamment avec l'ériodictyol et/ou la lutéoline comme
substrat
et une nnéthylation en position 4', de préférence sélectionnée parmi l'enzyme
comprenant une séquence choisie parmi SEQ ID NO : 117 et les polypeptides
comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité
de
séquence avec cette séquence et présentant une activité 0-nnéthyltransférase ;
- une séquence d'acide nucléique hétérologue codant pour une flavone synthase
(FNS) comprenant une séquence choisie parmi SEQ ID NO: 37 et un polypeptide
comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité
de
séquence avec cette séquence et présentant une activité flavone synthase ;
- une séquence d'acide nucléique hétérologue ou endogène codant pour une S-
adénosylnnéthionine synthétase (SAMT) comprenant une séquence choisie parmi
SEQ ID NO: 81 et un polypeptide ayant au moins 60, 70, 80, 85,90 ou 95%
d'identité
de séquence avec cette séquence et présentant une activité S-
adénosylnnéthionine
synthétase ;
- une séquence d'acide nucléique hétérologue codant pour une tyrosine
annnnonia
lyase (TAL) capable de produire de l'acide p-counnarique à partir de la
tyrosine et
comprenant une séquence choisie parmi SEQ ID NO: 41 et les polypeptides
comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité
de
séquence avec cette séquence et présentant une activité tyrosine annnnonia
lyase ;
- une séquence d'acide nucléique hétérologue codant pour une 4-counnarate-
CoA
ligase (4CL) capable de produire de le counnaryl-CoA à partir de l'acide p-
counnarique et de Coenzyme A et comprenant une séquence choisie parmi SEQ ID
NOs: 123, 125 et 45 et les polypeptides comprenant une séquence ayant au moins
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
104
60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec une de ces séquences et
présentant une activité 4-counnarate-CoA ligase, de préférenceconnprenant une
séquence choisie parmi SEQ ID NO: 45 et les polypeptides comprenant une
séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec
cette séquence et présentant une activité 4-counnarate-CoA ligase ;
- une séquence d'acide nucléique hétérologue codant pour une chalcone
synthase
(CHS) capable de produire de la naringénine-chalcone à partir du 4-counnaroyl-
CoA
et de nnalonyl-CoA et comprenant une séquence choisie parmi SEQ ID NO: 53 et
les
polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 %
d'identité de séquence avec cette séquence et présentant une activité chalcone
synthase ;
- une séquence d'acide nucléique hétérologue codant pour une chalcone
isonnérase
(CHI) capable de produire la naringénine à partir de la naringénine chalcone
et
comprenant une séquence choisie parmi SEQ ID NO: 61 et les polypeptides
comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité
de
séquence avec cette séquence et présentant une activité chalcone isonnérase ;
- une séquence d'acide nucléique hétérologue codant pour une phénylalanine
amnnonia lyase (PAL) et comprenant une séquence choisie parmi SEQ ID NOs: 65
et
77 et les polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85,
90
ou 95 % d'identité de séquence avec une de ces séquences et présentant une
activité phénylalanine annnnonia lyase, de préférence comprenant une séquence
choisie parmi SEQ ID NO: 65 et les polypeptides comprenant une séquence ayant
au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec cette séquence
et
présentant une activité phénylalanine annnnonia lyase ; et
- une séquence d'acide nucléique hétérologue codant pour une cinnannate 4-
hydroxylase (C4H) et comprenant une séquence choisie parmi SEQ ID NO : 79 et
les
polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 %
d'identité de séquence avec cette séquence et présentant une activité
cinnannate
4-hydroxylase, capables de produire de l'acide p-counnarique à partir de la
phénylalanine.
Dans un mode de réalisation particulier, le vecteur comprend
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
105
- une séquence d'acide nucléique codant pour une 0-nnéthyltransférase, en
particulier capable de méthyler l'ériodictyol et/ou la lutéoline en position
4' et une
séquence d'acide nucléique hétérologue codant pour une flavonoïde 3'-
nnonooxygénase (F3'H), en particulier capable d'ajouter un hydroxyle en
position 3'
de la naringénine et/ou de l'apigénine ; ou
- une séquence d'acide nucléique hétérologue codant pour une 0-méthyl-
transférase (OMT), en particulier capable de méthyler l'ériodictyol et/ou la
lutéoline
en position 4'; une séquence d'acide nucléique hétérologue codant pour une
flavonoïde 3'-monooxygénase (F3'H), en particulier capable d'ajouter un
hydroxyle
en position 3' de la naringénine et/ou de l'apigénine ; et une séquence
d'acide
nucléique hétérologue codant pour une cytochrome P450 réductase ; ou
- une séquence d'acide nucléique hétérologue codant pour une 0-méthyl-
transférase (OMT), en particulier capable de méthyler l'ériodictyol et/ou la
lutéoline
en position 4' ; et une séquence d'acide nucléique hétérologue codant pour une
flavone synthase (FNS), capable de transformer la naringénine en apigénine,
l'ériodictyol en lutéoline et/ou l'hespérétine en diosmétine, de préférence de
transformer l'ériodictyol en lutéoline; ou
- une séquence d'acide nucléique hétérologue codant pour une 0-
méthyltransférase
capable de méthyler l'ériodictyol et/ou la lutéoline en position 4' ; une
séquence
d'acide nucléique hétérologue codant pour une flavonoïde 3'-monooxygénase
(F3'H) capable d'hydroxyler en position 3' la naringénine et/ou l'apigénine ;
une
séquence d'acide nucléique hétérologue codant pour une cytochrome P450
réductase ; et une séquence d'acide nucléique hétérologue codant pour flavone
synthase (FNS) capable de transformer la naringénine en apigénine,
l'ériodictyol en
lutéoline et/ou l'hespérétine en diosmétine, de préférence de transformer
l'ériotdictyol en lutéoline.
Le vecteur peut ainsi comprendre plusieurs séquences d'acide nucléique
choisies parmi
celles-ci, notamment 1, 2, 3, 4, 5, 6, 7, 8, 9 ou 10 séquences d'acide
nucléique choisies
parmi celles-ci.
Le vecteur peut notamment comprendre des combinaisons de séquences codantes
particulières telles que décrites ci-dessus.
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
106
Les vecteurs comprennent des séquences codantes hétérologues dans la mesure où
les
séquences codantes peuvent être optimisées pour le microorganisme hôte, être
sous le
contrôle de promoteur hétérologue et/ou peuvent combiner des séquences
codantes qui
ne proviennent pas du même organisme d'origine et/ou qui ne sont pas présentes
dans le
même arrangement.
Le vecteur peut être toute séquence d'ADN dans laquelle il est possible
d'insérer des acides
nucléiques étrangers, les vecteurs permettant d'introduire de l'ADN étranger
dans le
microorganisme hôte. Le vecteur peut être par exemple un plasmide, un
phagemide, un
cosmide, un chromosome artificiel, notamment un YAC, ou un BAC.
Les vecteurs d'expression peuvent comprendre des séquences nucléiques codant
pour des
marqueurs de sélection. Les marqueurs de sélection peuvent être des gènes de
résistance
à un ou plusieurs antibiotiques ou des gènes d'auxotrophie. Le gène
d'auxotrophie peut
par exemple être URA3, LEU2, HIS3 ou TRP1. Le gène de résistance aux
antibiotiques peut
par exemple être de préférence un gène de résistance à l'ampicilline, à la
kanamycine, à
l'hygromycine, à la généticine et/ou la nourséothricine.
L'introduction de vecteurs dans un microorganisme hôte est un procédé
largement connu
de l'homme du métier. Plusieurs méthodes sont notamment décrites dans
Current
Protocols in Molecular Biology , 13.7.1-13.7.10; ou encore dans Ellis T. et
al., Integrative
Biology, 2011, 3(2), 109- 118.
Le microorganisme hôte peut être transformée/transfectée de manière
transitoire ou
stable et l'acide nucléique, la cassette ou le vecteur peut être contenu dans
celui-ci sous
forme d'épisome ou sous forme intégré dans le génome du microorganisme hôte.
Le vecteur d'expression peut également comprendre une ou plusieurs séquences
permettant l'insertion ciblée du vecteur, de la cassette d'expression ou de
l'acide nucléique
dans le génome du microorganisme hôte.
Toutes ou une partie des cassettes d'expression comprenant les séquences
nucléiques
codant pour les enzymes telles que décrites ci-dessus ou une combinaison de
certaines de
celles-ci peuvent être insérées dans le/un chromosome du microorganisme
recombinant.
A contrario, toutes ou une partie des cassettes d'expression comprenant les
séquences
nucléiques codant pour les enzymes telles que décrites ou une combinaison de
certaines
de celles-ci peuvent être conservées sous forme épisomale, notamment sous
forme de
plasmide.
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
107
Éventuellement, le microorganisme peut comprendre plusieurs copies de
séquences
nucléiques codant pour une enzyme telle que décrite précédemment. Notamment,
il peut
comprendre 2 à 10 copies, par exemple 2, 3, 4, 5, 6, 7, 8, 9 ou 10 copies
d'une séquence
nucléique codant pour une enzyme telle que décrite précédemment.
La présente invention est relative à une méthode de préparation d'un
microorganisme
selon la présente invention comprenant l'introduction de séquences d'acide
nucléique
codant pour une flavanone 7-0-bêta-D-glucosyltransférase (UGT), en particulier
capable
d'ajouter un glucose en position 7 de l'hespérétine et la diosnnétine ; pour
une 6"-O-
rhannnosyltransferase (RhaT), en particulier capable de transférer un
rhannnose en position
6 du glucose de l'hespérétine-7-0-glucoside et/ou la diosnnétine-7-0-
glucoside; et pour
une UDP-glucose 4,6-déshydratase/UDP-4-kéto-6-désoxy-D-glucose 3,5-
épinnerase/UDP-
4-kéto-L-rhannnose-réductase (RHM), en particulier capable de produire de
l'UDP-
rhamnose dans le microorganisme et la sélection des microorganismes comprenant
lesdites séquences nucléiques.
La méthode peut comprendre en outre l'introduction une ou plusieurs séquences
d'acide
nucléique choisies parmi :
- une séquence d'acide nucléique hétérologue codant pour une flavonoïde 3'-
nnonooxygénase (F3'H) capable d'hydroxyler en position 3' la naringénine et/ou
l'apigénine ; de préférence de Perilla frutescens var. crispa, Petunia x
hybrida,
Callistephus chinensis, Gerbera hybrida, Citrus sinensis, Citrus clementina,
Osteospermum hybrid cultivar, Phanerochaete chrysosporium, Streptomyces
avermitilis ou Pilosella officinarum, en particulier Perilla frutescens var.
crispa,
Petunia x hybrida, Callistephus chinensis, Gerbera hybrida, Citrus sinensis ou
Pilosella officinarum, de préférence une F3'H comprenant une séquence choisie
parmi SEQ ID NOs: 1, 3, 5, 7, 9, 11, 13, 15, 17, 19 et 21 et les polypeptides
comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité
de
séquence avec une de ces séquences et présentant une activité flavonoïde 3'-
nnonooxygénase, de préférence sélectionnée parmi les enzymes comprenant une
séquence choisie parmi SEQ ID NOs: 1, 5, 7, 11, 17 et 19 et les polypeptides
comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité
de
séquence avec une de ces séquences et présentant une activité flavonoïde 3'-
nnonooxygénase, en particulier une F3'H comprenant une séquence sélectionnée
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
108
parmi les SEQ ID NOs: 5, 7, 17 et les polypeptides comprenant une séquence
ayant
au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec cette séquence
et
présentant une activité flavonoïde 3'-nnonooxygénase, de préférence une F3'H
comprenant une séquence choisie parmi SEQ ID NO: 7 et les polypeptides
comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité
de
séquence avec la séquence SEQ ID NO: 7 et présentant l'activité flavonoïde 3'-
nnonooxygénase ;
- une séquence d'acide nucléique hétérologue codant pour une cytochrome P450
réductase (CPR) ; de préférence une CPR de Saccharomyces cerevisiae, ou de
plante,
par exemple de Catharanthus roseus ou d'Arabidopsis thaliana ; de préférence
une
CPR comprenant une séquence choisie parmi SEQ ID NOs: 23, 25, 27, 29 et 31 et
les
polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 %
d'identité de séquence avec une de ces séquences et présentant une activité
cytochrome P450 réductase, de préférence parmi les enzymes comprenant une
séquence choisie parmi SEQ ID NOs: 23, 25, 29 et 31 et les polypeptides
comprenant
une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence
avec une de ces séquences et présentant une activité cytochrome P450
réductase,
en particulier une CPR comprenant une séquence choisie parmi SEQ ID NO: 25 et
les polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou
95
% d'identité de séquence avec cette séquences et présentant une activité
cytochrome P450 réductase;
- une séquence d'acide nucléique hétérologue codant pour une 0-
nnéthyltransférase
(OMT) capable de nnéthyler l'ériodictyol et/ou la lutéoline en position 4'; de
préférence une OMT d'Arabidopsis thaliana ou d'Homo sapiens, de préférence une
OMT comprenant une séquence choisie parmi SEQ ID NOs: 87 et 89 et les
polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 %
d'identité de séquence avec une de ces séquences et présentant une activité 0-
nnéthyltransférase, notamment avec l'ériodictyol et/ou la lutéoline comme
substrat
et une nnéthylation en position 4', de préférence sélectionnée parmi l'enzyme
comprenant une séquence choisie parmi SEQ ID NO 89 et les polypeptides
comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité
de
séquence avec cette séquence et présentant une activité 0-nnéthyltransférase ;
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
109
- une séquence d'acide nucléique hétérologue codant pour une flavone synthase
(FNS) capable de transformer la naringénine en apigénine, l'ériodictyol en
lutéoline
et/ou l'hespérétine en diosnnétine, de préférence de transformer l'ériodictyol
en
lutéoline; de préférence une FNS d'Arabidopsis thaliana, Lonicera japonica,
Lonicera macranthoides, Medicago truncatula, Oryza sativa, Petroselinum
crispum,
Populus deltoides, Zea mays, Callistephus chinensis, Apium graveolens,
Medicago
truncatula, Cuminum cyminum, Aethusa cynapium, Angelico archangelica, Conium
maculatum, Camellia sinensis, Cynara cardunculus var scolymus, Saussurea
medusa, Plectranthus barbatus, Scutellaria baicalensis, Dorcoceras
hygrometricum,
Antirrhinum majus, Perilla frutescens var crispa, Dahlia pinnata ou
Erythranthe
lewisii, en particulier d'Arabidopsis thaliana, Lonicera japonica, Lonicera
macranthoides, Medicago truncatula, Oryza sativa, Petroselinum crispum,
Populus
deltoides ou Zea mays, de préférence de Lonicera japonica, Lonicera
macranthoides, et Petroselinum crispum; de préférence une FNS comprenant une
séquence choisie parmi SEQ ID NOs: 33, 35, 37, 127, 129, 131, 133, 135, 137,
139,
141, 143, 145, 147, 149, 151, 153, 155, 157 et 159 et les polypeptides
comprenant
une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence
avec une de ces séquences et présentant une activité flavone synthase, de
préférence parmi les enzymes comprenant une séquence choisie parmi SEQ ID NOs:
33, 35 et 37 et les polypeptides comprenant une séquence ayant au moins 60,
70,
80, 85, 90 ou 95 % d'identité de séquence avec une de ces séquences et
présentant
une activité flavone synthase, de préférence une flavone synthase (FNS)
comprenant une séquence choisie parmi SEQ ID NO: 37 et un polypeptide
comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité
de
séquence avec cette séquence et présentant une activité flavone synthase;
- une séquence d'acide nucléique hétérologue ou endogène codant pour une S-
adénosylnnéthionine synthétase (SAMT) ; en particulier de Saccharomyces
cerevisiae, par exemple une SAMT comprenant une séquence choisie parmi SEQ ID
NO: 81 et un polypeptide comprenant une séquence ayant au moins 60, 70, 80,
85,
90 ou 95 % d'identité de séquence avec cette séquence et présentant une
activité
S-adénosylnnéthionine synthétase ;
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
110
- une séquence d'acide nucléique hétérologue codant pour une tyrosine
ammonia
lyase (TAL) capable de produire de l'acide p-counnarique à partir de la
tyrosine; de
préférence de Rhodotorula glutinis ou Flayobacterium johnsoniae ; en
particulier
une TAL comprenant une séquence choisie parmi SEQ ID NOs: 39 et 41 et les
polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 %
d'identité de séquence avec une de ces séquences et présentant une activité
tyrosine ammonia lyase ; de préférence une TAL comprenant une séquence choisie
parmi SEQ ID NO: 41 et les polypeptides comprenant une séquence ayant au moins
60, 70, 80, 85, 90 ou 95% d'identité de séquence avec cette séquence et
présentant
une activité tyrosine ammonia lyase;
- une séquence d'acide nucléique hétérologue codant pour une 4-coumarate-
CoA
ligase (4CL) capable de produire de le coumaryl-CoA à partir de l'acide p-
coumarique et de Coenzyme A; de préférence d'Arabidopsis thaliana,
Petroselinum
crispum ou Streptomyces clayuligerus ; une 4CL comprenant une séquence choisie
parmi SEQ ID NOs: 43, 45, 47 et 49 et les polypeptides comprenant une séquence
ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec une de
ces
séquences et présentant une activité 4-coumarate-CoA ligase, et de préférence
une
4CL comprenant une séquence sélectionnée parmi la SEQ ID NO: 45 et les
polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 %
d'identité de séquence avec cette séquence et présentant une activité 4-
coumarate-CoA ligase;
- une séquence d'acide nucléique hétérologue codant pour une chalcone
synthase
(CHS) capable de produire de la naringénine-chalcone à partir du 4-coumaroyl-
CoA
et de malonyl-CoA ; de préférence de Citrus sinensis, de Hordeum yulgare ou de
Streptomyces clayuligerus, en particulier une CHS comprenant une séquence
choisie parmi SEQ ID NOs: 51, 53, 55 et 57 et les polypeptides comprenant une
séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec
une de ces séquences et présentant une activité chalcone synthase, de
préférence
parmi les enzymes comprenant une séquence choisie parmi SEQ ID NOs: 53 et 55
et les polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90
ou
95 % d'identité de séquence avec une de ces séquences et présentant une
activité
chalcone synthase, et de manière plus particulièrement préférée une CHS
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
111
comprenant une séquence choisie parmi SEQ ID NO: 53 et les polypeptides
comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité
de
séquence avec cette séquences et présentant une activité chalcone synthase ;
- une séquence d'acide nucléique hétérologue codant pour une chalcone
isomérase
(CHI) capable de produire la naringénine à partir de la naringénine chalcone;
de
préférence d'Arabidopsis thaliana ou Streptomyces clavuligerus, en particulier
une
CHI comprenant une séquence choisie parmi SEQ ID NOs: 59 et 61 et les
polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 %
d'identité de séquence avec une de ces séquences et présentant une activité
chalcone isomérase, et de préférence une CHI comprenant une séquence choisie
parmi la SEQ ID NO: 61 et les polypeptides comprenant une séquence ayant au
moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec cette séquence et
présentant une activité chalcone isomérase ;
- une séquence d'acide nucléique hétérologue codant pour une phénylalanine
ammonia lyase (PAL), en particulier une PAL comprenant une séquence choisie
parmi SEQ ID NOs: 63, 65, 77 et les polypeptides comprenant une séquence ayant
au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec une de ces
séquences et présentant une activité phénylalanine ammonia lyase, en
particulier
une PAL comprenant une séquence choisie parmi SEQ ID NO: 65 et les
polypeptides
comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité
de
séquence avec cette séquences et présentant une activité phénylalanine ammonia
lyase ; et
- une séquence d'acide nucléique hétérologue codant pour une cinnamate 4-
hydroxylase (C4H), en particulier une C4H comprenant une séquence choisie
parmi
SEQ ID NOs: 67, 69, 79 et les polypeptides comprenant une séquence ayant au
moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec une de ces
séquences
et présentant une activité cinnamate 4-hydroxylase, capables de produire de
l'acide
p-coumarique à partir de la phénylalanine, en particulier une C4H comprenant
une
séquence choisie parmi SEQ ID NO: 79 et les polypeptides comprenant une
séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec
cette séquence et présentant une activité cinnamate 4-hydroxylase ;
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
112
- une séquence d'acide nucléique codant pour une sous-unité 4-
hydroxyphénylacetate 3-nnonooxygénase oxygénase (HpaB), de préférence
comprenant une séquence choisie parmi SEQ ID NO: 83 et les polypeptides
comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité
de
séquence avec celle-ci et présentant une activité 4-hydroxyphénylacetate 3-
nnonooxygénase oxygénase, et une séquence d'acide nucléique codant pour une
sous-unité 4-hydroxyphénylacetate 3-nnonooxygénase réductase (HpaC), de
préférence comprenant une séquence choisie parmi SEQ ID NO: 85 et les
polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 %
d'identité de séquence avec celle-ci et présentant une activité 4-
hydroxyphénylacetate 3-nnonooxygénase réductase; ou une séquence d'acide
nucléique codant pour une 4-nnéthoxybenzoate 0-dénnéthylase capable de
convertir la tyrosine en L-Dopa et également l'acide p counnarique en acide
caféique, de préférence comprenant une séquence choisie parmi SEQ ID NOs: 73
et
75 et les polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85,
90
ou 95 % d'identité de séquence avec une de ces séquences et présentant une
activité L-tyrosine hydrolase; ou une séquence d'acide nucléique hétérologue
codant pour une p-counnarate 3-hydroxylase capable de convertir l'acide p-
counnarique en acide caféique, de préférence comprenant une séquence SEQ ID
Nos : 71 et les polypeptides comprenant une séquence ayant au moins 60, 70,
80,
85, 90 ou 95 % d'identité d'une séquence avec une de ces séquences et
présentant
une activité P-counnarate 3 hydroxylase, et
- une séquence d'acide nucléique codant pour une dihydroxyphénylalanine
annnnonia-lyase (DAL).
Selon un mode de réalisation préféré, la méthode comprend l'introduction :
(i) une séquence d'acide nucléique hétérologue codant pour une flavanone 7-0-
bêta-
D-glucosyltransférase (UGT) d'Arabidopsis thaliana, de Scutellaria baicalensis
ou
d'Homo sapiens, de préférence d'Arabidopsis thaliana ou de Scutellaria
baicalensis,
de préférence une flavanone 7-0-bêta-D-glucosyltransférase (UGT) sélectionnée
parmi les enzymes comprenant une séquence choisie parmi SEQ ID NOs: 113, 115,
91, 93, 95, 97, 99 et 101 et les polypeptides comprenant une séquence ayant au
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
113
moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec une de ces
séquences
et présentant une activité flavanone 7-0-bêta-D-glucosyltransférase ; de
manière
particulière une flavanone 7-0-bêta-D-glucosyltransférase (UGT) comprenant une
séquence choisie parmi SEQ ID NOs: 113 et 95 et les polypeptides comprenant
une
séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec
une de ces séquences et présentant une activité flavanone 7-0-bêta-D-
glucosyltra nsférase;
(ii) une séquence d'acide nucléique hétérologue codant pour une 6"-O-
rhamnosyltransferase (RhaT) du genre Citrus ou de Petunia hybrida, de
préférence
Citrus sinensis, Citrus maxima, ou Citrus clementina, de manière encore plus
préférée Citrus sinensis ou Citrus clementina, de préférence une RhaT
comprenant
une séquence choisie parmi SEQ ID NOs: 103, 105 et les polypeptides comprenant
une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 %d'identité de séquence
avec
une de ces séquences et présentant une activité 6"-O-rhamnosyltransferase, en
particulier une 6"-O-rhamnosyltransferase (RhaT) comprenant une séquence
choisie parmi SEQ ID NO: 103 et les polypeptides comprenant une séquence ayant
au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec cette séquence
et
présentant une activité 6"-O-rhamnosyltransferase; et
(iii) une séquence d'acide nucléique hétérologue codant pour une UDP-glucose
4,6-
déshydratase/UDP-4-kéto-6-désoxy-D-glucose 3,5-
épimerase/UDP-4-kéto-L-
rhamnose-réductase (RHM) de Citrus sinensis ou d'Arabidopsis thaliana, de
préférence une RHM comprenant une séquence choisie parmi SEQ ID NOs: 107, 109
et 111 et les polypeptides comprenant une séquence ayant au moins 60, 70, 80,
85,
90 ou 95 % d'identité de séquence avec une de ces séquences et présentant une
activité UDP-glucose 4,6-déshydratase/UDP-4-kéto-6-désoxy-D-glucose 3,5-
épirnerase/UDP-4-kéto-L-rhamnose-réductase, en particulier une RHM
comprenant une séquence sélectionnée parmi SEQ ID NO: 107 et les polypeptides
comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité
de
séquence avec cette séquence et présentant une activité UDP-glucose 4,6-
déshydratase/UDP-4-kéto-6-désoxy-D-glucose 3,5-
épimerase/UDP-4-kéto-L-
rhamnose-réductase ;
et d'au moins une séquence d'acide nucléique choisie parmi :
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
114
- d'une séquence d'acide nucléique hétérologue codant pour une 0-
nnéthyltransférase (OMT) capable de nnéthyler l'ériodictyol et/ou la lutéoline
en position
4'; de préférence, une OMT de Citrus clementina, Citrus sinensis,
d'Arabidopsis thaliana ou
d'Homo sapiens, de préférence une OMT comprenant une séquence choisie parmi
SEQ ID
NOs: 117, 119, 87 et 89 et les polypeptides comprenant une séquence ayant au
moins 60,
70, 80, 85, 90 ou 95 % d'identité de séquence avec une de ces séquences et
présentant une
activité 0-méthyltransférase, notamment avec l'ériodictyol et/ou la lutéoline
comme
substrat et une méthylation en position 4', de préférence une OMT comprenant
une
séquence choisie parmi SEQ ID NOs: 117 et 119 et les polypeptides comprenant
une
séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec
une de ces
séquences et présentant une activité 0-méthyltransférase, et manière tout
particulièrement préférée une OMT comprenant une séquence choisie parmi SEQ ID
NO:
117 et les polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85,
90 ou 95
% d'identité de séquence avec cette séquence et présentant une activité 0-
méthyltransférase;
- d'une séquence d'acide nucléique hétérologue codant pour une flavonoïde 3'-
monooxygénase (F3'H) capable d'hydroxyler en position 3' la naringénine et/ou
l'apigénine
et comprenant une séquence sélectionnée parmi les SEQ ID NOs: 7, 11, 17 et
121, et les
polypeptides comprenant une séquence ayant au moins 75%, au moins 80%, au
moins 85%,
au moins 90% ou au moins 95% d'identité avec l'une de ces séquences et
présentant
l'activité flavonoïde 3'-monooxygénase, de préférence une enzyme comprenant
une
séquence sélectionnée parmi les SEQ ID NOs: 7, 17 et 121, et les polypeptides
comprenant
une séquence ayant au moins 75%, au moins 80%, au moins 85%, au moins 90% ou
au
moins 95% d'identité avec l'une de ces séquences et présentant l'activité
flavonoïde 3'-
monooxygénase, et de manière tout particulièrement préférée une enzyme
comprenant
une séquence sélectionnée parmi la SEQ ID NO: 7, et les polypeptides
comprenant une
séquence ayant au moins 75%, au moins 80%, au moins 85%, au moins 90% ou au
moins
95% d'identité avec cette séquence et présentant l'activité flavonoïde 3'-
monooxygénase ;
et
- une séquence d'acide nucléique hétérologue codant pour une cytochrome P450
réductase (CPR) et comprenant une séquence choisie parmi SEQ ID NOs: 23, 25,
27, 29 et
31 et les polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85,
90 ou 95
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
115
% d'identité de séquence avec une de ces séquences et présentant une activité
cytochrome
P450 réductase, de préférence parmi les enzymes comprenant une séquence
choisie parmi
SEQ ID NOs: 23, 25 et 29 et les polypeptides comprenant une séquence ayant au
moins 60,
70, 80, 85, 90 ou 95 % d'identité de séquence avec une de ces séquences et
présentant une
activité cytochrome P450 réductase, en particulier une CPR comprenant une
séquence
choisie parmi SEQ ID NO: 25 et les polypeptides comprenant une séquence ayant
au moins
60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec cette séquences et
présentant une
activité cytochrome P450 réductase; et
- une séquence d'acide nucléique hétérologue codant pour une FNS comprenant
une séquence choisie parmi SEQ ID NOs: 33, 35, 37, 127, 129, 131, 133, 135,
137, 139, 141,
143, 145, 147, 149, 151, 153, 155, 157 et 159 et les polypeptides comprenant
une séquence
ayant au moins 60,70, 80, 85, 90 ou 95% d'identité de séquence avec une de ces
séquences
et présentant une activité flavone synthase, en particulier une flavone
synthase (FNS) et
comprenant une séquence choisie parmi SEQ ID NOs: 33, 35, 37 et les
polypeptides
comprenant une séquence ayant au moins 60, 70,80, 85, 90 ou 95% d'identité de
séquence
avec une de ces séquences et présentant une activité flavone synthase, de
préférence une
flavone synthase (FNS) comprenant une séquence choisie parmi SEQ ID NO: 37 et
un
polypeptide comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 %
d'identité
de séquence avec cette séquence et présentant une activité flavone synthase;
et
- une séquence d'acide nucléique codant pour une S-adénosylméthionine
synthétase (SAMT) ; en particulier de Saccharomyces cerevisiae, par exemple
une SAMT
comprenant une séquence choisie parmi SEQ ID NO: 81 et un polypeptide
comprenant une
séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec
cette
séquence et présentant une activité S-adénosylméthionine synthétase, et
- une séquence d'acide nucléique hétérologue codant pour une tyrosine ammonia
lyase (TAL) de Rhodotorula glutinis ou Flavobacterium johnsoniae ; en
particulier une TAL
comprenant une séquence choisie parmi SEQ ID NOs: 41 et 39 et les polypeptides
comprenant une séquence ayant au moins 60, 70,80, 85, 90 ou 95% d'identité de
séquence
avec une de ces séquences et présentant une activité tyrosine ammonia lyase ;
de
préférence une TAL comprenant une séquence choisie parmi SEQ ID NO: 41 et les
polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90 ou 95 %
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
116
d'identité de séquence avec cette séquence et présentant une activité tyrosine
annnnonia
lyase ;
- une séquence d'acide nucléique hétérologue codant pour une 4-counnaroyl-CoA
ligase (4CL) d'Arabidopsis thaliana, Citrus clementina, Petroselinum crispum
ou
Streptomyces clavuligerus ; en particulier, une 4CL comprenant une séquence
choisie parmi
SEQ ID NOs: 123, 125, 45, 43, 47 et 49 et les polypeptides comprenant une
séquence ayant
au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec une de ces
séquences et
présentant une activité 4-counnarate-CoA ligase ; de préférence une 4CL
comprenant une
séquence sélectionnée parmi les SEQ ID NOs: 123, 125 et 45 et les polypeptides
comprenant une séquence ayant au moins 60, 70,80, 85, 90 ou 95% d'identité de
séquence
avec une de ces séquences et présentant une activité 4-counnarate-CoA ligase ;
de manière
tout particulièrement préférée une 4CL comprenant une séquence sélectionnée
parmi la
SEQ ID NO: 45 et les polypeptides comprenant une séquence ayant au moins 60,
70, 80, 85,
90 ou 95 % d'identité de séquence avec cette séquence et présentant une
activité 4-
counnarate-CoA ligase, et
- une séquence d'acide nucléique hétérologue codant pour une chalcone synthase
(CHS) de Citrus sinensis, de Hordeum vulgare ou de Streptomyces clavuligerus,
en
particulier une CHS comprenant une séquence choisie parmi SEQ ID NOs: 53, 51,
55 et 57
et les polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90
ou 95 %
d'identité de séquence avec une de ces séquences et présentant une activité
chalcone
synthase, de préférence une CHS comprenant une séquence choisie parmi SEQ ID
NO: 53
et les polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85, 90
ou 95 %
d'identité de séquence avec cette séquence et présentant une activité chalcone
synthase;
et
- une séquence d'acide nucléique hétérologue codant pour une chalcone
isonnérase (CHI) d'Arabidopsis thaliana ou Streptomyces clavuligerus, en
particulier une CHI
comprenant une séquence choisie parmi SEQ ID NOs: 61 et 59 et les polypeptides
comprenant une séquence ayant au moins 60, 70,80, 85, 90 ou 95% d'identité de
séquence
avec une de ces séquences et présentant une activité chalcone isonnérase ; de
préférence
une CHI comprenant une séquence choisie parmi la SEQ ID NO: 61 et les
polypeptides
comprenant une séquence ayant au moins 60, 70,80, 85, 90 ou 95% d'identité de
séquence
avec cette séquence et présentant une activité cha Icone isonnérase .
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
117
De préférence, la méthode comprend l'introduction de toutes ces séquences.
De préférence, la méthode comprend en outre l'introduction :
- une séquence d'acide nucléique hétérologue codant pour une phénylalanine
ammonia
lyase (PAL), en particulier une PAL comprenant une séquence choisie parmi SEQ
ID NOs:
63, 65 et 77, de préférence SEQ ID NO: 65 et 77, et les polypeptides
comprenant une
séquence ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec
une de ces
séquences et présentant une activité phénylalanine ammonia lyase, et de
manière plus
particulièrement préférée une phénylalanine ammonia lyase (PAL) comprenant une
séquence choisie parmi la SEQ ID NO: 65 et les polypeptides comprenant une
séquence
ayant au moins 60, 70, 80, 85, 90 ou 95 % d'identité de séquence avec cette
séquence et
présentant une activité phénylalanine ammonia lyase ; et
- une séquence d'acide nucléique hétérologue codant pour une cinnamate 4-
hydroxylase
(C4H), en particulier une C4H comprenant une séquence choisie parmi SEQ ID
NOs: 67, 69,
79 et les polypeptides comprenant une séquence ayant au moins 60, 70, 80, 85,
90 ou 95
% d'identité de séquence avec une de ces séquences et présentant une activité
cinnamate
4-hydroxylase ; et de manière tout particulièrement préférée une cinnamate 4-
hydroxylase
(C4H) comprenant une séquence choisie parmi la SEQ ID NO: 79 et les
polypeptides
comprenant une séquence ayant au moins 60, 70,80, 85, 90 ou 95% d'identité de
séquence
avec cette séquence et présentant une activité cinnamate 4-hydroxylase.
De préférence, la méthode comprend l'introduction de combinaisons de séquences
codantes particulières telles que décrites ci-dessus.
Production de la diosmine et/ou de l'hespéridine
La présente invention est relative à l'utilisation d'un microorganisme selon
la présente
invention pour la production de la diosmine et/ou de l'hespéridine. Dans un
premier mode
de réalisation préféré, elle est relative à l'utilisation d'un microorganisme
selon la présente
invention pour la production de la diosmine. Dans un deuxième mode de
réalisation
préféré, elle est relative à l'utilisation d'un microorganisme selon la
présente invention
pour la production de l'hespéridine. Dans un mode de réalisation préféré, elle
est relative
à l'utilisation d'un microorganisme selon la présente invention pour la
production de la
diosmine et de l'hespéridine.
La présente invention est également relative à une méthode de production de la
diosmine
et/ou de l'hespéridine comprenant la mise en culture d'un microorganisme selon
la
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
118
présente invention, notamment dans des conditions permettant ou favorables à
la
production de la diosnnine et/ou de l'hespéridine et facultativement la
récupération et/ou
purification de la diosnnine et/ou de l'hespéridine produite.
Les conditions de culture du microorganisme selon l'invention peuvent être
adaptées selon
les techniques classiques, bien connues de l'homme du métier.
Le microorganisme est cultivé dans un milieu de culture approprié. Le terme
milieu de
culture approprié désigne d'une manière générale un milieu de culture
apportant les
nutriments essentiels ou bénéfiques à la maintenance et/ou à la croissance
dudit
microorganisme, tels que les sources carbonées ; les sources azotées telles
que le sulfate
d'ammonium ; les sources de phosphores, par exemple, le potassium phosphate
monobasique ; les oligo-éléments, par exemple, les sels de cuivre, d'iodure,
de fer, de
magnésium, de zinc ou de molybdate ; les vitamines et autres facteurs de
croissance tels
que des acides aminés ou autres promoteurs de croissance. Un antimousse peut
être
ajouté selon les besoins. Selon l'invention, ce milieu de culture approprié
peut être
chimiquement défini ou complexe. Le milieu de culture peut ainsi être de
composition
identique ou similaire à un milieu synthétique, tel que défini par Verduyn et
al., (Yeast.
1992. 8:501-17), adapté par Visser et al., (Biotechnology and bioengineering.
2002.
79:674-81), ou commercialement disponible tel que le milieu YNB (Yeast
Nitrogen Base,
MP Biomedicals ou Sigma-Aldrich). Notamment, le milieu de culture peut
comprendre une
source de carbone simple, tel le glucose, le fructose, le xylose, l'éthanol,
le glycérol, le
galactose, le saccharose, la cellulose, la cellobiose, l'amidon, les polymères
de glucose, les
mélasses, ou les sous-produits de ces sucres.
De préférence, la production de diosmine et/ou d'hespéridine par le
microorganisme selon
l'invention est obtenue sans apport de naringénine, d'apigénine,
d'ériodictyol, de lutéoline,
d'hespérétine et/ou de diosmétine dans le milieu de culture, de préférence
sans apport de
naringénine, de d'apigénine, d'ériodictyol, de lutéoline, d'hespérétine et de
diosmétine
dans le milieu de culture.
Selon l'invention, tout mode de culture permettant la production à l'échelle
industrielle de
molécules d'intérêt peut être envisagé. Avantageusement, la culture se fait en
bioréacteurs, notamment en mode batch, fed-batch, chemostat et/ou culture
continue.
Une alimentation contrôlée en vitamines au cours du procédé peut également
être
bénéfique à la productivité (Alfenore et al., Appl Microbiol Biotechnol. 2002.
60:67-72).
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
119
La culture est généralement conduite en bioréacteurs, avec de possibles étapes
de
précultures solides et/ou liquides en Erlennneyers, avec un milieu de culture
approprié.
D'une manière générale, les conditions de culture des microorganismes selon
l'invention
sont aisément adaptables par l'homme du métier, en fonction du microorganisme.
Par
exemple, la température de culture est notamment comprise pour les levures
entre 20 C
et 40 C, de préférence entre 28 C et 35 C, et plus particulièrement d'environ
30 C pour S
cerevisiae.
Le microorganisme selon la présente invention peut être cultivé pendant 1 à 30
jours, et
de préférence de 1 à 10 jours.
Un microorganisme selon la présente invention est capable de produire de la
diosmine
et/ou de l'hespéridine en une quantité minimale de 1 mg/I de milieu de
culture, de
préférence de 10, 20, 25, 30, 35, 40, 45, 50, 55, 60, 65, 70, 75, 80, 85, 90,
95, 100 mg/I de
milieu de culture, éventuellement de 150, 200, 250, 300, 350, 400, 450, 500,
600, 700, 800,
900 ou 1000 mg/I de milieu de culture.
DESCRIPTION DES FIGURES
Figure 1 Description des voies métaboliques de production d'hespéridine et
de la
diosmine.
Figure 2 : Production d'ériodictyol à partir de naringénine par la souche
FL_405 (F3'H4 +
CPR2). Souche témoin : CF235. Observation de la disparition du pic de
naringénine et
apparition d'un pic d'ériodictyol dans la souche FL-405.
Figure 3 : Production de lutéoline à partir d'apigénine par la souche FL_405
(F3'H4 + CPR2).
Souche témoin : CF235. Observation de la disparition du pic d'apigénine et
apparition d'un
pic de lutéoline dans la souche FL-405.
Figure 4: Production d'Apigénine à partir de naringénine par la souche 5C744
(FNSII1 +
CPR2). Souche témoin : CF234. Observation de la disparition du pic de
naringénine et
apparition d'un pic d'apigénine dans la souche.
Figure 5 : Production de lutéoline à partir d'ériodictyol 5C744 (FNSII1 +
CPR2). Souche
témoin : CF234. Observation de la disparition du pic d'ériodictyol et
apparition d'un pic de
lutéoline dans la souche.
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
120
Figure 6 : Production d'ériodictyol et de lutéoline par la souche SC1500.
Souche témoin :
CF237. Observation des pics d'ériodictyol et de lutéoline.
Figure 7 : Production d'hespérétine à partir d'ériodictyol par les souches SC
1612 (MET +
SAM) et SC 1614 (MET + SAM). Souche témoin : CF235. Observation de la
disparition du pic
d'ériodictyol et apparition d'un pic d'hespérétine dans les souches.
Figure 8: Production de diosnnétine à partir de lutéoline par la souche SC
1612 (MET + SAM)
et SC 1614 (MET + SAM). Souche témoin : CF235. Observation de la disparition
du pic de
lutéoline et apparition d'un pic de diosnnétine dans les souches.
Figure 9 : Production de diosnnétine à partir d'hespérétine par la souche
5C744 (FNSII +
CPR). Souche témoin : CF234. Observation de la disparition du pic
d'hespérétine et
apparition d'un pic de diosnnétine dans la souche.
Figure 10: Production de d'hespérétine à partir d'ériodictyol par E. coli EC26
(MET + SAM).
Souche témoin : E. coli MH1. Observation de la disparition du pic
d'ériodictyol et apparition
d'un pic d'hespérétine dans la souche.
Figure 11 : Production de diosnnétine à partir de lutéoline par E. coli EC26
(MET + SAM).
Souche témoin : E. coli MH1. Observation de la disparition du pic de lutéoline
et apparition
d'un pic de diosnnétine dans la souche.
Figure 12: Production de diosnnétine à partir d'hespérétine par E. coli EC30
(FNSII). Souche
témoin : E. coli MH1. Observation de la disparition du pic d'hespérétine et
apparition d'un
pic de diosnnétine dans la souche.
Figure 13 : Production d'hespérétine et de diosnnétine par la souche 5C1508.
Souche
témoin : CF237. Observation des pics d'hespérétine de de diosnnétine.
Figure 14: Production d'hespéridine à partir d'hespérétine par la souche FL
547 (GT + RHM
+ RHAT). Souche témoin : CF 233. Observation de la disparition du pic
d'héspérétine et
apparition du pic d'hespéridine.
Figure 15: Production de diosnnine à partir de diosnnétine par la souche FL
547 (GT + RHM
+ RHAT). Souche témoin : CF 233. Observation de la disparition du pic de
disométine et
apparition du pic de diosmine.
Figure 16: Production d'hespérédine à partir d'hespérétine par E. coli EC38,
EC45 et EC47
(GT + RHM + RHAT). Souche témoin : E. coli MH1. Observation de la disparition
du pic
d'héspérétine et apparition du pic d'hespéridine.
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
121
Figure 17 : Production de diosmine à partir de diosmétine par E. coli EC38,
EC45 et EC47
(GT + RHM + RHAT). Souche témoin : E. coli MH1. Observation de la disparition
du pic de
diosmétine et apparition du pic de diosmine.
Figure 18 : Production d'hespéridine et de diosmine par les souches 5C1509,
5C1530,
5C1529, 5C1568 et 5C2410. Souche témoin : CF237. Observation des pics
d'hespéridine et
de diosmine.
Figure 19 : Production d'ériodictyol et de lutéoline par les souches 5C2424,
5C2425,
5C2426, 5C2427, 5C2428 et 5C1500. Souche témoin: CF237.
Figure 20: Production d'hespérétine et d'homoériodictyol par les souches
5C2147, 5C2151,
5C1612 et 5C1614. Souche témoin: CF235.
Figure 21 : Production de diosmétine et de chrysoériol par les souches 5C2147,
5C2151,
5C1612 et 5C1614. Souche témoin: CF235.
Figure 22: Production de d'hespérétine à partir d'ériodictyol par E. coli EC41
(MET + SAM).
Souche témoin : E. coli MH1. Observation de la disparition du pic
d'ériodictyol et apparition
d'un pic d'hespérétine dans la souche.
Figure 23: Production de d'hespérétine à partir d'ériodictyol par E. coli EC43
(MET + SAM).
Souche témoin : E. coli MH1. Observation de la disparition du pic
d'ériodictyol et apparition
d'un pic d'hespérétine dans la souche.
Figure 24 : Production de diosmétine à partir de lutéoline par E. coli EC43
(MET + SAM).
Souche témoin : E. coli MH1. Observation de la disparition du pic de lutéoline
et apparition
d'un pic de diosmétine dans la souche.
Figure 25 : Production d'hespérétine et de diosmétine par la souche 5C2408.
Souche
témoin: CF237. Observation des pics d'hespérétine de de diosmétine.
Figure 26 : Production d'hespérétine et de diosmétine par la souche 5C2409.
Souche
témoin: CF237. Observation des pics d'hespérétine de de diosmétine.
Figure 27 : Production d'hespérétine et de diosmétine par les souches 5C2408,
5C2409 et
5C1508. Souche témoin: CF237.
Figure 28 : Production d'hespéridine et de diosmine par les souches 5C1579,
5C1584,
5C1621 et 5C1626.
Figure 29: Production de diosmétine à partir de naringénine par les souches
5C2429 à
5C2434, 5C2436 à 5C2444, 5C2446 à 5C2454, 5C2456 à 5C2464 et 5C2466.
CA 03129605 2021-08-09
WO 2020/165189
PCT/EP2020/053503
122
[Tableau 1] DESCRIPTION DES SEQUENCES
SEQ ID Description
NO
39 Séquence d'acides aminés de la tyrosine annnnonia lyase de
Flavobacterium
johnsoniae
40 Séquence nucléique codant pour la tyrosine annnnonia lyase de
Flavobacterium johnsoniae
41 Séquence d'acides aminés de la tyrosine annnnonia lyase de
Rhodotorula
glu tinis
42 Séquence nucléique codant pour la tyrosine annnnonia lyase de
Rhodotorula
glu tinis
43 Séquence d'acides aminés de la 4-counnarate-CoA ligase
d'Arabidopsis
thaliana
44 Séquence nucléique codant pour la 4-counnarate-CoA ligase
d'Arabidopsis
thaliana
45 Séquence d'acides aminés de la 4-counnarate-CoA ligase de
Petroselinum
crispum
46 Séquence nucléique codant pour la 4-counnarate-CoA ligase de
Petroselinum
crispum
47 Séquence d'acides aminés de la 4-counnarate-CoA ligase de
Petroselinum
crispum
48 Séquence nucléique codant pour la 4-counnarate-CoA ligase de
Petroselinum
crispum
49 Séquence d'acides aminés de la 4-counnarate-CoA ligase de
Streptomyces
clavuligerus
50 Séquence nucléique codant pour la 4-counnarate-CoA ligase de
Streptomyces
clavuligerus
51 Séquence d'acides aminés de la chalcone synthase de Hordeum
vulgare
52 Séquence nucléique codant pour la chalcone synthase de Hordeum
vulgare
53 Séquence d'acides aminés de la chalcone synthase de Citrus
sinensis
54 Séquence nucléique codant pour la chalcone synthase de Citrus
sinensis
55 Séquence d'acides aminés de la chalcone synthase de Citrus
sinensis
56 Séquence nucléique codant pour la chalcone synthase de Citrus
sinensis
57 Séquence d'acides aminés de la chalcone synthase de Streptomyces
clavuligerus
58 Séquence nucléique codant pour la chalcone synthase de
Streptomyces
clavuligerus
59 Séquence d'acides aminés de la chalcone isonnérase de Streptomyces
clavuligerus
60 Séquence nucléique codant pour la chalcone isonnérase de
Streptomyces
clavuligerus
61 Séquence d'acides aminés de la chalcone isonnérase d'Arabidopsis
thaliana
62 Séquence nucléique codant pour la chalcone isonnérase
d'Arabidopsis
thaliana
33 Séquence d'acides aminés de la flavone synthase de Lonicera
japonica
34 Séquence nucléique codant pour la flavone synthase de Lonicera
japonica
35 Séquence d'acides aminés de la flavone synthase de Lonicera
macranthoides
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
123
36 Séquence nucléique codant pour la flavone synthase de Lonicera
macranthoides
37 Séquence d'acides aminés de la flavone synthase de Petroselinum
crispum
38 Séquence nucléique codant pour la flavone synthase de Petroselinum
crispum
1 Séquence d'acides aminés de la flavonoïde 3'-nnonooxygénase de
Perilla
frutescens var. crispa
2 Séquence nucléique codant pour la flavonoïde 3'-nnonooxygénase de
Perilla
frutescens var. crispa
3 Séquence d'acides aminés de la flavonoïde 3'-nnonooxygénase de
Phanerochaete chrysosporium
4 Séquence nucléique codant pour la flavonoïde 3'-nnonooxygénase de
Phanerochaete chrysosporium
Séquence d'acides aminés de la flavonoïde 3'-nnonooxygénase de Petunia x
hybrida
6 Séquence nucléique codant pour la flavonoïde 3'-nnonooxygénase de
Petunia
x hybrida
7 Séquence d'acides aminés de la flavonoïde 3'-nnonooxygénase de
Callistephus
chinensis
8 Séquence nucléique codant pour la flavonoïde 3'-nnonooxygénase de
Callistephus chinensis
9 Séquence d'acides aminés de la flavonoïde 3'-nnonooxygénase de
Callistephus
chinensis
Séquence nucléique codant pour la flavonoïde 3'-nnonooxygénase de
Callistephus chinensis
11 Séquence d'acides aminés de la flavonoïde 3'-nnonooxygénase de
Gerbera
hybrida
12 Séquence nucléique codant pour la flavonoïde 3'-nnonooxygénase de
Gerbera
hybrida
13 Séquence d'acides aminés de la flavonoïde 3'-nnonooxygénase de
Osteospermum hybrid cultivar
14 Séquence nucléique codant pour la flavonoïde 3'-nnonooxygénase de
Osteospermum hybrid cultivar
Séquence d'acides aminés de la flavonoïde 3'-nnonooxygénase de Citrus
clementina
16 Séquence nucléique codant pour la flavonoïde 3'-nnonooxygénase de
Citrus
clementina
17 Séquence d'acides aminés de la flavonoïde 3'-nnonooxygénase de
Citrus
sinensis
18 Séquence nucléique codant pour la flavonoïde 3'-nnonooxygénase de
Citrus
sinensis
19 Séquence d'acides aminés de la flavonoïde 3'-nnonooxygénase de
Pilosella
officin arum
Séquence nucléique codant pour la flavonoïde 3'-nnonooxygénase de Pilosella
officin arum
21 Séquence d'acides aminés de la flavonoïde 3'-nnonooxygénase de
Streptomyces avermitilis
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
124
22 Séquence nucléique codant pour la flayonoïde 3'-nnonooxygénase de
Streptomyces avermitilis
23 Séquence d'acides aminés de la cytochrome P450 réductase de
Catharanthus
rose us
24 Séquence nucléique codant pour la cytochrome P450 réductase de
Catharanthus rose us
25 Séquence d'acides aminés de la cytochrome P450 réductase de
Saccharomyces cerevisiae
26 Séquence nucléique codant pour la cytochrome P450 réductase de
Saccharomyces cerevisiae
27 Séquence d'acides aminés de la cytochrome P450 réductase
chimérique
28 Séquence nucléique codant pour la cytochrome P450 réductase
chimérique
29 Séquence d'acides aminés de la cytochrome P450 réductase
d'Arabidopsis
thaliana
30 Séquence nucléique codant pour la cytochrome P450 réductase
d'Arabidopsis
thaliana
31 Séquence d'acides aminés de la cytochrome P450 réductase
d'Arabidopsis
thaliana
32 Séquence nucléique codant pour la cytochrome P450 réductase
d'Arabidopsis
thaliana
63 Séquence d'acides aminés de la phénylalanine annnnonia lyase de
Citrus
sinensis
64 Séquence nucléique codant pour la phénylalanine annnnonia lyase de
Citrus
sinensis
65 Séquence d'acides aminés de la phénylalanine annnnonia lyase de
Citrus
sinensis
66 Séquence nucléique codant pour la phénylalanine annnnonia lyase de
Citrus
sinensis
67 Séquence d'acides aminés de la cinnannate 4-hydroxylase de Citrus
sinensis
68 Séquence nucléique codant pour la cinnannate 4-hydroxylase de
Citrus sinensis
69 Séquence d'acides aminés de la cinnannate 4-hydroxylase de Citrus
sinensis
70 Séquence nucléique codant pour la cinnannate 4-hydroxylase de
Citrus sinensis
71 Séquence d'acides aminés de la counnarate 3-hydroxylase de
Saccharothrix
espanaensis
72 Séquence nucléique codant pour la counnarate 3-hydroxylase de
Saccharothrix
espanaensis
73 Séquence d'acides aminés de la 4-nnéthoxybenzoate 0-dénnéthylase
de Beta
vulgaris
74 Séquence nucléique codant pour la 4-nnéthoxybenzoate 0-
dénnéthylase de
Beta vulgaris
75 Séquence d'acides aminés de la 4-nnéthoxybenzoate 0-dénnéthylase
de
Rhodopseudomonas pal ustris
76 Séquence nucléique codant pour la 4-nnéthoxybenzoate 0-
dénnéthylase de
Rhodopseudomonas pal ustris
77 Séquence d'acides aminés de la phénylalanine annnnonia lyase
d'Arabidopsis
thaliana
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
125
78 Séquence nucléique codant pour la phénylalanine annnnonia lyase
d'Arabidopsis thaliana
79 Séquence d'acides aminés de la cinnannate 4-hydroxylase
d'Arabidopsis
thaliana
80 Séquence nucléique codant pour la cinnannate 4-hydroxylase
d'Arabidopsis
thaliana
81 Séquence d'acides aminés de la S-adénosylnnéthionine synthétase de
Saccharomyces cerevisiae
82 Séquence nucléique codant pour la S-adénosylnnéthionine synthétase
de
Saccharomyces cerevisiae
83 Séquence d'acides aminés de la 4-hydroxyphénylacetate 3-
nnonooxygénase
oxygénase d'Escherichia coui
84 Séquence nucléique codant pour la 4-hydroxyphénylacetate 3-
nnonooxygénase oxygénase d'Escherichia coui
85 Séquence d'acides aminés de la 4-hydroxyphénylacetate 3-
nnonooxygénase
réductase d'Escherichia coui
86 Séquence nucléique codant pour la 4-hydroxyphénylacetate 3-
nnonooxygénase réductase d'Escherichia coui
87 Séquence d'acides aminés de la 0-méthyltransférase d'Arabidopsis
thaliana
88 Séquence nucléique codant pour la 0-méthyltransférase
d'Arabidopsis
thaliana
89 Séquence d'acides aminés de la 0-nnéthyltransférase d'Homo sapiens
90 Séquence nucléique codant pour la 0-méthyltransférase d'Homo
sapiens
91 Séquence d'acides aminés de la flavanone 7-0-bêta-D-
glucosyltransférase
d'Arabidopsis thaliana
92 Séquence nucléique codant pour la flavanone 7-0-bêta-D-
glucosyltransférase
d'Arabidopsis thaliana
93 Séquence d'acides aminés de la flavanone 7-0-bêta-D-
glucosyltransférase de
Scutellaria baicalensis
94 Séquence nucléique codant pour la flavanone 7-0-bêta-D-
glucosyltransférase
de Scutellaria baicalensis
95 Séquence d'acides aminés de la flavanone 7-0-bêta-D-
glucosyltransférase de
Scutellaria baicalensis
96 Séquence nucléique codant pour la flavanone 7-0-bêta-D-
glucosyltransférase
de Scutellaria baicalensis
97 Séquence d'acides aminés de la flavanone 7-0-bêta-D-
glucosyltransférase de
Scutellaria baicalensis
98 Séquence nucléique codant pour la flavanone 7-0-bêta-D-
glucosyltransférase
de Scutellaria baicalensis
99 Séquence d'acides aminés de la flavanone 7-0-bêta-D-
glucosyltransférase
d'Homo sapiens
100 Séquence nucléique codant pour la flavanone 7-0-bêta-D-
glucosyltransférase
d'Homo sapiens
101 Séquence d'acides aminés de la flavanone 7-0-bêta-D-
glucosyltransférase
d'Homo sapiens
102 Séquence nucléique codant pour la flavanone 7-0-bêta-D-
glucosyltransférase
d'Homo sapiens
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
126
103 Séquence d'acides aminés de la 6-0-rhannnosyltransferase de Citrus
clementina
104 Séquence nucléique codant pour la 6-0-rhannnosyltransferase de
Citrus
clementina
105 Séquence d'acides aminés de la 6-0-rhannnosyltransferase de Citrus
sinensis
106 Séquence nucléique codant pour la 6-0-rhamnosyltransferase de
Citrus
sinensis
107 Séquence d'acides aminés de l'UDP-glucose 4,6-déshydratase/UDP-4-
kéto-6-
désoxy-D-glucose 3,5-épimerase/UDP-4-kéto-L-rhamnose-réductase de Citrus
sinensis
108 Séquence nucléique codant pour l'UDP-glucose 4,6-déshydratase/UDP-
4-
kéto-6-désoxy-D-glucose 3,5-épimerase/UDP-4-kéto-L-rhamnose-réductase
de Citrus sinensis
109 Séquence d'acides aminés de l'UDP-glucose 4,6-déshydratase/UDP-4-
kéto-6-
désoxy-D-glucose 3,5-épinnerase/UDP-4-kéto-L-rhamnose-
réductase
d'Arabidopsis thaliana
110 Séquence nucléique codant pour l'UDP-glucose 4,6-déshydratase/UDP-
4-
kéto-6-désoxy-D-glucose 3,5-épimerase/UDP-4-kéto-L-rhamnose-réductase
d'Arabidopsis thaliana
111 Séquence d'acides aminés de l'UDP-glucose 4,6-déshydratase/UDP-4-
kéto-6-
désoxy-D-glucose 3,5-épimerase/UDP-4-kéto-L-rhamnose-réductase
d'Arabidopsis thaliana
112 Séquence nucléique codant pour l'UDP-glucose 4,6-déshydratase/UDP-
4-
kéto-6-désoxy-D-glucose 3,5-épimerase/UDP-4-kéto-L-rhamnose-réductase
d'Arabidopsis thaliana
113 Séquence d'acides aminés de la flavanone 7-0-bêta-D-
glucosyltransférase de
Citrus sinensis
114 Séquence nucléique codant pour la flavanone 7-0-bêta-D-
glucosyltransférase
de Citrus sinensis
115 Séquence d'acides aminés de la flavanone 7-0-bêta-D-
glucosyltransférase de
Citrus clementina
116 Séquence nucléique codant pour la flavanone 7-0-bêta-D-
glucosyltransférase
de Citrus clementina
117 Séquence d'acides aminés de la 0-nnéthyltransférase de Citrus
clementina
118 Séquence nucléique codant pour la 0-nnéthyltransférase de Citrus
clementina
119 Séquence d'acides aminés de la 0-nnéthyltransférase de Citrus
sinensis
120 Séquence nucléique codant pour la 0-nnéthyltransférase de Citrus
sinensis
121 Séquence d'acides aminés de la flavonoïde 3'-nnonooxygénase
d'Arabidopsis
thaliana
122 Séquence nucléique codant pour la flavonoïde 3'-nnonooxygénase
d'Arabidopsis thaliana
123 Séquence d'acides aminés d'une 4-counnarate-CoA ligase
d'Arabidopsis
thaliana
124 Séquence nucléique codant pour une 4-counnarate-CoA ligase
d'Arabidopsis
thaliana
125 Séquence d'acides aminés d'une 4-counnarate-CoA ligase de Citrus
clementina
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
127
126 Séquence nucléique codant pour la 4-counnarate-CoA ligase de
Citrus
clementina
127 Séquence d'acides aminés de la flavone synthase d'Angelico
archangelica
128 Séquence nucléique codant pour la flavone synthase d'Angelico
archangelica
129 Séquence d'acides aminés de la flavone synthase de Cynara
cardunculus var.
scolymus
130 Séquence nucléique codant pour la flavone synthase de Cynara
cardunculus
var. scolymus
131 Séquence d'acides aminés de la flavone synthase de Perilla
frutescens var.
crispa
132 Séquence nucléique codant pour la flavone synthase de Perilla
frutescens var.
crispa
133 Séquence d'acides aminés de la flavone synthase de Dahlia pinnata
134 Séquence nucléique codant pour la flavone synthase de Dahlia
pinnata
135 Séquence d'acides aminés de la flavone synthase de Callistephus
chinensis
136 Séquence nucléique codant pour la flavone synthase de Callistephus
chinensis
137 Séquence d'acides aminés de la flavone synthase d'Apium graveolens
138 Séquence nucléique codant pour la flavone synthase d'Apium
graveolens
139 Séquence d'acides aminés de la flavone synthase de Medicago
truncatula
140 Séquence nucléique codant pour la flavone synthase de Medicago
truncatula
141 Séquence d'acides aminés de la flavone synthase de Cuminum cyminum
142 Séquence nucléique codant pour la flavone synthase de Cuminum
cyminum
143 Séquence d'acides aminés de la flavone synthase d'Aethusa cynapium
144 Séquence nucléique codant pour la flavone synthase d'Aethusa
cynapium
145 Séquence d'acides aminés de la flavone synthase de Conium
maculatum
146 Séquence nucléique codant pour la flavone synthase de Conium
maculatum
147 Séquence d'acides aminés de la flavone synthase de Camellia
sinensis
148 Séquence nucléique codant pour la flavone synthase de Camellia
sinensis
149 Séquence d'acides aminés de la flavone synthase de Saussurea
medusa
150 Séquence nucléique codant pour la flavone synthase de Saussurea
medusa
151 Séquence d'acides aminés de la flavone synthase de Plectranthus
barbatus
152 Séquence nucléique codant pour la flavone synthase de Plectranthus
barbatus
153 Séquence d'acides aminés de la flavone synthase de Scutellaria
baicalensis
154 Séquence nucléique codant pour la flavone synthase de Scutellaria
baicalensis
155 Séquence d'acides aminés de la flavone synthase de Dorcoceras
hygrometricum
156 Séquence nucléique codant pour la flavone synthase de Dorcoceras
hygrometricum
157 Séquence d'acides aminés de la flavone synthase d'Antirrhinum
malus
158 Séquence nucléique codant pour la flavone synthase d'Antirrhinum
malus
159 Séquence d'acides aminés de la flavone synthase d'Erythranthe
lewisii
160 Séquence nucléique codant pour la flavone synthase de
d'Erythranthe lewisii
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
128
EXEMPLES
Matériels et méthodes
Souches
Les levures utilisées dans les exemples ont été obtenues à partir de
Saccharomyces
cerevisiae FY1679-28A (Tettelin et al., 1995 https://doi.org/10.1016/S1067-
2389(06)80008-7). Cette levure possède une quadri auxotrophie pour l'uracile,
le
tryptophane, l'histidine et la leucine. Les souches de bactéries utilisées
dans les exemples
ont été obtenues à partir de Escherichia con M H1.
Standards
Les standards ont été acquis chez le fournisseur Extrasynthèse, France
(Naringénine,
Apigénine, Eriodictyol, Lutéoline, Hespérétine, Hespéridine, Diosnnétine et
Diosnnine).
Clonage des gènes
Les gènes optimisés pour s'exprimer dans la levure ont été synthétisés par
Eurofins
genonnics, Ebersberg, Allemagne ou Bionnatik, Cambridge, Canada ou Twist
Biosciences,
San Francisco, USA ou DC Biosciences, Dundee, UK. Par PCR, le gène cpr2 (SEQ
ID NO: 26)
de S. cerevisiae a été amplifié à partir de l'ADN génonnique.
Les gènes obtenus par synthèse ou par PCR comprennent à l'extrémité 5' et 3',
un site de
restriction Bbsl (GAAGAC) ou Bsal (GGTCTC).
Tous les gènes, les promoteurs et les ternninateurs ont été clonés restriction
dans le vecteur
pSBK pour l'expression dans la levure ou dans le vecteur pSB1K3 pour
l'expression dans E.
cou. Les promoteurs et ternninateurs (Wargner et al., 2015 D01:
10.1016/j.fgb.2015.12.001)
ont été récupérés par PCR à partir de l'ADN génonnique de la levure S.
cerevisiae ou de E.
cou.
Le vecteur pSBK comprend un marqueur de sélection de la levure URA, ou LEU ou
TRP ou
HIS et le vecteur pSB1K3 comprend un marqueur de résistance à la kanannycine.
Conditions de culture
Les souches ont été cultivées dans 1 ml de milieu minimum nitrogen base
(Dutscher,
Brunnath, Fr) supplénnenté de glucose à 20 g/I pour les levures et dans 1 ml
de M9
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
129
supplénnenté de glucose à 4 g.I-1 pour E. cou i en plaque 24 puits (Starlab,
Orsay, Fr) à 30 C
pendant 72h sous agitation continu à 200 RPM. Dans certains cas, de la
naringénine ou de
l'apigénine a été ajoutée à une concentration de 100 nng.I-1 pour déterminer
l'activité des
F3'H, de la naringénine ou de l'ériodictyol a été ajoutée à une concentration
de 100 nng.I-1
pour déterminer l'activité des FNSII, de l'ériodictyol ou de la lutéoline a
été ajoutée à une
concentration de 100 nng.I-1 pour déterminer l'activité des MET, de
l'hespérétine ou de la
diosnnétine a été ajoutée à une concentration de 100 nng.I-1 pour déterminer
l'activité des
GT, et de l'hespérétine 7-0-glucioside et ou de la diosnnétine 7-0-glucoside
pour
déterminer l'activité des RHM et RHAT.
Chaque souche a été inoculée à DO 0,2 à partir d'une pré-culture de 24h
cultivée dans les
mêmes conditions.
Méthode analytique
Préparation des échantillons : Les cultures de 1 nnL sont congelées à -80 C
puis lyophilisées
pendant 12h à 0,10 nnbar. Les échantillons sont ensuite repris dans 1 nnL de
dinnéthylsulfoxyde (DMSO), agités pendant 30 secondes à 1000 rpnn puis
centrifugés
pendant 5 min à 3000 rpnn à température ambiante. Après centrifugation, un
volume connu
de surnageant est ajouté à un volume connu d'un mélange d'étalons internes
solubilisés
dans du méthanol.
Les concentrations finales des étalons internes sont :
= Diosnnine C13 0,5 nng/L
= Diosnnetine C13 0,015 mg/L.
Analyse par UHPLC-TQ : Les échantillons ont été analysés par une UHPLC
Vanquish-H
(Thernno) couplée à un triple-quadripôle Quantis (Thernno). La colonne est une
colonne
Waters Acquity UPLC@ USST3 (8 gin 2,1 X 100 mm) associée à une pré-colonne
HSST3 1,8
'Inn 2,1 X 5 nnnn.
La phase mobile A est une solution de 0,1 % d'acide formique dans de l'eau de
qualité
LC/MS et la phase mobile B est une solution de 0,1% d'acide formique dans de
l'acétonitrile
pur de qualité LC/MS . La température de la colonne est de 50 C et la
température du
passeur d'échantillons à 10 C.
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
130
Deux conditions chronnatographiques ont pu être utilisés pour la détection des
flavonoïdes
d'intérêt :
[Tableau 2] Conditions chronnatographiques méthode 1
Temps (min) Débit (ml/min) Phase mobile A (%) Phase mobile B (%)
0 0,5 73 27
8 0,5 73 27
[Tableau 3] Conditions chronnatographiques méthode 2
Temps (min) Débit (ml/min) Phase mobile A (%) Phase mobile B (%)
0 0,5 83 17
3,75 0,5 83 17
4 0,5 73 27
8,5 0,5 73 27
11,0 0,5 50 50
13,0 0,5 0 100
13,5 0,5 83 17
15,0 0,5 83 17
Les ions suivis et les conditions de fragmentation pour les molécules
d'intérêts sont :
[Tableau 4] Pour la méthode 1
Molécules Temps de Polarité Ion Ion fils Énergie de RF lens
Standard
rétention précurseur collision (V) interne de
(min) référence
Naringénine 3,3 Négative 271,0 119,0 27 169 Diosmétine
150,9 18 169 013
Apigénine 3,5 Négative 269,0 117,1 35 201 Diosmétine
150,9 24 201 013
Eriodictyol 1,9 Négative 287,1 135,1 26 147 Diosmétine
150,9 14 147 013
Lutéoline 2,1 Négative 285,0 133,0 34 213 Diosmétine
013
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
131
[Tableau 5] Pour la méthode 2
Molécules Temps de Polarité Ion Ion fils Énergie de RF lens
Standard
rétention précurseur collision (V) interne de
(min) référence
Naringénine 7,8 Négative 271,0 119,0 27 169 Diosmétine
150,9 18 169 __ 013
Apigénine 8,2 Négative 269,0 117,1 35 201 Diosmétine
150,9 24 201 __ 013
Eriodictyol 6,2 Négative 287,1 135,1 26 147 Diosmétine
150,9 14 147 __ 013
Lutéoline 6,6 Négative 285,0 133,0 34 213 Diosmétine
150,9 25 213 __ 013
Hespérétine 8,7 Négative 301,0 164,0 24 169 Diosmétine
150,9 17 169 __ 013
Diosmétine 9,1 Négative 299,0 256,0 30 192 Diosmétine
284,1 21 192 __ 013
Hespérétine- 4,9 Négative 463,2 286,0 32 141
Diosmine 013
7-0-
301,1 15 141
Glucoside
Hespéridine 3,9 Négative 609,2 286,0 42 211
Diosmine C13
301,1 25 211
Diosmétine- 5,0 Négative 461,1 284,0 36 194
Diosmine 013
7-0-
299,1 10 194
Glucoside
Diosmine 4,0 Négative 607,2 284,0 50 274
Diosmine 013
299,0 25 274
F3'H
_
Des constructions pour chacune des F3'H ont été réalisées dans un vecteur
portant le
marqueur de sélection URA (Tableau 6). Des constructions comportant chacune
SAM2 et
un seul des différents CPR ont été créés dans un vecteur portant le marqueur
de sélection
LEU (Tableau 7). Deux vecteurs comportant uniquement le marqueur de sélection
URA ou
LEU ont aussi été créé comme témoin. Les gènes marqueurs permettent de repérer
et de
sélectionner les cellules ayant intégré le gène d'intérêt.
CA 03129605 2021-08-09
WO 2020/165189
PCT/EP2020/053503
132
[Tableau 6] Liste des différentes constructions F3'H testées.
Noms Genes assemblés Marqueurs
FL 23 F3'H de Perilla frutescens var. crispa (SEQ ID NO : 2) URA
FL 24 F3'H de Phanerochaete chrysosporium (SEQ ID NO : 4) URA
FL 25 F3'H de Petunia x hybrida (SEQ1D NO :6) URA
FL 26 F3'H de Callistephus chinensis (SEQ ID NO : 8) URA
FL 27 F3'H de Callistephus chinensis (SEQ ID NO : 10) URA
FL 28 F3'H de Gerbera hybrida (SEQ ID NO : 12) URA
FL 29 F3'H d'Osteospermum hybrid cultivar (SEQ ID NO: 14) URA
FL 30 F3'H de Citrus clementina (SEQ ID NO: 16) URA
FL 31 F3'H de Citrus sinensis (SEQ ID NO: 18) URA
FL 32 F3'H de Pilosella officinarum (SEQ ID NO : 20) URA
FL 1031 F3'H d'Arabidopsis thaliana (SEQ ID NO: 122) URA
TT URA - URA
[Tableau 7] Listes des constructions réalisées avec les différents CPR
Noms Genes assemblés Marqueurs
FL 121 CPR de Catharanthus roseus (SEQ ID NO : 24),
LEU
(CPR + SAM) SAM de Saccharomyces cerevisiae (SEQ ID NO: 82)
FL 274 CPR chimérique (SEQ ID NO : 28),
LEU
(CPR + SAM) SAM de Saccharomyces cerevisiae (SEQ ID NO: 82)
FL 275 ATR d'Arabidopsis thaliana (SEQ ID NO : 30),
LEU
(ATR + SAM) SAM de Saccharomyces cerevisiae (SEQ ID NO: 82)
FL 401 CPR de Saccharomyces cerevisiae (SEQ ID NO: 26),
LEU
(CPR + SAM) SAM de Saccharomyces cerevisiae (SEQ ID NO: 82)
FL 463 ATR d'Arabidopsis thaliana (SEQ ID NO : 32),
LEU
(ATR + SAM) SAM de Saccharomyces cerevisiae (SEQ ID NO: 82)
TT LEU LEU
CA 03129605 2021-08-09
WO 2020/165189
PCT/EP2020/053503
133
Plusieurs souches ont été créées avec respectivement toutes les F3'H listées
dans le tableau
6 pour qu'elles soient chacune testées avec toutes les constructions du
tableau 7.
Ces différents assemblages permettent de vérifier l'activité enzymatique des
F3'H et
permettent également de déterminer les couples F3'H ¨ CPR les plus efficaces.
Par exemple, la souche FL 405 possède les constructions FL 26 et FL 401.
La souche témoin (sans les gènes) possédant les constructions TT URA et TT LEU
est
nommée CF235.
FNSII
Pour chacune des FNSII suivantes, des constructions dans un vecteur TRP ont
été effectués
(Tableau 8). Les mêmes vecteurs avec le marqueur de sélection LEU contenant
chacun
SAM2 et un CPR différent ont été utilisés pour tester les FNSII (Tableau 9).
[Tableau 8] Constructions comportant les différentes FNSII testées.
Noms Genes assemblés Marqueurs
TAL de Rhodotorula glutinis (SEQ ID NO : 42),
FL 620 4CL de Petroselinum crispum (SEQ ID NO : 46),
(TAL + 4CL+ CHS + CHI + CHS
de Citrus sinensis (SEQ ID NO: 54), TRP
FNSII) CHI d'Arabidopsis thaliana (SEQ ID NO : 62)
FNSII de Lonicera japonica (SEQ ID NO : 34)
TAL de Rhodotorula glutinis (SEQ ID NO : 42),
FL 621 4CL de Petroselinum crispum (SEQ ID NO : 46),
(TAL + 4CL+ CHS + CHI + CHS
de Citrus sinensis (SEQ ID NO: 54), TRP
FNSII) CHI d'Arabidopsis thaliana (SEQ ID NO : 62)
FNSII de Lonicera macranthoides (SEQ ID NO : 36)
FL 112 TAL de Flavobacetrium jonhsoniae (SEQ ID NO:
40), TRP
(TAL + 4CL+ CHS + CHI +
FNSII) 4CL de Petroselinum crispum (SEQ ID NO : 46),
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
134
CHS de Citrus sinensis (SEQ ID NO: 54),
CHI d'Arabidopsis thaliana (SEQ ID NO : 62)
FNSII de Petroselinum crispum (SEQ ID NO : 38)
TT TRP - TRP
[Tableau 9] Listes des constructions réalisées avec les différents CPR
Noms Genes assemblés Marqueurs
FL 121 CPR de Catharanthus roseus (SEQ ID NO: 24),
LEU
(CPR + SAM) SAM de Saccharomyces cerevisiae (SEQ ID NO : 82)
FL 274 CPR chimérique (SEQ ID NO : 28),
LEU
(CPR + SAM) SAM de Saccharomyces cerevisiae (SEQ ID NO : 82)
FL 275 ATR d'Arabidopsis thaliana (SEQ ID NO : 30),
LEU
(ATR + SAM) SAM de Saccharomyces cerevisiae (SEQ ID NO : 82)
FL 401 CPR de Saccharomyces cerevisiae (SEQ ID NO : 26),
LEU
(CPR + SAM) SAM de Saccharomyces cerevisiae (SEQ ID NO : 82)
FL 463 ATR d'Arabidopsis thaliana (SEQ ID NO : 32),
LEU
(ATR + SAM) SAM de Saccharomyces cerevisiae (SEQ ID NO : 82)
TT LEU - LEU
Plusieurs souches ont été créées avec respectivement chacune des constructions
des FNSII
listées dans le tableau 8 et chacune des constructions des CPR du tableau 9.
Ces différents assemblages permettent de vérifier l'activité enzymatique des
FNSII et
permettent également de déterminer les FNSII les plus efficaces.
Par exemple, la souche SC 744 possède les constructions FL 620 et FL 401.
La souche témoin (sans les gènes) possédant les constructions TT TRP et TT LEU
est nommé
CF234.
Des constructions similaires ont été réalisées pour tester les FNSII des SEQ
ID Nos : 127,
129, 131, 133, 135, 137, 139, 141, 143, 145, 147, 149, 151, 153, 155, 157 et
159.
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
135
LEVURE JUSQU'A ERIODICTYOL/LUTEOLINE
Des souches comportant la voie jusqu'à l'ériodictyol et la lutéoline ont
également été
testées :
- la souche SC1500 comprend les constructions FL 26, FL 602, FL 808 et FL 822;
- la souche SC2424 comprenant les constructions FL 1031 + FL 602 + FL 822 +
TT HIS ;
- la souche SC2425 comprenant les constructions FL 26 + FL 602 + FL 822 +
TT HIS ;
- la souche SC2426 comprenant les constructions FL 31 + FL 602 + FL 822 +
TT HIS ;
- la souche SC2427 comprend les constructions FL 1031, FL 602, FL 808 et FL
822; et
- la souche SC2428 comprenant les constructions FL 31 + FL 602 + FL 808 + FL
822.
[Tableau 10] Listes des constructions utilisées des souches comportant la voie
jusqu'à
l'ériodictyol et la lutéoline
Noms Genes assemblés Marqueurs
FL 26
F3'H de Callistephus chinensis (SEQ ID NO : 8) URA
(F3'H)
FL 1031
F3'H de Arabidopsis thaliana (SEQ ID NO: 122) URA
(F3'H)
FL 31 (F3'H) F3'H de Citrus sinensis (SEQ ID NO : 18) URA
TAL de Rhodotorula glutinis (SEQ ID NO : 42),
FL 602 4CL de Petroselinum crispum (SEQ ID NO : 46),
(TAL + 4CL + CHS + CHS de Citrus sinensis (SEQ ID NO: 54), TRP
CHI + FNS) CHI d'Arabidopsis thaliana (SEQ ID NO :62)
FNSII de Petroselinum crispum(SEQ ID NO : 38)
FL 808 PAL d'Arabidopsis thaliana (SEQ ID NO : 78),
HIS
(PAL + C4H) C4H d'Arabidopsis thaliana (SEQ ID NO : 80),
FL 822 CPR de Catharanthus roseus (SEQ ID NO: 24), LEU
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
136
(CPR + CAF) CAF de Rhodopseudonnonas palustris (SEQ ID NO : 76)
TT HIS HIS
La souche témoin (sans les gènes) possédant les constructions TT URA, TT TRP,
TT HIS et TT
LEU est nommé CF237.
MET:
Afin de tester chacune des MET, des constructions ont été effectuées et sont
présentées
dans le tableau 11. Les gènes marqueurs permettent de repérer et de
sélectionner les
cellules ayant intégré le gène d'intérêt.
[Tableau 11] Listes des constructions réalisées pour tester les différentes
MET
Noms Genes assemblés Marqueurs
FL 121 CPR de Catharanthus roseus (SEQ ID NO: 24),
LEU
(CPR + SAM) SAM de Saccharomyces cerevisiae (SEQ ID NO : 82)
FL 266 F3'H de Callistephus chinensis (SEQ ID NO :8),
URA
(F3'H + MET) MET d'Arabidopsis thaliana (SEQ ID NO : 88)
FL 268 F3'H de Callistephus chinensis (SEQ ID NO :8),
URA
(F3'H + MET) MET d'Homo sapiens (SEQ ID NO : 90)
FL 469 F3'H de Callistephus chinensis (SEQ ID NO :8)
URA
(F3'H + MET) MET de Citrus clementina (SEQ ID NO: 118)
FL 475 F3'H de Callistephus chinensis (SEQ ID NO :8)
URA
(F3'H + MET) MET de Citrus sinensis (SEQ ID NO: 120)
Quatre souches SC1612, SC1614, SC2147 et SC2151 ont été créés, avec FL 121 et
FL 266
pour SC1612, FL 121 et FL 268 pour SC1614, FL 475 et FL 121 pour SC2147 et FL
469 et FL
121 pour SC2151 pour la conversion de l'ériodictyol en hespérétine afin de
déterminer
quelle MET est la plus efficace.
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
137
La souche témoin (sans les gènes) possédant les constructions TT LEU et TT URA
est
nommée CF235.
F3'H, MET, FNS, CPR: PRODUCTION DE DIOSMETINE A PARTIR DE NARINGENINE
[Tableau 12] Liste des constructions utilisées pour tester les enzymes dans
Saccharomyces
cerevisiae (SC)
Noms Genes assemblés Marqueurs
FL 121 CPR de Catharanthus roseus (SEQ ID NO : 24),
LEU
(CPR + SAM) SAM de Saccharomyces cerevisiae (SEQ ID NO: 82)
TT LEU LEU
FL_26
F3'H de Callistephus chinensis (SEQ ID NO :8), URA
(F3'H)
FL 1031
F3'H d'Arabidopsis thaliana (SEQ ID NO :122), URA
(F3'H)
FL 1111 FNSII de Petroselinum crispum (SEQ ID NO : 33)
TRP
(FNS + MET) MET de Citrus clementina (SEQ ID NO: 118)
FL 1112 FNSII d'Angelico archangelica (SEQ ID NO: 128)
TRP
(FNS + MET) MET de Citrus clementina (SEQ ID NO: 118)
FNSII de Cynara cardunculus var. scolymus (SEQ ID NO:
FL 1113
130) TRP
(FNS + MET)
MET de Citrus clementina (SEQ ID NO: 118)
FL 1114 FNSII de Perilla frutescens var. crispa (SEQ ID NO : 132)
TRP
(FNS + MET) MET de Citrus clementina (SEQ ID NO: 118)
FL 1115 FNSII de Dahlia pinnata (SEQ ID NO: 134)
TRP
(FNS + MET) MET de Citrus clementina (SEQ ID NO: 118)
FL 1116 FNSII de Petroselinum crispum (SEQ ID NO : 33)
TRP
(FNS + MET) MET de Citrus sinensis (SEQ ID NO: 120)
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
138
FNSII de Cynara cardunculus var. scolymus (SEQ ID NO:
FL 1118
130) TRP
(FNS + MET)
MET de Citrus sinensis (SEQ ID NO: 120)
FL 1119 FNSII de Perilla frutescens var. crispa (SEQ ID NO: 132)
TRP
(FNS + MET) MET de Citrus sinensis (SEQ ID NO: 120)
FL 1120 FNSII de Dahlia pinnata (SEQ ID NO: 134)
TRP
(FNS + MET) MET de Citrus sinensis (SEQ ID NO: 120)
Les souches suivantes ont été construites :
SC2429 : FL 1111 + FL 1031 + FL 121 SC2451 : FL 1113 + FL 26 + FL 121
SC2430 : FL 1112 + FL 1031 + FL 121 SC2452 : FL 1114 + FL 26 + FL 121
5 SC2431 : FL 1113 + FL 1031 + FL 121 25 SC2453 : FL 1115 + FL 26 + FL 121
SC2432 : FL 1114 + FL 1031 + FL 121 SC2459 : FL 1111 + FL 26 + TT LEU
SC2433 : FL 1115 + FL 1031 + FL 121 SC2460 : FL 1112 + FL 26 + TT LEU
SC2439 : FL 1111 + FL 1031 + TT LEU SC2461 : FL 1113 + FL 26 + TT LEU
SC2440 : FL 1112 + FL 1031 + TT LEU SC2462 : FL 1114 + FL 26 + TT LEU
10 SC2441 : FL 1113 + FL 1031 + TT LEU 30 SC2463 : FL 1115 + FL 26 + TT LEU
SC2442 : FL 1114 + FL 1031 + TT LEU SC2454 : FL 1116 + FL 26 + FL 121
SC2443 : FL 1115 + FL 1031 + TT LEU SC2456 : FL 1118 + FL 26 + FL 121
SC2434 : FL 1116 + FL 1031 + FL 121 SC2457 : FL 1119 + FL 26 + FL 121
SC2436 : FL 1118 + FL 1031 + FL 121 SC2458 : FL 1120+ FL 26 + FL 121
15 SC2437 : FL 1119 + FL 1031 + FL 121 35 SC2464 : FL 1116 + FL 26 + TT LEU
SC2438 : FL 1120+ FL 1031 + FL 121 SC2466 : FL 1118 + FL 26 + TT LEU
SC2444 : FL 1116 + FL 1031 + TT LEU SC2467 : FL 1119 + FL 26 + TT LEU
SC2446 : FL 1118 + FL 1031 + TT LEU SC2468 : FL 1120 + FL 26 + TT LEU
SC2447 : FL 1119 + FL 1031 + TT LEU
20 SC2448 : FL 1120 + FL 1031 + TT LEU
SC2449 : FL 1111 + FL 26 + FL 121
SC2450 : FL 1112 + FL 26 + FL 121
CA 03129605 2021-08-09
WO 2020/165189
PCT/EP2020/053503
139
La souche témoin (sans les gènes) possédant les constructions TT URA, TT TRP,
TT HIS et
TT LEU est nommé CF237.
E. COLI JUSQU'A HESPERETINE/DIOSMETINE
[Tableau 13] Liste des constructions utilisées pour tester les enzymes dans E.
coui
Noms Genes assemblés
SAM de Saccharomyces cerevisiae (SEQ ID NO : 82)
EC26 (SAM + MET)
MET d'Homo Sapiens (SEQ ID NO : 90)
SAM de Saccharomyces cerevisiae (SEQ ID NO : 82)
EC41 (SAM + MET)
MET de Citrus clementina (SEQ ID NO : 118)
SAM de Saccharomyces cerevisiae (SEQ ID NO : 82)
EC43 (SAM + MET)
MET de Citrus sinensis (SEQ ID NO : 120)
EC30 (FNSII) FNSII de Petroselinum crispum (SEQ ID NO : 38)
LEVURE JUSQU'A HESPERETINE/DIOSMETINE
Trois souches comportant la voie jusqu'à hespérétine/diosnnétine ont également
été
testées. La souche SC1508 comprend les constructions FL 121 + FL 268 + FL 602
+ FL 808
du tableau 14. La souche SC2408 comprend les constructions FL 121 + FL 469 +
FL 602 + FL
808 du tableau 14. La souche SC2409 comprend les constructions FL 121 + FL 475
+ FL 602
+ FL 808 du tableau 14.
[Tableau 14] Listes des constructions utilisées dans les exemples.
Noms Genes assemblés
Marqueurs
FL 121 CPR de Catharanthus roseus (SEQ ID NO : 24),
LEU
(CPR + SAM) SAM de Saccharomyces cerevisiae (SEQ ID NO : 82)
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
140
FL 268 F3'H de Catilistephus chinensis (SEQ ID NO : 8),
URA
(F3'H + MET) MET de Homo Sapiens (SEQ ID NO : 90)
FL 469 F3'H de Callistephus chinensis (SEQ ID NO :8)
URA
(F3'H + MET) MET de Citrus clementina (SEQ ID NO: 118)
FL 475 F3'H de Callistephus chinensis (SEQ ID NO :8)
URA
(F3'H + MET) MET de Citrus sinensis (SEQ ID NO: 120)
TAL de Rhodotorula glutinis (SEQ ID NO : 42),
FL 602 4CL de Petroselinum crispum (SEQ ID NO : 46),
(TAL + 4CL + CHS + CHS de Citrus sinensis (SEQ ID NO : 54), TRP
CHI + FNSII) CHI d'Arabidopsis thaliana (SEQ ID NO : 62)
FNSII de Petroselinum crispum (SEQ ID NO : 38)
FL 808 PAL d'Arabidopsis thaliana (SEQ ID NO : 78),
HIS
(PAL + C4H) C4H d'Arabidopsis thaliana (SEQ ID NO : 80),
TT LEU LEU
TT URA URA
TT TRP TRP
TT HIS HIS
La souche témoin (sans les gènes) possédant les constructions TT LEU, TT URA,
TT TRP et
TT HIS est nommée CF237.
GT
Afin de tester chacune des GT, des constructions ont été effectuées et sont
présentées
dans le tableau 15. Les gènes marqueurs permettent de repérer et de
sélectionner les
cellules ayant intégré le gène d'intérêt.
[Tableau 15] Listes des constructions utilisées pour tester les différentes GT
Noms Genes assemblés
Marqueurs
CA 03129605 2021-08-09
WO 2020/165189
PCT/EP2020/053503
141
F3'H de Callistephus chinensis (SEQ ID NO : 8),
FL 545 MET de Citrus sinensis (SEQ ID NO: 120),
(F3'H + MET + GT + RHM + GT d'Arabidopsis thaliana (SEQ ID NO : 92), URA
RHAT) RHM de Citrus sinensis (SEQ ID NO: 108)
RHAT de Citrus clementina (SEQ ID NO: 104)
F3'H de Callistephus chinensis (SEQ ID NO : 8),
FL 546 MET de Citrus sinensis (SEQ ID NO: 120),
(F3'H + MET + GT + RHM + GT de Scutellaria baicalensis (SEQ ID NO : 94),
URA
RHAT) RHM de Citrus sinensis (SEQ ID NO: 108)
RHAT de Citrus clementina (SEQ ID NO: 104)
F3'H de Callistephus chinensis (SEQ ID NO : 8),
FL 547 MET de Citrus sinensis (SEQ ID NO: 120),
(F3'H + MET + GT + RHM + GT de Scutellaria baicalensis (SEQ ID NO: 96), URA
RHAT) RHM de Citrus sinensis (SEQ ID NO: 108)
RHAT de Citrus clementina (SEQ ID NO: 104)
F3'H de Callistephus chinensis (SEQ ID NO : 8),
FL 548 MET de Citrus sinensis (SEQ ID NO: 120),
(F3'H + MET + GT + RHM + GT de Scutellaria baicalensis (SEQ ID NO : 98),
URA
RHAT) RHM de Citrus sinensis (SEQ ID NO: 108
RHAT de Citrus clementina (SEQ ID NO: 104)
F3'H de Callistephus chinensis (SEQ ID NO : 8),
FL 549 MET de Citrus sinensis (SEQ ID NO: 120),
(F3'H + MET + GT + RHM + GT d'Homo sapiens (SEQ ID NO: 100), URA
RHAT) RHM de Citrus sinensis (SEQ ID NO: 108)
RHAT de Citrus clementina (SEQ ID NO: 104)
FL 550 F3'H de Callistephus chinensis (SEQ ID NO : 8),
(F3'H + MET + GT + RHM + MET de Citrus sinensis (SEQ ID NO: 120), URA
RHAT) GT d'Homo sapiens (SEQ ID NO: 102),
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
142
RHM de Citrus sinensis (SEQ ID NO: 108)
RHAT de Citrus clementina (SEQ ID NO: 104)
F3'H de Callistephus chinensis (SEQ ID NO : 8),
FL 554 MET de Citrus sinensis (SEQ ID NO: 120),
(F3'H + MET + GT + GT de Citrus sinensis (SEQ ID NO: 114), URA
UDPRHA + RHAT)
UDPRHA de Citrus sinensis (SEQ ID NO: 108),
RHAT de Citrus clementina (SEQ ID NO: 104)
F3'H de Callistephus chinensis (SEQ ID NO : 8),
FL 556 MET de Citrus sinensis (SEQ ID NO: 120),
(F3'H + MET + GT + GT de Citrus clementina (SEQ ID NO: 116), URA
UDPRHA + RHAT)
UDPRHA de Citrus sinensis (SEQ ID NO: 108),
RHAT de Citrus clementina (SEQ ID NO: 104)
TT URA - URA
Les différentes constructions avec les différents GT vont permettre de
vérifier l'activité
enzymatique des GT et permettre également de déterminer les GT les plus
efficaces.
La souche témoin (sans les gènes) possédant la construction TT URA est nommée
CF233.
RHM
Afin de tester chacune des RHM, des constructions ont été effectuées et sont
présentées
dans le tableau 16.
[Tableau 16] Listes des constructions utilisées pour tester les différentes
RHM.
Noms Genes assemblés Marqueurs
FL 545 F3'H de Callistephus chinensis (SEQ ID NO : 8), URA
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
143
(F3'H + MET + GT + MET de Citrus sinensis (SEQ ID NO: 120),
RHM + RHAT)
GT d'Arabidopsis thaliana (SEQ ID NO : 98),
RHM de Citrus sinensis (SEQ ID NO: 108),
RHAT de Citrus clementina (SEQ ID NO: 104)
F3'H de Callistephus chinensis (SEQ ID NO : 8),
FL 512 MET d'Homo sapiens (SEQ ID NO: 90),
(F3'H + MET + GT + GT d'Arabidopsis thaliana (SEQ ID NO : 98), URA
RHM + RHAT) RHM d'Arabidopsis thaliana (SEQ ID NO: 110),
RHAT de Citrus clementina (SEQ ID NO: 104)
TT URA URA
Les différentes constructions avec les différents RHM vont permettre de
vérifier l'activité
enzymatique des RHM et permettre également de déterminer les RHM les plus
efficaces.
La souche témoin (sans les gènes) possédant la construction TT URA est nommée
CF233.
RHAT
Afin de tester chacune des RHAT, des constructions ont été effectuées et sont
présentées
dans le tableau 17.
[Tableau 17] Listes des constructions utilisées pour tester les différentes
RHAT
Noms Genes assemblés Marqueurs
F3'H de Callistephus chinensis (SEQ ID NO: 8),
FL 512 MET d'Homo sapiens (SEQ ID NO: 90),
(F3'H + MET + GT3+ GT d'Arabidopsis thaliana (SEQ ID NO : 92), URA
RHM + RHAT) RHM d'Arabidopsis thaliana (SEQ ID NO: 110)
RHAT de Citrus clementina (SEQ ID NO: 104)
FL 165 F3'H de Callistephus chinensis (SEQ ID NO : 8), URA
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
144
(F3'H + MET + GT + MET d'Homo sapiens (SEQ ID NO: 90),
RHM + RHAT)
GT d'Arabidopsis thaliana (SEQ ID NO : 92),
RHM d'Arabidopsis thaliana (SEQ ID NO: 110)
RHAT de Citrus sinensis (SEQ ID NO: 106)
TT U RA' URA
Les différents assemblages effectués avec les différentes RHAT vont permettre
de vérifier
l'activité enzymatique des RHAT et également permettre de déterminer les RHAT
les plus
efficaces.
La souche témoin (sans les gènes) possédant la construction TT URA est nommée
CF233.
E.COLI JUSQU'A HESPERIDINE/DIOSMINE
[Tableau 18] Liste de la souche créée pour tester la voie dans E. coui
Noms Genes assemblés
GT d'Arabidopsis thaliana (SEQ ID NO : 92),
EC38
RHM de Citrus sinensis (SEQ ID NO: 108)
(GT + RHM + RHAT)
RHAT de Citrus clementina (SEQ ID NO : 104)
GT de Citrus sinensis (SEQ ID NO: 114),
EC45 UDPRHA de Citrus sinensis (SEQ ID NO : 108),
(GT + UDPRHA + RHAT)
RHAT de Citrus clementina (SEQ ID NO : 104)
GT de Citrus clementina (SEQ ID NO: 116),
EC47 UDPRHA de Citrus sinensis (SEQ ID NO : 108),
(GT + UDPRHA + RHAT)
RHAT de Citrus clementina (SEQ ID NO : 104)
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
145
LEVURE JUSQU'A HESPERIDINE/DIOSMINE
Neuf souches comportant la voie complète ont également été créées.
La souche SC1509 comprend les constructions FL 121 + FL 511 + FL 602 + FL 808.
La souche SC1530 comprend les constructions FL 121 + FL 603 + FL 602 + FL 808.
La souche SC1529 comprend les constructions FL 121 + FL 554 + FL 602 + FL 808.
La souche SC1568 comprend les constructions FL 121 + FL 556 + FL 602 + FL 808.
La souche SC2410 comprend les constructions FL 121 + FL 1100 + FL 602 + FL
808.
La souche SC1579 comprend les constructions FL 401 + FL 547 + FL 602 + FL 828.
La souche SC1584 comprend les constructions FL 401 + FL 554 + FL 602 + FL 828.
La souche SC1621 comprend les constructions FL 401 + FL 556 + FL 602 + FL 828.
La souche SC1626 comprend les constructions FL 401 + FL 603 + FL 602 + FL 828.
[Tableau 19] Listes des constructions utilisées pour créer les souches jusqu'à
hespéridine/diosnnine.
Noms Genes assemblés Marqueurs
FL 121 CPR de Catharanthus roseus (SEQ ID NO : 24),
LEU
(CPR + SAM) SAM de Saccharomyces cerevisiae (SEQ ID NO : 82)
FL 401 CPR de Saccharomyces cerevisiae (SEQ ID NO : 26),
LEU
(CPR + SAM) SAM de Saccharomyces cerevisiae (SEQ ID NO : 82)
F3'H de Callistephus chinensis (SEQ ID NO : 8),
FL 547
MET de Citrus sinensis (SEQ ID NO: 120),
(F3'H + MET + GT + URA
GT de Scutellaria baicalensis (SEQ ID NO: 96),
RHM + RHAT)
RHM de Citrus sinensis (SEQ ID NO: 108)
CA 03129605 2021-08-09
WO 2020/165189
PCT/EP2020/053503
146
RHAT de Citrus clementina (SEQ ID NO : 104)
F3'H de Callistephus chinensis (SEQ ID NO : 8),
FL 511 MET d'Homo sapiens (SEQ ID NO: 90),
(F3'H + MET + GT + GT d'Arabidopsis thaliana (SEQ ID NO : 92), URA
RHM + RHAT) RHM de Citrus sinensis (SEQ ID NO: 108)
RHAT de Citrus clementina (SEQ ID NO: 104)
F3'H de Callistephus chinensis (SEQ ID NO : 8),
FL 603 MET de Citrus clementina (SEQ ID NO: 118),
(F3'H + MET + GT + GT de Citrus sinensis (SEQ ID NO: 114), URA
UDPRHA + RHAT) UDPRHA de Citrus sinensis (SEQ ID NO: 108),
RHAT de Citrus clementina (SEQ ID NO: 104)
F3'H de Callistephus chinensis (SEQ ID NO : 8),
FL 554 MET de Citrus sinensis (SEQ ID NO: 120),
(F3'H + MET + GT + GT de Citrus sinensis (SEQ ID NO: 114), URA
UDPRHA + RHAT) UDPRHA de Citrus sinensis (SEQ ID NO: 108),
RHAT de Citrus clementina (SEQ ID NO: 104)
F3'H de Callistephus chinensis (SEQ ID NO : 8),
FL 556 MET de Citrus sinensis (SEQ ID NO: 120),
(F3'H + MET + GT + GT de Citrus clementina (SEQ ID NO: 116), URA
UDPRHA + RHAT) UDPRHA de Citrus sinensis (SEQ ID NO: 108),
RHAT de Citrus clementina (SEQ ID NO: 104)
FL 1100 F3'H de Callistephus chinensis (SEQ ID NO : 8), URA
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
147
(F3'H + MET + GT + MET de Citrus clementina (SEQ ID NO: 118),
UDPRHA + RHAT)
GT de Citrus clementina (SEQ ID NO: 116),
UDPRHA de Citrus sinensis (SEQ ID NO: 108),
RHAT de Citrus clementina (SEQ ID NO: 104)
TAL de Rhodotorula glutinis (SEQ ID NO : 42),
FL 602 4CL de Petroselinum crispum (SEQ ID NO : 46),
(TAL + 4CL + CHS + CHS de Citrus sinensis (SEQ ID NO : 54), TRP
CHI + FNS) CHI d'Arabidopsis thaliana (SEQ ID NO : 62)
FNSII de Petroselinum crispum (SEQ ID NO : 38)
FL 808 PAL d'Arabidopsis thaliana (SEQ ID NO : 78),
HIS
(PAL + C4H) C4H d'Arabidopsis thaliana (SEQ ID NO : 80),
FL 828 PAL de Citrus sinensis (SEQ ID NO : 66),
HIS
(PAL + C4H) C4H d'Arabidopsis thaliana (SEQ ID NO : 80),
TT LEU LEU
TT URA URA
TT TRP TRP
TT HIS HIS
La souche témoin (sans les gènes) possédant les constructions TT LEU, TT URA,
TT TRP et
TT HIS est nommée CF237.
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
148
RESULTATS
F3'H
Les tableaux 20 et 21 ci-dessous présente la production d'ériodictyol (tableau
20) et de
lutéoline (tableau 21) obtenue en cultivant les souches comprenant les F3'H
listées dans le
tableau 6 et les constructions du tableau 7, en présence respectivement de
naringénine et
d'apigénine.
[Tableau 20] Concentration en Eriodictyol (en nng.I-1)
F3'H SANS CPR CPR CPR CPR ATR ATR
SE ID No (TT LEU) (SEQ ID No (SEQ ID No (SEQ ID No (SEQ ID No (SEQ
ID No
Q ) (
24; FL121) 26; FL401) 28; FL274) 30; FL275) 32;
FL463)
2 (FL23) 35,5 2,9 42,1 4,3 49,9 4,2 43,6 4,2
38,8 4,1 43,3 5,1
4 (FL24) 1 0,8 6,4 0,5 4,0 0,5 4,7 0,3 5,1
0,4 5,3 0,4
6 (FL25) 115,2 3,2 76,8 4,2 42,3 2,6
70,2 8,6 71,1 8,7 71,3 7,4
8 (FL26) 108,3 4,0 71,1 7,1 89,2 9,5
87,4 5,0 75,8 5,2 90,0 6,1
(FL27) 28,8 1,2 57,7 2,6 69,3 10,6 79,1 4,2
52,3 0,5 69,7 2,3
12 (FL28) 108,0 2,0 7,0 1,4 9,1 5,9 4,6 0,3
7,4 2,8 9,2 0,6
14 (FL29) 119,9 1,1 39,9 4,7 56,1 16,3 64,8
4,1 36,8 4,4 46,1 5,5
16 (FL30) < LQ 76,3 2,6 70,9 6,2 70,4 4,4 58,5
10,9 76,9 1,7
18 (FL31) 107,3 8,0 82,3 17,2 102,2 7,1 98,8
5,9 96,6 4,7 101,3 4,0
(FL32) 33,7 4,0 68,9 2,7 81,5 3,4 63,6 3,7
69,5 0,9 69,7 1,1
122
(FL1031) 4,8 0,3 60,5 3,4 34,4 2,8 25,8 5,8
59,0 1,7 40,0 9,5
LQ: inférieur à la limite de quantification.
10 Les différentes souches sont bien capables de produire de l'ériodictyol
à partir de la
naringénine, en concentrations différentes suivant les F3'H et le CPR utilisés
(voir Figure 2).
[Tableau 21] Concentration en Lutéoline (en nng.I-1)
3' S S C CPR CPR CPR ATR ATR
H AN PR
SE ID N
F
(SEQ ID No (SEQ ID No (SEQ ID No (SEQ ID No (SEQ
ID No
Q o) ( (TT LEU)
24; FL121) 26; FL401) 28; FL274) 30; FL275) 32;
FL463)
2 (FL23) 3,5 0,1 11,7 0,7 9,1 2,1 11,01 0,4
10,8 1,7 10,2 1,4
4 (FL24) < LQ < LQ < LQ < LQ < LQ < LQ
6 (FL25) 10,2 0,9 12,8 0,7 7,8 1,4 11,9 0,8
10,1 1,2 12,9 1,4
8 (FL26) 9,5 0,4 13,2 1,1 8,2 0,7 10,9 0,7
12,2 0,4 12,1 0,7
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
149
(FL27) < LQ 2,5 0,3 < LQ 0,5 0 2,7 0,1
2,77 0,4
12 (FL28) 12,1 0,4 13,3 1,2 14,7 1,8 14,1
1,7 12,5 3,8 15,3 0,9
14 (FL29) 1,5 0,1 0,6 0,04 1,1 0,2 0,8
0,03 0,7 0,06 1,0 0,08
16 (FL30) 0,5 0,02 1,3 0,1 2,5 1,5 1,6
0,1 1,5 0,5 2,0 0,1
18 (FL31) 12,2 0,7 13,2 0,8 13,7 1,2 12,7
0,4 14,0 1,8 12,7 0,6
(FL32) 1,2 0,2 9,9 1,4 2,8 0,4 4,3 0,1
11,0 0,9 9,3 1,8
122
(FL1031) 0,4 0,1 10,9 0,1 3,0 0,6 3,0
0,9 11,4 0,4 9,5 1,6
LQ : inférieur à la limite de quantification.
Les différentes souches sont bien capables de produire de la lutéoline à
partir d'apigénine,
en concentrations différentes suivant les F3'H et le CPR utilisés (voir Figure
3).
5 FNS
Les tableaux 22 et 23 ci-dessous présente la production d'apigénine (tableau
22) et de
lutéoline (tableau 23) obtenue en cultivant les souches comprenant les FNSII
listées dans
le tableau 8 et les constructions du tableau 9, en présence respectivement de
naringénine
et d'ériodictyol.
10 [Tableau 22] Concentration en Apigénine (en nng.I-1)
FNSII SANS CPR CPR CPR CPR ATR ATR
(SEQ ID No) TT LEU) (SEQ ID No (SEQ ID No (SEQ ID No (SEQ ID No (SEQ ID No
(
24; FL121) 26; FL401) 28; FL274) 30;
FL275) 32; FL463)
34 (FL620) 11,6 0,3 34,7 1,0 47,6 5,7 37,7
1,6 50,5 1,5 51,3 3,4
36 (FL621) 3,5 0,1 35,6 0,2 14,9 1,3 16,4
1,4 29,8 3,9 33,2 1,5
38 (FL112) 2,9 0,1 40,7 1,2 41,4 1,5 34,2
1,7 38,0 0,9 43,5 0,0
[Tableau 23] Concentration en Lutéoline (en nng.I-1)
FNSII SANS CPR CPR CPR CPR ATR ATR
(SEQ ID (SEQ
ID No (SEQ ID No (SEQ ID No (SEQ ID No (SEQ ID No
(U LEU) No) 24;
FL121) 26; FL401) 28; FL274) 30; FL275) 32; FL463)
34
(FL620) 2,5 0,2 1,4 0,3 7,8
1,4 4,5 0,9 4,5 2,9 8,5 0,8
36
0,2 0,2 1,5 0,1 1,3 0,2 0,9 0,1 1,3
0,3 1,2 0,2
(FL621)
38
(FL112) 0,2 0,0 4,5 1,9 2,3 0,5 2,6
1,2 1,4 0,0 1,6 0,0
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
150
Les différentes souches sont bien capables de produire de l'apigénine et de la
lutéoline à
partir de naringénine et d'ériodictyol, en concentrations différentes suivant
le FNS utilisé
(Figures 4 et 5).
Des résultats similaires ont été obtenus avec les FNSII des SEQ ID Nos : 127,
129, 131, 133,
135, 137, 139, 141, 143, 145, 147, 149, 151, 153, 155, 157 et 159.
F3'H, MET, FNS, CPR : PRODUCTION DE DIOSMETINE A PARTIR DE NARINGENINE
Les résultats pour la production de diosnnétine à partir de naringénine par
les souches
SC2429 à SC2434, SC2436 à SC2444, SC2446 à SC2454, SC2456 à SC2464 et SC2466 à
SC2468 sont présentés dans la figure 29.
Toutes les souches sont capables de produire de la diosnnétine à partir de
naringénine. La
production de diosnnétine est largement augmentée par l'ajout d'une CPR.
LEVURE JUSQU'A ERIODICTYOL/LUTEOLINE
Les souches SC2424, SC2425, SC2426, SC2427, SC1500 et SC2428 possédent toutes
les
enzymes de la voie jusqu'à l'ériodyctiol et la lutéoline et sont capables de
produire de la
lutéoline et de l'ériodictyol à partir de glucose.
Les résultats de la souche SC1500 correspondent à la figure 6, où les pics de
l'ériodictyol
et de la lutéoline sont observés. Des résultats similaires sont obtenus pour
les souches
SC2424, SC2425, SC2426, SC2427, et SC2428. La production d'ériodictyol et de
lutéoline
pour chacune des souches SC2424, SC2425, SC2426, SC2427, SC1500 et SC2428 est
présentée sur la figure 19.
Il est à noter que l'ajout des enzymes PAL et C4H à la voie de biosynthèse
permet d'obtenir
des concentrations d'ériodictyol et de lutéoline nettement plus importantes
Celles-ci
peuvent être jusqu'à 6 fois plus importantes que les concentrations obtenues
avec les
souches contenant les mêmes enzymes à l'exception de PAL et C4H (cf. figure
19, par
exemple en comparant la souche SC2425 sans PAL/C4H et la souche SC1500 avec
PAL/C4H
ou la souche SC2426 sans PAL/C4H et la souche SC2428 avec PAL/C4H).
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
151
MET
_
Les résultats pour la production de l'hespérétine et de diosnnétine à partir
d'ériodictyol et
de lutéoline par les souches SC1612, SC1614, SC2147 et SC2151 sont présentés
respectivement dans les figures 7, 8, 20 et 21.
Les souches de levure SC1612, SC1614, SC2147 et SC2151 sont bien capables de
produire
de l'hespérétine et/ou la diosnnétine.
A partir d'ériodictyol, les souches SC2147, SC2151 et SC1612 sont capables de
produire
spécifiquement de l'hespérétine, c'est-à-dire de nnéthyler spécifiquement
l'hydroxyle en
position 4' de l'ériodictyol (Figure 20). La souche SC1614 produit quant à
elle un mélange
d'hespérétine et d'honnoériodictyol.
De manière remarquable, la souche SC2151 est par ailleurs capable de produire
de l'ordre
de 40 nng/L d'hespérétine (Figure 20). Les souches SC2147, SC1612 et SC1614
sont quant à
elles capables de produire de la diosnnétine à partir de lutéoline (Figure
21).
FNSII
Les résultats pour la production de la diosnnétine à partir de l'hespérétine
par la souche
SC744 sont présentés sur la figure 9.
La souche de levure SC744 est bien capable de produire de la diosnnétine à
partir
d'hespérétine.
E. COLI JUSQU'A HESPERETINE/DIOSMETINE
Les résultats pour la production de l'hespérétine à partir d'ériodictyol par
la souche EC26,
EC41 et EC43 sont présentés dans les figure 10, 22 et 23 et la production de
diosnnétine à
partir de lutéoline par les souches EC26 et EC43 sont présentés dans les
figures 11 et 24.
Les souches E. cou i EC26, EC41 et EC43 sont bien capables de produire de
l'hespérétine
et/ou de la diosnnétine.
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
152
Les résultats pour la production de diosnnétine à partir d'hespérétine par la
souche EC30
sont présentés sur la figure 12.
La souche E. cou i EC30 est bien capable de produire de la diosnnétine à
partir d'hespérétine.
LEVURE JUSQU'A HESPERETINE/DIOSMETINE
Les résultats pour la production d'hespérétine et de diosnnétine à partir de
glucose des
souches SC1508, SC2408 et SC2409 sont présentés sur les figures 13, 25 et 26.
Les souches de levure SC1508, SC2408 et SC2409 possédant toutes les enzymes de
la voie
jusqu'à l'hespérétine et la diosnnétine sont capables de produire de
l'hespérétine et/ou de
la diosnnétine à partir de glucose (Figure 27). De manière remarquable, la
souche SC2408
produit de l'ordre de 25 nng/L d'hespérétine et environ 5 nng/L de
diosnnétine.
GT
[Table 24] Concentration en hespérétine, hespérétine 7-0-Glucoside et en
hespéridine
suivant les GT utilisées (en nng.I-1)
Hespérétine Hespérétine7-0-Glucoside Hespéridine
milieu 90 8,1 N/D N/D
Témoin - 84,2 4,6 N/D N/D
FL 545 - GT SEQ ID No 92 59,2 1,1 7,1 0,2 70,4 2,2
FL 546- GT SEQ ID No 94 85,1 1,4 N/D 22,2 1,2
FL 547 - GT SEQ ID No 96 N/D 6 0,5 109,9 4,8
FL 548- GT SEQ ID No 98 46,7 2,4 N/D 71,9 2,1
FL 554 - GT SEQ ID No
114 46,7 1,7 fi 70,4 5,0
FL 556 - GT SEQ ID No
116 107,0 3,0 fi 11,3 1,1
fi quantité non déterminée
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
153
[Table 25] Concentration en diosnnétine, diosnnétine 7-0-Glucoside et en
diosnnine suivant
les GT utilisées (en nng.I-1)
Diosmetine Diosmetine 7-o-Glucoside Diosmine
milieu 82,1 3,9 N/D N/D
Témoin - 75,3 6,1 N/D N/D
FL 545 - GT SEQ ID No 92 67,1 2,6 N/D 21,8 0,4
FL 546- GT SEQ ID No 94 61,9 3,3 N/D 23,2 3,2
FL 547- GT SEQ ID No 96 2,4 0,4 7,1 0,5 122,8 8,5
FL 548 - GT SEQ ID No 98 17,7 0,7 N/D 76 4,1
FL 554 - GT SEQ ID No
114 61,4 4,2 7,1 1,1 108,5 8,3
FL 556 - GT SEQ ID No
116 138,6 3,5 N/D N/D
Les différentes souches sont bien capables de produire de l'hespéridine et/ou
de la
diosnnine à partir d'hespérétine et de diosnnétine, en concentrations
différentes suivant les
GT utilisées.
RHM
[Table 26] Concentration en hespérétine, en hespérétine 7-0-Glucoside et en
hespéridine
suivant les RHM utilisées (en nng.I-1)
Hespérétine Hespérétine7-0-Glucoside Hespéridine
milieu 90 8,1 N/D N/D
Témoin - 84,2 4,6 N/D N/D
FL 545 - RHM SEQ ID No 108 59,2 1,1 7,1 0,3 70,4
2,2
FL 512 - RHM SEQ ID No 110 45,6 2,4 N/D 82,2 5,2
[Table 27] Concentration en diosnnétine, en diosnnétine 7-0-Glucoside et en
diosnnine
suivant les RHM utilisées (en nng.I-1)
Diosmetine Diosmetine 7-o-Glucoside Diosmine
milieu 82,1 3,9 N/D N/D
Témoin - 75,3 6,1 N/D N/D
FL 545 - RHM SEQ ID No 108 67,1 2,6 N/D 21,8 0,4
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
154
FL 512- RHM SEQ ID No 110 61,4 3,6 N/D 23,7 1
Les différentes souches sont bien capables de produire de l'hespéridine et de
la diosnnine
à partir d'hespérétine et de diosnnétine, en concentrations différentes
suivant les RHM
utilisées.
RHAT
_
[Table 28] Concentration en hespérétine, hespérétine 7-0-Glucoside et en
hespéridine
suivant les RHAT utilisées (en nng.I-1)
Hespérétine Hespérétine7-0-Glucoside Hespéridine
milieu 90 8,1 NID NID
Témoin - 84,2 4,6 NID NID
FL 512 - RHAT SEQ ID No 104 45,6 2,4 NID 82,2 5,2
FL 165 - RHAT SEQ ID No 106 46 1,5 8,1 0,2 69,8 1,4
[Table 29] Concentration en diosnnétine, diosnnétine 7-0-Glucoside et en
diosnnine suivant
les RHAT utilisées ( en nnünnol)
Diosmetine Diosmetine 7-o-Glucoside Diosmine
milieu 82,1 3,9 NID NID
Témoin - 75,3 6,1 NID NID
FL 512 - RHAT SEQ ID No 104 61,4 3,6 NID 23,7 1
FL 165 - RHAT SEQ ID No 106 66,7 2,5 NID 23 1,5
Les différentes souches sont bien capables de produire de l'hespéridine et de
la diosnnine
à partir d'hespérétine et de diosnnétine, en concentrations différentes
suivant les GT, les
RHM et les RHAT utilisées.
Les résultats pour la production de l'hespéridine et de la diosnnine à partir
d'hespérétine et
de diosnnétine par la souche FL 547 sont présentés respectivement dans les
figures 14 et
15.
CA 03129605 2021-08-09
WO 2020/165189 PCT/EP2020/053503
155
Les levures testées avec les différentes constructions sont bien capables de
produire de
l'hespéridine et de la diosnnine.
E.COLI JUSQU'A HESPERIDINE/DIOSMINE
Les résultats pour la production d'hespéridine à partir d'hespérétine par les
souches EC38,
EC45 et EC47 sont présentés dans la figure 16.
Ces souches sont bien capables de produire de l'hespéridine à partir
d'hespérétine.
Les résultats pour la production de diosnnine à partir de diosnnétine par les
souche EC38,
EC45 et EC47 sont présentés sur la figure 17.
Ces souches sont bien capables de produire de la diosnnine à partir de
diosnnétine.
LEVURE JUSQU'A H ESPERI DINE/DIOSMI NE
Les résultats pour la production d'hespéridine et de diosnnine à partir de
glucose par les
souches SC1509, SC1530, SC1529, SC1568 et SC2410 sont présentés sur la figure
18. Les
résultats pour la production d'hespéridine et de diosnnine par les souches
SC1579, SC1584,
SC1621 et SC1626 sont présentés sur la figure 28.
Toutes les souches possédant toutes les enzymes de la voie sont capables de
produire de
l'hespéridine et/ou de la diosnnine.