Note: Descriptions are shown in the official language in which they were submitted.
~ wo 94/24300 216 0 6 9 7 PcT/FRs4/on4ls
-
1 .
ENSEMBLE DE TRANSPOSITION POUR LE TRANSFERT DE GENE CHEZ LES EUCARYOTES.
20 La présente invention a pour objet un ensemble de transposition permettant le transfert
de gènes d'intérêt dans le génome d'une cellule ou d'un organisme eucaryote. Un tel
ensemble est particulièrement utile à des fins de thérapie génique.
De nombreux éléments pouvant être mis en oeuvre par l'intégration de gènes d'intérêt
2s dans le génome eucaryote, ont été décrits dans les publications de l'art antérieur. Les
éléments classiques sont soit des vecteurs intégratifs, par exemple des vecteursrétroviraux soit des vecteurs non-intégratifs, par exemple des vecteurs adénoviraux.
Toutefois ces vecteurs de l'art antérieur ne présentent pas que des avantages.
30 En effet, les vecteurs rétroviraux s'intègrent au hasard de façon non-spécifique dans le
génome cellulaire. Ainsi, le risque de mutagenèse insertionnelle liée soit à l'inactivatiori
de gènes essentiels à la cellule, soit à l'activation d'oncogènes pouvant donner lieu à
une prolifération tumorale, n'est pas à négliger avant d'envisager leur utilisation en
thérapie génique humaine.
3s
En ce qui concerne les vecteurs non-intégratifs, ils présentent des inconvénients liés à
une instabilité du fait de leur non-intégration dans le génome cellulaire, ce qui nécessite
leur atlmini~tration régulièrement renouvelée. Dans le cadre d'une thérapie génique
wo 9~/24300 21 6 0 6 9 7 PCT/FR94/00419
destinée à l'homme, ceci risque de poser à long terme des problèmes d'immunisation
contre le virus recombinant, des patients régulièrement traités.
Un troisième type de méthode, plus récemment décrite, permet le transfert de gènes
s d'intéret par recombinaison homologue dans un site défini du génome cellulaire.
Cependant, la technique de recombinaison homologue se heurte encore à de
nombreuses difficultés techniques. De plust des événements de recombinaison non-homologue peuvent également se produire, de sorte que le risque de mutagenèse
insertionnelle subsiste.
D'autre part, l'art antérieur a depuis longtemps établi le schéma d'organisation et
d'expression de l'ADN ribosomal (ADNr) chez les euca~otes (Reeder, Trends Genet.1990, 6, 390-395). D'une manière générale, l'ADNr est constitué par des copies
multiples d'unités de transcription arrangées en tandems. Une unité transcriptionnelle
5 est composée des gènes codant respectivement pour les ARNr 18S ou 16S, 5-8S et28S ou 26S, qui entrent dans la constitution du ribosome (ci-après désigné gène ARNr
18S etc.). Chaque gène est séparé du suivant par des séquences transcrites, dont le rôle
exact n'est pas encore défini. Les trois gènes ARNr compris dans une unité
transcriptionnelle sont placés sous le contrôle d'un promoteur unique situé en amont
20 dans la région non-transcrite qui sépare une unité de la suivante. Les unités d'ADNr
sont transcrites par l'A~N polymérase I en une longue molécule d'ARNr précurseur(pré ARNr), qui est ensuite maturé pour produire les trois principaux types d'ARNr
associés aux sous-unités ribosomates.
2s De nombreuses publications ont signalé la présence fréquente d'éléments génétiques
étrangers dans l'AONr d'eucaryotes. Parmi ces éléments génétiques, on peut citer des
introns de classe I ou Il et des rétroposons, par exemple de classe Rl, R2 ou R3. La
présence de ces éléments étrangers peut éventuellement inactiver une fraction des
unités transcriptionnelles de l'ADNr. Cela ne semble pas avoir de conséquences graves
30 pour la vie des organismes les contenant. Ce phénomène a surtout été décrit chez la
drosophile.
Les introns de classes I et 11 sont définis par l'existence de séquences conservées
forrnant des éléments structuraux caractéristiques de chacune des classes tels que
35 définis par Cech et Bass (1986, Annu. Rev. Biochem. 55, 599-629). Plusieurs a uteurs
ont observé que certains introns de classes I et II sont mobiles. Ils peuvent être copiés
et transférés spécifiquement dans des copies de gènes qui en sont dépourws (intron~)
Ce processus de transposition, au moins dans la plupart des cas, s'avère être spécifique
du point de we de la région d'insertion.
WO 91/24300 21 6 0 6 9 7 PCT/FR94/00419
3
Parmi la soixantaine d'introns de classe I caractérisés jusqu'à présent, la majorité est
localisée dans les gènes des mitochondries et des chloroplastes. L'art antérieurmentionne, dans trois cas seulement, la présence d'introns mobiles de classe I dans les
gènes nucléaires. Ces introns ont été mis en évidence au niveau des gènes ARNr de
trois espèces d'eucaryotes inférieurs, respectivement les gènes ARNr 26S de plusieurs
souches de Te~ahyme~7a (Kan et Gall, 1982, Nucl. Acids Res., 10, 2809-2821) et de la
souche carolil?a de Physar~m~ polycephal~m (Muscarella et Vogt, 1989, Cell, 56,
443-454; cet intron mobile étant désigné ci-après intron 3) et enfin dans le gène ARNr
o 16S de P~7e1~mocystis calillii (Edman et al., 1988, Nature, 33~, 519-522). Jusqu'à
présent, on a pas pu mettre en évidence la présence d'introns interrompant les gènes
ARNr d'eucaryotes supérieurs.
L'intron 3 de Physar1~m polycepha~1m1 est le mieux caractérisé. Sa mobilité a été
démontrée expérimentalement (Muscarella et Vogt, 1989, Cell, 56 443-454). Cet
intron code en partie pour une protéine de 160 acides aminés (Muscarella et al., 1990,
Mol. Cel. Biol., 10, 3386-3396). Le codon d'initiation de la traduction putatif est situé
en amont de l'intron, à l'extrémité 3' de la séquence exonique adjacente en 5' de l'intron.
La protéine codée par l'intron 3, est une endonucléase qui reconnaît une séquence cible
d'au moins 18 paires de bases (pb) présente dans la région d'insertion et est capable de
cliver cette séquence exactement au niveau du site d'insertion de l'intron 3 (Muscarella
et Vogt, 1989, Cell, 56 443-454).
La séquence cible reconnue par l'endonucléase comprend la séquence suivante:
5'CTATGACTCTCTTAAGGTAGCCAAA3'
On suppose que la transposition de l'intron 3 est initiée par la reconn~iss~nc.e de la
séquence cible particulière par l'endonucléase spécifiquement codée par l'intron 3,
suivie d'une coupure des deux brins de la molécule d'ADN au niveau de cette séquence
particulière, libérant ainsi le site d'insertion. Puis par un échange de fibres impliquant
les séquences flanquantes en 5' et 3' du site d'insertion, copie de la séquence intronique
et recombinaison, I'intron 3 est inséré dans la copie du gène ARNr intron-, de manière
précise et site-spécifique. Une fois l'intron 3 inséré dans une copie du gène intron~, la
c séquence cible reconnue par l'endonucléase est interrompue par la séquence intronique
de la manière suivante:
5'CTATGACTCTCT intron 3 TAAGGTAGCCAAA3'
D'autre part, des rétroposons semblent également être présents dans l'ADNr de certains
eucaryotes. D'une manière générale, les rétroposons forment un groupe très vaste et
très hétérogène, notamment au niveau de leur séquence nucléotidique. Par rétroposon,
wo 94/24300 PcT/FRg~mo4l9
4 2f 60~ 7
on entend des éléments apparentés aux rétrovirus, mais dépourvus de "long terminal
repeats" (LTR). Ils comprennent un ou des cadre(s) de lecture ouvert(s) susceptible(s)
de coder pour des protéines présentant une homologie avec des protéines rétrovirales,
comme par exemple la reverse-transcriptase (Jakubczak et al., 1991, Proc. Natl. Acad.
5 Sci. USA, 88, 3295-3299).
Chez un certain nombre d'eucalyotes non-mammifères, notamment les insectes, on atrouvé des rétroposons présentant une spécificité d'insertion remarquable, localisée au
niveau des gènes ARNr. La mobilité de certains d'entre eux a été observée. Plusieurs
0 familles (notamment Rl, R2 et R3) ont été définies en fonction de leur région
d'insertion spécifique dans les gènes ARNr.
Les rétroposons des classes Rl et R2 sont notamment illustrés par les rétroposons
RlBm et R2Bm de Bombyx mori (Xiong et Eickbush, 19~8, Cell, 55, 235-246; Xiong
lS et Eickbush, 1988, Mol. Cell. Biol., 8, 114-123; Xiong et al., 1988, Nucl. Ac. Res.,
16, 10561-10573) et les rétroposons RlDm et R2Dm de Drosophila melanogaster
(Jakubczak et al., 1990, J. Mol. Biol., 212, 37-52). Ils contiennent un cadre de lecture
ouvert susceptible de coder pour une protéine de grande taille dont la partie centrale
présente une homologie avec la famille des réverse-transcriptases. Xiong et Eickbush
20 (1988, Cell, 55, 235-246) rapportent que la protéine codée par le rétroposon R2Bm de
Bombyx mori présente en outre une activité endonucléase. Cette nuclé~ce reconnâît et
clive une séquence cible contenue dans la région d'insertion de ~2Bm située dans le
gène ARNr 28S du génome de l'insecte.
2s Une nouvelle classe de rétroposons ribosomaux, désignée R3, a récemment été mise en
évidence. On n'a pas encore caractérisé de protéine codée par cet élément. Cependant,
la région d'insertion spécifique de l'élément R3 dans l'ADNr de Scaria coprophila a été
décrite (Kerrebrock et al., J. Mol. Biol., 1989, 210, 1-13).
30 A côté des rétroposons, il existe un grand nombre d'éléments génétiques mobiles qui
ont été parfois désignés sous le nom de rétroposons par les auteurs les décrivant, mais
dont on n'a pas encore montré qu'ils codent pour une protéine présentant une
homologie avec la famille des protéines rétrovirales, notamment la reverse-
transcriptase. On a également trouvé ce type d'éléments au niveau de séquences bien
3s définies de gènes ARNr des organismes les contenant (voir par exemple Back et al.,
1984, EMBO J., 3, 2523-2529).
De manière surprenante, on a maintenant trouvé que d'une part les séquences cibles
reconnues par les endonucléases codées par les deux éléments généti~ues mobiles pour
WO 94/24300 216 0 ~ 9 7 PCTtFR94/00419
- 5
lesquels les données sont disponibles (intron 3 de Physarum polycephalun? et
rétroposon R2Bm de Bomb~x mori) et d'autre part les régions d'insertion de certains
éléments génétiques mobiles dont on n'a pas encore montré qu'ils codent pour uneendonucléase et qui ont été divulguées dans l'art antérieur (Jakubczak et al., 1991,
s Proc. Natl. Acad. Sci. USA, 88, 3295-3299; Paskwitz et Collins, 1989, Nucleic Acid
Res., 17, 8125-8133; Kerrebrock et al., 1989, J. Mol. Biol., ~10, 1-13; Kan et Gall,
1982, Nucleic Acid Res., 10, 2809-2821; Edman et al., 1988, Nature, 263, 519-522),
sont conservées dans les gènes ARNr de divers mammifères, notamment l'homme et la
souris. Ainsi, les séquences des régions d'insertion des introns mobiles de Tetrahymena,
lo de Pne2~n70cystis cari~7ii et de Physa~ m polycephalunt, et des rétroposons R2Bm de
Bon1byx mori, et R3 de Scaria c~pl ophila sont conservées à l'identique dans les gènes
ARNr de mammifères. Par contre, les séquences de la région d'insertion des
rétroposons RlDm et RlBm sont homologues mais non identiques à une séquence
présente dans le gène ARNr 28S de mammifère.
Par conséquent, la présente invention a pour objet un ensemble de transposition destiné
au transfert d'un fragment d'ADN d'intérêt dans le génome d'une cellule hôte eucaryote,
qui comprend une cassette d'intégration, essentiellement constituée d'un élémentgénétique mobile au sein duquel est inséré ledit fragment d'ADN d'intérêt; ladite
20 cassette d'intégration étant capable de s'intégrer d'une façon site-spécifique dans un site
d'insertion spécifique situé dans l'ADN nucléaire ribosomal de ladite cellule hôte.
Un ensemble de transposition selon l'invention présente l'avantage de permettre
l'intégration d'un fragment d'ADN d'intérêt dans une région définie et non essentielle du
25 génome cellulaire. Un tel ensemble est notamment utile à des fins de thérapie génique.
Aux fins de la présente invention, la cassette d'intégration doit comporter un élément
génétique mobile qui possède la capacité de s'intégrer par un méc~ni~me site-
spécifique, dans de l'ADNr nucléaire, en particulier au niveau du gène ARNr 28S, 18S
30 ou 5-8S. De manière plus précise, cet élément génétique mobile doit s'intégrer dans un
site d'insertion situé dans une région d'insertion dont la séquence est conservée à
l'identique ou non dans l'ADNr nucléaire de la cellule-hôte.
Pour une meilleure compréhension, on précise que le terme "site d'insertion" définit
3s l'endroit entre 2 nucléotides où s'insère un élement génétique mobile. De même, par
"région d'insertion", on entend les séquences nucléotidiques en 5' et 3' du sited'insertion qui sont requises pour la transposition site-spécifique. D'une manière
générale, chaque élément génétique mobile possède une région d'insertion qui lui est
spécifique. Comme mentionné précédemment pour les rétroposons RlBm et RlDm, il
W O 94/24300 ~ ` PCT/~R9~/00419
6 216~97
n'est pas exigé que la région d'insertion dans la cellule hôte comporte à l'identique les
séquences de la région d'insertion naturelle (dans l'organisme d'origine). Ainsi, la
séquence de la région d'insertion dudit element génétique mobile dans la cellule hôte
eucaryote présentera un degré d'llomologie avec la séquence de la région d'insertion
5 naturelle supérieur à 80%, de manière avantageuse supérieur à 90% et de préférence
supérieur à 95%.
L'élément génétique mobile est avantageusement sélectionné parmi des introns mobiles
de classe I ou II, et des rétroposons tels que les rétroposons de classe Rl R2 ou R3.
Un élément génétique mobile peut être constitué par un fragment fonctionnel ou un
variant dudit élément. Par fragment fonctionnel, on entend tout fragment ayant
conservé la capacité à s'intégrer de l'élément complet. Un variant peut comporter une
ou plusieurs mutations par rapport à la séquence nucléotide naturelle de llélément,
5 notamment la substitution, addition ou délétion d'un ou plusieurs nucléotide(s), à
condition que ces mutations n'en altèrent pas la fonction.
De manière plus particulière, l'élément génétique mobile est sélectionné parmi:
- l'intron 3 de Physamm polycephalum, souche Carolina, un fragment ou
un variant dudit intron,
- l'intron mobile de classe I de Te~rahymena, un fragment ou un variant
dudit intron,
- I'intron mobile de classe I de P~et~mocysfis cari)~ii, un fragment ou un
variant dudit intron,
- le rétroposon R2Bm de Bombyx mori, un fragment ou un variant dudit
rétroposon et,
- le rétroposon R3 de Scaria coprophila, un fragment ou un variant dudit
rétroposon.
La présence dans cet elément génétique mobile, de tout ou partie d'un cadre de lecture
ouvert susceptible de coder pour une intégrase, est une caractéristique préférée.
D'une manière générale, par intégrase, on entend une protéine présentant une activité
35 enzymatique lui permettant de participer directement ou indirectement à la
transposition de l'élément génétique mobile spécifiquement au niveau de son sited'insertion dans l'ADNr nucléaire d'une cellule eucaryote.
Avantageusement, l'intégrase peut être constituée notamment par:
WO 94/24300 21 6 0 6 9 7 PCT/FR9`1/00419
(I) une protéine présentant une activite endonucléase capable de reconnâître uneséquence cible spécifique inclue dans la région d'insertion de l'élément génétique mobile
qui code pour elle et de cliver ladite séquence cible, ou
(2) une protéine présentant une homologie avec la famille des réverse-transcriptases.
On peut citer, par exemple, I'endonucléase codée en partie par l'intron 3 de Physarum
popycepha~7~m Cette endonucléase initie la transposition de l'intron 3 en reconnaissant
sa séquence cible et clivant la molécule d'ADN au niveau de cette séquence spécifique.
0 On peut citer également la protéine codée par le rétroposon R2Bm de Bombyx mori.
L'activité endonucléase de ladite protéine est probablement impliquée dans la
transposition de R2Bm au niveau de sa région d'insertion spécifique mais par un
mécanisme qui n'est pas connu à ce jour.
15 Le fragment d'ADN d'intérêt peut être introduit dans l'élément génétique mobile par les
techniques classiques du génie génétique. 11 peut être intégré dans ou en dehors du
cadre de lecture ouvert codant pour une éventuelle intégrase.
Selon un aspect particulièrement avantageux de l'invention, le fragment d'ADN d'intérêt
20 est inséré dans le cadre de lecture ouvert, afin d'empêcher l'expression de l'intégrase.
Ceci introduit une sécurité pour éviter la propagation incontrôlée de la c~ssette
d'intégration dans le génome de la cellule hôte.
Dans ce cas, I'intégrase spécifique de l'élément génétique mobile mis en oeuvre dans
25 I'ensemble de transposition selon l'invention, devra être fournie à la cellule hôte de
manière transitoire pendant un temps suffisamment long pour permettre la transposition
de la cassette d'intégration dans sa région d'insertion spécifique. Les moyens de fournir
en fral7s une protéine à une cellule eucaryote sont nombreux et connus de l'homme de
l'art.
Par exemple, I'ensemble de transposition selon l'invention peut être introduit dans un
vecteur (tel que défini ci-après), comprenant en outre une c~csette d'expression de
I'intégrase spécifique de l'élément génétique mobile présent dans l'ensemble de
transposition.
D'une manière alternative, on peut transfecter parallèlement la cellule hôte eucaryote
avec un vecteur helper comprenant une cassette d'expression de l'intégrase ou fournir à
la cellule hôte, I'intégrase sous forme purifiée.
wo 94/24300 216 ~ ~ 9 7 PCT/~R94/(10419
Le fragment d'ADN d'intérêt peut être tout fragment capable d'être transcrit en ARN
pour produire un ARN anti-sens, par exemple une séquence d'A~N complémentaire
d'un gène pathogène susceptible de former un duplex avec un transcrit pathogène afin
d'inhiber la traduction en protéine pathogène. Un gène pathogène est:
5 (1) un gène qui n'est pas présent dans les cellules eucaryotes, par exemple un gène
présent dans le génome d'un organisme pathogène (bactérie, virus ou parasite), ou
(2) un gène homologue ou un gène homologue muté, par exemple un oncogène, qui est
présent mais normalement pas exprimé dans les cellules eucaryotes normales et dont
l'expression anormalement induite peut causer une maladie comme le cancer.
De façon alternative, le fragment d'ADN d'intérêt peut coder pour une protéine
d'intérêt et de manière préférée, une protéine dont l'absence d'expression ou
l'expression en quantité anormale ou sous forme mutante est associée à une maladie
génétique.
s
Le fragment d'ADN d'intérêt peut coder pour une protéine mature ou un précurseur de
celle-ci. Dans le premier cas, il comprend la séquence codante pour une protéinemature permettant l'expression de la protéine de façon intrace.~ ire. Dans le dernier
cas, le fragment d'ADN d'intérêt peut également inclure une séquence signal permettant
20 la sécrétion de ladite protéine de la cellule hôte. Le fragment d'ADN d'interêt peut
coder pour une protéine chimérique provenant de la fusion de séquences d'origines
diverses.
Les exemples de protéines pouvant être codées par le fragment d'ADN d'intérêt
25comprennent:
- des cytokines, comme l'interféron~alpha, I'interféron gamma et différents
types de facteurs de croissance,
- des récepteurs membranaires, comme les récepteurs impliqués dans la
transmission des signaux de la surface à l'intérieur des cellules et les
récepteurs reconnus par des organismes pathogènes, comme le
récepteur CD4 présent à la surface des Iymphocytes T et reconnu par la
glycoprotéine d'enveloppe du virus VIH (Virus d'Immunodéficience
Humaine),
- des enzymes, comme les ribonucléases et la thymidine-kinase (TK) du
virus simplex de l'herpès de type I (HSV-I). Cette dernière présente une
af~lnité supérieure par rapport à l'enzyme TK de cellule mammifère pour
certains analogues de nucleosides, comme l'acyclovir ou le gancyclovir.
L'enzyme TK-HSV- I convertit les analogues en précurseurs de
WO 94124300 216 0 6 9 7 PCT/FR94/00419
nucléotides. Ces précurseurs toxiques seront donc incorporés dans
l'ADN des cellules en état de réplication. Cette incorporation permet de
tuer spécifiquement les cellules en division, comme les cellules
cancéreuses,
s - des inhibiteurs d'une activité enzymatique, comme l'alphal antitrypsine,
I'antithrombine III, la protéine C et les inhibiteurs de protéases
spécifiques d'un organisme pathogène,
- des facteurs de coagulation, comme le facteur VIII, le facteur IX et la
thrombine,
lo - des protéines participant aux canaux ioniques, comme la protéine CFTR
(Cystic Fibrosis Transmembrane Conductance Regulator),
- des protéines capables d'inhiber l'activité d'une protéine produite par un
gène pathogène, comme l'antigène supresseur de tumeur p53,
- des variants de protéines pathogènes mutés de façon à altérer leur
fonction biologique, comme des mutants trans-dominants de la protéine
de régulation TAT du virus VIH capables de compétition avec la
protéine virale native pour la liaison à la séquence cible, empêchant
I'activation de l'expression des gènes viraux et,
- des épitopes antigéniques permettant d'accroître l'immunité de la cellule
hôte.
Le fragment d'ADN d'intérêt peut être muté afin de permettre l'expression d'une
protéine d'intérêt dont les propriétés biologiques sont modifiées, par exemple un
variant de l'alphal antitrypsine dont le résidu méthionine en position 358 du site actif a
25 été remplacé par une leucine. Un tel variant est fonctionnel dans des conditions
d'oxydation telle que des conditions d'inflammation.
Une fois la cassette d'intégration introduite dans l'ADNr de la cellule hôte, le fragment
d'ADN d'intérêt peut être placé sous la dépendance du promoteur de l'unité
30 transcriptionnelle de l'ADNr de la cellule hôte. De manière alternative, le fragment
d'ADN d'intérêt peut être placé sous le contrôle d'éléments d'expression appropriés
insérés dans l'élément génétique mobile. Expression signifie ici transcription en ARN
et/ou traduction de cet ARN en protéine.
3s Selon cette alternative, les éléments de contrôle de l'expression comprennentnotamment un promoteur approprié. De tels promoteurs sont bien connus de l'hommede l'art et sont insérés en amont du fragment d'ADN d'intérêt par les techniquesconventionnelles du génie génétique.
WO 94/24300 21 ~ 0 6 9 7 PCT/FR94/00419
Le promoteur retenu peut être un promoteur reconnu par l'ARN polymérase I, de
façon à favoriser l'expression du fragment d'ADN d'intérêt, après intégration de la
cassette d'intégration dans les gènes A}~Nr de la cellule hôte eucaryote. Par exemple, le
fragment d'ADN d'intérêt peut être placé sous le contrôle des éléments de régulation
5 compris dans les régions non-transcrites de l'ADNr impliqués dans la transcription des
gènes ARNr. De tels promoteurs seront choisis de manière à être fonctionnels dans la
cellule hôte eucaryote qui a été retenue.
De manière alternative, le promoteur retenu peut être un promoteur reconnu par l'ARN
o polymérase II. De tels promoteurs sont bien connus de l'homme de l'art. Le promoteur
peut être isolé d'un gène cellulaire ou d'un virus. Il peut être ubiquitaire permettant une
expression permanente du fragment d'ADN dlintérêt dans tous les types de cellules
hôtes. Le terme promoteur inclut egalement un promoteur régulable, par exemple un
promoteur tissu-spécifique. On peut citer notamment le promoteur du gène HMG
5 (Hydroxy-Methyl-Glutaryl coenzyme A réductase), du gène TK du virus HSV-I, du
virus SV40 (Simian virus 40), les promoteurs EIII et MLP (Major Late Promoter) de
l'adénovirus, le LTR du virus MoMuLV (Moloney Murine Leukemia Virus), le
promoteur du gène FIX conférant une expression foie-spécifique ou le promoteur des
gènes immunoglobulines spécifique des cellules lymphocytaires.
De manière avantageuse, llensemble de transposition comprend outre une c~sette
d'intégration des eléments permettant ou favorisant l'intégration site-spécifique de cette
cassette. Selon un mode particulier de l'inven~ion, notamment lorsque l'élément
génétique mobile mis en oeuvre est un intron mobile de classe I ou II, I'ensemble de
25 transposition selon l'invention comprend en outre:
- en 5' de la cassette d'intégration une région d'au plus 10kb, présentant
au moins 80% d'homologie avec les séquences du gène ARNr de la
cellule hôte immédiatement adjacentes en 5' audit site d'insertion; et
- en 3' de la cassette d'intégration une région d'au plus 10kb, présentant
au moins 80% d'homologie avec les séquences du gène ARNr de la
cellule hôte immédiatement adjacentes en 3' audit site d'insertion.
En effet, il peut être avantageux de placer ladite cassette d'intégration dans un
3s environnement ADNr, particulièrement lorsque la transposition implique un phénomène
d'échange de fibres comme cela a éte montré notamment pour l'intron 3. L'échange de
fibres implique les séquences donneuses intron+ (de l'ensemble de transposition) et les
séquences receveuses intron~ (de la cellule hote). Seule la l~csette d'intégration sera
transposée dans le génome de la cellule hôte. Les séquences du gène ARNr
WO 9~/24300 21 6 0 6 9 7 PCT/FR94/00419
immédiatement adjacentes en S' et 3' du site d'insertion interviennent uniquement dans
le processus de transposition.
Afin de favoriser la transposition de la cassette d'intégration, on préférera avoir une
homologie parfaite entre les séquences donneuses et receveuses impliquées dans
l'échange de fibre. Ainsi, un ensemble de transposition préféré comprendra en outre:
- en 5' de la cassette d'intégration une région d'au plus IOkb, présentant
100% d'homologie avec les séquences du gène ARNr de la cellule hôte
o immédiatement adjacentes en S' audit site d'insertion; et
- en 3' de la cassette d'intégration une région d'au plus 10kb, présçnt~nt
100% d'homologie avec les séquences du gène ARNr de la cellule hôte
immédiatement adjacentes en 3' audit site d'insertion.
15 Les séquences du gène ARNr immédiatement adjacentes en 5' et 3' au site d'insertion
peuvent être isolées selon les techniques classiques du génie génétique, par exemple par
clonage ou PCR (Polymerase Chain Reaction) ou synthèse chimique. De manière plusparticuljère, ces séquences auront une longueur d'au plus lOkb, de manière avantageuse
d'au plus 3kb et de préférence d'au plus 0,4 kb.
La présente invention concerne également un moyen d'introduction d'un ensemble de
transposition selon l'invention dans une cellule eucaryote. De tels moyens consistant à
introduire un acide nucléique dans une cellule eucaryote sont généralement connus de
l'homme de l'art.
Aux fins de la présente invention, I'ensemble de transposition selon l'invention peut être
introduit dans un moyen d'introduction sélectionné parmi les véhicules de délivrance de
type liposomes et lipides cationiques synthétiques et les vecteurs de clonage etd'expression classiquement utilisés dans les cellules eucaryotes. L'encapsulation dans un
30 véhicule de délivrance et les techniques de clonage dans des vecteurs constituent des
techniques classiques connues de l'homme de l'art. Mais d'autres protocoles permettant
d'introduire un acide nucléique dans une cellule peuvent également être employés,
comme par exemple la precipitation au phosphate de calcium, la technique du DEAEdextrane, I'injection directe de l'acide nucléique dans une cellule ou le bombardement
35 de microparticules d'or couvertes d'acide nucléique dans les cellules d'un animal.
L'acide nucléique peut être introduit sous forme superenroulee, circulaire ou linéaire.
D'une manière avantageuse, le moyen d'introduction selon l'invention est un vecteur
comprenant les éléments appropriés à sa maintenance dans la cellule hôte pendant un
wo 94/24300 , 2 ~ 7 PCT/FRg4/00419
temps suffisamment long pour permettre le transfert de la cassette d'intégration dans la
région d'insertion. Un tel vecteur doit être capable d'entrer dans une cellule eucaryote
supérieure, de rester de préférence sous forme extrachromosomique et de se maintenir
dans la cellule pendant un temps suffisamment long pour permettre la transposition de
5 la cassette d'intégration dans le génome de la cellule hôte. Un tel vecteur peut être sous
la forme d'un plasmide ou d'un vecteur viral.
De préférence, le moyen d'introduction selon l'invention est un vecteur de type non-
intégratif. On peut citer un vecteur dérivé du virus de l'herpès simplex ou d'uno adénovirus. D'une manière particulièrement préférée, le moyen d'introduction selon
l'invention est un vecteur dérivé d'un adénovirus, comme par exemple l'adénovirus de
type 5.
Selon un mode avantageux et comme évoqué précédemment, le moyen d'introduction
5 selon l'invention peut contenir en outre une cassette d'expression permettant la
production de l'intégrase spécifique de l'ensemble de transposition.
Une telle c~csette d'expression comprend le fragment d'ADN codant pour ladite
intégrase, placé sous le contrôle d'éléments appropriés permettant son exy~ession. Par
20 éléments appropriés permettant son expression, on entend l'ensemble des éléments
permettant la transcription dudit fragment d'ADN en ARNm et la traduction de
l'ARNm en protéine. Ces éléments comportent notamment un promoteur approprié, depréférence un promoteur reconnu par l'ARN polymérase II et pennettant une
expression forte et ubiquitaire, par exemple le promoteur SV40.
Le moyen d'introduction selon l'invention peut en outre comprendre une cassette
d'expression permettant l'expression d'un gène de sélection, perrnettant la détection et
l'isolement des cellules hôtes comprenant ledit moyen d'introduction. Dans le contexte
de l'invention, le gène codant pour le marqueur de sélection peut etre sous le contrôle
30 d'éléments appropries permettant son expression dans la cellule hôte, tels que définis ci-
dessus.
L'invention est également relative à un procédé i~7 l~itro de transfert d'un fragment
d'ADN d'intérêt dans le génome d'une cellule hôte eucaryote au niveau de l'ADN
3s nucléaire ribosomal, selon lequel on introduit dans ladite cellule hôte un ensemble de
transposition selon l'invention ou un moyen d'introduction selon l'invention.
Avantageusement, on peut en outre fournir dans le procédé i~l vitro selon l'invention,
I'intégrase spécifique de l'ensem~le de transposition selon l'invention à la cellule hôte.
~ WO 94/24300 21 6 113 6 9 7 PCT/FR94/0041~
L'invention a également pour objet une cellule eucaryote comprenant un ensemble de
transposition selon l'invention ou un moyen d'introduction selon l'invention. Ladite
cellule sera de manière avantageuse une cellule de mammifère, et de manière préférée
5 une cellule humaine.
La présente invention concerne également une composition pharmaceutique
comprenant à titre d'agent thérapeutique, un ensemble de transposition selon
l'invention, un moyen d'introduction selon l'invention ou une cellule selon l'invention, en
o association avec un support acceptable d'un point de vue pharmaceutique.
La composition pharmaceutique selon l'invention, est en particulier destinée au
traitement préventif ou curatif de maladies telles que:
- des maladies génétiques, comme par exemple l'hémophilie ou la
mucoviscidose,
- des cancers, comme par exemple ceux induits par des oncogènes ou des
virus,
- des maladies rétrovirales, comme par exemple le SIDA (syndrome de
l'immunodéficience acquise), et
- des maladies virales récurrentes, comme par exemple les infections
virales provoquées par le virus de l'herpès.
Une composition pharmaceutique selon l'invention peut être fabriquée de manière
2s conventionnelle. En particulier, on associe une quantité thérapeutiquement efficace d'un
agent thérapeutique à un support tel qu'un diluant. Une composition selon l'invention
peut être administrée par n'importe quelle voie conventionnelle en usage dans ledomaine de l'art, en particulier par voie sous-cutanée, par voie intramuscul~ire, par voie
intraveineuse ou par voie intra-trachéale. L'administration peut avoir lieu en dose
30 unique ou répétée une ou plusieurs fois après un certain délai d'intervalle. La voie
d'administration et le dosage appropriés varient en fonction de divers paramètres, par
exemple, de l'individu traité ou du fragment d'ADN d'intérêt. Une composition
pharmaceutique peut comprendre en outre un adjuvant acceptable d'un point de vuepharmaceutique.
3S
Avantageusement, la composition pharmaceutique selon l'invention comprend en outre
une intégrase ou une cassette d'expression de l'intégrase.
WO94/24300 216 0 6 9 7 pcT~ lon4ls
14
L'invention s'etend égaiement à une méthode de traitement selon laquelle on administre
une quantité thérapeutiquement ef~lcace d'un ensemble de transposition selon
l'invention un moyen d'introduction selon l'invention ou une cellule selon l'invention à
un patient ayant besoin d'un tel traitement.
s
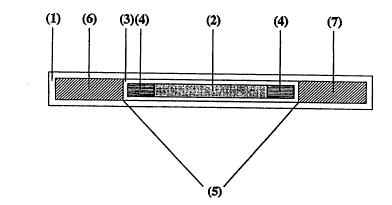
L'invention est illustree ci-après en référence à la figure 1 qui représente
schématiquement un ensemble de transposition (I) (grande boîte blanche) qui
comprend de 5' vers 3': les séquences du gène ARNr (6) (boîte hachurée) en 5' du site
d'insertion (5) (traits noirs) une cassette d'intégration (3) (petite boîte blanche)
o constituée d'un élément génétique mobile (4) (boîte rayée horizontalement) au sein
duquel est inséré un fragment d'ADN d'intérêt (2) (boîte pointillée) et les séquences du
gène ARNr (7) (boîte hachurée) en 3' du site d'insertion (5).
EXEMPLES:
EXEMPLE 1: Construction d'un ensemble de transposition pour l'inté~ration site-
spécifique dans les ~ènes ARNr 28S d'une cellule humaine. mettant en oeuvre l'intron 3
de Phvsan~m polycephal1~n1 souche Carolina.
20 L'exemple se réfère à un ensemble de transposition qui comporte
(I) l'intron 3 modifié de manière à créer un site unique de restriction XhoI dans
la séquence intronique. Le site X77oI permettra d'introduire un fragment d'ADN
d'intérêt quelconque. En ef~et le fragment d'ADN d'intérêt peut être isolé de manière à
présenter des e~l~é"~i~és cohésives compatibles avec les exl~e"~;~és générées par une
25 digestion XhoI (fragment de restriction XhoI ou SQII OU XhoI-Sa/I ou addition de
linkers XhoI et/ou Sa/I ou mutagenèse dirigée afin de créer les sites X77oI et/ou Sal~
requis). D'une manière alternative, le site de clonage X77oI peut être traité par exemple
à la nucléase de haricot Mung afin de générer des eA~ré~-ilés franches entre lesquelles
on peut cloner tout fragment d'ADN d'intérêt à bouts francs. Le fragment d'ADN de
30 0,94 kb comportant l'intron 3 peut être isolé notamment par clonage, PCR ou
synthétiséchimiquement et
(2) les séquences immédiatement adjacentes en 5' et 3' au site d'insertion de
l'intron 3. Ces séquences sont isolées du gène ARNr 28S humain comprenant la région
d'insertion de l'intron 3 parfaitement conservée. Ainsi l'intron mobile sera placé dans un
3s environnement ADNr. L'échange de fibres impliquera des séquences parfaitementhomologues puisque toutes deux d'origine humaine (séquences donneuses de la
cassette d'intégration et séquences receveuses de la cellule humaine hôte). Les
séquences immédiatement adjacentes en 5' et 3' peuvent etre isolées not~mment par
clonage, PCR ou synthétisees chimiquement.
WO 9~/24300 PCT/FR94/00419
15 ~160697
1. Clonage par PCR des séquences immédiatement adjacentes en 5' et 3' au site
d'insertion de l'intron 3 à partir du ~ène humain ARNr 28S.
5 Des amorces ont été définies à l'aide de la séquence du gène ARNr 28S humain inclus
dans la banque de donnée DNAstar (référence: HUMRGM, la séquence reportée allantde la position + I à la position + 5025). On a localisé le site d'insertion et la région
d'insertion de l'intron 3 (tels que reportés dans Muscarella et Vogt, 1989, Cell, 56, 443-
454). Le site d'insertion est compris entre les nucléotides 3742 et 3743 du gène ARNr
0 28S humain.
Un premier fragment d'ADN correspondant au fragment allant de. Ia position 2324 à la
position 3742 du gène ARNr 28S humain est généré par PCR. L'amorce correspondantau brin codant (position 2324 à 2349) est reportée dans la SEQ ID NO: 1 Elle
lS comporte en outre une extrémité S' libre portant les sites Hi~7dIII et NheI. L'amorce
reverse est décrite dans la SEQ ID NO: 2 et comporte une extrémité 5' incluant les
sites SpeI et X~aI. L'amplification est réalisée avec la polymérase de Thermus
Aquaticus (Perkin Elmer Cetus) selon les conditions standards indiquées par le
fabricant. On utilise comme matrice du DNA humain préparé classiquement. Le produit
20 amplifié est vérifié sur gel d'agarose et séquencé.
On isole également par PCR, le deuxième fragment de gène ARNr 28S humain. Il s'agit
de la portion de gène allant de la position 3743 à 4438. Les deux oligonucléotides mis
en oeuvre sont reportés dans les SEQ ID NO: 3 et 4. La première amorce comporte à
25 son e"llé.,.ilé 5' un site SpeI tandis que l'amorce reverse contient de 5' vers 3' les sites
EcoR~, NheI et Bg/lI. La réaction d'amplification est réalisée à partir de l'ADNgénomique humain préparé classiquement, dans les conditions standards courammentemployées par l'homme du métier. Le fragment PCR ainsi généré est vérifié sur gel
d'agarose et séquencé.
Le fragment comprenant Ikb de séquences humaines immédiatement adjacentes en S'
au site d'insertion de l'intron 3, est digéré par les enzymes Hi ?dIII et SpeI. Le fragment
comprenant les séquences immédiatement adjacentes en 3' est digéré par SpeI et
EcoRI. Les deux fragments sont ensuite ligués dans le plasmide pUC 19 (Yanisch-
35 Perron et al., 1985, Gene, 33, 103-119) préalablement digéré par Hi~7dIII et EcoRI. Le
mélange de ligation est transformé dans la souche Escherichia coli SK (E. coli 5K)
donnant lieu au plasmide pHREI.
2. Clona~e de l'intron 3 de Phvsar7/m polvcephal1~nt modifié.
WO 94/24300 _ 21 6 0 6 9 7 PCl`~FR9~/00419
16
On isole l'intron 3 (0,94kb) par la technique PCR. Les amorces sont établies à partir
des données de séquence reportées dans la littérature (Muscarella et al, 1990, Mol.
Cell. Biol., 10, 3386-3996; Johansen et al, 1992, Curr. Genet., 22, 297-304). Les
amorces sont décrites dans les SEQ ID NO: 5 et 6. Elles comprennent toutes deux un
site Kp~7I dans leur région 5'.
La réaction d'amplification est ef~ectuée à partir d'une banque d'ADN génomique de
Physarum polycephal~ , souche Calo1i~1a (Muscarella et Vogt, 1989, Cell, 56, 443-
0 454), en appliquant les conditions standards connues de l'homme de métier. Aprèsamplification, la taille du fragment obtenu est contrôlée sur gel d'agarose et sa
séquence vérifiée
Le fragment PCR est digéré par Kp~rI et cloné dans le site Kp~I du vecteur pUCl915 pour donner prNEI.
Le vecteur pINEI est digéré par NheI, site unique situé dans l'intron 3 de Physarum
polycephal~ . Le vecteur pINEI linéarisé est traité à la nucléase de haricot Mung et
ligué à un linker XhoI (Stratagène). Le vecteur en résultant est désigné pINEII et
20 comporte donc un site uni~ue XhoI pour l'insertion d'un fragment d'ADN d'intérêt.
3. Insertion de l'intron 3 de PhY.sart~m polYcephal~m (XhoI) dans un environnement
gène ARNr 28S humain
25 Le fragment Kp~I obtenu par digestion de prNEII est traité à la polymérase de T4 pour
donner un fragment à bouts francs dont les eAII é~n;lés 5' et 3' correspondent
exactement à celles de l'intron.
Parallèlement, pHREI est digéré par les enzymes X~aI et SpeI, puis traité a la nucléase
30 de haricot Mung (selon la technique reportée dans Sambrook et al, 1989, Cold Spring
Harbor Press). Ceci génère un grand fragment comportant la majorité du vecteur
pHREI, dont les e,~l~é~ és franches correspondent respectivement aux nucléotides en
position 3742 et 3743 du gène ARNr 28S humain.
35 Le fragment issu de prNEII comportant l'intron 3 de Physan~m polycephal~m est ligué
au vecteur pHREI traité comme indiqué ci-dessus, pour donner le vecteur pHREII.
Après introduction d'un fragment d'ADN d'intérêt, le vecteur pH~EI comprendra donc
un ensemble de transposition permettant l'insertion d'une cassette d'intégration (intron 3
co"~prenant un fragment d'ADN d'intérêt) dans le gène ARNr 28S humain.
WO 94/24300 PCT/FR94/00419
~ 17 _ 2160697
EXEMPLE 2: Construction d'un vecteur adénoviral pour l'intégration du gène
néomycine (neo) dans le ~énome humain.
On construit un vecteur adénoviral comportant:
(1) I'intron 3 de Physartm1 polycephal1~m (Xho+) au niveau duquel on introduit
une première cassette d'expression permettant l'expression du gène neo,
(2) une deuxième cassette d'expression, celle de l'endonucléase site-specifique
IPpo, codée en partie par l'intron 3. La deuxième cassette d'expression
permettra la production de l'endonucléase IPpo pendant un temps suffisamment
long pour pennettre la transposition de la cassette d'intégration dans le génomede la cellule hôte. Le codon initiateur de la traduction de IPpo se situe dans la
séquence exonique en 5' de l'intron 3 dans le gène ARNr 26S de Physarum
polycephal~l/n. La séquence immédiatement adjacente en 5' au site d'insertion
isolée du gène ARNr 28S humain ne contient pas l'ATG initiateur présent dans
la région équivalente chez Physarum, et
(3) Les éléments appropriés à la maintenance du vecteur dans la cellule hôte
pendant un temps suffisamment long pour permettre la transposition de la
cassette d'intégration.
1. Construction de la cassette d'expression de IPpo.
2s Un fragment de 167 nucléotides contenant la région poly A de SV40 (Fitzgerald et
Shenk, 1981, Cell, 2~, 251-260) est synthétisé sous forme d'un fragment HiodIII-EcoPl à l'aide d'un synthétiseur automatique (Milligen 7500). Un site BglII est
introduit juste en amont du site ~coRI.
On isole un fragment Hfndlll-BamHI de pMSG-CAT (Pharmacia) comprenant le
promoteur précoce du virus SV40. On introduit le fragment synthétique HindIII-EcoRI
et le fragment HindllI-Ban7HI dans un dérivé du vecteur pUCl9 (HindIII) digéré par
- BamHI et EcoRl. Le dérivé est obtenu après digestion de pUCl9 par HindIII et
traitement à la polymérase de T4. Le vecteur obtenu est désigné pSV40EI.
Un fragment d'ADN comportant la séquence codant pour la nucléase IPpo est isolé
sous forme d'un fragment PCR. La séquence reportée dans l'art antérieur (Muscarella
et al., 1990, Mol. Cell. Biol, 10, 3386-3396) a permis de définir deux amorces, qui sont
respectivement décrites dans les SEQ ID NO: 7 et 8. Les 2 oligonucléotides
wo 9~/24300 21 6 ~ 6 ~ 7 PCT~Rg~/00419
comportent un site ~i~7dIII à leur extrémité 5'. L'amplification est effectuée dans les
conditions standards connues de l'homme de métier à partir d'une banque d'ADN
génomique de Phy.sar~/m p~lycepJ?ah~m, souche Carolina.
5 Le fragment PCR généré, après vérification sur gel d'agarose et sequençage, est digéré
par Hi~tdIlI et introduit dans le site unique Hi~ldIII du vecteur pSV40EI. On identifie
un clone présentant une orientation correcte de la séquence codant pour IPpo vis à vis
du promoteur SV40. Le clone est désigné pSV40EII.
0 2. Clona~e de la cassette d'expression de l'endonucléase IPpo dans le vecteur pHREII.
Le vecteur pHREII est digéré par BglI] et traité à la polymérase de T4. Le vecteur
pSV40EII est digéré par les enzymes PstI et Bg/II. Le fragment PstI-Bg~I comportant
la cassette d'expression de IPpo, est traité à la nucléase de haricot Mung. Puis on ligue
5 le fragment ainsi traité au vecteur pHREII donnant lieu à p~EIII.
3. Introduction de la cassette d'expression d'un fra~ment d'ADN d'intérêt dans
l'intron 3.
20 La première cassette d'expression du fragment d'ADN d'intérêt comprend en séquence
de 5' vers 3' respectivement:
(1) le promoteur TK du virus HSV,
(2) le gène néomycine, et
(3) la région polyA du virus SV40.
Le fragment XhoI-SalI de l,lkb comprenant ladite cassette d'e,.pres~ion est isolé du
plasmide pMCI neo (Stratagène), est cloné dans le vecteur p~EIII linéarisé par X~7oI.
Le vecteur en résultant est désigné pHRE IV.
30 4. Clonage dans un vecteur adénoviral et infection de cellules hôtes.
On isole du vecteur pHRElV le fragment NheI comprenant l'ensemble de transposition
(intron 3 dans lequel est introduit la première cassette d'e~p.ession neo, flanqué des
séquences adjacentes 5' et 3' du gène ARN 28S humain) et la deuxième c~sette
35 d'expression de IPpo. Le fragment purifié est traité à la polymérase de T4 avant d'etre
inséré dans le vecteur pXCX2 (Spessot et al., 1989, Virology, 168, 378-387).
Le vecteur en résultant est utilisé pour générer un adénovirus recombinant par les
méthodes conventionnelles, comme par exemple, la méthode reportée dans Spessot et
WO 9~/24300 PCT/FR94/00419
19 2160~97
al. (1989, Virology, IC8, 378-387). Le vecteur adénoviral recombinant est construit à
partir d'un mutant de délétion de l'adénovirus de type 5 (Thimmappaye et al. 1982,
Cell, 31, 543-551). Le vecteur adenoviral recombinant ainsi obtenu est utilisé pour
infecter des cellules humaines en culture.
s
On cultive les cellules dans les conditions conventionnelles connues de l'homme de l'art.
48 heures après l'infection, les cellules sont placées dans un milieu de culture contenant
de la néomycine.
0 La présence du gène neo dans les cellules peut être contrôlée par leur phénotype de
résistance à la néomycine et vérifiée par Southern blot. L'insertion site-spécifique de la
cassette d'intégration dans le génome cellulaire peut être vérifiée par PCR. On utilisera
de préférence, une amorce complémentaire à une séquence du gène ARNr 28S humain
non présente dans l'ensemble de transposition (en 5' de la position 2324 ou en 3' de la
15 position 4438) et une amorce complémentaire à une séquence de la cassette
d'intégration normalement non présente dans le génome cellulaire (par exemple del'intron 3 ou de la cassette d'expression du gène neo). On obtiendra un fragment PCR
d'une taille définie uniquement en cas d'intégration site-spécifique de la cassette
d'intégration dans le gène ARNr 28S humain.
EXEMPLE 3: Construction d'un vecteur plasmidique (épisomal) réplicatif pour
l'inté~ration du gène néomycine dans le génome humain.
Le fragment Nhel de l'exemple 2.4 est inséré dans le vecteur pREP4 (Grager et al.,
25 1989, Gene, 81, 385-294; Invitrogen Corporation, British Bio-Technology Products
LTD, Oxon, UK; reference V 004-50) au niveau du site unique NheI.
Le vecteur en résultant est transfecté dans une lignée de cellules humaines en culture
comme indiqué dans Yates et al. (1985, Nature, 313, 812-815). A titre indicatif, on
30 mentionne la lignée 293, lignee de rein embryonnaire humain, disponible à l'ATCC. 48
heures après la transfection les cellules sont placées dans un milieu de culture sélectif.
On vérifie la présence du gène neo intégré dans le génome cellulaire comme décrit dans
I'exemple 2.4.
W 094/24300 PCT~R94/00419
216~697 ~
o
LISTE DE SEQUENCES
(1) INFORMATION GENERALE:
(i) DEPOSANT:
(A) NOM: Transgene S.A.
(B) RUE: 11 rue de Molsheim
(C) VILLE: Strasbourg
(E) PAYS: France
(F) CODE POSTAL: 67082
(G) TELEPHONE: (33) 88 27 91 00
(H) TELECOPIE: (33) 88 22 58 07
(ii) TITRE DE L' INVENTION: Ensem~le de transposition destine au
transfert d'un gene d'interet dans le genome d'une cellule
eucaryote.
(iii) NOMBRE DE SEQUENCES: 8
(iv) FORME LISIBLE PAR ORDINATEUR:
(A) TYPE DE SUPPORT: Tape
(B) ORDINATEUR: IBM PC compatible
(C) SYSTEME D' EXPLOITATION: PC-DOS/MS-DOS
(D) LOGICIEL: PatentIn Release #1.0, Version #1.25 (OEB)
(2) INFORMATION POUR LA SEQ ID NO: 1:
(i) CARACTERISTIQUES DE LA SEQUENCE:
(A) LONGUEUR: 44 paires de bases
(B) TYPE: acide nucl~ique
(C) NOMBRE DE BRINS: simple
(D) CONFIGURATION: linéaire
(ii) TYPE DE MOLECULE: ADN (génomique)
(iii) HYPOTHETIQUE: NON
(iii) ANTI-SENS: NON
(vi) ORIGINE:
(A) ORGANISME: oligonucleotide de synthese
(xi) DESCRIPTION DE LA SEQUENCE: SEQ ID NO: 1:
GGGAAAAGCT TGCTAGCGGA TCTTGGTGGT AGTAGCAAAT ATTC 44
(2) INFORMATION POUR LA SEQ ID NO: 2:
(i) CARACTERISTIQUES DE LA SEQUENCE:
(A) LONGUEUR: 42 paires de bases
(B) TYPE: acide nucléique
(C) NOMBRE DE BRINS: simple
(D) CONFIGURATION: linéaire
(ii) TYPE DE MOLECULE: ADN (génomique)
(iii) HYPOTHETIQUE: NON
(iii) ANTI-SENS: OUI
(vi) ORIGINE:
(A) ORGANISME: oligonucleotide de synthese
~ W 09~/2430n _ 21 6 0 G 9 7 PCT~R94/00419
(xi) DESCRIPTION DE LA SEQUENCE: SEQ ID NO: 2:
CCCCAAACTA GTTCTAGAGA GAGTCATAGT TACTCCCGCC GT 42
(2) INFORMATION POUR LA SEQ ID NO: 3:
(i) CARACTERISTIQUES DE LA SEQUENCE:
(A) LONGVEUR: 37 paires de bases
(B) TYPE: acide nucléique
(C) NOMBRE DE BRINS: simple
~ (D) CONFIGURATION: linéaire
(ii) TYPE DE MOLECULE: ADN (génomique)
(iii) HYPOTHETIQUE: NON
(iii) ANTI-SENS: NON
(vi) ORIGINE:
(A) ORGANISME: oligonucleotide de synthese
(xi) DESCRIPTION DE LA SEQUENCE: SEQ ID NO: 3:
CCCCAAACTA GTAAGGTAGC CAAATGCCTC GTCATCT 37
(2) INFORMATION POUR LA SEQ ID NO: 4:
(i) CARACTERISTIQUES DE LA SEQUENCE:
(A) LONGUEUR: 48 paires de bases
(B) TYPE: acide nucléique
(C) NOMBRE DE BRINS: simple
(D) CONFIGURATION: linéaire
(ii) TYPE DE MOLECULE: ADN (génomique)
(iii) HYPOTHETIQUE: NON
(iii) ANTI-SENS: OUI
(vi) ORIGINE:
(A) ORGANISME: oligonucleotide de synthese
(xi) DESCRIPTION DE LA SEQUENCE: SEQ ID NO: 4:
AAAAGGGAAT TCGCTAGCAG ATCTCTGCTT CACAATGATA GGAAGAGC 48
(2) INFORMATION POUR LA SEQ ID NO: 5:
(i) CARACTERISTIQUES DE LA SEQUENCE:
(A) LONGUEUR: 33 paires de bases
(B) TYPE: acide nucléique
(C) NOMBRE DE BRINS: simple
(D) CONFIGURATION: linéaire
(ii) TYPE DE MOLECULE: ADN (génomique)
(iii) HYPOTHETIQUE: NON
(iii) ANTI-SENS: NON
(vi) ORIGINE:
(A) ORGANISME: oligonucleotide de synthese
W 094/24300 , _ 21 ~ 0 6 9 7 PCT~R9~/00419
(xi) DESCRIPTION DE LA SEQUENCE: SEQ ID NO: 5:
TTTGGGGTAC CCACCCCCTT AAATATGGCG CTC 33
(2) INFORMATION POUR LA SEQ ID NO: 6:
(i) CARACTERISTIQUES DE LA SEQUENCE:
(A) LONGUEUR: 31 paires de bases
(B) TYPE: acide nucléique
(C) NOMBRE DE BRINS: simple
(D) CONFIGURATION: lineaire
(ii) TYPE DE MOLECULE: ADN (génomique)
(iii) HYPOTHETIQUE: NON
(iii) ANTI-SENS: OUI
(vi) ORIGINE:
(A) ORGANISME: oligonucleotide de synthese
(xi) DESCRIPTION DE LA SEQUENCE: SEQ ID NO: 6:
TTTGGGGTAC CGCTGATTCC AAACTCGGGT G 31
(2) INFORMATION POUR LA SEQ ID NO: 7:
(i) CARACTERISTIQUES DE LA SEQUENCE:
(A) LONGUEUR: 30 paires de bases
(B) TYPE: acide nucléique
(C) NOMBRE DE BRINS: simple
(D) CONFIGURATION: linéaire
(ii) TYPE DE MOLECULE: ADN (génomique)
(iii) HYPOTHETIQUE: NON
(iii) ANTI-SENS: NON
(vi) ORIGINE:
(A) ORGANISME: oligonucleotide de synthese
(xi) DESCRIPTION DE LA SEQUENCE: SEQ ID NO: 7:
CCCCAAAGCT TAAACAAACC ACCGCATGGA 30
(2) INFORMATION POUR LA SEQ ID NO: 8:
(i) CARACTERISTIQUES DE LA SEQUENCE:
(A) LONGUEUR: 34 paires de bases
(B) TYPE: acide nucléique
(C) NOMBRE DE BRINS: simple
(D) CONFIGURATION: linéaire
(ii) TYPE DE MOLECULE: ADN (génomique)
(iii) HYPOTHETIQUE: NON
(iii) ANTI-SENS: OUI
(vi) ORIGINE:
(A) ORGANISME: oligonucleotide de synthese
2 1 ~ ~ PCT~R94/00419
~ W 094/24300 ~ U ~ 6 9 7
23
(xi) DESCRIPTION DE LA SEQUENCE: SEQ ID NO: 8:
CCCAAAAGCT TCAGTGCTCT GGATGTTAAA ATGG