Note: Descriptions are shown in the official language in which they were submitted.
CA 02204jll 1997-Oj-Oj
WO 96/14409 ~ ~ 1ir~SJ'~,1464
PRODUCTION DE PEPTIDES RECOMBINANTS
ANAL06UES DE PEPTIDES NATURELS HYDROPHOBES
La présente invention se rapporte à des peptides recombinants,
analogues de peptides na~urels, et ayant conservé l'ac~ivité biologique de
5 ces peptides naturels. Ccs peptides peuvent être e.~;primés par différents
types de cellules et leur production est améliorée par rapport à celle du
peptide naturel.
Par peptide on entend toutc substance composée d'un enchaînement
d'acides aminés, c'est-à-dire oligopeptide, polypeptide et protéine.
Les peptides possèdent une grande variétc de propriétés
biologiques, et pour éviter les problèmes notamment de contamination liés
au~; techniques de purification à partir de produits biologiques, on a été
amené à les produire par génie génétique.
Toutefois, la production de peptides par voie recombinante se heurte
à des difficultés liées au système d'expression et à la cellule hote. En effet,
pour faciliter leur récupération, il est souhaitable que les pcptides soient
e,~;crétés à travers la membrane cellulaire, afin de les obtenir dans le
milieu e~;tracellulaire ou, dans le cas de bactérie Gram négatif, dans
l'espace périplasmique. On peut introduire dans une cellulc, cn particulier
~0 une bactérie, un système permcttant l'e.~pression du peptide sous forme
fusionnée a~ec une séquencc d'ancrage mcmbranaire, de manière à
obtenir le produit de fusion lié de manièrc covalen~c, à la surface
mem branaire.
C'est ainsi que dans le cadre de la prcparation de vaccin contre le
VRS, les demandeurs ont mis au point un procédé de technique d'ADN
recombinant permettant de modifier ponctuellemellt par mutagénèse
dirigée, des nucléotides dans Ull gène codant pour une séquence
pol) peptidique, utile notamment pour l'obtention dc vaccins par voie
orale contre le virus respiratoire syncitial (VRS) .
Le ~irus respiratoire s~ncytial (VRS) est 1~ c~use la plus frequente
de maladies respiratoires chez Ic nouveau-nc: bronchopneumopathies
(bronchiolites). I.'O~IS cstimc chaquc anlléc ~0 millions dc cas atleints du
VRS, dont 160 000 dccès dans le monde entier. Il c~iste dcu~; sous groupes du
virus (sous groupes A ct 13).
3~ Le VRS est classc dans la ramille d~s l'aram~ ~;oviridac, genre
pneumovirus comportant un gellomc ARN 11011 segmenté, de polarité
né~ative, codant pour 10 pro~cincs spécifiques.
- ~ -
CA 02204~11 1997-0~-0~
WO 96/14409 PCTtl;~95/01464
Il n'existe pas actuellement de vaccin disponible, contre le VRS. Les
vaccins à virus inactivé se sont montrés inefficaces et ont même parfois
aggravé les infections des nourrissons. Dans les années 60, les tentatives
de vaccination avec le VRS inactivé à la formaline ont conduit à l'échec:
au lieu de conférer une protection lors de la réinfection due au VRS, le
vaccin a eu pour effet d'aggraver la maladie chez l'enfant.
La demande WO 87/0~185 a proposé d'utiliser des protéines
structurales du VRS en vue d'un VaCCill, comme les protéines d'enveloppc
appelées protéine F (protéine de fusion) ou protéine G, une glycoprotéine
de 22 Kd, une protéine de 9,5 Kd, ou la protéine majeure de capside
(protéine N).
1~ demande WO ~9/0~935 décrit les propriétés de protection de la
protéine F entière du VRS, éventuellement modifiée sous forme
monomérique ou désacétylée.
Une série de fragments de la protéine F a été clonée en vue de
rechercher leurs propriétés neutralisantes.
Toutefois les vaccins immunitaires testés ~i ce jour se sont montrés
inefficaces ou ont induit une pathologie pulmonaire ( bronchiolite ou
péribronchite) .
A l'heure actuelle il n'e~;iste pas de traitement de fond des
infections dues au VRS.
Les infeccions au VRS des voies aéricnncs supérieures: le
traitement repose essentiellement sur lcs médications symptomatiques
identiques à celles des autres infcctions virales.
Les infections au VRS des voies aériennes inférieurcs: le traitement
chez les nourrissons repose sur le maintiell d'une hydratation correcte,
I'aspiration des sécrétions et l'admillistration d'o~;ygène si besoin. Un effet
positif a été observé ~vec la ribavirine, nucléotide actif in vitro contre le
VRS.
Afin de faciliter l'administration du VaCCill, il serait souhaitable de
disposer d'un produit ac(if par voie oralc, générallt UllC bonlle immunité,
avec des effets secondaires redui~s.
CA 02204~11 1997-0~-0~
WO 96/14409 PCT/IiRg5/01464
Les demandeurs ont construit un système vecteur original, appelé
encore vecteur navette, qui est fonctionnel dans Escherichia coli et
Staphylococcus ~ylosus: le vecteur renferme un signal séquence de
sécrétion S, et une région Xl~l d'ancrage membranaire d'origine de la
S protéine A du Staphylococcus aureus, avec un site de clonage entre S et XM
permettant d'insérer un ou plusieurs gènes.
Afin de faciliter le passage transmembranaire du peptide,
l'hydrophobicité de la molécule doit dans certains cas être modifiée.
Cependant, ces modifications ne doivent pas altérer les propriétés
10 biologiques, notamment immunogènes du produit.
C'est pourquoi la présente invention a pour objet un procédé de
production d'un peptide recombinant biologiquement actif, analogue d'un
peptide naturel présentant au moins une région hydrophobe, caractérisé
en ce qu'il comprend une étape dans laquelle on introduit dans une cellule
15 une construction d'ADN, codallt pour un peptide dont la séquence d'acides
aminés diffère de la séquence du peptide naturel par au moins une
modification dans ladite région hydrophobe, et comportant des éléments
assurant l'e.~pression et la sécrétion dudit peptide par la cellule,
et en ce que, après culture des cellules, on récupère le peptide et/ou les
2~) cellules portant ledit peptide recombinallt.
De préférence, la séqucnce d'acides amines est modifiée au moins
dans une région différente de la région transmembranairc du peptide
naturel.
La modification devra de préférencc intcrvenir dans une région
25 non essentielle pour l'activité biologique d'intérêt du peptide, qui doit être
maintenue.
Ce procédé met Cll jeu une construction d'ADN recombinallt dans
laquelle une séquence signal de secrétion fonctionnelle est liée à un gène
de structure qui a été altéré afin de modifier la structure qui permette au
30 produit recombinant de traverser la membrane de la cellule hôte, alors
que le produit recombinall~ du gène de structurc d'origine ne l'est pas
~ quant il est lié à la même séquence signal dc secrétion; et les modificatiolls
structurelles du produit recombinant devraient être réalisées par génic
~ génétique en altérant la localisation du produit recombinant c.~;primé dans
35 une cellule hôte.
CA 02204~11 1997-0~-0~
WO 96/14409 PCI'1:~95/01464
Les modifications, selon un aspect de l'invention, visent à modifier
l'hydrophobicité du produit recombinant.
L'invention a donc pour objet un procédé de production de peptide
recombinant dans lequel les modifications structurelles du gène
5 conduisent à un peptide dans lequel au moins un acide aminé hydrophobe
de la séquence du peptide naturel est remplacé par un acide aminé non
hydrophobe. Dans un autre mode de réalisation dans le peptide
recombinant, au moins un acide aminé hydrophobe est délété de la
séquence du peptide naturel.
Avantageusement, I'acide aminé hydrophobe est choisi dans le
groupe suivant: Tryptophane, Phénylalanine, Proline, Valine, Alanine,
Isoleucine, Leucine et Méthionine.
Les modifications structurelles du gène peuvent être faites par
insertion de nucléotides, ou par délétion de nucléotides.
Les constructions dans lesquelles les modifications structurelles du
gène sont faites par substitution de nucléotides par mutagénèse dirigée
sont également comprises dans l'invention.
Les modifications structurelles du gène pourront changer la
localisation de telle manière que le produit recombinant soit e,~;posé à la
surface membranaire de la cellule par une liaison covalente à la partie
d'ancrage membranaire.
Dans un autre mode de mise en oeuvre, les modifications
structurelles du gène peuvent changer la localisation de telle manière que
le produit recombinant soit sécrété dans le milieu de culture.
L'invention a donc également pour objet la construction d'ADN qui
comprend une séquence signal de sécrétion liée de manière
opérationnelle à la séquence d'ADN codant pour le peptide recombinant, et
assurant la translocation dudit peptide et sa sécrétion e~;tracellulaire.
Selon un autre aspect, I'invention se rapporte à un procédé
caractérisé en ce que la construction d'ADN comprcnd une séquence
signal liée de manière opérationnelle à la séquence d'ADN codant pour
ledit peptide et permettant la translocation du peptide ~i travers la
membrane de la cellule hôte et son ancrage membranaire.
CA 02204~11 1997-0~-0~
WO 96/14409 PCT/FRg5/01464
Des peptides recombinants susceptibles d'être obtenus par le
procédé caractérisés en ce qu'ils diffèrent du peptide naturel par au moins
une modification dans la région hydrophobe du peptide naturel sont un
autre des objets de l'invention. Ils peuvent se présenter ancrés à 1~
5 surface de la cellule hôte. Ces peptides seront notammcnt choisis parmi les
analogues d'une protéine de structure du VRS ou d'un fragment d'une telle
protéine; plus particulièrement, le peptide recombinant comprend une
séquence analogue de la protéine G du VRS, sous groupc A ou B, notamment
comprise entre les résidus 130 et 230 de la protéine G du VRS en présentant
10 au moins 80 % d'homologie.
La protéine G est une glycoprotéine d'enveloppe du VRS, de poids
moléculaire compris entre 81 et 90 Kd, pauvre en méthionine. La séquence
de la protéine G diffère pour les sous-groupes A et B du VRS; les termes
~séquence de la protéine G" lorsqu'ils sont employés dans la présente
15 demande, doivent s'entendre comme se référant à la fois à la séquence du
sous-groupe A ou du sous-groupe B, lorsque cela n'est pas précisé
différemment.
La Demanderesse a mis en évidence que la séquence comprise entre
les acides aminés 130 et 230 de la protéine G naturellc est particulièrement
20 appropriée pour induire une protcction efricacc contrc l'infection par le
VRS, sous-groupes A et 13, sans induire les pathologies observées avec les
vaccins basés sur le virus cntier inactivé par Ic formol, ou observécs avec
les protéines F et G entières.
Les moyens permettant l'expression du polypeptide sont COIlllUS de
2S l'homme du métier et sont adaplés en fonction de la bactérie utilisée.
De préférence, la séquence d'ADI~ esl introduite sous forme d'un
plasmide, tel qu'un plasmide navette.
Les protéines du VRS ont été à ce jour c~;primées d~ns différents
systèmes d'expression comme le virus de la vaccine, les baculovirus ou les
30 adénovirus. Cependan~, des problèmes potcntiels SOIIt associés à la
présence de particules viralcs résiduelles.
CA 02204~11 1997-0~-0~
WO g6/14409 PCT/I;R95/01464
Dans un de ses modes de mise en oeuvre, le procédé selon la présente
invention utilise une bactérie commensale de l'homme, apathogène et
comestible. En particulier, la bactérie peut appartenir au genre
Staphylococcus. Staphylococcus ~;ylosus qui esI une bactérie utilisée dans
I'industrie alimentaire depuis de nombreuses années, et peut être
administrée, vivante, par voie orale. Des systèmes d'e~;pression d'épitopes
hétérologues à la surface de S. xylosus ont été notamment décrits par
N'guyen et al dans Gene, 1993, 12~: 89-9~.
De préférence, le polypeptide hétérologue est exprimé à la surface
de la membrane de la bactérie, sous une conformation essentiellement
identique à celle de l'épitope correspondant de la protéine G naturelle.
La présentation de la protéine recombinante à la surface
membranaire de la bactérie dépend de sa nature chimique et de son
enchaînement peptidique.
On peut u~iliser la séquence naturelle de la protéine G du VRS et
introduire une séquence d'ADN codant pour un peptide comportant la
séquence ID n~ 1 ou la séquence ID n~ 2.
Selon un aspect de l'invelltion, dans la séquence correspondant à la
séquence comprise entre les acides aminés 130 et 230 de la protéine G,
I'acide aminé cystéine en positions 173 et/ou 186 a été remplacé par Ull
acide aminé ne forman~ pas de pont disulrure, en particulier la serille.
Une telle mutation fa~orise la rormation du pont disulfure en~re Ies
résidus cystéine restant en positions 176 et 182, qui est critique pour
l'immunogénicilé de la séquence; elle évite la formation de ponts
disulfures désordonnés.
Des peptides utiles pour la mise en oeuvre d'un tel procédé sont ceu~;
comportant notamment l'une des séquences ID n~ 3 ou ID n~ -l.
Selon un autre aspect de l'invelltioll dans la séquence du
polypeptide hétérologue correspolldallt à la protéine G du VRS, les acides
~20 aminés phénylalanine correspolldallI au.~; pOSitiOllS lG3, 165, lG8 et/ou 170
de la séquence de la protéille G sont remplaces par Ull acide~ aminé polaire,
en particulier la sérine
Cette modification peut être associée au.~; mutations precédemmen~
citées. Un tel polypeptide peut notamment présenter la séquence ID n~ 5.
CA 02204jll 1997-Oj-Oj
WO 96/14409 PCT/FR95/01464
Lors de la mise en oeuvre du procédé, la suppression de la région
hydrophobique située en amont de la boucle critique formée par le pont
disulfure entre les acides aminés cystéine en positions 176 et 182, permet à
la protéine recombinante de mieu.~; traverser la membrane bactérienne et
5 d'e~cposer correctement la partie immunodominante à la surface
membranaire.
Selon encore un autre aspect de l'invention, dans la séquence
peptidique correspondant à la protéine G du VRS, la séquence comprise
entre les acides aminés numérotés 162 et 170 est délétée.
Plus particulièrement la séquence du peptide hétérologue exprimée
dans la bactérie peut comporter la séquence ID n~ 6.
L'invention comprend également une bactérie e~primant un
peptide ou une protéine, susceptibles d'être obtenus par le procédé décrit
dans la présente demande. Ladite bactérie peut être utilisée vivante ou
tuée.
Les polypeptides ou les bactéries présentant une ou plusieurs des
caractéristiques ci-dessus sont utiles à titre de médicament.
L'invention comprend des compositions pharmaceutiques,
caractérisées en ce qu'elles comprennent un polypeptide ou unc bactérie
selon l'invention en mélange avec des adjuvants pharmaceu~iquement
acceptables
Le vaccin oral à base de vecteur vi~ant doit comprcndre la protéine
modifiée qui présente la conformation optimale pour induire la mcilleure
protection contre le VRS.
C'est pourquoi la présente invention à également pour objet une
application d'une telle composition pharmaccutiquc à la préparation d'un
vaccin par voie orale destiné à prévenir Ics infcctiolls provoquées par le
virus respiratoire syncytial.
Enfin, I'invention a pour objet les séquences nucléotidiques codant
pour un peptide recombinallt analogue d'un peplide naturel cel que décrit
précédemment; ccs séquenccs peuvent comportcr en ou~re des élémen~s
assurant l'e:;pressioll du pcptidc dans unc ou plusicurs cellules hotes
spécifiques. Ces éléments permettcnt de cibler les cellules dans lesquelles
CA 02204~11 1997-0~-0~
WO 96/14409 PCT/FR95/01464
la construction va s'exprimer lors de son administration à un mammifère,
humain ou animal. Il peut s'agir de constructions d'ADN ou d'ARN, qui
seront de préférence incorporées dans un vecteur. Des vecteurs
a~ o~liés sont notamment des plasmides ou des virus du ~ype Adenovirus,
5 qui pourront être formulés dans des compositions pharmaceutiques avec
des excipients acceptables.
Selon l'un de ses aspects, I'invention a pour objet des séquences
nucléotidiques codant pour un polypeptide porté par une séquence
peptidique comprise entre les résidus aminoacides 130 et 230 de la protéine
10 G du virus respiratoire ou pour un polypeptide présentant au moins 80 %
d'homologie avec ladite séquence peptidique, et comportant en outre les
moyens permettant l'expression du polypeptide à la surface de la
membrane d'une bactérie non pathogène du genre Staphylococcus.
Une séquence d'ADN qui comprend
1~ - une séquence signal de sécrétion fonctionnelle,
- une séquence d'ADN codant pour un peptide recombinant
analogue d'un peptide naturel, la séquence de peptide
recombinant présentant au moins une modification dans la région
hydrophobe non trallsmembranaire du peptide naturel,
2~) fait partie de l'invention.
Le procédé selon l'invention comprend les étapes suivantes:
a. transformer les cellules hôtes avec une constructioll d'ADN
recombinant renfermant une séquence signal qui est liée dc manière
opérationnelle à un gène structurel, et que ce dernier soit modifié de telle
~5 sorte que le produit recombinant peut être translocaté à travers la
membrane de la cellule hôte;
b. fermenter ladite cellule hôte pour e:~primer le produit
recombinant;
c. récupérer les protéines extracellulaires, sécrétées par les cellules
30 transformées par les constructions.
CA 02204~11 1997-0~-05
WO 96/14409 PCI~/FR95/01464
L'invention comprend également une cellule recombinante qui
contient une séquence d'ADN ou une construction telle que définie
précédemment
Cette cellule hôte peut être une bactérie, Gram+ ou Gram-, une
5 cellule de levure ou une cellule de m~mmifère.
Des bactéries particulièrement adaptées sont choisies dans le groupe
comprenant Escherichia coli, Staphylococcus ,~ylosus, Staphylococcus
carnosus.
La séquence d'ADN peut être intégrée dans le chromosome de la
10 bactérie, Gram positif ou Gram négatif
Cette construction d'ADN recombinant peut renfermer le gène
codant pour la protéine G de l'amino-acide 130 à 230 du VRS humain sous-
groupe A fusionnée en amont et/ou en aval de celle du sous-groupe B.
Un type de construction peut être réalisé avec le gène codant pour
la protéine de l'amino-acide 130 à 230 du VRS bovin appartenant au sous-
groupe A, ou sous sous-groupe B.
Les exemples qui suivent sont destinés à illustrer l'invention sans
aucunement en limiter la portée~
Dans ces e~;emples, on se référera au~ figures suivantes:
- Figure 1: Construction par assemblage de gènes du plasmide pRlT28G2av;
- Figure 2: 1) Construction du gène G2 substitué par des résidus serine;
2) Construction du gène G2 délété des résidus 162 à 170;
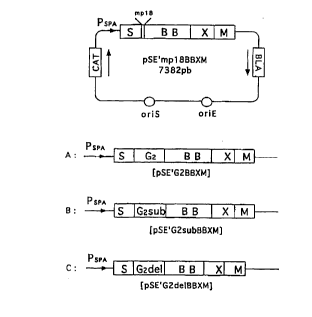
- Figure 3: Schéma des constructions de vecteurs navettes, en haut
25 pSE'mlpl8BBXM, en (A) vecteur pSE'G2BBXM; (B) vecteur pSE'G2subBBXM;
et (C) vecteur pSE'G2delBBXM;
- Figure 4 :Analyse par cytométrie de flu~ des protéines recombinantes de
surface;
- Figure 5: Analyse par SDS-Page des protéines e~:traites de la membrane
30 de S. Xylosus recombinant;
- Figure 6: Schéma du principe de construction dcs vecteurs de sécrétion à
partir des vecteurs navcttes respectifs pSE'G2subBBXI~1 e~ pSE'G2delBBXl~I
dont les descriptions sont détaillées dans l'e.~;emple ~. Sont illustrés les
produits sécrétés à partir du milieu de culture dc S. .~;ylosus portant des
35 vecteurs dont le codon Stop (T) a été inséré en amont de la région
d'ancrage membranaire Xl~
CA 02204~11 1997-0~-05
WO 96tl4409 P~l/r~!i5lol464
- Figure 7: Analyse par SDS-Page et immunoblot des protéines de fusion
secrétées par S. ~losus;
- Figure 8: Analyse par cytométrie de ~flux des protéines secrétées par S.
xylosus portant différents vecteurs navettes.
s
EXEMP~l~S
E~EMPLE 1:
I) Construction de G2 Par assembla~e de gènes svnthétiaues:
Le gène codant pour la région en amino-acides 130-230 de la
glycoprotéine G du virus RS, nommé G2, où nous avons en plus changé
deux résidus Cys en position 173 et 186 en Ser par rapport à la séquence
d'origine, est obtenu par les techniques d'assemblage de gènes en phase
solide (Stahl S. et coL,1993. BioTechniques, 14:12~ 431). La séquence des
oligonucléotides a été optimisée en combinant les codons usuels de
bactéries telles que Ecoli et Staphylococcus.
Les oligonucléotides ont été synthétisés par la chimie des
phosphoramidites sur le s~nthétiseur d'ADN automatique (Gene Assembler
Plus, Pharmacia Biotech) selon les recommandations du constructeur. Les
oligonucléotides à fixer en phase solide sont biotin)~lés en extrémité 5' par
le réactif Biotin-on phosphoramidite (Clontech). Les autres
oligonucléotides sont phosphorylés en 5' par le réactif Phosphate-on
amidite (Clontech) selon le protocole de Clontech. Les oligonucléotides sont
déprotégés et SOIIt purifiés selon les recommandations de Pharmacia. Les
oligonucléotides biotinylés sont purifiés par chromatographie liquide sur
phase réverse (colonne PEP RPC, Pharmacia).
Le gène est assemblé en deux parties: G2am (amont) de l'aa 130 à
177 avec deux sites de restriction BamHI et Pstl en 5' et 3' du gène, G2av
(aval) de l'aa 177 à 230 avec deux sites de restriction Pstl et Baml{l en 5' et
en 3' du gène. Le gène G2 est reconstitué en ligaturant les deu~: fragments
G2am et G2av par le site Pstl.
a) Assembla~e de ~ène de ('.2am (Fl(.URI~ l):
Dans un micro tube, deu\ oligonucléotides complémentaires dont
l'un est biotinylé en 5': T~11 B = 5'-Biotin-CCGGATCCT ~TGACCGTGA ~-3'
-
CA 02204~11 1997-0~-05
WO 96tl4409 rCI~/FRg~i/01464
et TH2 = S '-G l l l l l GGTT TTCACGGTCA TAGGATCCGG-3 ' sont hybridés et
immobilisés sur des billes magnétiques couplées à la streptavidine (Dynal,
Oslo, Norway). Le double brin immobilisé comprend un site de restriction
BamHI et le brin complémentaire à celui qui est biotinylé possède 6 à 15
nucléotides de dépassement de son côté 5' (phosphorylé) permettant à
l'oligonucléotide suivant de venir s'hybrider. L'hybridation de ce dernier
oligonucléotide est effectuée en élevant la température du milieu à 70 C
pour éviter la formation de structures secondaires. La ligature est faite en
ajoutant de la T4 D~IA ligasc (Gibco BRL). Ainsi le gène est construit
successivement en prenant la précaution à chaque cycle de rincer le
support solide afin d'élimincr l'e~icès d'oli~onucléotides non liés avant
d'ajouter l'oligonucléotide suivant. Le dernier double brin ligaturé
contient un site de restriction Pstl.
Le double brin est ensuite libéré de son support solide en le digérant
avec les enzymes de restrictions BamHI et Pstl puis ligaturé dans le
vecteur de clonage et de séquençage prit28 (Hultman et col., 1988,
Nucleosides Nucleotides 7:629-638~ digéré par les memes enzymes: le
vecteur résultant est pRlT28G2am de taille 30G7pb. La séquence
nucléotique de G2am est déterminée par séquencage d'ADN sur le
séquenceur automatique ABI, selon les recomandations du constructeur
(Applied Biosystem).
b) Assembla~e de ~eène de G7av (FIGURE 1):
De la même manière lc gène G2av est asscmblé en phase solide sur
billes magnétiques par les deu~; premiers oligonucléotides
complémentaires dont l'un est biotin~lé en 5': TH13B =
(5'-Biotin-TGTGAAGCl~ AGTCGACCGG l l-l'G-3') Ct Tl-112 = (S'-GCCGACCACC
AAACCGGTCG ACTAAGCTTC ACA-3'), ainsi de SUitC. Le double brin est libéré
du support solide par digestion enzymatique successivement par Pstl et
Hindlll, cloné dans pRlT28: le vecteur résultant est nommé pRlT28G2av de
~aille 3091pb. La séquence nucléotique de G2av est déterminée par
- séquençage d'ADN sur lc scquenceur automatiquc ABI, selon les
recomandations du constructeur (Applied Biosystem).
CA 02204511 1997-05-05
WO 96/14409 PCT/FR95/01464
c) Construction du ~ène G2 = G2am + G2av:
Les deu~; fragments G2am et G2a~ sont ligaturés par le site de
restriction Pstl et le gène constitué est cloné dans pRlT28 par les sites
BamHI en 5' et ~lindlll en 3': Ic vecteur résultant est nommé pRlT28G2 de
taille 3220pb. La séquence nucléotique de G2 est déterminée par
séquen~age d'ADN sur le séquenceur automatique ABI, selon les
recomandations du constructeur (Applied Biosystem). Le fragment G2 est
digéré par BamHI et Hindlll et cloné dans le vecteur navette: pSE'GZBBXM
(7666 pb) (FIGURE 3)
Liste des oligonucléotides nécessaires pour l'~ssemblage de gènes
G2:
BamHI
THlB (20mer): 5'-Biotin-CCGGATCCT ATGACCGTGA A-3'
BamHI
TH2 (30mer): 5'~11111~iGTTTTCACGGTCATAGC,ATCCGG-3'
TH3 (28mer): 5'-AACCAAAAAC ACCACGACCA CCCAGACC-3'
TH I (3 lmer): 5'-Gl~TGCTCGG CTGGGTCTGG GTGGTCGTGG T-3'
TH5 (3 lmer): 5'-CAGCCGAGCA AACCGACCAC CAAACAGCGTC-3'
TH6 (29mer): 5'-CGG 1 11~ 1 I C l'GACGCTGTT TGGTGGTCG-3'
TH7 (29mer): 5'-AGAACAAACC GCCGMCAAA CCGAACAAC-3'
TH8 (3~mer): 5' CITCGAAATG GAAATCGTl G TTCG(~ I I I(; I l'CGG3'
TH9 (27mer): 5'-GATlTCCATTTCGAAGTGTTCAACrl'C-3'
Pst I
lH 10 (33mer): 5'-l'GCTGCAC.AT GCTGCTCGGC ACGMGTTGA ACA-3 '
Pst I
THll (22mer):5'-GTGCCGAGCAGCATCTGCAC.CA-3'
I lindlll
THl2 (33mer): 5'-GCCGACCACC AAACCGGTCG ACl'AAC.Cl IC ACA-3'
CA 02204511 1997-05-OC,
WO 96/14409 PCT/FRg5/01464
Hindlll
TH13B (19mer): 5'-Biothl-CCCTGTGAAG CTTGGTTTG-3'
TH14 (32mer): 5'-CATMACCGC AGACCACCAA ACCGAAAGAA GT-3'
TH15 (32mer): 5'-GTGGTCGGCACTT(_I I ICGGTTTGGTGGTCTG-3'
5 TH16 (3lmer): 5'-AAAACCGACC TTCMAACCA CCMMAAGA T-3'
TH17 (31mer): 5'-CGGTI IATGA I~-I I I I I IGGTGGI I I I~MG-3'
TH18(30mer): 5'-GGGCMAAM ACCACGACCA MCCGACCAA-3'
TH19 (31mer): 5'~TCGGI I-I I I TGGTCG~I I I GGTCGTGGTTT-3'
TH20 (34mer): 5'-GGGCGATCAG CAMCGTATC CCGMCMAA MCC-3'
TH2 1 (33mer): 5'-TTTTGCCCGG I I I I l I GTTC GGGATACGTT TGC-3'
Pst I
TH22(25mer): 5'-ATC TGCAGCMCA ACCCGACCTG CT-3'
Pst I
TH23 (34mer): 5'-TGATCGCCCA GCAGGTCGGG I I~ CTGC AGAT-3'
II) Construction de G2sub par muta~énèse diri~ée:
Le fragment de gène où les quatre résidus Phenylalanine en
position 163, 165, 168 et 170 sont remplacés par des Scrines, est généré par
amplification génique (PCR), à partir du gène G2 inséré dans le vecteur
pRlT28, en utilisant comme couples d'amorces RIT27/TNG73 et
RIT28/TNG72 (voir FIGURE 2.(1)). Les amorccs l NG72 ct TNG73 sont
complémentaires d'unc région de 19 nucléotides rcnfermant trois des
quatres Phénylalanine:
Rit27 : S'-GCTTCCGGCr CGT~ I ~ I I (; I GTG-3 '
Rit28: 5'-MAGGGGGAT GTGCl'GCMG GCG-3'
TNG72 5'- C CAT rcc GM GTG TCC MC I~C GTG CCG AGC AG-3'
TNG73 3 '- GC TTG CTA AGG GTA AGG CTT CAC AGG TTG-5 '
D'une part, I'amorce TNG73 introduit les trois premières mutations
(TTC en TCC) sur le fragment en amont amplifié avec RIT27. D'autre part
I'amorce TNG72 introduit les Irois dernières mu~alions (TTC en TCC) sur Ic
fragment en aval amplific avec RIT28. Cinq cycles de tempcratures (96 c,
15 sec; 50~c, 1 min; 72 c, I min) suivis dc Villgl c~clcs (9G c, lS sec; 60 c,
15 sec; 72 c, 15 sec) Lcs dcu:; fragments amplifiés sont mélangés dans un
CA 02204~11 1997-0~-0~
WO 96/14409 PCT/FR95/01464
11
seul tube dilué dans du tampon de PCR sans amorces et la réaction
d'e~tension est faite en cinq cycles de températures (96 c, 15 sec; 51 c, 30
sec; 72 c, 1 min). Le produit d'extension est dilué a 1/100 d~ns du tampon
PCR contellant les amorces RIT27 et RIT28 et l'amplification génique est
faite en 30 cycles de températures (96 c, 15 sec; 54 c, 15 sec; 72 c, 30 sec).
Le fragment est ensuite digéré avcc les enzymes de restrictions
BamHI/Hindlll et cloné dans le vecteur prit2 8 di~éré par les mêmes
enzymes: pRlT28G2sub. La séquence nucléotiquc de G2Sub est déterminée
par séquencage d'ADN sur l'appareil de séquenceur automatique ABI, selon
les recomandations du constructeur (Applied Biosystem). Le fragment
G2sub est digéré par Baml-ll et Hindlll et cloné dans le vecteur navette:
pSE'G2subBBXM (7666 pb)(FlGURE 3).
111) Construction de G2del: (voir FIGURE 2.(2))
De la même manière, le fragment de gène G2 délété de la partie
renfermant les I résidus Phénylalanine de l'a~ 162 ;i l'aa 170 est généré
par PCR en deu~i fragments: en amont avec les amorces R11 27/TI-1 18 d'une
part et en aval avec RIT28/T1111 d'autre part. Lcs amorces TH11 et TH48
sont complémentaires de 13 nucléotides.
TH11 5'-GTGCCGAGCA GCATC1'GCAG CA-3'
3'-GG( I1~;IIIGGCT~r.lrGCACGGCrCCICGl:5' = n~
Cinq cycles de tempcratures (96 c, 15 scc; 52 c, 1 min; 72 c, 1 min)
suivis de vingt cycles (96 c, 15 sec; 60 c, 15 sec; 72 c, 15 sec) Les deu~;
fragments amplifiés SOllt mclangés dans un seul tubc dilué dans du tampon
de PCR sans amorce et la réaction d'e,~;tension est faite en cinq cvcles de
températures (96 ~ c, 15 sec; ~2 ~ c, 30 sec; 72 ~ c, 1 min). Lc produit
d'e.~tension est dilué à 1/100 dans du tampon PCR contenallt les amorces
RIT27 et RIT28 et l'ampliricatioll géniquc est faite en 30 cvcles de
températures (96 c, 15 sec; 60 c, 15 scc; 72 c, 30 sec). Ie fr~gmcnt est
ensuite digéré avec Ics en~yMcs de rcstrictions l~ml-ll/l-lindlll ct CIOIlC
CA 02204~11 1997-0~-0~
WO 96/14409 PCTtFR95/01464
dans le vecteur prit28 digéré par les memes enzymes: pRlT28G2del. La
séquence nucléotique de G2del est déterminée par séquencage d'ADN sur
I'appareil de séquenceur automatique ABI, selon les recomandations du
constructeur (Applied Biosystem). Le fragment G2del est digéré par Bam~ll
et HindIII et cloné dans le vecteur navette: pSE'G2delBBXM (7639
pb)(FlGURE 3).
IV) Construction du vecteur navette:
Un linker d'oligonucléotides ( 5 '-AGCTTGGCTG TTCCGCCATG
GCTCGAG-3', avec le brin complémentaire) est inséré dans le site Hindlll du
plasmide pSZZmpl8XM (I{ansson et col, 1992, J.Bacteriol 17~: ~239-12~5),
créant ainsi deux sites de restriction supplémentaires Ncol et Xhol en aval
du site Hindlll du vecteur résultant pSZZmpl8(Xhol)XM. Un fragment de
gène codant 198 acides aminés, nommé BB, de la région de fi.~;ation du
sérum albumine de la protéine G streptococcique (Nygren et col, ]988.
J.~lol.Reconig., 1:69-71), est généré par l'CR avec les amorces ( 1 =
5'-CCGAATICAA GCITAGATGC TCrAGCMAA GCCAA~3' et 2 = 5'~CCCrGCAGT
TAGGATCCCT CGAGAGGTAA TGCAGCTAAA Al-l-lCATC-3') sur le template
plasmidique pSPG1 (Guss et col, 1986,E~IBO J., 5: 1567-1575). Le fragment
est digéré avec Hindlll et Xhol et cloné en aval du site multiple de clonage
mpl8 du vecteur pSZZmpl8(Xhol)X~I; le vecteur résultant
pSZZmpl8(Xhol)BBXI~I est digére par Notl el l-lindlll. Le fragment
renfermant ZZ est remplacé p~r Ull autre fragmellt digéré par les mêmes
enzymes de restriction du vec~eur pE'mpl8 (Sophia l-lober, non publié). Le
vecteur navette résultant est nommé pSE'mpl8BBXI\1 (EIGURE 3).
EXEMPLE 2:
Extraction et analvse de protéilles membranaire~s des ~S. .~lo.sus
recombinants:
Dans un erlenme~er de 1 litre, Oll inocule 250 ml de milieu (7,5 g de
TSB, 12,5 g de Yeast e.~;tract) contell;lllt du Chloramphellicol (20 ~g/ml)
~vec 5 ml de préculture (o~ ernight) de S. ~-~losus transformé par le
vecteur navette pSE'G2BBX~1 ou pSE'G2subBBX~1 ou pSE'G2delB13X~I selon le
CA 02204jll 1997-0j-0j
WO 96/14409 PCT/FR95/01464
16
protocole de Gotz et col, 1981, J Bacteriol., 1~5:7 1-81. Incuber sous agitationà température = 32~ C pendant une durée de 6 heures. Centrifuger le milieu
à 5000 rpm, 12 min e~ à température = 1~ C. Le culot bactérien est
resuspendu dans 40 ml de TST, on rajoute 200 IJI de solution contenant de la
S lysostaphine (1 mg/ml) et 200 IJI de Iysozymc (50 mg/ml). Incuber
pendant une heure à température = 37~ C sous agitation modérée. Soniquer
la solution pendant 2 min, avec l'appareil Vibra cell équipé d'une sonde
dont la puissance est réglée à 7. Centriruger à 13 500 rpm, pendant 20 min,
à température = 4~ C. Les protéines sont purifiées par affinité: le
10 surnageant est passé sur colonne d'affinité IlSA-Sépharose (Sérum
Albumine Humaine). Après avoir rincé la colonne, les protéines sont
éluées avec un tampon acide pH 2,7 et Iyophilisées.
Les protéines sont séparées sur deu.~ gels SDS-PAGE (12%) identiques
avec les marqueurs de tailles moléculaires standard (Gibco BRL)
15 précolorés. Un gel est coloré au Bleu de Coomassie. Le deuxième est
transféré sur membrane ProblotT~1 (Applied Biosystem) pour
l'immunoblot avec l'anticorps spécifique anti-G 1 (obtenu à partir du
sérum de lapin immunisé avec le peptide Gl(aal7 1-187) selon les
protocoles courants d'immunisation). Voir Figure 5.
EXEMPLE 3:
Analvse r)ar C~tométrie dc flu~; (FACScallT~I) dcs protéinc~s recombinalltcs
à la surface de S. ~ivlosus:
Les cultures des bactérics rccombinalltcs ~S. xylosus SOllt fai~es
25 comme décrit précédemment. Pour constituer une solution stoc~;,
resuspend la bactérie dans une SOlUIiOIl de PBS ~i 0,1% de Sodium Azide
(p/v) à la concentration finale estimée par densilc oplique (600nm) égale
à l'unité. On aliquote 30 1ll de solution stocl; dans chaque puits cdnique
d'une plaque de microtitrcs, Oll centrifuge à 550 g pendant 10 minutes à ~~
30 C. On resuspend Ic culot bactérien dans un volumc dc 150 IJI de SOlU~iOIl de
PBS contenant du ~scrum lapill polvclollal allti-G_ (~ilrc 1/1 ~t~() 000) diluc
CA 02204~11 1997-0~-05
WO 96/14409 PCTIF'R95/01464
au 1/200ème, incubation pendant 30 minutes. On rince deu~ fois les
cellules bactériennes avec du PBS et on incube dans 150 ~I d'une solution
de PBS renfermant de l'anti-lapin FITC ~Sigma) dilué au 1/lOOème pendant
une durée de 30 minutes. Après avoir rincé les cellules deu~; fois avec du
5 tampon PBS, on resuspend dans un tube Falcon contenant 1 ml de tampon
PBS-Paraformaldéhyde lYo tp/v). Les échantillons préparés sont analysés
sur l'appareil FACScanTM (Becton Dic};inson). La distribution de
fluorescence de chaque suspension cellulaire est analysée par le logiciel
LYSIS IITM et est représentée par des histogrammes de fluorescence. Voir
10 Figure 4.
EXEMPLE 4:
Modulation secrétion-insertion en secrétion:
A partir des différents vecteurs navettes, il est possible d'insérer des
15 codons de terminaison en amont de la région codant pour l'ancrage
membranaire X~l, comme le montre la figure G. Un site unique de
restriction Xhol entre BB et XM a été utilisé pour insérer un double brin
d'oligonucléotides codant pour trois codons terminaison (Ter) dans les deu.~;
sens d'orientation, avec introduction d'un site de restricliOn Aat ll:
Aat ll l'er Tcr Ter -->
5'-TC GAC GTC T~A TGA TAA TT~ TCA TTA G-3'
3'-G CAG ATT ACT Al~ MT AGT AAT CAG CT-5'
<-- Ter Ter Ter
25 Les vecteurs pSE'G2subBBXI~I et pSE'G2delBBX~I SOllt ainsi digérés par Xho I
et sont ligaturés avec le double brin d'oligonucléotides pre~lablement
phosphorylés en 5'. Les vecteurs résultants sont respectivement
pSE'G2subBB[Ter]X~I (7693 pb) et pSE'G2delBB [ler~X~I (766G pb). La culture
des E.coli ou des S.,~ylosus transFormées respectivement avec ces deu.~;
30 vecteurs suivie d'une purirication sur colonnc d'~ffinité (I-ISA Sépharose)
permet d'obtenir des protéillcs G2subB13 e~ G2delBI3. La figure 7 montre: en
CA 02204~11 1997-0~-0~
WO 96/14409 PCT/1i~95/01464
18
A) le gel SDS-PAGE, la séparation des protéines secrétées à partir des
S.~ylosus, dans des conditions réduites, en 1 et en 2 représentent
respectivement les protéines G2subBB et G2delBB à la taille attendue: 35,23
Kda et 34,28 Kda; en B) I'immunoblot des protéines montre que l'anticorps
S spécifique à la région G(aal7~-187) ou Gl du VRS reconnaît bicn les deux
protéines secrétées. Très peu de dégradations protéolytiques ont été
observées. De plus, I'analyse l:ACSCAN avec l'anticorps polyclonal anti-
lapin, anti-BB a été réaliséc sur les différents S..~;,vlosus. 1~ figure 8 montre
que les spectres de S.,~;ylosus portant des vecteurs navettes pSE'mpl8BBXM,
10 pSE'G2subBBXM et pSE'G2delBBXM sont déplacés dans l'axe de l'intensité de
fluorescence vers la droite, c'est-a-dire vers la présence des an~igènes
hétérologues à la surface de la bactérie. Alors que les spectres de S..~losus
portant des vecteurs navettes pSE'G2subBBTerXI~1 et pSE'G2delBB l'erXM n'y
sont pas déplacés, ceci indique que les antigènes hétérologues SOllt absents
15 à la surface de la baclérie et que l'on les a re~rouvés et purifiés à partir du
milieu de culture.
CA 02204511 1997-05-05
WO 96/14409 PCT/FR9~i101464
19
LISTE DE SEQUENCES
(1) INFORMATIONS GENERALES:
(i) DEPOSANT:
(A) NOM: PIERRE FABRE MEDICAMENT
(B) RUE: 45 PLACE ABEL GANCE
(C) VILLE: BOULOGNE
(E) PAYS: FRANCE
(F) CODE POSTAL: 92100
(ii) TITRE DE L' INVENTION: PRODUCTION DE PEPTIDES ANALOGUES DE
PEPTIDES ~YDROPHOBES, PEPTIDE RECOMBINANT, SEQUENCE D'ADN
CORRESPONDANTE
(iii) NOMBRE DE SEQUENCES: 6
(iv) FORME DECHIFFRABLE PAR ORDINATEUR:
(A) TYPE DE SUPPORT: Floppy disk
(B) ORDINATEUR: IBM PC compatible
(C) SYSTEME D' EXPLOITATION: PC-DOS/MS-DOS
(D) LOGICIEL: PatentIn Release #1.0, Version #1.30 (OEB)
(vi) DONNEES DE LA DEMANDE ANTERIEURE:
(A) NUMERO DE LA DEMANDE: FR 94133~7
(B) DATE DE DEPOT: 07-NOV-1994
(2) INFORMATIONS POUR LA SEQ ID NO: 1:
(i) CARACTERISTIQUES DE LA SEQUENCE:
(A) LONGUEUR: 303 paires de bases
(B) TYPE: nucléotide
(C) NOMBRE DE BRINS: simple
(D) CONFIGURATION: linéaire
(ii) TYPE DE MOLECULE: ADN
(ix) CARACTERISTIQUE:
(A) NOM/CLE: CDS
(B) EMPLACEMENT:1..303
CA 02204~11 1997-0~-0~
WO 96114409 PCTIIiR95/01464
(xi) DESCRIPTION DE LA SEQUENCE: SEQ ID NO: 1:
ACC GTG AAA ACC AAA AAC ACC ACG ACC ACC CAG ACC CAG CCG AGC AAA48
Thr Vol Lys Thr Lys Asn Thr Thr Thr Thr Gln Thr Gln Pro Ser Lys
1 5 10 15
CCG ACC ACC AAA CAG CGT CAG AAC AAA CCG CCG AAC AAA CCG AAC AAC96
Pro Thr Thr Lys Gln Arg Gln Asn Lys Pro Pro Asn Lys Pro Asn Asn
20 25 30
GAT TTC CAT TTC GAA GTG TTC AAC TTC GTG CCG TGC AGC ATC TGC AGC144
Asp Phe His Phe Glu Val Phe Asn Phe V~l Pro Cys Ser Ile Cys Ser
35 40 45
AAC AAC CCG ACC TGC TGG GCG ATC TGC AAA CGT ATC CCG AAC AAA AAA192
Asn Asn Pro Thr Cys Trp Ala Ile Cys Lys Arg Ile Pro Asn Lys Lys
50 55 60
CCG GGC AAA AAA ACC ACG ACC AAA CCG ACC AAA AAA CCG ACC TTC AAA240
Pro Gly Lys Lys Thr Thr Thr Lys Pro Thr Lys Lys Pro Thr Phe Lys
65 70 75 80
ACC ACC AAA AAA GAT CAT AAA CCG CAG ACC ACC AAA CCG AAA GAA GTG288
Thr Thr Lys Lys Asp His Lys Pro Gln Thr Thr Lys Pro Lys Glu Vol
85 90 95
CCG ACC ACC AAA CCA 303
Pro Thr Thr Lys Pro
100
(2) INFORMATIONS POUR LA SEQ ID NO: 2:
(i) CARACTERISTIQUES DE LA SEQUENCE:
(A) LONGUEUR: 303 paires de b~ses
(B) TYPE: nucléotide
(C) NOMBRE DE BRINS: simple
(D) CONFIGURATION: liné~ire
(ii) TYPE DE MOLECULE: ADN
(ix) CARACTERISTIQUE:
(A) NOM/CLE: CDS
(B) EMPLACEMENT:1..303
CA 02204~11 1997-0~-0~
WO 96/14409 PCT/FRg5/01464
(xi) DESCRIPTION DE LA SEQUENCE: SEQ ID NO: 2:
ACC GCG CAG ACC AAA GGC CGT ATC ACC ACC AGC ACC CAG ACC AAC AAA48
Thr Ala Gln Thr Lys Gly Arg Ile Thr Thr Ser Thr Gln Thr Asn Lys
1 S 10 15
CCG AGC ACC AAA AGC CGT AGC AAA AAC CCG CCG AAA AAA CCG AAA GAT96
Pro Ser Thr Lys Ser Arg Ser Lys Asn Pro Pro Lys Lys Pro Lys Asp
20 25 30
GAT TAC CAC TTC GAA GTG TTC AAC TTC GTG CCC TGC AGC ATC TGC GGC144
Asp Tyr His Phe Glu Val Phe Asn Phe Val Pro Cys Ser Ile Cys Gly
35 40 45
AAC AAC CAG CTG TGC AAA AGC ATC TGC AAA ACC ATC CCG AGC AAC AAA192
Asn Asn Gln Leu Cys Lys Ser Ile Cys Lys Thr Ile Pro Ser Asn Lys
50 55 60
CCG AAA AAG AAA CCG ACC ATC AAA CCG ACC AAC AAA CCG ACC ACC AAA240
Pro Lys Lys Lys Pro Thr Ile Lys Pro Thr Asn Lys Pro Thr Thr Lys
65 70 75 80
ACC ACC AAC AAA CGT GAT CCG AAA ACC CCG GCG AAA ATG CCG AAG AAG288
Thr Thr Asn Lys Arg Asp Pro Lys Thr Pro Ala Lys Met Pro Lys Lys
85 90 95
GAA ATC ATC ACC AAC 303
Glu Ile Ile Thr Asn
100
C2) INFORMATIONS POUR LA SEQ ID NO: 3:
(i) CARACTERISTIQUES DE LA SEQUENCE:
(A) LONGUEUR: 303 paires de bases
(B) TYPE: nucléotide
(C) NOMBRE DE BRINS: simple
(D) CONFIGURATION: linéaire
(ii) TYPE DE MOLECULE: ADN
(ix) CARACTERISTIQUE:
(A) NOM/CLE: CDS
(B) EMPLACEMENT:1..303
CA 02204~11 1997-0~-0~
WO 96/14409 PCI~/FRgS/01464
(xi) DESCRIPTION DE LA SEQUENCE: SEQ ID NO: 3:
ACC GTG AAA ACC AAA AAC ACC ACG ACC ACC CAG ACC CAG CCG AGC AAA48
Thr Val Lys Thr Lys Asn Thr Thr Thr Thr Gln Thr Gln Pro Ser Lys
1 5 10 15
CCG ACC ACC AAA CAG CGT CAG AAC AAA CCG CCG AAC AAA CCG AAC AAC96
Pro Thr Thr Lys Gln Arg Gln Asn Lys Pro Pro Asn Lys Pro Asn Asn
20 25 30
GAT TTC CAT TTC GAA GTG TTC AAC TTC GTG CCG AGC AGC ATC TGC AGC144
Asp Phe His Phe Glu Val Phe Asn Phe Val Pro Ser Ser Ile Cys Ser
35 40 45
AAC AAC CCG ACC TGC TGG GCG ATC AGC AAA CGT ATC CCG AAC AAA AAA192
Asn Asn Pro Thr Cys Trp Ala Ile Ser Lys Arg Ile Pro Asn Lys Lys
50 55 60
CCG GGC AAA AAA ACC ACG ACC AAA CCG ACC AAA AAA CCG ACC TTC AAAZ40
Pro Gly Lys Lys Thr Thr Thr Lys Pro Thr Lys Lys Pro Thr Phe Lys
65 70 75 80
ACC ACC AAA AAA GAT CAT AAA CCG CAG ACC ACC AAA CCG AAA GAA GTG288
Thr Thr Lys Lys Asp His Lys Pro Gln Thr Thr Lys Pro Lys Glu Val
85 90 95
CCG ACC ACC AAA CCA 303
Pro Thr Thr Lys Pro
100
(2) INFORMATIONS POUR LA SEQ ID NO: 4:
(i) CARACTERISTIQUES DE LA SEQUENCE:
(A) LONGUEUR: 303 paires de bases
(B) TYPE: nucléotide
(C) NOMBRE DE BRINS: simple
(D) CONFIGURATION: linéaire
(ii) TYPE DE MOLECULE: ADN
(ix) CARACTERISTIQUE:
(A) NOM/CLE: CDS
(B) EMPLACEMENT:1..303
CA 02204~11 1997 - 0~ - 05
WO 96/14409 PCTIFRg5/01464
Cxi) DESCRIPTION DE LA SEQUENCE: SEQ ID NO: 4:
ACC GCG CAG ACC AAA GGC CGT ATC ACC ACC AGC ACC CAG ACC AAC AAA48
Thr Ala Gln Thr Lys Gly Arg Ile Thr Thr Ser Thr Gln Thr Asn Lys
1 S 10 15
CCG AGC ACC AAA AGC CGT AGC AAA AAC CCG CCG AAA AAA CCG AAA GAT96
Pro Ser Thr Lys Ser Arg Ser Lys Asn Pro Pro Lys Lys Pro Lys Asp
Z0 25 30
GAT TAC CAC TTC GAA GTG TTC AAC TTC GTG CCC AGC AGC ATC TGC GGC144
Asp Tyr His Phe Glu Val Phe Asn Phe Val Pro Ser Ser Ile Cys Gly
35 40 45
AAC AAC CAG CTG TGC AAA AGC ATC AGC AAA ACC ATC CCG AGC AAC AAA192
Asn Asn Gln Leu Cys Lys Ser Ile Ser Lys Thr Ile Pro Ser Asn Lys
50 55 60
CCG AAA AAG AAA CCG ACC ATC AAA CCG ACC AAC AAA CCG ACC ACC AAA240
Pro Lys Lys Lys Pro Thr Ile Lys Pro Thr Asn Lys Pro Thr Thr Lys
65 70 75 80
ACC ACC AAC AAA CGT GAT CCG AAA ACC CCG GCG AAA ATG CCG AAG AAGZ88
Thr Thr Asn Lys Arg Asp Pro Lys Thr Pro Ala Lys Met Pro Lys Lys
85 90 95
GAA ATC ATC ACC AAC 303
Glu Ile Ile Thr Asn
100
(2) INFORMATIONS POUR LA SEQ ID NO: 5:
(i) CARACTERISTIQUES DE LA SEQUENCE:
(A) LONGUEUR: 303 paires de bases
(B) TYPE: nucléotide
(C) NOMBRE DE BRINS: simple
(D) CONFIGURATION: linéaire
(ii) TYPE DE MOLECULE: ADN
(ix) CARACTERISTIQUE:
(A) NOM/CLE: CDS
(B) EMPLACEMENT:1..303
CA 02204~11 1997-0~-0~
WO 96/14409 PCT/FR95/01464
24
(xi) DESCRIPTION DE LA SEQUENCE: SEQ ID NO: S:
ACC GTG AAA ACC AAA AAC ACC ACG ACC ACC CAG ACC CAG CCG AGC AAA48
Thr Val Lys Thr Lys Asn Thr Thr Thr Thr Gln Thr Gln Pro Ser Lys
1 5 10 15
CCG ACC ACC AAA CAG CGT CAG AAC AAA CCG CCG AAC AAA CCG AAC AAC96
Pro Thr Thr Lys Gln Arg Gln Asn Lys Pro Pro Asn Lys Pro Asn Asn
20 25 30
GAT TCC CAT TCC GAA GTG TCC AAC TCC GTG CCG AGC AGC ATC TGC AGC144
Asp Ser His Ser Glu Val Ser Asn Ser Val Pro Ser Ser Ile Cys Ser
35 40 45
AAC AAC CCG ACC TGC TGG GCG ATC AGC AAA CGT ATC CCG AAC AAA AAA192
Asn Asn Pro Thr Cys Trp Ala Ile Ser Lys Arg Ile Pro Asn Lys Lys
50 55 60
CCG GGC AAA AAA ACC ACG ACC AAA CCG ACC AAA AAA CCG ACC TTC AAA240
Pro Gly Lys Lys Thr Thr Thr Lys Pro Thr Lys Lys Pro Thr Phe Lys
65 70 75 80
ACC ACC AAA AAA GAT CAT AAA CCG CAG ACC ACC AAA CCG AAA GAA GTG288
Thr Thr Lys Lys Asp His Lys Pro Gln Thr Thr Lys Pro Lys Glu Val
85 90 95
CCG ACC ACC AAA CCA 303
Pro Thr Thr Lys Pro
100
(2) INFORMATIONS POUR LA SEQ ID NO: 6:
(i) CARACTERISTIQUES DE LA SEQUENCE:
CA) LONGUEUR: 276 paires de bases
(B) TYPE: nucléotide
(C) NOMBRE DE BRINS: simple
(D) CONFIGURATION: linéaire
(ii) TYPE DE MOLECULE: ADN
(ix) CARACTERISTIQUE:
(A) NOM/CLE: CDS
(B) EMPLACEMENT:1..276
CA 02204~11 1997-0~-0~
WO 96/14409 PCT/FR95/01464
(xi) DESCRIPTION DE LA SEQUENCE: SEQ ID NO: 6:
ACC GTG AAA ACC AAA AAC ACC ACG ACC ACC CAG ACC CAG CCG AGC AAA48
Thr Val Lys Thr Lys Asn Thr Thr Thr Thr Gln Thr Gln Pro Ser Lys
1 5 10 15
CCG ACC ACC AAA CAG CGT CAG AAC AAA CCG CCG AAC AAA CCG AAC AAC96
Pro Thr Thr Lys Gln Arg Gln Asn Lys Pro Pro Asn Lys Pro Asn Asn
20 25 30
GTG CCG AGC AGC ATC TGC AGC AAC AAC CCG ACC TGC TGG GCG ATC AGC144
Val Pro Ser Ser Ile Cys Ser Asn Asn Pro Thr Cys Trp Ala Ile Ser
35 40 45
AAA CGT ATC CCG AAC AAA AAA CCG GGC AAA AAA ACC ACG ACC AAA CCG192
Lys Arg Ile Pro Asn Lys Lys Pro Gly Lys Lys Thr Thr Thr Lys Pro
50 55 60
ACC AAA AAA CCG ACC TTC AAA ACC ACC AAA AAA GAT CAT AAA CCG CAG240
Thr Lys Lys Pro Thr Phe Lys Thr Thr Lys Lys Asp His Lys Pro Gln
65 70 75 80
ACC ACC AAA CCG AAA GAA GTG CCG ACC ACC AAA CCA 276
Thr Thr Lys Pro Lys Glu Val Pro Thr Thr Lys Pro
CA 02204~11 1997-0~-0~
WO 96/14409 PCT/F~g5/01464
26
LEGENDE DES FIC'.URES
Figure 5:
A) Coloration au bleu de Comassie: Gel SDS-Page des proteines de
~sion extraites de membrane bacterienne et purifiees par affinite sur colonne Albumine des
differents constructions:
- Puits HW: Marqueurs de taille moleculaire (en Kda).
- puits 1: S.xylosus [pSE'G2BBXM].
- puits 2: S.xylosus [pSE'G2subBBXM].
- puits 3: S.xylosus [pSE'G2delBBXM].
B) Immunoblot des proteines de fusion extraites de membrane bacterienne e~ purifiees
par affinité sur colonne Albumine des differen~s construc~ions avec anticorps polyclonal
apin an~i-Gl.
- Puits HW: Marqueurs de taille moleculaire precolores (en Kda).
- puits 1: S.xylosus [pSE'G2BBXM~.
- puits 2: S.xylosus [pSE'G2subBBXM].
- puits 3: S.xylosus [pSE'G2delBBXM].
Figure 7:
A) Coloration au bleu de Comassie: Gel SDS-Page des proteines de
fusion secré~ees et purifees par affinité sur colonne Albumine des differentes
constructions:
- puits 1: S.xylosus [pSE'G2delBB] (34,28 Kda).
- puits 2: S.xylosus [pSE'G2subBB] (35,23 Kda).
- puits HW: Marqueurs de taille moleculaire (en Kda).
B) Immunoblot des protéines de fusion secretées et purifees par affinite sur colonne
Alburnine des différentes constructions avec anticorps polyclonal lapin anti-Gl(VRS).
- puits 1: S.xylosus [pSE'G2delBB] (34,28 Kda).
- puits 2: S.xylosus [pSE'G2subBB] (35,23 Kda).
- puits HW: Marqueurs de taille moleculaire précoiores (en Kda).