Note: Descriptions are shown in the official language in which they were submitted.
CA 02347121 2001-04-09
WO 00/22120 PCT/FR99/02465
POLYPEPTIDE (MBP1) CAPABLE D'INTERAGIR AVEC LES MUTANTS ONCO6ENIQUES DE LA
PROTEINE P53
La présente invention concerne le domaine de la biologie et de la régulation
du cycle cellulaire. Plus particulièrement, la présente invention concerne de
nouveaux
polypeptides capables d'interagir spécifiquement avec les formes oncogéniques
de la
protéine p53.
La protéine p53 sauvage intervient dans la régulation du cycle cellulaire et
dans le maintien de l'intégrité du génome de la cellule. Cette protéine, dont
la
fonction principale est d'être un activateur de la transcription de certains
gènes, est
susceptible de bloquer la cellule en phase G 1 du cycle cellulaire lors de
l'apparition
de mutations au cours de la réplication du génome, et d'enclencher un certain
nombre
de processus de réparation de l'ADN. Ce blocage en phase G 1 est dû
principalement à
l'activation du gène p21/WAFI. De plus, en cas de mauvais fonctionnement de
ces
processus de réparation ou en cas d'apparition d'évènements mutationnels trop
nombreux pour être corrigés, cette protéine est capable d'induire le phénomène
de
mort cellulaire programmée, appelé apoptose.
De cette façon, la protéine p53 agit comme un supresseur de tumeur, en
éliminant les cellules anormalement différenciées ou dont le génome a été
endommagé.
La protéine p53 comporte 393 acides aminés, qui définissent 5 domaines
fonctionnels (voir Figure 1 )
- le domaine activateur de la transcription, constitué par les acides aminés 1
à
73, capable de lier certains facteurs de la machinerie générale de
transcription comme
la protéine TBP. Ce domaine est aussi le siège d'un certain nombre de
modifications
post-traductionnelles. Il est également le siège de nombreuses interactions de
la
protéine p53 avec de nombreuses autres protéines et notamment avec la protéine
cellulaire MDM2 ou la protéine EBNAS du virus d'Epstein-Ban (EBV), capables de
CA 02347121 2001-04-09
WO 00/22120 PCT/FR99/02465
2
bloquer la fonction de la protéine sauvage. De plus, ce domaine possède des
séquences d'acides aminés dites PEST de susceptibilité à la dégradation
protéolytique.
- le domaine de liaison à l'ADN, localisé entre les acides aminés 73 et 315.
La
conformation de ce domaine central de p53 régule la reconnaissance de
séquences
d'ADN spécifiques de la protéine p53. Ce domaine est le siège de deux types
d'altérations affectant la fonction de la protéine sauvage :
(i) l'interaction avec des protéines bloquant la fonction de la protéine
p53 comme l'antigène 'grand T' du virus SV40 ou les protéines virales E6 des
virus
HPV 16 et HPV 18 capables de provoquer sa dégradation par le système de
fubiquitine. Cette dernière interaction ne peut se faire qu'en présence de la
protéine
cellulaire E6ap (enzyme E3 de la cascade de fubiquitinilation).
(ü) les mutations ponctuelles qui affectent la fonction de la protéine
p53 et dont la quasi-totalité observée dans les cancers humains sont
localisées dans
cette région.
- le signal de localisation nucléaire, constitué des acides aminés 315 à 325,
indispensable au bon adressage de la protéine dans le compartiment où elle va
exercer
sa principale fonction.
- le domaine d'oligomérisation, constitué des acides aminés 325 à 355. Cette
région 325 à 355 forme une structure de type : feuillet !3 (326-334)-coude
(335-336}-
hélice a (337-355). Les altérations de fonctions localisées dans cette région
sont
essentiellement dues à l'interaction de la protéine sauvage avec les
différentes formes
mutantes qui peuvent conduire à des effets variables sur la fonction de la
protéine
sauvage.
- le domaine de régulation, constitué des acides aminés 365 à 393, qui est le
siège d'un certain nombre de modifications post-traductionnelles
(glycosylations,
CA 02347121 2001-04-09
WO 00/22120 PCT/FR99/02465
3
phosphorylations, fixation d'ARN,...) qui modulent la fonction de la protéine
p53 de
façon positive ou négative. Ce domaine joue un rôle extrêmement important dans
la
modulation de l'activité de la protéine sauvage.
Le fonctionnement de la protéine p53 peut être perturbé de différentes façons
- par le blocage de sa fonction par un certain nombre de facteurs comme par
exemple l'antigène 'grand T' du virus SV40, la protéine EBNAS du virus
d'Epstein-
Barr, ou la protéine cellulaire MDM2.
- par la déstabilisation de la protéine par augmentation de sa susceptibilité
à la
protéolyse, notamment par interaction avec la protéine E6 des virus du
papillome
humain HPV 16 et HPV 18, qui favorise l'entrée de la p53 dans le cycle
d'ubiquitinilation. Dans ce cas l'interaction entre ces deux protéines ne peut
se faire
que par la fixation préalable d'une protéine cellulaire, la protéine E6ap dont
le site de
fixation est mal connu.
- par des mutations ponctuelles au niveau du gène de Ia protéine p53.
- par délétion d'un ou des deux allèles de p53
Les deux derniers types de modifications sont retrouvés dans environ 50% des
différents types de cancer. A cet égard, les mutations du gène de la proéine
p53
repertoriées dans les cellules cancéreuses touchent une très grande partie du
gène
codant pour cette protéine, et ont pour résultats des modifications variables
du
fonctionnement de cette protéine. On peut cependant noter que ces mutations
sont en
grande majorité localisées dans la partie centrale de la protéine p53 dont on
sait
qu'elle est la région de contact avec les séquences génomiques spécifiques de
la
protéine p53. Ceci explique pourquoi la plupart des mutants de la protéine p53
ont
comme principale caractéristique de ne plus pouvoir se fixer aux séquences
d'ADN
que reconnaît la protéine sauvage et ainsi de ne plus pouvoir exercer leur
rôle de
facteur de transcription.
CA 02347121 2001-04-09
WO 00/22120 PCT/FR99/02465
4
On regroupe actuellement l'ensemble de ces modifications dans deux catégories
- les mutants dits faibles, dont le produit est une protéine non-
fonctionnelle,
qui, dans Ie cas de mutation sur un seul des deux allèles, n'affecte pas le
fonctionnement de la protéine sauvage codée par l'autre allèle. Le principal
représentant de cette catégorie est le mutant H273 spécifique du syndrome
familial de
Li-Fraumeni d'hypersensibilité aux affections cancéreuses.
- les mutants dominant-oncogéniques, dont le produit est une protéine qui a
perdu la capacité de se lier à l'ADN et qui participe activement à la
transformation
néoplasique. Les mutants de cette catégorie ont perdu leur capacité
transactivatrice et
sont plus stables que la protéine sauvage. Ils sont incapables d'inhiber la
transformation des fibroblastes embryonnaires de rat et ils fonctionnent comme
oncogènes en coopérant avec la forme activée de RAS dans la transformation de
fibroblastes embryonnaires de rat (Eliyahu et al, Nature 312 ( 1984) 646 /
Parada et al,
Nature 312 ( 1984) 649). Ce comportement peut être expliqué par deux
mécanismes
différents non exclusifs l'un de l'autre ;
(i) ces mutants génèrent une protéine non-fonctionnelle, qui, dans le
cas de mutation sur un seul des deux allèles et par interaction avec la
protéine
sauvage, est capable de bloquer le fonctionnement de celle-ci par formation
d'oligomères mixtes non-actifs qui ne peuvent plus se fixer aux séquences
d'ADN
spécifiques de la protéine sauvage. Un tel mécanisme est invoqué dans le cas
où l'on
observe la transformation maligne des cellules après transfection des mutants
en
présence de p53 endogène.
(ü) ces mutants peuvent de plus présenter un phénotype "gain de
fonction". Leur expression dans des cellules non tumorigènes n'exprimant pas
de p53
endogène conduit à l'apparition de tumeurs chez la souris athymique (Dittmer
et al,
Nature Genetics 4 { 1993) 42). Ces mutants sont capables d'activer la
trancription des
gènes comme MDR ou PCNA n'ayant pas de séquences consensus reconnues par
CA 02347121 2001-04-09
WO 00/22120 PCT/FR99/02465
p53 ; activation qui se fait probablement par recrutement des facteurs de
transcription
spécifiques des mutants et qui peut participer à l'apparition du phénotype
tumoral
(Chin et a1, Science 255 ( 1992} 459; Deb et al, J. Virol. 66 ( 1992) 6164).
Enfin, il a
été rapporté récemment que ces mutants peuvent perturber l'attachement de
certaines
5 régions de l'ADN (MAR/SAR) au réseau de la matrice nucléaire (Müller et al,
Oncogene 12 ( 1996) 1941 ).
De nombreux partenaires cellulaires ont été décrits pour la protéine p53.
Certains intéragissent aussi bien avec les conformations sauvage et mutées de
la
protéine et d'autres sont spécifiques de l'une ou l'autre des conformations
(Iwabuchi
et al, Proc. Natl. Acad. Sci. USA 91 (1994) 6098). I1 est concevable que
certaines de
ces propriétés 'gain de fonction' puissent être médiées par des partenaires
protéiques
spécifiques des mutants de p53, cependant de tels partenaires n'ont encore
jamais été
identifiés. L'identification de tels partenaires permettrait de nouvelles
approches dans
les thérapies anti-cancéreuses basées sur la modification ou le contrôle de
ces
interactions et sur l'obtention de composés capables d'interférer dans
l'interaction de
ces partenaires protéiques avec les différentes formes de p53. La présente
invention
satisfait ce besoin et apporte en outre d'autres avantages.
Dans le but d'étudier ce phénotype "gain de fonction" susceptible d'impliquer
des interactions protéine-protéine spécifiques de ce type de mutant, le
système
double-hybride a été utilisé pour rechercher des partenaires spécifiques du
mutant
H 175, principal représentant de cette catégorie de mutants. Une banque de
cDNA
d'embryon de souris, fusionnée à la séquence du domaine de transactivation de
GAL4
(TA), a été criblée dans la souche de levure YCM 17 en utilisant comme
protéine
appât le domaine 73-393 du mutant H 175 fusionné au domaine de liaison à l'ADN
de
Gal4 (DB). Ce criblage a permis d'isoler deux cDNA codant pour deux protéines
différentes : la protéine MBP 1 et la Fibuline-2..
Les interactions entre ces deux protéines et le mutant H 175 de la protéine
p53
ont pu être confirmées en cellules mammifères et des effets fonctionnels ont
pu être
CA 02347121 2001-04-09
WO 00/22120 PCT/FR99/02465
6
démontrés, aussi bien sur des propriétés de la forme mutée de la p53 que sur
des
propriétés de la forme sauvage.
La présente invention résulte donc de la mise en évidence par la demanderesse
de nouveaux polypeptides capables d'interagir spécifiquement avec différentes
formes de la protéine p53. Plus précisément la présente invention résulte de
l'identification, l'isolement et la caractérisation d'une nouvelle protéine et
du gène
correspondant, la dite protéine étant caractérisée en ce qu'elle est capable
d'interagir
spécifiquement avec les formes oncogéniques de p53 et avec les mutants H175 et
6281 en particulier. Cette protéine est appelée MBP 1 pour p53 Mutant Binding
Protein. La présente invention résulte également de la mise en évidence qu'une
autre
protéine, la fibuline2, est capable d'interagir spécifiquement avec les formes
oncogéniques de p53 et avec les mutants H I 75 et G28I en particulier.
La présente invention résulte également de la découverte des propriétés
particulières de ces nouveaux partenaires protéiques de p53 qui de manière
inattendue
s'avèrent également être capables de bloquer les effets anti-prolifératifs de
la forme
sauvage de p53.
Ces nouveaux partenaires protéiques de p53 présentent en outre une synergie
d'action très importante avec les mutants oncogéniques de p53, cette synergie
s'exerce aussi bien pour la coopération oncogénique avec la forme activée de
la
protéine Ras que sur l'effet prolifératif des formes mutées de p53.
De plus et indépendamment de toute interaction avec p53, ces polypeptides
présentent un effet positif sur la croissance cellulaire. En outre, un de ces
partenaires,
la protéine MBP1, présente les caractéristiques d'un oncogène immortalisant en
coopérant avec la forme activée de la protéine Ras pour la transformation
cellulaire.
De part la spécificité et les effets synergiques que présentent ces nouveaux
partenaires de p53 vis à vis de certaines formes mutées de p53, ces
polypeptides
constituent une cible thérapeutique de choix pour le traitement des cancers
liés aux
CA 02347121 2001-04-09
WO 00/22120 PCT/FR99/02465
7
mutations de la protéine p53.
En outre, ces polypeptides qui présentent des propriétés oncogéniques
intrinsèques, constituent de nouvelles cibles potentielles pour le traitement
du cancer
en général.
Un premier objet de l'invention concerne donc des polypeptides capables
d'interagir spécifiquement avec les formes oncogéniques de p53. Ces
polypeptides
sont en outre capables de stimuler la croissance cellulaire et de bloquer les
effets
anti-prolifératifs de la forme sauvage de p53.
Selon un premier mode de réalisation, ces polypeptides comprennent tout ou
partie d'une séquence choisie parmi les séquences polypeptidiques SEQ ID
N° 9
(fragment C-terminal de MBP 1 mucine) ou SEQ ID N° I 6 (MBP I mutine)
ou un
dérivé de celles-ci.
Selon un autre mode de réalisation, ces polypeptides comprennent tout ou
partie d'une séquence choisie parmi les séquences polypeptidiques SEQ ID
N°31
(fragment C-terminal MBP I humaine) ou SEQ ID N°22 (MBP I humaine) ou
un
dérivé de celles-ci.
Enfin selon encore un autre mode de réalisation, ces polypeptides
comprennent tout ou partie de la séquence polypeptidique SEQ ID N° 33
(fragment
C-terminal Fibuline-2 mutine) ou un dérivé de celle-ci.
De manière préférée les polypeptides de l'invention sont représentés par la
séquence polypeptidique SEQ ID N°22 ou ses dérivées
Au sens de la présente invention, le terme séquence polypeptidique dérivée
désigne toute séquence polypeptidique différant de la séquence considérée,
obtenue
par une ou plusieurs modifications de nature génétique et/ou chimique, et
possédant
la capacité d'interagir avec les formes mutées oncogéniques de p53. Par
modification
CA 02347121 2001-04-09
WO 00/22120 PCT/FR99/02465
de nature génétique et/ou chimique, on peut entendre toute mutation,
substitution,
délétion, addition et/ou modification d'un ou plusieurs résidus. De tels
dérivés
peuvent être générés dans des buts différents, tels que notamment celui de
modifier
leurs propriétés de liaison au formes mutées oncogéniques de p53, ou
d'augmenter
leur éfficacité thérapeutique ou de réduire leurs effets secondaires, ou celui
de leur
conférer de nouvelles propriétés pharmacocinétiques et/ou biologiques.
A cet égard, un autre objet de l'invention concerne les séquences
polypeptidiques qui présentent des fonctions biologiques comparables à celles
des
polypeptides selon l'invention et notamment la capacité à interagir avec les
formes
mutées oncogéniques de p53 et qui présentent un degré d'identité d'au moins 80
et de préférence au moins 90 % avec la séquence polypeptidique SEQ ID
N° 16 ou la
séquence polypeptidique SEQ ID N°22 ou la séquence polypeptidique SEQ
ID N°33.
De préférence, les séquences polypeptidiques selon l'invention présentent au
moins 95 % et de préférence encore au moins 97 % d'identité avec la séquence
polypeptidique SEQ ID N° 16 ou la séquence polypeptidique SEQ ID
N°22 ou la
séquence polypeptidique SEQ ID N°33.
De manière plus particulièrement préférée, les séquences polypeptidiques
selon l'invention présentent au moins 98 % d'identité et de préférence encore
au
moins 99 % d'identité avec la séquence polypeptidique SEQ ID N° 16 ou
la
séquence polypeptidique SEQ ID N°22 ou Ia séquence polypeptidique SEQ
ID N°33.
Le terme séquence polypeptidique dérivée comprend également les fragments
des séquences polypeptidiques indiquées ci-dessus. De tels fragments peuvent
être
générés de différentes façons. En particulier, ils peuvent être synthétisés
par voie
chimique, sur la base des séquences données dans la présente demande, en
utilisant
les synthétiseurs peptidiques connus de (homme du métier. Ils peuvent
également
être synthétisés par voie génétique, par expression dans un hôte cellulaire
d'une
séquence nucléotidique codant pour le peptide recherché. Dans ce cas, la
séquence
CA 02347121 2001-04-09
WO 00/22120 PCT/FR99/02465
9
nucléotidique peut être préparée chimiquement en utilisant un synthétiseur
d'oligonucléotides, sur la base de la séquence peptidique donnée dans la
présente
demande et du code génétique. La séquence nucléotidique peut également être
préparée à partir des séquences données dans la présente demande, par coupures
enzymatiques, ligature, clonage, etc, selon les techniques connues de l'homme
du
métier, ou par criblage de banques d'ADN avec des sondes élaborées à partir de
ces
séquences.
Un autre objet de la présente invention concerne les séquences nucléotidiques
SEQ ID N° 15, SEQ ID N°21 et SEQ ID N°32 codant
respectivement pour les
séquences polypeptidiques présentées dans les séquences SEQ ID N° 16,
ou SEQ ID
N°22 ou SEQ ID N°33.
Selon un mode particulier de l'invention, les séquences nucléotidiques
comprennent tout ou partie de la séquence SEQ ID N° 15 ou SEQ ID
N° 21 ou de
leurs dérivées.
Selon un autre mode de l'invention, les séquences nucléotidiques
comprennent tout ou partie de la séquence nucléotidique SEQ ID N° 32
(cDNA
correspondant au fragment C-term de fibuline-2 murine) ou de ses dérivées.
Selon encore un autre mode, les séquences nucléotidiques comprennent la
séquence SEQ ID N°23(cDNA MBPI murine , séquence partielle), ou la
séquence
SEQ ID N°30 (cDNA correspondant au fragment C-term de MBP1
humaine).
Selon un mode préféré, la séquence nucléotidique est représentée par la
séquence SEQ ID N° 21 ou ses dérivées.
Au sens de la présente invention, le terme séquence nucléotidique dérivée
désigne toute séquence différant de la séquence considérée en raison de la
dégénérescence du code génétique, obtenue par une ou plusieurs modifications
de
CA 02347121 2001-04-09
WO 00/22120 PCT/FR99/02465
nature génétique et/ou chimique, ainsi que toute séquence hybridant avec ces
séquences ou des fragments de celles-ci et codant pour un polypeptide selon
l'invention. Par modification de nature génétique et/ou chimique, on peut
entendre
toute mutation, substitution, délétion, addition et/ou modification d'un ou
plusieurs
5 résidus.
Le terme séquence nucléotidique dérivée comprend également les séquences
homologues à la séquence considérée, issues d'autres sources cellulaires et
notamment de cellules d'origine humaine, ou d'autres organismes.
A cet égard la présente invention concerne toute séquence nucléotidique qui
10 présente au moins 70 % d'identité et de préférence au moins 85 % d'identité
avec la
séquence nucléotidique SEQ ID N°21 ou la séquence nucléotidique SED ID
N° 15 ou
la séquence nucléotidique SEQ ID N°32.
De préférence, la séquence nucléotidique selon l'invention présente au moins
90 % et de préférence encore au moins 93 % d'identité avec la séquence
nucléotidique SEQ ID N°21 ou la séquence nucléotidique SED ID N°
15 ou la
séquence nucléotidique SEQ ID N°32.
De manière plus particulièrement préférée, les séquences selon l'invention
présentent au moins 95 % et de préférence encore 97 %, voire 98 % ou même 99
d'identité avec la séquence nucléotidique SEQ ID N°21 ou Ia séquence
nucléotidique
SED ID N° 15 ou la séquence nucléotidique SEQ ID N°32.
De telles séquences homologues peuvent être obtenues par des expériences
d',hybridation. Les hybridations peuvent être réalisées à partir de banques
d'acides
nucléiques, en utilisant comme sonde, la séquence native ou un fragment de
celle-ci,
dans des conditions variables d'hybridation.
CA 02347121 2001-04-09
WO 00/22I20 PCT/FR99/02465
11
Un autre objet de l'invention concerne des séquences nucléotidiques capables
de s'hybrider dans des conditions de stringence élevée avec les séquences
nuciéotidiques définies ci-avant.
A cet égard, le terme condition de stringence élevée signifie que
l'hybridation
se produit si les séquences nucléotidiques présentent au moins 95 % et
préférentiellement au moins 97 % d'identité.
Comme indiqué ci-avant, de telles séquences peuvent être notamment
utilisées comme sondes de détection avec du RNA ou du cDNA ou du DNA
génomique pour isoler des séquences nucléotidiques codant pour des
polypeptides
selon l'invention. De telles sondes ont généralement au moins 15 bases. De
préférence, ces sondes font au moins 30 bases et peuvent avoir plus de 50
bases. De
manière préférée, ces sondes ont entre 30 et 50 bases.
Les séquences nucléotidiques selon l'invention peuvent être d'origine
artificielle ou non. Il peut s'agir de séquences génomiques, d'ADNc, d'ARN, de
séquences hybrides ou de séquences synthétiques ou semi-synthétiques. Ces
séquences peuvent être obtenues par exemple par criblage de banques d'ADN
(banque d'ADNc, banque d'ADN génomique) au moyen de sondes élaborées sur la
base de séquences présentées ci-avant. De telles banques peuvent être
préparées â
partir de cellules de différentes origine par des techniques classiques de
biologie
moléculaire connues de l'homme du métier. Les séquences nucIéotidiques de
l'invention peuvent également être préparées par synthèse chimique ou encore
par
des méthodes mixtes incluant la modification chimique ou enzymatique de
séquences obtenues par criblage de banques. D'une manière générale les acides
nucléiques de l'invention peuvent être préparés selon toute technique connue
de
l'homme du métier.
Au sens de la présente invention la dénomination formes oncogéniques ou
forme mutées oncogéniques de p53 désigne les mutants dominant-oncogéniques,
CA 02347121 2001-04-09
WO 00/22120 PCT/FR99/02465
12
dont le produit est une protéine qui a perdu la capacité de se lier à l'ADN et
qui
participe activement à la transformation néoplasique. Les mutants de cette
catégorie
ont perdu leur capacité transactivatrice et sont plus stables que la protéine
sauvage.
Les représentants de cette catégorie de mutants de p53 sont notamment les
formes
mutantes H 175, 6281, W248, et A 143.
Un autre objet de la présente invention concerne un procédé de préparation
des polypeptides selon l'invention selon lequel on cultive une cellule
contenant une
séquence nucléotidique selon l'invention, dans des conditions d'expression de
ladite
séquence et on récupère le polypeptide produit. Dans ce cas, la partie codant
pour
ledit polypeptide est généralement placée sous le contrôle de signaux
permettant son
expression dans un hôte cellulaire. Le choix de ces signaux (promoteurs,
terminateurs, séquence leader de sécrétion, etc.) peut varier en fonction de
l'hôte
cellulaire utilisé. Par ailleurs, les séquences nucléotidiques de l'invention
peuvent
faire partie d'un vecteur qui peut être à réplication autonome ou intégratif.
Plus
particulièrement, des vecteurs à réplication autonome peuvent être préparés en
utilisant des séquences à réplication autonome chez l'hôte choisi. S'agissant
de
vecteurs intégratifs, ceux-ci peuvent être préparés, par exemple, en utilisant
des
séquences homologues à certaines régions du génome de l'hôte, permettant, par
recombinaison homologue, l'intégration du vecteur.
La présente invention a également pour objet des cellules hôtes
transformées avec un acide nucléique comportant une séquence nucléotidique
selon
l'invention. Les hôtes cellulaires utilisables pour la production des peptides
de
l'invention par voie recombinante sont aussi bien des hôtes eucaryotes que
procaryotes. Parmi les hôtes eucaryotes qui conviennent, on peut citer les
cellules
animales, les levures, ou les champignons. En particulier, s'agissant de
levures, on
peut citer les levures du genre Saccharomyces, Kluyveromyces, Pichia,
Schwanniomyces, ou Hansenula. S'agissant de cellules animales, on peut citer
les
cellules d'insectes (SF9 ou SF21), les cellules COS, CHO, C127, de
neuroblastomes
CA 02347121 2001-04-09
WO 00/22120 PCT/FR99/02465
13
humains etc. Parmi les champignons, on peut citer plus particulièrement
Aspergillus
ssp. ou Trichoderma ssp. Comme hôtes procaryotes, on préfère utiliser les
bactéries
suivantes E.coli, Bacillus, ou Streptomyces.
Selon un mode préféré, les cellules hôtes sont avantageusement représentées
par des souches de levures recombinantes pour l'expression des acides
nucléiques de
l'invention ainsi que la production des protéines dérivées de ceux-ci.
Préférentiellement, les cellules hôtes comprennent au moins une séquence
ou un fragment de séquence choisis parmi les séquences nucléotidiques SEQ ID
N°I5, N°21, N°32, N°23 et N° 30 pour
la production des poIypeptides selon
l'invention.
Une autre application des séquences d'acides nucléiques selon l'invention
est Ia réalisation d'oligonucléotides antisens ou d'antisens génétiques
utilisables
comme agents pharmaceutiques. Les séquences antisens sont des oligonucléotides
de
petite taille, complémentaires du brin codant d'un gène donné, et de ce fait
capables
d'hybrider spécifiquement avec l'ARNm transcrit, inhibant la traduction en
protéine.
L'invention a ainsi pour objet les séquences antisens capables d'inhiber, au
moins
partiellement , l'expression de polypeptides capables d'interagir avec la p53
comme
la protéine MBP1 ou la fibuline2. De telles séquences peuvent être constituées
par
tout ou partie des séquences nucléotidiques définies ci-avant et peuvent être
obtenus
par fragmentation, etc. ou par synthèse chimique.
Les séquences nucléotidiques selon l'invention peuvent être utilisées pour le
transfert et la production in vitro, in vivo ou ex vivo de séquences antisens
ou pour
l'expression de protéines ou de polypeptides capables d'interagir avec la
protéine
p53.
CA 02347121 2001-04-09
WO 00/22120 PCT/FR99/02465
14
A cet égard les séquences nucléotidiques selon l'invention peuvent être
incorporées dans des vecteurs viraux ou non viraux, permettant leur
administration in
vitro, in vivo ou ex vivo.
Un autre objet de l'invention concerne en outre tout vecteur comprenant une
séquence nucléotidique définie ci-avant. Le vecteur de l'invention peut être
par
exemple un plasmide, un cosmide ou .tout ADN non encapsidé par un virus, un
phage, un chromosome artificiel, un virus recombinant etc. II s'agit de
préférence
d'un plasmide ou d'un virus recombinant.
A titre de vecteurs viraux conformes à l'invention on peut tout
particulièrement citer les vecteurs de type adénovirus, rétrovirus, virus
adéno-
associés, virus de l'herpès ou virus de la vaccine. La présente demande a
également
pour objet des virus recombinants défectifs comprenant une séquence nucléique
hétérologue codant pour un polypeptide selon l'invention.
L'invention permet également la réalisation de sondes nucléotidiques,
synthétiques ou non, capables de s'hybrider avec les séquences nucléotidiques
définies ci-avant ou des ARNm correspondants. De telles sondes peuvent être
utilisées in vitro comme outil de diagnostic, pour la détection des
polypeptides selon
l'invention et notamment de la protéine MBP1 humaine ou de la fibuline 2. Ces
sondes peuvent également être utilisées pour la mise en évidence d'anomalies
génétiques (mauvais épissage, polymorphisme, mutations ponctuelles, etc). Ces
sondes peuvent ausi être utilisées pour la mise en évidence et l'isolement de
séquences d'acides nucléiques homologues codant pour les polypeptides tels que
définis précédemment, à partir d'autres sources cellulaires et
préférentiéllement de
cellules d'origines humaines. Les sondes de (invention comportent généralement
au
moins 10 nucléotides, de préférence au moins 15 nucléotides, et de préférence
encore
au moins 20 nucléotides. Préférentiellement, ces sondes sont marquées
CA 02347121 2001-04-09
WO 00/22120 PCT/FR99/02465
préalablement à leur utilisation. Pour cela, différentes techniques connues de
(homme du métier peuvent être employées (marquage radioactif, enzymatique,
etc).
L'invention concerne également l'utilisation de sondes nucléotidiques,
synthétiques ou non, capables de s'hybrider avec les séquences nucléotidiques
codant
5 pour la protéine MBP 1 pour la réalisation de test de diagnostic de tissus
cancéreux .
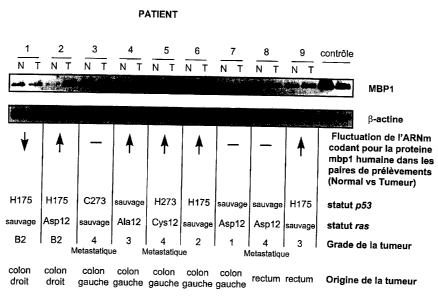
basés sur la detection du niveau d'expression de MBP1. A titre d'exemple de
sondes
nucléotidiques utilisables pour cette application, on peut citer notamment les
séquences SEQ ID N°27 et SEQ ID N°28. Ces sondes nucléotidiques
permettent de
détecter l'amplification de l'expression de la protéine MBP1. Ces sondes
peuvent être
10 des sondes ARN ou ADN. La présente invention met en évidence qu'une
amplification de l'ARN messager codant pour la protéine MBP 1 humaine peut-
être
décelée dans certains types de tumeurs humaines et notamment dans le cas de
cancers
du colon. A cet égard, l'invention concerne également un procédé de diagnostic
du
cancer comportant le fait de détecter l'amplification de l'expression du gène
codant
15 pour la protéine MBPI humaine.
Un autre objet de l'invention réside dans des anticorps ou fragments
d'anticorps polyclonaux ou monoclonaux dirigés contre un polypeptide tel que
défini
ci-avant. De tels anticorps peuvent être générés par des méthodes connues de
l'homme du métier. En particulier ces anticorps peuvent être préparés par
'immunisation d'un animal contre un polypeptide dont la séquence est choisie
parmi
les séquences SEQ ID N°9 (fragment C-terminal MBP1 murine) ou SEQ ID
N°31
(fragment C-terminal MBP 1 humaine) ou les séquences polypeptidiques SEQ ID
N°22 (MBP I humaine) ou SEQ ID N°33 (fragment C-term
Fibuline-2) ou tout
fragment ou dérivé de celles-ci, puis prélèvement du sang et isolement des
anticorps.
Ces anticorps peuvent également être générés par préparation d'hybridomes
selon les
techniques connues de l'homme de l'art.
L'invention a également pour objet des anticorps simple chaîne ScFv dérivés
des anticorps monoclonaux définis ci-avant. De tel anticorps simple chaîne
peuvent
CA 02347121 2001-04-09
WO 00/22120 PCT/FR99/02465
16
être obtenus selon les techniques décrites dans les brevet US 4,946,778, US
5,132,405 et US 5,476,786.
Les anticorps ou fragments d'anticorps selon l'invention peuvent notamment
être utilisés pour inhiber et/ ou révéler l'interaction entre la p53 et les
polypeptides
tels que définis ci-avant.
Un autre objet de la présente invention concerne un procédé d'identification
de composés capables de se lier aux polypeptides selon l'invention. La mise en
évidence et/ou l'isolement de ces composés, peut-être réalisée selon les
étapes
suivantes
- on met en contact une molécule ou un mélange contenant différentes
molécules, éventuellement non-identifiées, avec un polypeptide de l'invention
dans
des conditions permettant l'interaction entre ledit polypeptide et ladite
molécule dans
le cas où celle-ci possèderait une affinité pour ledit polypeptide, et,
- on détecte et/ou isole les molécules liées au dit polypeptide de
l'invention.
Selon un mode particulier, un tel procédé permet d'identifier des molécules
capables de s'opposer ou de bloquer l'activité de stimulation de la croissance
cellulaire des polypeptides selon l'invention et notamment de la protéine MBP
1
humaine ou Fibuline 2 ou des fragments dérivés de ces protéines. Ces molécules
sont
également susceptibles de présenter des propriétés anti-cancéreuses et de
s'opposer à
la fonction d'oncogènes immortalisants que présentent MBP 1 ou les
polypeptides
dérivés de MBP 1 qui coopèrent avec la forme activée de la protéine Ras pour
la
transformation cellulaire.
A cet égard, un autre objet de l'invention concerne (utilisation d'un ligand
identifié et/ou obtenu selon le procédé décrit ci-avant comme médicament. De
tels
CA 02347121 2001-04-09
WO 00/22120 PCT/FR99/02465
17
ligands sont en effet susceptibles de traiter certaines affections impliquant
un
dysfonctionnement du cycle cellulaire et notamment les cancers.
Un autre objet de la présente invention concerne un procédé d'identification
de composés capables de moduler ou d'inhiber totalement ou partiellement
l'interaction entre les formes mutées oncogènes de p53 et les polypeptides
selon
l' invention.
La mise en évidence etlou l'isolement de modulateurs ou de ligands capables
de moduler ou d'inhiber totalement ou partiellement l'interaction entre les
formes
mutées oncogènes de p53 et les polypeptides selon l'invention, peut-être
réalisée
selon les étapes suivantes
- on réalise la liaison d'une forme mutée de p53 ou d'un fragment de celle-ci
à
un polypeptide selon l'invention ; il peut s'agir des formes mutées de p53
telles que
H 175, 6281, W248, ou A 143 ou d'un fragment de celles-ci, il s'agit
préférentiellement de la forme H175 ou encore de la forme 6281.
- on ajoute un composé à tester pour sa capacité à inhiber la liaison entre la
forme mutée de p53 et les polypeptides selon l'invention ;
- on détermine si la forme mutée de p53 ou les polypeptides selon l'invention
sont déplacés de la liaison ou empêchés de se lier ;
- on détecte et/ou isole les composés qui empêchent ou qui gênent la liaison
entre la forme mutée de p53 et les polypeptides selon l'invention.
Dans un mode particulier, ce procédé de l'invention est adapté à la mise en
évidence et/ou l'isolement d'agonistes et d'antagonistes de (interaction entre
les
formes mutées de p53 et les polypeptides de l'invention. Toujours selon un
mode
particulier, l'invention fournit un procédé d'identificatïon de molécules
capables de
CA 02347121 2001-04-09
WO 00/22120 PCT/FR99/02465
18
bloquer l'interaction entre les formes mutées de p53 et la protéine MBP1
humaine ou
fibuline 2 humaine. Un tel procédé permet d'identifier des molécules capables
de
s'opposer aux effets de l'action des polypeptides selon l'invention avec les
formes
mutées de p53. En particulier de tels composés sont susceptibles de prévenir
la
coopération oncogénique entre la protéine MBP 1 et les formes mutantes
oncogéniques de p53 telle notamment H 175.
A cet égard, un autre objet de l'invention concerne l'utilisation d'un ligand
ou
d'un modulateur identifié etlou obtenu selon le procédé décrit ci-avant comme
médicament. De tels ligands ou modulateurs sont en effet susceptibles de
traiter
certaines affections impliquant un dysfonctionnement du cycle cellulaire et
notamment des cancers.
L'invention fournit également des composés non peptidiques ou non
exclusivement peptidiques utilisables pharmaceutiquement. Il est en effet
possible, à
partir des motifs protéiques actifs décrits dans la présente demande, de
réaliser des
molécules inhibitrices de l'interaction de MBP 1 ou de la fibuline2 avec les
formes
mutées oncogéniques de p53, ces molécules étant non exclusivement peptidiques
et
compatibles avec une utilisation pharmaceutique. A cet égard, l'invention
concerne
l'utilisation d'un polypeptide de l'invention tel que décrit ci-avant pour la
préparation
de molécules non-peptidiques, ou non exclusivement peptidiques, actives
pharmacologiquement, par détermination des éléments structuraux de ce
polypeptide
qui sont importants pour son activité et reproduction de ces éléments par des
structures non-peptidiques ou non exclusivement peptidiques. L'invention a
aussi
pour objet des compositions pharmaceutiques comprenant une ou plusieurs
molécules ainsi préparées.
L'invention a encore pour objet toute composition pharmaceutique
comprenant comme principe actif au moins un ligand obtenu selon l'un et/ou
l'autre
des procédés décrit ci-avant, et/ou au moins un anticorps ou fragment
d'anticorps,
CA 02347121 2001-04-09
WO 00/22120 PCT/FR99/02465
19
et/ou un oligonucléotide antisens, et/ou un composé non exclusivement
peptidiques
tels que décrits ci-avant.
Les compositions selon l'invention peuvent être utilisées pour moduler
l'interaction des formes mutées oncogènes de p53 avec les polypeptides MBP 1
ou
Fibuline 2 et de ce fait peuvent être utilisées pour moduler la prolifération
de certain
type cellulaires. Plus particulièrement ces compositions pharmaceutiques sont
destinées au traitement des maladies impliquant un dysfonctionnement du cycle
cellulaire et notamment au traitement des cancers. II s'agit en particulier
des cancers
associés à la présence de mutants oncogéniques de p53.
D'autres avantages de la présente invention apparaîtront à la lecture des
exemples qui
suivent et qui doivent être considérés comme illustratifs et non limitatifs.
Légende des Figures
Figure I . Domaines fonctionnels de la protéine p53 sauvage. TA : Domaine
activateur de la transcription; DNB : domaine de liaison à l'ADN; NLS : signal
de
localisation nucléaire; OL : domaine d'oligomérisation;1ZEG : domaine de
régulation.
Figure 2 : Interaction entre la protéine C-mbp I et les protéines p53 et H 175
en
cellules mammifères.
Figure 3 : Interaction entre la protéine C-fibulin2 et les protéines p53 et H
175 en
cellules mammifères.
Figure 4 : Comparaison des séquences protéiques codées par les ADNc mMBP I
(mucine) et hMBP 1 (humaine).
Figure 5 : Effets comparés des protéines C-mbp 1 et MBP 1 mucine sur la
croissance
CA 02347121 2001-04-09
WO 00/22120 PCT/FR99/02465
cellulaire de cellules tumorales.
Figure 6 : Expression de l'ARNm codant pour la protéine MBPI chez la souris.
5 Figure 7 : Expression de l'ARNm codant pour la protéine MBPI dans différents
tissus humains.
Figure 8 : Expression de l'ARN messager codant pour la protéine MBP 1 humaine
dans des tumeurs du colon.
Exemple 1 - Construction des différents fragments nucléotidiques nécessaires
au
criblage
1-a - Construction du cDNA codant pour la p53 sauvage humaine
Le gène de la p53 humaine a été cloné par réaction d'amplification en chaine
(PCR) sur de l'ADN d'une banque de placenta humain (Clontech) en utilisant les
oligonucléotides 5'-1 et 3'-393.
Oligonucléotide 5'-1 (SEQ ID N° 1)
AGATCTGTATGGAGGAGCCGCAG
Oligonucléotide 3'-393 (SEQ ID N° 2)
AGATCTCATCAGTCTGAGTCAGGCCCTTC
Ce produit a ensuite été cloné directement après PCR dans le vecteur pCRII
(Invitrogène).
1-b - Construction des cDNA codant pour les différentes formes mutées de la
p53 humaine
CA 02347121 2001-04-09
WO 00/22120 PCT/FR99/02465
21
I-b.(i) - Construction du cDNA codant pour ie mutant H175 de la p53
humaine
Le cDNA portant une mutation ponctuelle sur l'acide aminé 175 de la protéine
p53
humaine (Arginine -> Histidine) a été obtenu par mutagénèse dirigée sur l'ADN
de~
p53 (décrit dans l'exemple I-a) au moyen du kit Amersham, en utilisant
l'oligonucléotide H 175 de séquence
Oligonucléotide H 175 3' (SEQ ID N° 3)
GGGGCAGTGCCTCAC
Ce fragment a été désigné H 175.
1-b. (ü) - Construction du cDNA codant pour le mutant W248 de la p53
humaine
Le cDNA portant une mutation ponctuelle sur l'acide aminé 248 de la protéine
p53
humaine (Arginine -> Tryptophane) a été obtenu par mutagénèse dirigée sur
l'ADN
de p53 (décrit dans l'exemple I-a) au moyen du kit Amersham, en utilisant
l'oligonucléotide W248 de séquence
Oligonucléotide W248 3' (SEQ ID N° 4)
GGGCCTCCAGTTCAT
Ce fragment a été désigné W248.
I-b (iii) - Construction du cDNA codant pour le mutant H273 de la p53
humaine
Le cDNA portant une mutation ponctuelle sur (acide aminé 273 de la protéine
p53 humaine (Aspartate -> Histidine) a été obtenu par mutagénèse dirigée sur
l'ADN
de p53 (décrit dans l'exemple 1-a) au moyen du kit Amersham, en utilisant
l'oligonucléotide H273 de séquence
CA 02347121 2001-04-09
WO 00/22120 PC'T/FR99/02465
22
Oligonucléotide H273 3' (SEQ ID N° 5)
ACAAACATGCACCTC
Ce fragment a été désigné H273.
1-b (iv) - Construction du cDNA codant pour le mutant 6281 de la p53
humaine
Le cDNA portant une mutation ponctuelle sur l'acide aminé 281 de la protéine
p53
humaine (Asparagine -> Glycine) a été obtenu par mutagénèse dirigée sur l'ADN
de
p53 (décrit dans l'exemple 1-a) au moyen du kit Amersham, en utilisant
l'oligonucléotide 6281 de séquence
Oligonucléotide 6281 3' (SEQ ID N° 6)
GCGCCGGCCTCTCCC
Ce fragment a été désigné 6281.
1-c - Construction des cDNA codant pour les fragments 73-393 de la p53
humaine sauvage et du mutant H175
1-c (i) - Construction du cDNA codant pour le fragment 73-393 de la p53
humaine sauvage
Cet exemple décrit la construction d'un cDNA codant pour les acides aminés 73
à 393
de la protéine p53 humaine sauvage (73-393wt).
Ce cDNA a été obtenu par réaction d'amplification en chaine (PCR) sur l'ADN de
p53 (décrit dans l'exemple 1-a) avec l'oligonucléotide 3'-393 (SEQ ID
N° 2) et
l'oligonucléotide 5'-73 suivant
S'-73 (SEQ ID N° 7)
AGATCTGTGTGGCCCCTGCACCA
CA 02347121 2001-04-09
WO 00/22120 PCT/FR99/02465
23
I-c (ü) - Construction du cDNA codant pour le fragment 73-393 du mutant
H 175
Cet exemple décrit la construction d'un cDNA codant pour les acides aminés 73
à 393
du mutant H 175 de la protéine p53 humaine (73-393H 175).
Ce cDNA a été obtenu par réaction d'amplification en chaire (PCR) sur l'ADN du
mutant (décrit dans l'exemple I-b) avec les oligonucléotides 3'-393 (SEQ ID
N° 2) et
5'-73 (SEQ ID N° 7).
Exemple 2 - Construction des vecteurs d'expression dans la levure des
fragments
73-393wt et 73-393H175 fusionnés au domaine de liaison à l'ADN de la protéine
Gal4 et des différentes formes de la p53 humaine entière (sauvage et mutée)
fusionnées au domaine d'activation de la transcription de la protéine Gal4
Cet exemple décrit la construction de vecteurs permettant l'expression dans la
levure des fragments 73-393wt et 73-393H175 sous forme d'une fusion avec le
domaine de liaison à l'ADN de la protéine Gal4 (DB) de la levure S. cerevisiae
pour
leur utilisation dans le système double-hybride et pour le criblage de banques
de
cDNA fusionnés au domaine d'activation de la transcription (transactivateur)
de la
même protéine Gal4 (TA).
2-a - Construction des vecteurs d'expression de levure des fragments 73-393wt
et
73-393H175 fusionnés au domaine de liaison à l'ADN de la protéine Gal4
Les fragments 73-393wt et 73-393H175 ont été clonés dans le vecteur pPC97
(Chevray et al, Proc. Natl. Acad. Sci. USA 89 ( 1992) 5789) en utilisant le
site de
reconnaissance par l'enzyme de restriction Bgl II.
Les produits de ces constructions portent les noms suivants:
CA 02347121 2001-04-09
WO 00/22120 PCT/FR99/02465
24
73-393wt dans pPC97 --> (plasmide pMA 1 ) --> DB-wt
73-393H 175 dans pPC97 --> (plasmide pEC 16)--> DB-H 175
2-b - Construction des vecteurs d'expression de levure des différentes formes
de
la p53 humaine entière (sauvage et mutée) fusionnées au domaine d'activation
de la transcription de la protéine Gal4
Les différentes formes de la p53 humaine entière (sauvage et mutée) ont été
clonés
dans le vecteur pPC86 (Chevray et al, Proc. Natl. Acad. Sci. USA 89 ( 1992)
5789) en
utilisant le site de reconnaissance par l'enzyme de restriction Bgl II.
Les produits de ces constructions portent les noms suivants:
p53 dans pPC86 --> (plasmide pEC 10)--> TA-wt
H 175 dans pPC86 --> (plasmide pEC20)--> TA-H 175
H273 dans pPC86 --> (plasmide pEC87)--> TA-H273
6281 dans pPC86 --> (plasmide pEC88)--> TA-G28I
Exemple 3 - Clonage par le système double-hybride des partenaires de la
protéine H175, et caractérisation de cette interaction en terme de spécificité
dans
la levure
Cet exemple décrit l'obtention des partenaires de la protéine H175 par le
système double-hybride en utilisant la banque de cDNA d'embryon de souris
pPC67
(Chevray et al, Proc. Natl. Acad. Sci. USA 89 ( 1992) 5789}, et la
caractérisation, à
l'aide du même système double-hybride, de ces partenaires en terme de
spécificité
d'interaction avec les différentes formes de la protéine p53 humaine (sauvage
et
mutée).
3-a - Isolement des partenaires de la protéine H175
3-a (i) - Génotype de la souche YCM 17
CA 02347121 2001-04-09
WO 00/22120 PCT/FR99/02465
La souche YCM17 utilisée pour l'isolement des partenaires et pour la
caractérisation de leur interaction avec les différentes formes de la protéine
p53
humaine par le système double-hybride est une souche de levure du genre
Saccharomyces cerevisiae qui possède le génotype suivant:
5 MATa, ~gal4, ~ga180, lys2, his3, trpl, leu2, ade2, ura3, canl, metl6:: URA3
pGALI-.
10 LacZ.
Cette souche de levure permet de détecter une réponse positive en système
double-hybride par l'apparition du phénotype Ura+ et/ou du double phénotype
Ura+/LacZ+,
10 3-a (ü) - Génotype de la souche TG 1
La souche TG1 utilisée pour la puri5cation des ADN plasmidiques est une souche
de
bactérie du genre E.coli qui possède le génotype suivant:
supE, hsdDS, thi, D(lac-proAB), F'[tra D36 proA+B+ lacIq lacZDMlS]
3-a (iii) - Construction de la souche YMA1
15 La souche YCM17 a été transformée par la méthode de Gietz et al (Yeast 11
(1995)
355) avec 1 ~g du plasmide pMA 1 permettant ainsi l'obtention de la souche YMA
1
qui exprime la protéine DB-H 175.
3-a (iv) - Isolement des partenaires de la protéine H 175
La souche YMA 1 a été transformée par la même méthode que celle utilisée dans
20 l'exemple C 1.3 en utilisant 100pg d'ADN de la banque pPC67, permettant
l'obtention de 3,5.10' transformants parmi lesquels 404 présentent le
phénotype Ura+
et 14 le double phénotype Ura+/LacZ+.
L'ADN plasmidique contenu dans les 14 clones présentant le double phénotype
Ura+/LacZ+ a été isolé par la méthode de Ward (Nucl. Acids Res. 18 ( 1990}
5319)
CA 02347121 2001-04-09
WO 00/22120 PCT/FR99/02465
26
avant d'être utilisé pour transformer la souche TG 1. Les plasmides
correspondants
issus de la banque ont ensuite été purifiés et regroupés en deux sous-groupes
de
plasmides différents contenant chacun un cDNA codant pour deux protéines
différentes:
- un cDNA codant pour la partie C-terminale (SEQ ID N° 8) d'un nouveau
gène.
- un cDNA codant pour la partie C-terminale de la fibuline 2 marine (acides
aminés 863 à 1195 (SEQ ID N° 32)) (Pan et al, J. Cell. Biol. 123 (1993)
1269).
Les protéines codées par ces deux cDNA sont appelées respectivement C-mbp 1
(mbp
- 'p53 Mutant Binding Protein') et C-fibuline2, les protéines de fusion avec
le
domaine d'activation de la transcription de Gal4 sont nommées TA-C-mbpl et TA-
C
fibuline2 et les plasmides correspondants sont nommés TA -C-MBP 1 et TA-C-FIB2
3 - b - Caractérisation de l'interaction entre les protéines C-mbpl et C-
~buline2
et la protéine H175 dans la levure
3 - b (i) - Caractérisation de la spécificité de l'interaction entre la
protéine
DB-H 175 et les protéines TA-C-mbp 1 et TA-C-fibuline2
Dans le but de tester la spécificité des interactions décrites dans l'exemple
3-a (iv),
les plasmides pPC86, TA-C-MBP1 et TA-C-FIB2 ont été réintroduits dans la
souche
YCM17 par co-transformation avec différents plasmides : le plasmide pPC97
codant
pour la protéine DB, le plasmide pMA I codant pour la protéine DB-H 175, le
plasmide pEC 10 codant pour la protéine DB-wt et le plasmide pPC76 codant pour
une protéine de fusion entre le domaine de liason à l'ADN de la protéine Gal4
et un
fragment de la protéine Fos humaine (acides aminés 132 à 211 ) (Chevray et al,
Proc.
Natl. Acad. Sci. USA, 89 ( 1992) 5789) (DB-Fos). Après la co-transformation,
les
différents clones obtenus ont été testés pour les phénotypes associés aux
gènes URA3
et LacZ
CA 02347121 2001-04-09
WO 00/22120 PCT/FR99/02465
27
Les résultats de cette expérience sont présentés dans Ie Tableau 1.
TA TA-C-mbp 1 TA-C-fibuline2
DB Ura - / LacZ- Ura - / LacZ- Ura - / LacZ-
DB-H 175 Ura - / LacZ- Ura + I LacZ+ Ura + I LacZ+
DB-wt Ura - / LacZ- Ura - / LacZ- Ura - / LacZ-
DB-Fos Ura - / LacZ- Ura - / LacZ- Ura - / LacZ-
Tableau l: Spécificité de l'interaction entre la protéine DB-H175 et les
protéines TA-
C-mbp 1 et TA-C-fibuline2
Ces résultats montrent que l'interaction entre la protéine DB-H175 et les
protéines TA-C-mbp 1 et TA-C-fibuline2 est spécifique et qu'une telle
interaction ne
peut être obtenue ni avec la protéine DB seule, ni avec la protéine DB-wt ni
avec Ia
protéine contrôle DB-Fos.
3-b (ü} - Construction de protéines de fusion entre le domaine liaison à l'ADN
de Gal4 et les protéines C-mbpl et C-fibuline2
Les cDNA codant pour C-mbp i et C-fibuline2 ont été extraits des plasmides
TA-C-MBP 1 et TA-C-FIB2, puis clonés dans le vecteur pPC97 en utilisant Ies
sites
de reconnaissance par les enzymes de restriction Sal I et Not I.
Les protéines de fusion avec le domaine de liaison à l'ADN de Gal4 ainsi
obtenues sont appelées respectivement DB-C-mbp 1 et DB-C-fibuline2 et les
plasmides correspondants DB-C-MBP 1 et DB-C-FIB2.
CA 02347121 2001-04-09
WO 00/22120 PCT/FR99/02465
28
3-b (iii) Caractérisation de la spécificité de l'interaction entre les DB-C-
mbpl
et DB-C-fibuline2 et les protéines TA-H 175 et TA-6281
Dans le but de vérifier que l'interaction potentielle entre les protéines C-
mbpl
et C-fibuline2 et la protéine H175 n'est pas un artefact dû à la fusion de
l'un ou
l'autre des partenaires avec l'un ou l'autre des domaines de Gal4, et de
confirmer la
spécificité de l'interaction avec la forme mutante de la protéine p53, une
nouvelle
expérience d'interaction dans la levure a été effectuée en utilisant des
fusions
différentes de celles de l'exemple 3-b (i).
Dans cette expérience, les protéines DB-C-mbp 1 et DB-C-fibuline2 ont été
testées contre les fusions du domaine d'activation de la transcription de Gal4
avec les
formes entières de la protéine p53 (sauvage ou mutante) décrites dans
l'exemple 2-b,
en utilisant une souche de levure différente de la souche YCM17, la souche
PCY2
(Chevray et al, Proc. Natl. Acad. Sci. USA 89 (1992) 5789).
Les résultats de cette expérience sont présentés dans le Tableau 2.
TA TA-wt TA-H175 TA-H273 TA-6281
DB LacZ - LacZ - LacZ - LacZ - LacZ -
DB-C-mbp 1 LacZ - LacZ - LacZ + LacZ - LacZ +
DB-C- LacZ - LacZ - LacZ + LacZ - LacZ +
fibuline2
Tableau 2: Spécificité de l'interaction entre les protéines DB-C-mbpl et DB-C-
fibuline2 et les protéines TA-H 175 et TA-6281
Ces résultats permettent d'une part de confirmer l'interaction observée lors
du
criblage. D'autre part, ces résultats mettent en évidence, Ia spécificité de
l'interaction
entre les protéines C-mbp 1 et C-Rbuline2 et certaines formes mutées de la
protéine
p53. En ce qui concerne cette spécificité, il est intéressant de noter, que
ces protéines
CA 02347121 2001-04-09
WO 00/Z2120 PCT/FR99/02465
29
n'interagissent pas avec le mutant H273. En effet, ce mutant présente une
conformation équivalente à celle de la protéine p53 sauvage car il est reconnu
par
l'anticorps PAb 1620 qui est spécifique de la forme sauvage et pas par
l'anticorps
PAb 240 qui est spécifique de la forme mutée (Medcalf et al, Oncogene 7 (1992)
71).
Ainsi, l'ensemble des données obtenues dans la levures montrent clairement
que les deux protéines C-mbp I et C-fibuline2 sont des partenaires potentiels
spécifiques des mutants oncogéniques de la protéine p53.
Exemple 4 - Interaction entre les protéines C-mbpl, C-fibuline2 et les
différentes
formes de la protéine p53 en cellules mammifères
Cet exemple décrit la construction de plasmides pour l'expression des
différentes
protéines en cellules mammifères et la caractérisation de l'interaction entre
les
protéines C-mbpl et C-fibuline2 et les différentes formes de Ia protéine p53
en
cellules mammifères.
4-a Construction des plasmides d'expression des différentes protéines en
cellules
mammifères
4-a (i) Construction du vecteur d'expression pBFA 107
Cet exemple décrit la construction d'un vecteur permettant l'expression dans
des cellules mammifères de protéines portant une étiquette dérivée de la
protéine c-
myc (acides aminés 410-4I9) et reconnue par l'anticorps 9E10 (Oncogene
Science).
Cette construction a été effectuée en utilisant comme vecteur de base le
vecteur
d'expression mammifère pSV2, décrit dans DNA Cloning, A practical approach
Vol.2, D.M. Glover (Ed) IItL Press, Oxford, Washington DC, 1985.
Le cDNA comprenant la séquence codant pour l'étiquette c-myc ainsi qu'un
multisite de clonage (MCS) a été construit à partir des 4 oligonucléotides
suivants
CA 02347121 2001-04-09
WO 00/22120 PCT/FR99/02465
c-myc 5' (SEQ ID N° 10):
GATCCATGGAGCAGAAGCTGATCTCCGAGGAGGACCTGA
c-myc 3' (SEQ ID N° 11 ):
GATCTCAGGTCCTCCTCGGAGATCAGCTTCTGCTCCATG
5 MCS 5' (SEQ ID N° 12):
GATCTCGGTCGACCTGCATGCAATTCCCGGGTGCGGCCGCGAGCT
MCS 3' {SEQ ID N° 13):
CGCGGCCGCACCCGGGAATTGCATGCAGGTCGACCGA
Ces quatre oligonucléotides présentent des complémentarités deux à deux (c-
10 myc 5' / c-myc 3', MCS 5' / MCS 3') et des complémentarités chevauchantes
(c-myc
3' / MCS 5') permettant l'obtention de la séquence nucléotidique désirée par
simple
hybridation et ligation. Ces oligonucléotides ont été phosphorylés à l'aide de
la T4
kinase, puis hybridés tous ensemble et insérés dans le vecteur d'expression
pSV2
préalablement digéré par les enzymes de restriction Bgl II et Sac I. Le
vecteur
15 résultant est le vecteur pBFA 107.
4-a (ü) - Construction des plasmides d'expression des protéines C-mbpl et C-
fibuline2 étiquetées
20 Les cDNA codant pour les protéines C-mbp 1 et C-fibuline2 ont été extraits
des plasmides TA-C-MBP1 et TA-C-FIB2 et clonés dans le vecteur d'expression
mammifère pBFA 107 en utilisant les sites de reconnaissance par les enzymes de
restriction Sal I et Not I. On obtient ainsi les plasmides pBFA107-C-MBP1 et
pBFA 107-C-FIB2
4-a (iii) Construction des plasmides d'expression des différentes formes de la
protéine p53
CA 02347121 2001-04-09
WO 00/22120 PCT/FR99/02465
31
Les cDNA codant pour les différentes formes de la protéine p53 (wt, H175,
H273 et 6281 ) ont été insérés dans les vecteurs d'expression pSV2 et pcDNA3
(Invitrogen) en utilisant le site de reconnaissance par l'enzyme de
restriction Bgl II.
4 b - Interaction entre les protéines C-mbpl et C-fibuline2 et les différentes
formes de la protéine p53 en cellules mammifères
Cet exemple décrit la mise en évidence dans des cellules mammifères de
l'interaction entre les protéines C-mbpl et C-fibuline2 et les différentes
formes de la
protéine p53. Ces expériences ont été effectuées par transfection transitoire
et co-
immunoprécipitation dans les cellules H1299 (cellules tumorales de type 'Non
Small
Cell Lung Cancer') déficientes pour les deux allèles de la protéine p53
(Mitsudomi et
al, Oncogene 1 ( 1992) 171 ).
Les cellules (10~) sont ensemencées dans des boites de Pétri de 10 cm de
diamètre contenant 8 ml de milieu DMEM (Gibco BItL) additioné de 10% de sérum
de veau foetal inactivé à la chaleur, et cultivées sur la nuit dans un
incubateur à COZ
(5%) à 37°C. Les différentes constructions sont alors transfectées en
utilisant la
lipofectAMINE (Gibco BRL) comme agent de transfection de la façon suivante: 6
pg
de plasmide total (3 pg de chaque plasmide codant pour chacun des deux
partenaires)
sont incubés avec 20 ul de lipofectAMINE (Gibco BRL) pendant 30 min avec 3 ml
de milieu Opti-MEM (Gibco BRL) (mélange de transfection). Pendant ce temps,
les
cellules sont rincées deux fois au PBS puis incubées 4 h à 37°C avec le
mélange de
transfection, après quoi celui-ci est aspiré et remplacé par 8 ml de milieu
DMEM
additionné de 10% de sérum de veau foetal inactivé à la chaleur et les
cellules
remises à pousser à 37°C.
Vingt quatre heures après la transfection, les cellules sont lavées une fois
en
PBS puis grattées, lavées de nouveau deux fois en PBS et remises en suspension
dans
200p1 de tampon de lyse (HNTG: Hepes 50 mM pH 7,5, NaCI 150 mM, Triton X-100
1%, glycérol 10%) additionné d'inhibiteurs de protéases (Aprotinine 2 pg/ml,
CA 02347121 2001-04-09
WO 00/22120 PCT/FR99/02465
32
pepstatine 1 pg/ml, leupeptine 1 ~,g/ml, E64 2 p.g/ml et Pefabloc 1 mM),
incubées 30
min à 4°C et centrifugées 15 min à 15.000 rpm et 4°C. L'extrait
cellulaire ainsi
obtenu est soumis à une étape de 'pré-clearing' par incubation lh à 4°C
avec 16u1
d'un sérum de lapin pré-immun, puis 30 min à 4°C avec 200p1
d'immunoprecipitin
(Gibco BRL) préparée selon les recommandations du fournisseur. Par la suite,
l'extrait cellulaire ainsi nettoyé est séparé en 3 lots égaux dont chacun est
incubé la
nuit à 4°C avec un anticorps différent; 3 pg d'anticorps 9E10 (anti
myc), 1 ug
d'anticorps DO1 (anti p53) (Oncogene Science) et 1 p.g d'anticorps PAb416
(anti
SV40 T-Ag utilisé comme anticorps contrôle) (Oncogene Science). Ce mélange
[extrait cellulaire/anticorps) est ensuite additionné de 30p,1
d'immunoprecipitin et
incubé 30 min à 4°C avant d'être centrifugé 30 sec à 15.000 rpm. Le
culot contenant
l'immunoprecipitin est ensuite lavé deux fois par 1 ml de tampon HNTG
additionné
d'inhibiteurs de protéases, puis resuspendu dans 30 ~.1 de tampon de dépot sur
gel
d'acrylamide (Laemmli, Nature 227 (1970) 680) et incubé S min à 95°C.
Après
centrigugation 15 sec à 15.000 rpm, les surnageants sont déposés sur gel de
polyacrylamide en milieu dénaturant (Novex) et les protéines séparées en
utilisant le
système de migration XCell II (Novex) suivant les recommandations du
fournisseur,
puis transférées sur membrane de PVDF (NEN Life Science Products) à l'aide du
même système XCell II.
Les anticorps 9E10 et DOI utilisés pour la révélation des protéines
transférées
sont couplés à la biotine LCnHS (Pierce) suivant les recommandations du
fournisseur.
Les membranes de transfert sont tout d'abord incubées 1 h à 4°C
dans 10 ml
de tampon TTBSN (Tris-HCl 20 mM, pH 7,5, NaCI ISO mM, NaN3 0,02%, Tween 20
0,1%) additionné de 3% d'albumine bovine sérique (BSA) (TTBSN-BSA), puis 2 h à
température ambiante avec 10 ml d'une solution de TTBSN-BSA contenant
l'anticorps 9E10 hiotinylé (1 ug/ml). Après 6 lavages par 10 ml de tampon
TTBSN,
les membranes sont ensuite incubées 1 h à température ambiante avec 10 ml
d'une
solution de TTBSN-BSA contenant de l'ExtrAvidin-Peroxidase (Sigma Immuno
CA 02347121 2001-04-09
WO 00/22120 PCT/FR99/02465
33
Chemicals) au I/5000', lavées de nouveau 6 fois au TTBSN et traitées au
réactif ECL
(Amersham) pour la révélation des protéines par chemiluminescence. Les mêmes
membranes sont ensuite traitées par l'anticorps DO1 biotinylé après avoir été
préalablement deshybridées (Ellis et al, Nature 343 (1990) 377) et en suivant
le
même protocole que pour l'anticorps 9E10.
4 - b (i) - Interaction entre la protéine C-mbp 1 et les différentes formes de
la
protéine p53 en cellules mammifères
Dans cet exemple, les cellules H 1299 ont été transfectées avec les
combinaisons de plasmides suivantes avant immunoprécipitation et Western-blot
pBFA 107 / pBFA 107-C-MBP 1 (3 p,g) + pSV2 / pSV2-p53 (sauvage ou mutant)
(3 N~g)
De plus la combinaison suivante servant de contrôle a été effectuée
pBFA107-Sam68 (3 p,g) + pSV2-H175 (3 p.g)
Ce contrôle sert à examiner si H175 peut ou non interagir soit avec
l'étiquette
myc soit avec une fusion entre l'étiquette myc et une protéine quelconque, la
protéine
Sam68 décrite par Lock et al (Cell 84(1996)23), n'étant pas censée interagir
avec les
différentes formes de la protéine p53.
Les résultats de cette expérience qui sont présentés dans la Figure 2 montrent
que
- la protéine C-mbpl peut interagir avec la protéine H175 dans des cellules
mammifères
- cette interaction est bien spécifique de la protéine C-mbp 1 car la protéine
H 175 n'intragit pas avec le contrôle myc-Sam68
CA 02347121 2001-04-09
WO 00/22120 PCT/FR99/02465
34
4-b (ü} - Interaction entre la protéine C-fibuline2 et les différentes formes
de
la protéine pS3 en cellules mammifères
Dans cet exemple, les cellules H 1299 ont été transfectées avec les
combinaisons de plasmides suivantes avant immunoprécipitation et Western-blot
pBFA107 / pBFA107-C-FIB2 (3 pg) + pSV2 / pSV2-pS3 (sauvage ou mutant) (3 p,g)
Les résultats de cette expérience qui sont présentés dans la Figure 3 montrent
que la protéine C-fibuline2 peut interagir spécifiquement avec la protéine H
17S dans
des cellules mammifères.
La conclusion générale de ces expériences est que les protéines C-mbpl et C-
fibuline2 sont capables d'interagir spécifiquement en cellules mammifères avec
la
protéines H17S. Ces résultats de même que ceux obtenus dans la levure (exemple
3)
sont en accord avec
1/ la classification des mutants de la protéine pS3
- H 17S et 6281: dominants oncogéniques
- H273: mutant faible
2/ la classification des différentes formes de la protéine pS3 en terme de
conformation et de reconnaissance par des anticorps conformationnels
- H 17S et 6281 : conformation mutante, PAb 1620 - / PAb 240 +
- pS3 et H273 : conformation sauvage, PAb 1620 + / Pab 240 -
L'ensemble des ces données montrent que les protéines C-mbpl et C-
fibuline2 interagissent avec les formes de la protéine pS3 présentant une
conformation mutée, et qu'elles sont susceptibles d'avoir un effet sur des
fonctions
spécifiques des formes mutées de la protéine pS3.
De plus, de par la littérature, on sait qu'une fraction de la protéine pS3
peut
exhiber une conformation mutante dans les cellules mammifères, en particulier
CA 02347121 2001-04-09
WO 00122120 PCT/FR99102465
1/ la protéine p53, capable de se lier à l'ADN (Hupp et al, Nucl. Acids Res.
21
(1993) 3167), peut adopter une conformation mutante lorsqu'elle se lie à l'ADN
(Halazonetis et al, EMBO J. 12 ( 1993) I02 I )
2/ la protéine p53 peut adopter deux conformations différentes au cours du
5 cycle cellulaire; la conformation dite 'suppresseur' (conformation sauvage,
PAb 1620
+ / PAb 240 -) et la conformation dite 'promoteur' (conformation mutante, PAb
1620
- / PAb 240 +) (Mimer & Watson, Oncôgene 2 (1990) 1683).
On peut donc supposer que les protéines C-mbpl et C-fibuline 2 sont
10 également susceptibles d'avoir un effet sur des fônctions spécifiques de la
forme
sauvage de la protéine p53.
Exemple 5 - Effet de la protéine C-mbpl sur la coopération oncogénique entre
la
protéine H175 et la protéine Ras-Va112
Cet exemple décrit les effets de la protéine C-mbp I sur une propriété du
mutant oncogénique H 175, sa capacité à coopérer avec la forme mutée du proto-
oncogène Ras (Ras-Va112) dans la transformation oncogénique de fibroblastes
embryonnaires de rat
Les fibroblastes embryonnaires de rat (REF) ont été préparés à partir de rats
OFA (IFA-CREDO) selon la méthode décrite par C. Finlay {Methods in Enzymology
255 ( 1995) 389). Après décongélation, les cellules ( I,S.106) sont
ensemencées dans
des boites de Pétri de 10 cm de diamètre contenant 8 ml de milieu DMEM (Gibco
BRL) additioné de 10% de sérum de veau foetal et cultivées sur la nuit dans un
incubateur à CO, (5%) à 37°C, puis sont transfectées par les différents
mélanges de
plasmides (21 pg d'ADN) en utilisant le réactif CeIlPhect (Pharmacia) suivant
les
recommandations du fournisseur. 24 h après Ia fin de la transfection, les
cellules
contenues dans chacune des boites sont grattées puis réensemencées sur trois
boites
de Pétri de 10 cm et cultivées pendant 15 jours avant d'être colorées au
cristal violet
suivant le protocole décrit par C. Finlay (Methods in Enzymology 255 (1995)
389).
CA 02347121 2001-04-09
WO 00/22120 PCT/FR99/02465
36
Les foyers de transformation sont alors visualisés et comptés.
Les plasmides utilisés au cours de cette série d'expériences sont les
suivants:
- plasmide tampon: pSGS (Stratagene)
- plasmide d'expression de la protéine Ras-Va112: pEJ-Ras (Shih & Weinberg,
Cell 29 ( 1982) 161 )
- plasmide d'expression de la protéine c-myc entière: pSVc-mycl (Land et al,
Nature 304 ( 1983) 596)
- plasmide d'expression de la protéine H 175: pSV2-H 175 (exemple 4-a (iii))
- plasmide d'expression de la protéine C-mbpl: pBFA107-C-MBP1(exemple 4-a
(ü))
Chaque point de transfection contient un mélange de trois plasmides â raison
de 7 pg de chaque plasmide. Les résultats de deux expériences indépendantes
sont
reportés dans le Tableau 3.
CA 02347121 2001-04-09
WO 00/22120 PCT/FR99/02465
37
Expérience I Expérience 2
contrôle 0 0
Ras-Val 12 0 0
c-myc 0 NT
H 175 0 NT
C-mbp 1 0 NT
Ras-Va112 + c-myc 111 16
Ras-Val l 2 + c-myc + C-mbp113 12
1
Ras-Val 12 + H 175 0 16
Ras-Val 12 + C-mbp 1 0 3
Ras-Val 12 + H 175 + C-mbp13 30
1
Tableau 3. Effet de la protéine C-mbp 1 sur la coopération oncogénique entre
la
protéine H 175 et la protéine Ras-Val l 2 (NT: non testé , *: expérience
effectuée avec
3 wg de plasmide pSVc-myc 1 )
Les résultats de ces expériences montrent que
C-mbp 1 peut coopérer avec Ia forme activée de Ras pour la transformation
des REF
- il existe une synergie entre les protéines H 175 et C-mbp 1 dans la
coopération oncogénique avec Ras qui est spécifique de cette association car C-
mbp 1
ne présente aucun effet sur la coopération oncogénique Ras / c-myc.
Exempte 6 - Effet des protéines C-mbpl et C-fibuline2 et relation avec les
effets
des différentes formes de la protéine p53 sur la croissance cellulaire des
cellules
tumorales
CA 02347121 2001-04-09
WO 00/22120 PCT/FR99/02465
38
Cet exemple décrit les effets des protéines C-mbpl et C-fibuline2 sur la
croissance cellulaire des cellules tumorales et leur relation avec les effets
des
différentes formes de la protéine p53 sur cette même croissance cellulaire.
Ces effets des protéines C-mbpl et C-fibuline2 sur la croissance cellulaire
ont
été testés sur la lignée cellulaire H 1299 dans une expérience de formation de
colonies
résistantes à la néomycine suite à la transfection par des plasmides exprimant
ces
protéines.
Ces expériences de transfection ont été effectuées selon le protocole décrit
dans l'exemple 4-b en utilisant 105 cellules par point et 1,5 p.g d'ADN total.
Les plasmides utilisés au cours de cette série d'expériences sont les
suivants:
- plasmides d'expression des protéines p53 et H175: pSV2-p53 et pSV2-H175.
- plasmide d'expression de la protéine C-mbp 1: pBFA 107-C-MBP 1 (exemple 4-a
(ü))
- plasmide d'expression de la protéine C-fibuline2: pBFA107-C-FIB2 (exemple 4-
a
(ü))
- plasmide conférant la résistance à la néomycine: pSV2-Neo pour une quantité
totale
de 0,4 pg
Protocole de formation de colonies résistantes à la néomycine . 48h après
transfection, les cellules sont grattées et transferées dans 2 boites de Pétri
de 10 cm de
diamètre et remises en culture avec 10 ml de milieu DMEM additionné de 10% de
sérum de veau foetal inactivé à la chaleur et contenant 400 pg/ml de
généticine
(G418). Après une sélection de 15 jours en présence de 6418, le nombre de
colonies
NeoR est déterminé par comptage après coloration à la fuchsine.
Les résultat de ces expériences sont reportés dans les Tableaux 4 et 5.
CA 02347121 2001-04-09
WO 00/22120 PCT/FR99/02465
39
Nombre de colonies résistantes à la Néomycine
Protéine exprimée Expérience 1 Expérience 2 Expérience 3 moyenné
Vecteur 36 (1,00) 52 (1,00) 73 (I,00) 1,00
C-mbpl100ng 45 (1,25) 49 (1,06) 69 (0,95) 1,09
C-mbplSOOng S1 (1,42) 71 (1,37) 110(1,51) 1,43
C-mbpl1000ng 70( 1,94} 83 (1,60) 160 (2,19) 1,90
p53 7 (0,19) 12 (0,23) 10 (0,14) 0,19
sauvage
100ng
C-mbp 100ng 6 (0,17) 14 (0,27) 8 (0,11 ) 0,18
1
C-mbp SOOng 19 (0,53) 28 (0,54) 23 (0,32) 0,46
1
C-mbpl1000ng 32 (0,89) 50 (0,96) 51 (0,70) 0,85
p53 2 (0,06) 5 (0,10) 8 (0,11 ) 0,08
sauvage
200ng
C-mbp 100ng 2 (0,06) 4 (0,08) 6 (0,08) 0,08
1
C-mbp SOOng S (0,14) 8 (0,15) 16 (0,22) 0,17
1
C-mbpl1000ng 9 (0,25) 20 (0,38) 28 (0,38) 0,35
H175 0ng 41 (1,14) 47 (0,90) 61 (0,84) 0,96
C-mbpl100ng 33 (0,92) 65 (1,25) 70 (0,96) 1,04
C-mbplSOOng 67 (1,86) 101 (1,94) 123 (1,68) 1,83
C-mbpl1000ng 162 (4,50) 128 (2,46) 316 (4,33) 3,76
H175 39 (1,08) 60 (1,15) 66 (1,10) 1,11
200ng
C-mbpl100ng 43 (1,19) 54 (1,04) 75 (1,03) 1,10
C-mbplSOOng 59 (1,64) 129 (2,48) 163 (2,23) 2,12
C-mbp 1000ng 131 (3,64) 282 (5,42) 299 (4,10) 4,39
1
Tableau 4: Effet de la protéine C-mbp 1 sur la croissance cellulaire de
cellules
5 tumorales
CA 02347121 2001-04-09
WO 00/22120 PCT/FR99/02465
Nombre de colonies résistantes à la Néomycine
Protéine exprimée Expérience 1 Expérience 2 Expérience 3 moyenne
Vecteur 36 (1,00) 52 (1,00) 73 (1,00) 1,00
C-fibuline2 100ng35 (0,97) 56 (1,08) 80 (1,10) 1,05
C-fibuline2 SOOng48 (1,33) 68 (1,31) 102 (1,40) 1,35
C-fibuline2 1000ng60( 1,67) 87 (1,67) 194 (2,66) 2,00
p53 sauvage 100ng7 (0,19) 12 (0,23) 10 (0,14) 0,19
C-fibuline2 100ng10 (0,28) 11 (0,21 13 (0,18) 0,22
)
C-fibuline2 SOOngIS (0,42) 30 (0,58) 26 (0,36) 0,45
C-fibuline2 1000ng35 (0,97) 44 (0,85) 45 (0,62) 0,81
p53 sauvage 200ng2 (0,06) 5 (0,10) 8 (0,11 ) 0,09
C-fibuline2 1 3 (0,08) 6 (0,12) 6 (0,08) 0,09
OOng
C-fibuline2 500ng4 (0,1 I 10 (0, I 16 (0,22) 0,16
) 9)
C-fibuline2 1000ng10 (0,28) 18 (0,35) 28 (0,38) 0,34
H175 100ng 41 (1,14) 4? (0,90) 6I {0,84) 0,96
C-fibuline2 100ng47 (1,31) 54 (1,04) 84 (1,15) 1,17
C-fibuline2 SOOng84 (2,33) 95 (1,83) 156 (2,14) 2,10
C-fibuline2 1000ng143 (3,97) 138 (2,65) 270 (3,70) 3,44
H175 200ng 39 (1,08) 60 (1,15) 66 (1,10) 1,11
C-fibuline2 100ng51 (1,42) 63 (1,21) 80 (1,10) 1,24
C-fibuline2 SOOng74 (2,06) 146 (2,81) 142 (1,95) 2,27
C-fibuline2 1000ng158 (4,39) 230 (4,42) 284 (3,89) 4,23
Tableau 5: Effet de la protéine C-fibuline2 sur la croissance cellulaire de
cellules
tumorales
Les résultats de ces expériences montrent que
5 - les protéines C-mbp 1 et C-fibuline2 ont un effet positif sur la
croissance
cellulaire
- les protéines C-mbpl et C-fibuline2 sont capables de bloquer l'effet anti-
prolifératif de la protéine p53, et ce indépendamment de leur effet
prolifératif
CA 02347121 2001-04-09
WO 00/22120 PCT/FR99/02465
41
- l'effet prolifératif des protéines C-mbpl et C-fibuline2 est fortement
augmenté en présence de la protéine H175
Exemple 7 - Clonage des ADNc codant pour la forme entière des protéines
MBP1 murine et humaine.
Cet exemple décrit le clonage des ADNc codant pour la protéine MBP 1
murine entière et l'utilisation de ces dônnées pour le clonage d'un homologue
humain
de MBP 1.
7 a - Clonage de l'ADNc codant pour la forme entière de la protéine mbpl
murine
L'ADNc codant pour la partie C-terminale de la protéine mbp 1 murine a été
cloné par réaction d'amplification en chaine (PCR) sur l'ADN de la banque
SuperScript d'embryon murin (8,5 jours) (Gibco BRL) en utilisant
l'oligonucléotide
3'-mMBP 1 et l'oligonucléotide SP6 (Gibco BRL).
Oligonucléotide 3'-mMBP 1 (SEQ ID N° 14)
CGGTACTGGCAGAGGTAACTGG
Cette amplification a permis d'obtenir un produit unique présentant une taille
d'environ 800 paires de bases qui a ensuite été cloné directement après PCR
dans le
vecteur pCRII (Invitrogene) et séquencé. La séquence ainsi obtenue (SEQ ID
N° 23)
montre un recouvrement de 368 paires de bases avec C-MBP 1 (SEQ ID N°
8) avec
une identité stricte de séquence sur cette partie commune. De plus, en 5' de
cette
partie commune, on trouve une séquence additionnelle de 445 paires de bases
présentant une phase de lecture ouverte et un codon d'initiation de la
traduction.
Les deux fragments représentés par ces séquences ont ensuite été rassemblés
par une ligation à trois partenaires en utilisant les sites de reconnaissance
par les
enzymes de restriction EcoR I, Pst I et Not I et le plasmide pBC-SK+
CA 02347121 2001-04-09
WO 00/22120 PCT/FR99/02465
42
(STRATAGENE) permettant ainsi la reconstitution de l'ADNc MBP1 murin entier
(mMBP 1 ) (SEQ ID N° I 5).
7-b Clonage de l'ADNc codant pour la forme entière de la protéine mbpl
humaine
La séquence du gène murin. MBP 1 a été utilisée pour une recherche
d'homologie dans Genbank. Cette recherche a permis de montrer une forte
homologie avec la séquence d'une EST humaine (g1548384). A partir de cette
séquence, deux fragments d'ADNc ont été clonés par réaction d'amplification en
chaine (PCR} sur l'ADN de la banque SuperScript de testicule humain (Gibco
BRL)
en utilisant les oligonucléotides 3'-hMBPI et SP6 (Gibco BRL) d'une part, et
les
oligonucléotides 5'-hMBP 1 et T7 (Gibco BRL) d'autre part.
Oligonucléotide 3'-hMBP 1 (SEQ ID N° 17)
CTCCGCTCCGAGGTGATGGTC
Oligonucléotide 5'-hMBP 1 (SEQ ID N° 18)
TGTAGCTACTCCAGCTACCTC
Ces amplifications ont permis d'obtenir deux produits présentant des tailles
d'environ 1100 et 700 paires de bases qui ont ensuite été clonés directement
après
PCR dans le vecteur pCRII (Invitrogene) et séquencés. Les séquences ainsi
obtenues
(SEQ ID N° 19 et SEQ ID N° 20) montrent un recouvrement de 325
paires avec une
identité stricte de séquence sur cette partie commune.
Les deux fragments représentés par ces séquences ont ensuite été rassemblés
par une ligation à trois partenaires en utilisant les sites de reconnaissance
par les
enzymes de restriction EcoR I, Nco I et Not I et le plasmide pBC-SK+
(STRATAGENE) permettant ainsi la reconstitution de l'ADNc MBP1 humain entier
(hMBP 1 ) (SEQ ID N° 21 ) présentant une phase de lecture ouverte et un
codon
d'initiation de la traduction.
CA 02347121 2001-04-09
WO 00/Z2120 PCT/FR99/02465
43
La comparaison des séquences protéiques correspondant aux ADNc
précédemment obtenus (Exemples 7-a et 7-b) (Figure 4) montre une identité
stricte
de 95% dans la phase ouverte de lecture supposée (après le site de démarrage
de la
traduction (ATG) supposé). Par contre, une absence d'identité et une très
mauvaise
homologie sont observées entre les régions situées en amont de ce site de
démarrage
de la traduction (ATG) putatif. Ces données permettent donc de confirmer cette
position comme démarrage de la traduction et par là même que ces deux ADNc
codent bien pour les formes entières des protéines MBP 1 humaine (SEQ ID
N°22) et
marine (SEQ ID N° 16).
7 - c Construction des plasmides d'expression en cellules mammifères des
formes marine et humaine de la protéine mbpl
Les ADNc codant pour les formes marine et humaine de la protéine MBP 1
contenus dans le vecteur pBC SK+, ont été insérés dans les vecteurs
d'expression
pSV2 et pcDNA3 (Invitrogen) en utilisant les sites de reconnaissance par les
enzymes
de restriction Hind III et Not I.
Exemple 8 - Effets comparés des protéines C-mbpl et mbpl marine sur la
croissance cellulaire des cellules tumorales
Cet exemple décrit les effets comparés des protéines C-mbp 1 et mbp 1 marine
sur la croissance cellulaire des cellules tumorales et leur relation avec les
effets de la
protéine H 175 sur cette même croissance cellulaire.
Ces effets de la protéine mbp 1 marine sur la croissance cellulaire ont été
testés sur la lignée cellulaire H 1299 dans une expérience de formation de
colonies
résistantes à la néomycine suite à la transfection par des plasmides portant
les ADNc
codant pour ces protéines.
Ces expériences de transfection ont été effectuées selon le protocole décrit
dans l'exemple D2 en utilisant 105 cellules par point et 1,5 p.g d'ADN total.
CA 02347121 2001-04-09
WO 00/22120 PCT/FR99/02465
44
Les plasmides utilisés au cours de cette série d'expériences sont les
suivants:
- plasmide d'expression de la protéine H175: pSV2-H175.
- plasmide d' expression de la protéine C-mbp 1: pBFA 107-C-MBP 1 (exemple 4-a
(ü))
- plasmide d'expression de la protéine mbpl murine: pSV2-mMBPI (exemple 7-c)
- plasmide conférant la résistance à la néomycine: pSV2-Neo pour une quantité
totale
de 0,4 pg
Le protocole de formation de colonies résistantes à la néomycine utilisé est
celui décrit dans l'exemple 6. Les résultat de cette expérience sont présentés
dans le
Tableau 6 et la Figure 5.
Nombre de colonies résistantes à la Néomycine
Protéine exprimée Expérience 1 Expérience 2
Vecteur 61 ( 1,00) 71 ( 1,00)
C-mbp 100ng 67 ( I ,10)
1
C-mbplSOOng 96 ( 1,57)
C-mbp 1000ng 278 ( 4,56) 239 ( 3,37)
1
mbpl 100ng 94 ( 1,54)
mbp SOOng 128 ( 2,10)
1
mbpl 1000ng 419 ( 6,87) 562 ( 7,92)
HI75 65 ( 1,07) 69 ( 0,97)
200ng
C-mbp 1 OOng 72 ( 1,18 )
1
C-mbplSOOng 134 ( 2,20)
C-mbp 1 OOOng 397 ( 6,51 ) 341 ( 4,80)
1
mbpl 100ng 81 ( 1,33)
mbpl SOOng 206 ( 3,38)
mbp 1 OOOng 729 ( 11,95) 12 I 5 ( 17,11
1 )
CA 02347121 2001-04-09
WO 00/22120 PCT/FR99/02465
Tableau 6 : Effets comparés des protéines C-mbp 1 et mbp 1 murine sur la
croissance
cellulaire de cellules tumorales
Les résultats de cette expérience montrent que la protéine mbp 1 murine
5 présente les mêmes caractéristiques que la protéine C-mbpl, à savoir un
effet positif
sur la croissance cellulaire qui est fortement augmenté en présence de la
protéine
H 175.
De plus cet effet de la protéine mbpl est très fortement augmenté par rapport
à la protéine tronquée C-mbp 1.
Exemple 8 bis - Coopération oncogénique des protéines C-mbpl et mbpl
murine et mbpl humaine avec la protéine Ras-Va112
Cet exemple décrit les effets comparés des protéines C-mbp 1, mbp 1 murine et
mbpl humaine dans une expérience de coopération oncogénique avec la protéine
Ras-Va112.
Cette coopération oncogénique a été testée sur des fibroblastes embryonnaires
de rat suite à la transfection par des plasmides portant les ADNc codant pour
ces
protéines et suivant le protocole décrit dans l'exemple 5.
Les résultats de cette expérience sont présentés dans le Tableau 7.
CA 02347121 2001-04-09
WO 00/22120 PCT/FR99/02465
46
Expérience 1 Expérience 2
contrle 0 0
Ras-Val l 2 0 0
c-myc 0 0
H 175 0 0
C-mbp 1 0 0
mbp 1 0 0
Ras-Val I2 + c-myc 3 I 42
Ras-Val I2 + H 175 10 15
Ras-Va112 + C-mbp 1 4 4
Ras-Val 12 + mbp 1 murine 5 7
Ras-Va112 + mbpi humaine 6 6
Tableau 7: Coopération oncogénique des protéines C-mbp 1, mbp 1 murine et mbp
1
humaine avec la protéine Ras-Va112
Les résultats de cette expérience montrent que les protéine MBP 1 murine et
MBP 1 humaine présentent les mêmes caractéristiques que la protéine C-mbp 1, à
savoir la capacité de coopérer avec la protéine Ras-Va112 pour la
transformation de
fibroblastes embryonnaires de rat.
De façon intéressante, on note aussi que les fibroblastes ainsi transformés
présentent un aspect morphologique tout à fait particulier qui diffère de
celui obtenu
avec l' oncogène c-myc.
Exempte 9 - Expression de la protéine MBP1 chez la souris et dans les tissus
humains
CA 02347121 2001-04-09
WO 00/22120 PCT/FR99/02465
47
Cet exemple décrit l'étude de l'expression de l'ARN messager de MBP 1 chez
la souris et dans différents tissus humains.
9-a Préparation des sondes
Les sondes mMBP 1 et hMBP 1 sont constituées par les ADNc correspondants.
La sonde GAPDH (contrôle) a été générée par réaction d'amplification en
chaine (PCR) sur l'ADN de la banque SuperScript de testicule humain (Gibco
BRL)
(GAPDH) en utilisant les oligonucléotides suivants
Oligonucléotide sens-GAPDH (SEQ ID N° 24)
CGGAGTCAACGGATTTGGTCGTAT
Oligonucléotide antisens-GAPDH(SEQ ID N° 25)
AGCCTTCTCCATGGTGGTGAAGAC
Les sondes ont été radiomarquées au 'ZP-dCTP en utilisant le kit Rediprime
(Amersharn) et les recommandations du fournisseur, et les nucléotides non
incorporés
ont été éliminés par chromatographie sur des colonnes MicroSpin G-25
(Pharmacie
Biotech). Les Northern blots utilisés lors de cette expérience ont été obtenus
chez
Clontech. Les membranes ont été préhybridées avec la solution ExpressHyb
(Clontech) 45 minutes à 65°c, puis incubées 2 heures avec les sondes
radiomarquées
à 65°C, lavées trois fois avec du tampon 2xSSC deux fois avec du tampon
2xSSC
additionné de 0,1% de SDS et enfin lavées avec du tampon 0,2xSSC additionné de
0,1% de SDS jusqu'à disparition du bruit de fond. Les membranes ont ensuite
été
soumises a autoradiographie et une quantification du signal a été effectuée à
l'aide
d'un instantimager (Packard instruments).
9-b Expression de la protéine MBPI chez la souris
CA 02347121 2001-04-09
WO 00/22120 PCT/FR99/02465
48
Cet exemple décrit l'étude de l'expression de l'ARN messager de MBP 1 chez
la souris.
Les sondes utilisées dans cette expérience sont ies sondes mMBP 1 et GAPDH
. Les membranes utilisées dans cette expérience contiennent l'une des ARNm
d'embryon de souris obtenus à différents stages du développement, et l'autre
des
ARNm représentatifs de différents tissus de souris adulte.
Les résultats de cette expérience (Figure 6) indiquent clairement que:
1 - un transcript unique de 1,8 kb est détécté aussi bien dans les ARNm
d'embryon de
souris que dans les tissus de souris adulte.
2 - ce messager présente des variations de niveaux d'expression au cours du
développement, avec une forte abondance dans les stades précoces (7 jours)
puis une
diminution importante (I 1 jours) pour atteindre un niveau apparement
constant.
3 - ce messager est modérément exprimé dans l'ensemble des tissus adultes
testé à
l'exception d'une expression importante dans le poumon et les testicules.
Un tel niveau d'expression de transcript élevée dans une phase du
développement embryonnaire ainsi que dans des tissus présentant un taux de
croissance élevé, confirme l'implication du produit du gène MBP1 dans les
processus
de croissance cellulaire mise en évidence dans les exemples 5, 6 et 7.
9-c Expression de la protéine MBP1 dans les tissus humains
Cet exemple décrit l'étude de l'expression de l'ARN messager de MBP 1 dans
différents tissus humains.
Les sondes utilisées dans cette expérience sont les sondes hMBP 1 et GAPDH
Les membranes utilisées contiennent des ARNm représentatifs de différents
tissus
humain.
CA 02347121 2001-04-09
WO 00/22120 PCT/FR99/02465
49
Les résultats de cette expérience (Figure 7) indiquent clairement que:
1 - deux transcripts de I,5 et 1,8 kb sont détéctés dans les tissus humains.
2 - ces messagers sont modérément exprimés dans l'ensemble des tissus testés
et leur
profil d'expression est comparable à celui du messager murin (forte expression
dans
le poumon et les testicules).
Ces résultats montrent que:
- il peut exister deux formes différentes de la protéine mbp 1 humaine avec
possibilité d'épissage alternatif du messager.
- les ARNm codant pour la(les) protéines) mbp 1 humaines) tout comme leur
homologue murin présentent un niveau d'expression élevé dans des tissus à taux
de
croissance élevé, et que le(s) produits) du gène MBP 1 humain pourrait(ent)
donc
aussi être impliqués) dans les processus de croissance cellulaire.
Les résultats présentés dans les différents exemples montrent que les
protéines
C-mbp 1, MBP 1 et C-fibuline2 interagissent spécifiquement avec les formes
mutantes
de la protéine p53 et que ces interactions conduisent à une synergie entre ces
protéines et les mutants oncogéniques de la protéine p53 que ce soit pour la
coopération oncogénique avec la forme activée de la protéine Ras ou pour
l'effet
proliférati~
De plus, les protéines C-mbp I , MBP 1 et C-fibuline2 présentent une activité
proliférative intrinsèque, et la protéine C-mbp 1 agit comme un oncogène
immortalisant en coopérant avec la forme activée de la protéine Ras pour la
transformation cellulaire.
Les résultats présentés dans les différents exemples montrent que les
protéines
ou polypeptides C-mbpl, MBP1 et C-fibuline2 interagissent spécifiquement avec
les
formes mutantes de la protéine p53 et que ces interactions conduisent à une
synergie
entre ces protéines et les mutants oncogéniques de la protéine p53 que ce soit
pour la
CA 02347121 2001-04-09
WO 00/22120 PCT/FR99/02465
coopération oncogénique avec la forme activée de la protéine Ras ou pour
l'effet
prolifératif.
De plus, les protéines C-mbpl, MBP1 et C-fibuline2 présentent une activité
proliférative intrinsèque, et les protéines C-mbp 1 et MBP 1 agissent comme
des
5 oncogénes immortalisants en coopérant avec la forme activée de la protéine
Ras pour
la transformation cellulaire.
Ces propriétés confèrent à MBP1 un rôle potentiel d'oncogène. Dans au
moins un des test (exemple 8 bis) la protéine MBP 1 présente des propriétés
oncogéniques accrues par rapport au polypeptide c-MBP I .
10 Enfin, la forte homologie présentée par les protéines MBP1 humaine et
murine (95% d'identité stricte), et la similarité d'expression tissulaire de
leurs
messagers respectifs, permettent de conclure que la protéine MBP 1 humaine,
qui
pourrait être présente sous forme de deux variants différents (2 messagers
distincts),
possède(ent) des propriétés analogues à celles de son homologue W urine.
15 En conclusion, ces résultats décrivent la caractérisation d'une nouvelle
protéine murine, MBPI, et de son(ses) homologues) humaine(s), qui présente des
propriétés oncogéniques et qui interagit spécifiquement avec les formes mutées
de la
protéine p53. Cette interaction qui se traduit par un accroissement des
propriétés
oncogéniques de MBP1, pourrait constituer un élément fondamental de la
capacité
20 oncogénique de ces mutants de la protéine p53.
De telles propriétés semblent aussi partagées par une autre protéine
présentant
des homologies avec MBP1, la fibuline 2. Cette protéine faisant partie d'une
famille
plus large, on peut envisager que ces propriétés puissent être étendues à
l'ensemble
des membres de la famille des fibulines.
25 Ces interactions montrant une forte synergie entre les pouvoirs
oncogéniques
des protéines MBPI, fbuline2 et mutants p53, elles constituent un point
d'action
potentiel dans le traitement des cancers liés aux mutations de Ia protéine
p53. De
CA 02347121 2001-04-09
WO 00/22120 PCT/FR99/02465
51
plus, les protéines MBP 1 et fibuline2 qui présentent des propriétés
oncogéniques
intrinsèques constituent des cibles potentielles pour le traitement du cancer
en
général.
Exemple 10 - Localisation chromosomique du gène MBPI humain
La localisation chromosomique du gène MBPI a été effectué selon un
protocole en quatre étapes (Lichter èt al, Science 247 ( 1990) 64) (Heng et
al,
Chromosoma 102 ( 1993) 325) ( Kischkel et al, Cytogenet. Cell Genet. 82 (
1998) 95)
- marquage de l'ADNc à la biotine par nick-translation
- hybridation sur métaphases humaines normales (technologie en haute
résolution)
- révélation par la fluorescéine
- visualisation et interprétation sur microscope à épifluorescence
Cette étude d'hybridation de la sonde MBP 1 sur métaphases humaines a été
effectuée par analyse de 30 mitoses et a montré la présence d'un double spot
sur les
bras longs (bras q) des deux chromosomes 1 I en 11 q 13. De façon
intéressante, cette
région du chromosome 11 a été associée à un grand nombre de pathologies
- maladie de Mac Ardle (Lebo et al, Science 225 ( 1984) 57)
- Syndrome de Usher de type 1B (WeiI et al, Nature 374 (1995) 60)
- néoplasie endocrine de type I (Teh et al, J. Intern. Med. 238 (1995) 249)
- dystrophie de Best (Graff et al, Genomics 24 ( 1994) 425)
- diabète insulino-dépendent (Davies et al, Nature 37 I ( 1994) 130)
- spinocerebellar ataxia 5 (Ranum et al, Nature Genet. 8 ( 1994) 280)
- syndrome de Bardet-Biedl (Leppert et al, Nature Genet. 7 (1994) 108)
- ostéoporose (Gong et al, Am. J. Hum. Genet. 59 (1996} 146)
De plus cette région du chromosome 11 est aussi le site d'évènements
d'amplification associées à différentes de tumeurs solides (oesophage, tête et
cou,
CA 02347121 2001-04-09
WO 00/22120 PCT1FR99/02465
52
vessie, sein et poumon) ( Lammie & Peters, Cancer Cells 3 (1991) 413).
Le gène MBPI pourrait donc être non seulement associé à un certain nombre
de cancers mais aussi à un grand nombre de pathologies présentant des
désordres de
types rénaux, neuro-dégénératifs, osseux et autres. Parmi ces pathologies on
peut
citer notamment : les déficiences rénales aigues telles celles associées à la
maladie de
Mac Ardle, les retinis pigmentosa et certaines formes de cécité et de surdité
telles que
celles associées au Syndrome de Usher de type 1B, l'hyperthyroïdie telle la
forme
associée à la néoplasie endocrine de type I, les pathologies liées à un défaut
de
pigmentation rétinienne telles que celles rencontrées dans la dystrophie de
Best, le
diabète insulino-dépendant, les pathologies neurodégénératives teiles que
celles
associées à l'ataxie cérébrospinale 5, les dystrophies rétiniennes, les
désordres rénaux
telles que les formes rencontrées dans le syndrome de Bardet-Biedl, et
l'ostéoporose.
Exemple 11 - Expression de l'Ai2N messager codant pour la protéine MBPl
humaine dans des tumeurs du colon
Cet exemple décrit une analyse semi-quantitative de l'expression de l'ARN
messager codant pour la protéine MBP 1 humaine, effectuée en parallèle sur 9
tumeurs du colon et 9 prélèvements de tissus sains (colon) provenant des mêmes
patients.
Les prélèvements ont été congelés à -70°C immédiatement après
ressection et
l'ARN total a été préparé par homogénéisation de IOOmg de tissu en utilisant
la
solution RNA NOW (Ozyme) et le protocole recommandé par le fournisseur. Par la
suite, une synthèse d'ADNc a été effectuée à l'aide du kit First-Strand cDNA
Synthesis (Amersham Pharmacia Biotech) en utilisant 1,5 pg d'ARN total et
selon les
recommandations du fournisseur. Puis les gènes MBP 1 et (3-actine (contrôle)
ont été
amplifiés par PCR en utilisant une quantité d'ADNc pour laquelle le niveau de
produit de PCR est directement corrélable avec la concentration de substrat et
le
programme de cycles suivant
CA 02347121 2001-04-09
WO 00/22120 PCT/FR99/02465
53
1 cycle 2min à 95°C
30 cycles 30sec à 94
lmin à 45°C
1 min à 72°C
1 cycle 3min à 72°C
Les oligonucléotides utilisés pour ces amplifications sont les suivants
Oligonucléotide sens-MBP 1 (SEQ ID N° 27)
GCCCTGATGGTTACCGCAAGA
Oligonucléotide antisens-MBP 1 (SEQ ID N° 28)
AGCCCCCATGGAAGTTGACAC
Oligonucléotide sens-~3 -actin (SEQ ID N° 29)
GTGGGGCGCCCCAGGCACCA
Oligonucléotide antisens-~3-actin (SEQ ID N° 26)
CGGTTGGCCTTGGGGTTCAGGGGGG
Les produits de PCR ainsi générés ont ensuite été analysés par électrophorèses
sur gel d'agarose à 1%.
Les résultats présentés dans la figure 8 montrent clairement que l'ARN
messager codant pour la protéine MBP1 humaine est amplifié dans cinq des
tumeurs
étudiées en comparaison avec le tissu sain provenant du même patient, et ce,
quelque
soit le grade de la tumeur et indépendamment de leur statut concernant les
gènes Ras
et p53.
Les résultats de cet exemple montrent donc qu'une amplification de l'ARN
messager codant pour Ia protéine MBP 1 humaine peut-être décelée dans certains
CA 02347121 2001-04-09
WO 00/22120 PCT/FR99/02465
54
types de tumeurs humaines et soulignent donc un rôle potentiel de la protéine
MBP1
dans l'apparition et/ou le developpement de ces tumeurs.
CA 02347121 2001-04-09
WO 00/22120 PCT/FR99/02465
1
LISTE DE SEQUENCES
<110> Rh8ne-Poulenc Rorer
<120> Polypeptides capables d'interagir avec les mutants
oncogéniques de la protéine p53
<130> Séquences
<140>
<141>
<150> FR9812754
<151> 1998-10-12
<160> 33
<170> PatentIn Ver. 2.1
<210> 1
<211> 23
<212> ADN
<213> Séquence artificielle
<z2o>
<223> Description de la séquence artificielle:
oligonucléotide
<400> 1
agatctgtat ggaggagccg cag 23
<210> 2
<211> 29
<zi2> ADN
<213> Séquence artificielle
<220>
<223> Description de la séquence artificielle:
oligonucléotide3'-393 (p53)
<400> 2
agatctcatc agtctgagtc aggcccttc 2g
<210> 3
<211> 15
<212> ADN
<2i3> Séquence artificielle
<220>
<223> Description de la séquence artificielle:
oligonucléotide H175 3'
<400> 3
ggggcagtgc ctcac 15
<210> 4
CA 02347121 2001-04-09
WO 00/22120 PCT/FR99/02465
2
<211> 15
<212> ADN
<213> Séquence artificielle
<z2o>
<223> Description de la séquence artificielle:
oligonucléotide W248 3'
<400> 4
gggcctccag ttcat 15
<210> 5
<211> 15
<212> ADN
<213> Séquence artificielle
<220>
<223> Description de la séquence artificielle:
oligonucléotide H273 3'
<400> 5
acaaacatgc acctc 15
<210> 6
<211> 15
<212> ADN
<213> Séquence artificielle
<220>
<223> Description de la séquence artificielle:
oligonucléotide 6281 3'
<400> s
gcgccggcct ctccc 15
<210> 7
<211> 23
<212> ADN
<213> Séquence artificielle
<220>
<223> Description de la séquence artificielle:
oligonucléotide 5'-73
<400> 7
agatctgtgt ggcccctgca cca 23
<zlo> s
<211> 1021
<212> ADN
<213> Séquence artificielle
<220>
<221> CDS
<222> (1}..(885}
CA 02347121 2001-04-09
WO 00/22120 PCT/FR99/02465
3
<220>
<223> Description de la squence artificielle: fragment
C-term MBP1 murine
<400> 8
tgc acc tgc cct gat ggt tac cga aaa att gga ccc 48
gaa tgt gtg gac
Cys Thr Cys Pro Asp Gly Tyr Arg Lys Ile Gly Pro
Glu Cys Val Asp
1 5 10 15
ata gat gag tgt cgt tac cgc tat tgc cag cat cga 96
tgt gtg sac ctg
Ile Asp Glu Cys Arg Tyr Arg Tyr Cys Gln His Arg
Cys Val Asn Leu
20 25 30
ccg ggc tcc ttt cga tgc cag tgt gag cca ggc ttc 144
cag ttg gga cct
Pro Gly Ser Phe Arg Cys Gln Cys Glu Pro Gly Phe
Gln Leu Gly Pro
35 40 45
sac sac cgc tct tgt gtg gat gtg aat gag tgt gac 192
atg gga gcc cca
Asn Asn Arg Ser Cys Val Asp Val Asn Glu Cys Asp
Met Gly Ala Pro
50 55 60 '
tgt gag cag cgc tgc ttc sac tcc tat ggg acc ttc 240
ctg tgt cgc tgt
Cys Glu Gln Arg Cys Phe Asn Ser Tyr Gly Thr Phe
Leu Cys Arg Cys
65 70 75 80
sac cag ggc tat gag ctg cac cgg gat ggc ttc tcc 288
tgc agc gat atc
Asn Gln Gly Tyr Glu Leu His Arg Asp Gly Phe Ser
Cys Ser Asp Ile
85 90 95
gat gag tgc ggc tac tcc agt tac ctc tgc cag tac 336
cgc tgt gtc sac
Asp Glu Cya Gly Tyr Ser Ser Tyr Leu Cys Gln Tyr
Arg Cys Val Asn
100 105 110
gag cca ggc cga ttc tcc tgt cac tgc cca cas ggc 384
tac cag ctg ctg
Glu Pro Gly Arg Phe Ser Cys His Cys Pro Gln Gly
Tyr Gln Leu Leu
115 120 125
gct aca agg ctc tgc cas gat att gac gag tgt gaa 432
aca ggt gca cac
Ala Thr Arg Leu Cys Gln Asp Ile Asp Glu Cys Glu
Thr Gly Ala His
130 135 140
cas tgt tct gag gcc cas acc tgt gtc sac ttc cat 480
ggg ggt tac cgc
Gln Cys Ser Glu Ala Gln Thr Cys Val Asn Phe His
Gly Gly Tyr Arg
145 150 155 160
tgt gtg gac acc sac cgt tgt gtg gag ccc tat gtc 528
cas gtg tca gac
Cys Val Asp Thr Asn Arg Cys Val Glu Pro Tyr Val
Gln Val Ser Asp
165 170 175
sac cgc tgc ctc tgc cct gcc tcc aat ccc ctt tgt 576
cga gag cag cct
Asn Arg Cys Leu Cys Pro Ala Ser Asn Pro Leu Cys
Arg Glu Gln Pro
180 185 190
tca tcc att gtg cac cgc tac atg agc atc acc tca 624
gag cga agt gtg
Ser Ser IIe Val His Arg Tyr Met Ser Ile Thr Ser
Glu Arg Ser Val
195 200 205
cct gct gac gtg ttt cag atc cag gca acc tct gtc 672
tac cct ggt gcc
CA 02347121 2001-04-09
WO 00/22120 PCT/FR99/02465
4
Pro Ala Asp Val Phe Gln ile Gln Ala Thr Ser Val Tyr Pro Gly Ala
210 215 220
tac aat gcc ttt cag atc cgt tct gga aac aca cag ggg gac ttc tac 720
Tyr Asn Ala Phe Gln Ile Arg Ser Gly Asn Thr Gln Gly Asp Phe Tyr
225 230 235 240
att agg caa atc aac aat gtc agc gcc atg ctg gtc ctc gcc agg cca 768
Ile Arg Gln Ile Asn Asn Val Ser Ala Met Leu Val Leu Ala Arg Pro
245 250 255
gtg acg gga ccc cgg gag tac gtg ctg gac ctg gag atg gtc acc atg 816
Val Thr Gly Pro Arg Glu Tyr Val Leu Asp Leu Glu Met Val Thr Met
260 265 270
aat tcc ctt atg agc tac cgg gcc agc tct gta ctg aga ctc acg gtc 864
Asn Ser Leu Met Ser Tyr Arg Ala Ser Ser Val Leu Arg Leu Thr Val
275 280 285
2~ ttt gtg gga gcc tat acc ttc tgaagaccct cagggaaggg ccatgtgggg 915
Phe Val Gly Ala Tyr Thr Phe
290 295
gccccttccc cctcccatag cttaagcagc cccgggggcc tagggatgac cgttctgctt 975
aaaggaacta tgatgtgaag gacaataaag ggagaaagaa ggaaaa 1021
<210> 9
<211> 295
<212> PRT
<213> Squence artificielle
<223> Description de la
squence artificielle:
fragment
C-term MBP1 murine
<400> 9
Cys Thr Cys Pro Asp Gly LysIleGlyPro Glu ValAsp
Tyr Arg Cys
1 5 10 15
4flIle Asp Glu Cys Arg Tyr CysGlnHisArg Cys AsnLeu
Arg Tyr Val
20 25 30
Pro Gly Ser Phe Arg Cys GluProGlyPhe Gln GlyPro
Gln Cys Leu
35 40 45
Asn Asn Arg Ser Cys Val AsnGluCyaAsp Met AlaPro
Asp Val Gly
50 55 60
Glu Gln Arg Cys Phe Asn TyrGlyThrPhe Leu ArgCys
Ser Cys
5~ 65 75 80
70
Asn Gln Gly Tyr Glu Leu AspGlyPheSer Cys AspIle
His Arg Ser
85 90 95
Asp Glu Cys Gly Tyr Ser LeuCysGlnTyr Arg ValAsn
Ser Tyr Cys
100 105 110
Glu Pro Gly Arg Phe Ser CysProGlnGly Tyr LeuLeu
Cys His Gln
115 120 125
CA 02347121 2001-04-09
WO 00/22120 PCT/FR99/02465
Ala Thr Arg Leu Cys Gln Asp Ile Asp Glu Cys Glu Thr Gly Ala His
130 135 140
5 Gln Cys Ser Glu Ala Gln Thr Cys Val Asn Phe His Gly Gly Tyr Arg
145 150 155 160
Cys Val Asp Thr Asn Arg Cys Val Glu Pro Tyr Val Gln Val Ser Asp
1 O 165 170 175
Asn Arg Cys Leu Cys Pro Ala Ser Asn Pro Leu Cys Arg Glu Gln Pro
180 185 190
Ser Ser Ile Val His Arg Tyr Met Ser Ile Thr Ser Glu Arg Ser Val
195 200 205
Pro Ala Asp Val Phe Gln Ile Gln Ala Thr Ser Val Tyr Pro Gly Ala
210 215 220
Tyr Asn Ala Phe Gln Ile Arg Ser Gly Asn Thr Gln Gly Asp Phe Tyr
225 230 235 240
Ile Arg Gln Ile Asn Asn Val Ser Ala Met Leu Val Leu Ala Arg Pro
245 250 255
Val Thr Gly Pro Arg Glu Tyr Val Leu Asp Leu Glu Met Val Thr Met
260 265 270
Asn Ser Leu Met Ser Tyr Arg Ala Ser Ser Val Leu Arg Leu Thr Val
3~ 275 280 285
Phe Val Gly Ala Tyr Thr Phe
290 295
<210> lo
<211> 39
<212> ADN
4~ <213> Séquence artificielle
<220>
<223> Description de la séquence artificielle:
oligonucléotide c-myc 5'
<400> lo
gatccatgga gcagaagctg atctccgagg aggacctga 39
5~ <210> 11
<211> 39
<212> ADN
<213> Séquence artificielle
<z2o>
<223> Description de la séquence artificielle:
oligonucléotide c-myc 3'
<400> 11
CA 02347121 2001-04-09
WO 00/22120 PCT/FR99/02465
6
gatctcaggt cctcctcgga gatcagcttc tgctccatg 3g
<210> 12
<211> 45
<212> ADN
<213> Séquence artificielle
<220>
<223> Description de la séquence artificielle:
oligonucléotide MCS S'
c400> 12
gatctcggtc gacctgcatg caattcccgg gtgcggccgc gagct 45
<210> 13
<211> 37
<212> ADN
2~ <213> Séquence artificielle
<220>
<223> Description de la séquence artificielle:
oligonucléotide MCS 3'
<400> 13
cgcggccgca cccgggaatt gcatgcaggt cgaccga 37
<210> 14
<211> 22
<212> ADN
<213> Séquence artificielle
<220>
<223> Description de la séquence artificielle:
oligonucléotide 3'mMBPl
<400> 14
cggtactggc agaggtaact gg 22
<210> 15
<211> 1513
<212> ADN
<213> Sêquence artificielle
<220>
<221> CDS
5~ <222> (49)..(1377)
<220>
<223> Description de la séquence artificielle: MBP1
murine (séquence complète)
<400> 15
gctgtggcag aaacccctga cttctgccca ccacctccca gcctcagg atg ctc cct 57
Met Leu Pro
1
CA 02347121 2001-04-09
WO 00/22120 PCT/FR99/02465
7
ttt gcc tcc tgc ctc ccc ggg tct ttg ctg ctc tgg 105
gcg ttt ctg ctg
Phe Ala Ser Cys Leu Pro Gly Ser Leu Leu Leu Trp
Ala Phe Leu Leu
5 10 15
ttg ctc ttg gga gca gcg tcc cca cag gat ccc gag 153
gag ccg gac agc
Leu Leu Leu Gly Ala Ala Ser Pro Gln Asp Pro Glu
Glu Pro Asp Ser
20 25 30 35
tac acg gaa tgc aca gat ggc tat gag tgg gat gca 201
gac agc cag cac
Tyr Thr Glu Cys Thr Asp Gly Tyr Glu Trp Asp Ala
Asp Ser Gln His
40 45 50
tgc cgg gat gtc aac gag tgc ctg acc atc ccg gag 249
gct tgc aag ggt
Cys Arg Aap Val Asn Glu Cya Leu Thr Ile Pro Glu
Ala Cys Lya Gly
55 60 65
gag atg aaa tgc atc aac cac tac ggg ggt tat ttg 297
tgt ctg cct cgc
Glu Met Lys Cys Ile Asn His Tyr Gly Gly Tyr Leu
Cys Leu Pro Arg
70 75 ao
tct gct gcc gtc atc agt gat ctc cat ggt gaa gga 345
cct cca ccg cca
Ser Ala Ala Val Ile Sr Asp Leu His Gly Glu Gly
Pro Pro Pro Pro
85 90 95
gcg gcc cat gct caa caa cca aac cct tgc ccg cag 393
ggc tac gag cct
Ala Ala His Ala Gln Gln Pro Asn Pro Cys Pro Gln
GIy Tyr Glu Pro
100 105 110 115
gat gaa cag gag agc tgt gtg gat gtg gac gag tgt 441
acc cag gct ttg
Asp Glu Gln Glu Ser Cys Val Asp Val Asp Glu Cys
Thr Gln Ala Leu
120 125 130
cat gac tgt cgc cct agt cag gac tgc cat aac ctt 489
cct ggc tcc tac
His Asp Cys Arg Pro Ser Gln Asp Cys His Asn Leu
Pro Gly Ser Tyr
135 140 145
cag tgc acc tgc cct gat ggt tac cga aaa att gga 537
ccc gaa tgt gtg
Gln Cys Thr Cys Pro Asp Gly Tyr Arg Lys Ile Gly
Pro Glu Cys Val
4~ 150 155 160
gac ata gat gag tgt cgt tac cgc tat tgc cag cat 585
cga tgt gtg aac
Asp Ile Asp Glu Cys Arg Tyr Arg Tyr Cys Gln His
Arg Cys Val Aan
165 170 175
ctg ccg ggc tct ttt cga tgc cag tgt gag cca ggc 633
ttc cag ttg gga
Leu Pro Gly Ser Phe Arg Cys Gln Cys Glu Pro Gly
Phe Gln Leu Gly
180 185 190 195
5~ cct aac aac cgc tct tgt gtg gat gtg aat gag tgt 681
gac atg gga gcc
Pro Asn Asn Arg Ser Cys Val Asp Val Asn Glu Cys
Asp Met Gly Ala
200 205 210
cca tgt gag cag cgc tgc ttc aac tcc tat ggg acc 729
ttc ctg tgt cgc
Pro Cys Glu Gln Arg Cys Phe Asn Ser Tyr Gly Thr
Phe Leu Cys Arg
215 220 225
tgt aac cag ggc tat gag ctg cac cgg gat ggc ttc 777
tcc tgc agc gat
Cys Asn Gln Gly Tyr GIu Leu His Arg Asp Gly Phe
Ser Cys Ser Asp
CA 02347121 2001-04-09
WO 00/22120 PCT/FR99/02465
230 235 240
atc gat gag tgc ggc tac tcc agt tac ctc tgc cag tac cgc tgt gtc 825
Ile Asp Glu Cys Gly Tyr Ser Ser Tyr Leu Cys Gln Tyr Arg Cys Val
245 250 255
aac gag cca ggc cga ttc tcc tgt cac tgc cca caa ggc tac cag ctg 873
Asn Glu Pro Gly Arg Phe Ser Cys His Cys Pro Gln Gly Tyr Gln Leu
260 265 270 275
ctg gct aca agg ctc tgc caa gat att gac gag tgt gaa aca ggt gca 921
Leu Ala Thr Arg Leu Cys Gln Asp Ile Asp Glu Cys Glu Thr Gly Ala
280 285 290
cac caa tgt tct gag gcc caa acc tgt gtc aac ttc cat ggg ggt tac 969
His Gln Cys Ser Glu Ala Gln Thr Cys Val Asn Phe His Gly Gly Tyr
295 300 305
cgc tgt gtg gac acc aac cgt tgt gtg gag ccc tat gtc caa gtg tca 1017
Arg Cys Val Asp Thr Asn Arg Cys Val Glu Pro Tyr Val Gln Val Ser
310 315 320
gac aac cgc tgc ctc tgc cct gcc tcc aat ccc ctt tgt cga gag cag 1065
Asp Asn Arg Cys Leu Cys Pro Ala Ser Aan Pro Leu Cys Arg Glu Gln
325 330 335
cct tca tcc att gtg cac cgc tac atg agc atc acc tca gag cga agt 1113
Pro Ser Ser Ile Val Hie Arg Tyr Met Ser Ile Thr Ser Glu Arg Ser
340 345 350 355
gtg cct gct gac gtg ttt cag atc cag gca acc tct gtc tac cct ggt 1161
Val Pro Ala Asp Val Phe Gln Ile Gln Ala Thr Ser Val Tyr Pro Gly
360 365 370
gcc tac aat gcc ttt cag atc cgt tct gga aac aca cag ggg gac ttc 1209
Ala Tyr Asn Ala Phe Gln Ile Arg Ser Gly Asn Thr Gln Gly Asp Phe
375 380 385
tac att agg caa atc aac aat gtc agc gcc atg ctg gtc ctc gcc agg 1257
Tyr Ile Arg Gln Ile Asn Asn Val Ser Ala Met Leu Val Leu Ala Arg
390 395 400
cca gtg acg gga ccc cgg gag tac gtg ctg gac ctg gag atg gtc acc 1305
Pro Val Thr Gly Pro Arg Glu Tyr Val Leu Asp Leu Glu Met Val Thr
~rJ 405 410 415
atg aat tcc ctt atg agc tac cgg gcc agc tct gta ctg aga ctc acg 1353
Met Asn Ser Leu Met Ser Tyr Arg Ala Ser Ser Val Leu Arg Leu Thr
420 425 430 435
gtc ttt gtg gga gcc tat acc ttc tgaagaccct cagggaaggg ccatgtgggg 1407
Val Phe Val Gly Ala Tyr Thr Phe
440
gccccttccc cctcccatag cttaagcagc cccgggggcc tagggatgac cgttctgctt 1467
aaaggaacta tgatgtgaag gacaataaag ggagaaagaa ggaaaa 1513
CA 02347121 2001-04-09
WO 00/22120 PCT/FR99/02465
9
<210> 16
<211> 443
<212> PRT
<213> Séquence artificielle
<223> Description de la séquence artificielle: MBP1
murine (séquence complète)
<400> 16
Met Leu Pro Phe Ala Ser Cys Leu Pro Gly Ser Leu Leu Leu Trp Ala
1 s l0 15
Phe Leu Leu Leu Leu Leu Gly Ala Ala Ser Pro Gln Asp Pro Glu Glu
25 30
15 Pro Asp Ser Tyr Thr Glu Cys Thr Asp Gly Tyr Glu Trp Asp Ala Asp
35 40 45
Ser Gln His Cys Arg Asp Val Asn Glu Cys Leu Thr Ile Pro Glu Ala
50 55 60
Cys Lys Gly Glu Met Lys Cys Ile Aan His Tyr Gly Gly Tyr Leu Cjrs
65 70 75 80
Leu Pro Arg Ser Ala Ala Val Ile Ser Asp Leu His Gly Glu Gly Pro
85 90 95
Pro Pro Pro Ala Ala His Ala Gln Gln Pro Asn Pro Cys Pro Gln Gly
100 105 110
Tyr Glu Pro Asp Glu Gln Glu Ser Cys Val Asp Val Asp Glu Cys Thr
115 120 125
Gln Ala Leu His Asp Cys Arg Pro Ser Gln Asp Cya His Asn Leu Pro
130 135 140
Gly Ser Tyr Gln Cys Thr Cys Pro Asp Gly Tyr Arg Lys Ile Gly Pro
145 150 155 160
Glu Cya Val Asp Ile Asp Glu Cys Arg Tyr Arg Tyr Cya Gln His Arg
165 170 175
Cya Val Asn Leu Pro Gly Ser Phe Arg Cys Gln Cys Glu Pro Gly Phe
180 185 190
Gln Leu Gly Pro Asn Asn Arg Ser Cya Val Asp Val Asn Glu Cya Asp
195 200 205
Met Gly Ala Pro Cys Glu Gln Arg Cys Phe Asn Ser Tyr Gly Thr Phe
210 215 220
Leu Cya Arg Cys Asn Gln Gly Tyr Glu Leu Hia Arg Asp Gly Phe Ser
225 230 235 290
Cys Ser Asp Ile Asp Glu Cys Gly Tyr Ser Ser Tyr Leu Cys Gln Tyr
245 250 255
Arg Cys Val Aan Glu Pro Gly Arg Phe Ser Cys Hia Cys Pro Gln Gly
260 265 270
CA 02347121 2001-04-09
WO 00/22120 PCT/FR99/02465
Tyr Gln Leu Leu Ala Thr Arg Leu Cys Gln Asp Ile Asp Glu Cys Glu
275 280 285
Thr Gly Ala His Gln Cys Ser Glu Ala Gln Thr Cys Val Asn Phe His
5 290 295 300
Gly Gly Tyr Arg Cys Val Aap Thr Asn Arg Cys Val Glu Pro Tyr Val
305 310 315 320
10 Gln Val Ser Asp Asn Arg Cys Leu Cys Pro Ala Ser Asn Pro Leu Cys
325 330 335
Arg Glu Gln Pro Ser Ser Ile Val His Arg Tyr Met Ser Ile Thr Ser
340 345 350
Glu Arg Ser Val Pro Ala Aap Val Phe Gln Ile Gln Ala Thr Ser Val
355 360 365
Tyr Pro Gly Ala Tyr Asn Ala Phe Gln Ile Arg Ser Gly Asn Thr Gln
370 375 380
Gly Asp Phe Tyr Ile Arg Gln Ile Asn Aan Val Ser Ala Met Leu Val
385 390 395 400
Leu Ala Arg Pro Val Thr Gly Pro Arg Glu Tyr Val Leu Asp Leu Glu
405 410 415
Met Val Thr Met Asn Ser Leu Met Ser Tyr Arg Ala Ser Ser Val Leu
420 425 430
Arg Leu Thr Val Phe Val Gly Ala Tyr Thr Phe
435 440
<210> 17
<211> 21
<212> ADN
<213> Séquence artificielle
<220>
<223> Description de la séquence artificielle:
oligonucléotide 3'hMBPl
<400> 17
ctccgctccg aggtgatggt c 21
<210> 18
<211> 21
<212> ADN
<213> Séquence artificielle
<220>
<223> Description de la séquence artificielle:
oligonucléotide 5'hMBPl
<400> 18
tgtagctact ccagctacct c 21
CA 02347121 2001-04-09
WO 00/22120 PCT/FR99102465
11
<210> 19
<211> 1122
<212> ADN
<213> Séquence artificielle
<220>
<223> Description la squenceartificielle: cDNAP1
de MB
huma ine (squence partielle)
<400> 19
aagccagccgagccgccagagccgcgggccgcgggggtgtcgcgggcccaaccccaggat60
gctccctgc gcctcctgcctacccgggtctctactgctctgggcgctgctactgttgct120
cttgggatcagcttctcctcaggattctgaagagcccgacagctacacggaatgcacaga180
tggctatgagtgggacccagacagccagcactgccgggatgtcaacgagtgtctgaccat240
ccctgaggcctgcaagggggaaatgaagtgcatcaaccactacgggggctacttgtgcct300
gccccgctccgctgccgtcatcaacgacctacacggcgagggacccccgccaccagtgcc360
tcccgctcaacaccccaacccctgcccaccaggctatgagcccgacgatcaggacagctg420
tgtggatgtggacgagtgtgcccaggccctgcacgactgtcgccccagccaggactgcca480
taacttgcctggctcctatcagtgcacctgccctgatggttaccgcaagatcgggcccga540
gtgtgtggacatagacgagtgccgctaccgctactgccagcaccgctgcgtgaacctgcc600
tggctccttccgctgccagtgcgagccgggcttccagctggggcctaacaaccgctcctg660
tgttgatgtgaacgagtgtgacatgggggccccatgcgagcagcgctgcttcaactccta720
tgggaccttcctgtgtcgctgccaccagggctatgagctgcatcgggatggcttctcctg780
cagtgatattgatgagtgtagctactccagctacctctgtcagtaccgctgcgtcaacga840
gccaggccgtttctcctgccactgcccacagggttaccagctgctggccacacgcctctg900
ccaagacattgatgagtgtgagtctggtgcgcaccagtgctccgaggcccaaacctgtgt960
caacttccatgggggctaccgctgcgtggacaccaaccgctgcgtggagccctacatcca1020
3~
ggtctctgagaaccgctgtctctgcccggcctccaaccctctatgtcgagagcagccttc1080
atccattgtgcaccgctacatgaccatcacctcggagcggag 1122
<210> 20
<211> s84
<212> ADN
<213> Séquence artificielle
<220>
<223> Description la squenceartificielle:
de cDNA
MBPlhumain
(squence
partielle)
<400> 20
tgtagctactccagctacctctgtcagtaccgctgcgtcaacgagccaggccgtttctcc
60
tgccactgcccacagggttaccagctgctggccacacgcctctgccaagacattgatgag
120
tgtgagtctggtgcgcaccagtgctccgaggcccaaacctgtgtcaacttccatgggggc
180
taccgctgcgtggacaccaaccgctgcgtggagccctacatccaggtctctgagaaccgc
240
tgtctctgcccggcctccaaccctctatgtcgagagcagccttcatccattgtgcaccgc
300
tacatgaccatcacctcggagcggagcgtgcccgctgacgtgttccagatccaggcgacc
5~ 360
tccgtctaccccggtgcctacaatgcctttcagatccgtgctggaaactcgcagggggac
420
ttttacattaggcaaatcaacaacgtcagcgccatgctggtcctcgcccggccggtgacg
480
ggcccccgggagtacgtgctggacctggagatggtcaccatgaattccctcatgagctac
540
cgggccagctctgtactgaggctcaccgtctttgtaggggcctacaccttctgaggagca
600
ggagggagccaccctccctgcagctaccctagctgaggagcctgttgtgaggggcagaat
660
gagaaaggcaataaagggagaaag 684
<210> 21
<211> 1480
CA 02347121 2001-04-09
WO 00/22120 PCT/FR99/02465
12
<212> ADN
<213> Séquence artificielle
<220>
<221> CDS
<222> (59)..(1387)
<220>
<223> Description de la séquence artificielle: MHP1
humaine (sêquence complète?
<400> 21
aagccagccg agccgccaga gccgcgggcc.gcgggggtgt cgcgggccca accccagg 58
atg ctc ccc tgc gcc tcc tgc cta ccc ggg tct cta ctg ctc tgg gcg 106
Met Leu Pro Cys Ala Ser Cys Leu Pro Gly Ser Leu Leu Leu Trp Ala
1 5 10 15
ctg cta ctg ttg ctc ttg gga tca gct tct cct cag gat tct gaa gag 154
Leu Leu Leu Leu Leu Leu Gly Ser Ala Ser Pro Gln Asp Ser Glu Glu
20 25 3p
ccc gac agc tac acg gaa tgc aca gat ggc tat gag tgg gac cca gac 202
Pro Asp Ser Tyr Thr Glu Cys Thr Asp Gly Tyr Glu Trp Asp Pro Aap
35 40 45
agc cag cac tgc cgg gat gtc aac gag tgt ctg acc atc cct gag gcc 250
Ser Gln His Cys Arg Asp Val Asn Glu Cys Leu Thr Ile Pro Glu Ala
50 55 60
tgc aag ggg gaa atg aag tgc atc aac cac tac ggg ggc tac ttg tgc 298
Cys Lys Gly Glu Met Lys Cys Ile Asn His Tyr Gly Gly Tyr Leu Cys
65 70 75 80
ctg ccc cgc tcc gct gcc gtc atc aac gac cta cac ggc gag gga ccc 346
Leu Pro Arg Ser Ala Ala Val Ile Asn Asp Leu His Gly Glu Gly Pro
85 90 95
ccg cca cca gtg cct ccc gct caa cac ccc aac ccc tgc cca cca ggc 394
Pro Pro Pro Val Pro Pro Ala Gln His Pro Asn Pro Cys Pro Pro Gly
100 105 110
tat gag ccc gac gat cag gac agc tgt gtg gat gtg gac gag tgt gcc 442
Tyr Glu Pro Asp Asp Gln Asp Ser Cys Val Asp Val Asp Glu Cys Ala
115 120 125
cag gcc ctg cac gac tgt cgc ccc agc cag gac tgc cat aac ttg cct 490
Gln Ala Leu Hie Asp Cys Arg Pro Ser Gln Asp Cys His Asn Leu Pro
130 135 140
ggc tcc tat cag tgc acc tgc cct gat ggt tac cgc aag atc ggg ccc 538
Gly Ser Tyr Gln Cys Thr Cys Pro Asp Gly Tyr Arg Lys Ile Gly Pro
145 150 155 160
gag tgt gtg gac ata gac gag tgc cgc tac cgc tac tgc cag cac cgc 586
Glu Cys Val Asp Ile Aap Glu Cys Arg Tyr Arg Tyr Cys Gln His Arg
165 170 175
tgc gtg aac ctg cct ggc tcc ttc cgc tgc cag tgc gag ccg ggc ttc 634
CA 02347121 2001-04-09
WO OOf22120 PCT/FR99/02465
13
Cys Val Asn Leu Pro Gly Ser Phe Arg Cys Gln Cys Glu Pro Gly Phe
180 185 190
cag ctg ggg cct aac aac cgc tcc tgt gtt gat gtg aac gag tgt gac 682
Gln Leu Gly Pro Asn Asn Arg Ser Cys Val Asp Val Asn Glu Cys Asp
195 200 205
atg ggg gcc cca tgc gag cag cgc tgc ttc aac tcc tat ggg acc ttc 730
Met Gly Ala Pro Cys Glu Gln Arg Cys Phe Asn Ser Tyr Gly Thr Phe
210 215 220
ctg tgt cgc tgc cac cag ggc tat gag ctg cat cgg gat ggc ttc tcc 778
Leu Cys Arg Cya His Gln Gly Tyr Glu Leu His Arg Asp Gly Phe Ser
225 230 235 240
tgc agt gat att gat gag tgt agc tac tcc agc tac ctc tgt cag tac 826
Cys Ser Asp Ile Asp Glu Cya Ser Tyr Ser Ser Tyr Leu Cys Gln Tyr
245 250 255
cgc tgc gtc aac gag cca ggc cgt ttc tcc tgc cac tgc cca cag ggt 874
Arg Cys Val Asn Glu Pro Gly Arg Phe Ser Cys His Cys Pro Gln Gly
260 265 270
tac cag ctg ctg gcc aca cgc ctc tgc caa gac att gat gag tgt gag 922
Tyr Gln Leu Leu Ala Thr Arg Leu Cys Gln Asp Ile Asp Glu Cys Glu
275 280 285
tct ggt gcg cac cag tgc tcc gag gcc caa acc tgt gtc aac ttc cat 970
Ser Gly Ala His Gln Cys Ser Glu Ala Gln Thr Cys Val Asn Phe His
3~ 290 295 300
ggg ggc tac cgc tgc gtg gac acc aac cgc tgc gtg gag ccc tac atc 1018
Gly Gly Tyr Arg Cys Val Asp Thr Asn Arg Cys Val Glu Pro Tyr Ile
305 310 315 320
cag gtc tct gag aac cgc tgt ctc tgc ccg gcc tcc aac cct cta tgt 1066
Gln Val Ser Glu Asn Arg Cys Leu Cys Pro Ala Ser Asn Pro Leu Cys
325 330 335
cga gag cag cct tca tcc att gtg cac cgc tac atg acc atc acc tcg 1114
Arg Glu Gln Pro Ser Ser Ile Val His Arg Tyr Met Thr Ile Thr Ser
340 345 350
gag cgg agc gtg ccc gct gac gtg ttc cag atc cag gcg acc tcc gtc 1162
Glu Arg Ser Val Pro Ala Asp Val Phe Gln Ile Gln Ala Thr Ser Val
355 360 365
tac ccc ggt gcc tac aat gcc ttt cag atc cgt gct gga aac tcg cag 1210
Tyr Pro Gly Ala Tyr Asn Ala Phe Gln Ile Arg Ala Gly Aan Ser Gln
370 375 380
ggg gac ttt tac att agg caa atc aac aac gtc agc gcc atg ctg gtc 1258
Gly Asp Phe Tyr Ile Arg Gln Ile Asn Asn Val Ser Ala Met Leu Val
385 390 395 400
ctc gcc cgg ccg gtg acg ggc ccc cgg gag tac gtg ctg gac ctg gag 1306
Leu Ala Arg Pro Val Thr Gly Pro Arg Glu Tyr Val Leu Asp Leu Glu
405 410 415
CA 02347121 2001-04-09
WO 00/22120 PCT/FR99/02465
14
atg gtc acc atg aat tcc ctc atg agc tac cgg gcc agc tct gta ctg 1354
Met Val Thr Met Asn Ser Leu Met Ser Tyr Arg Ala Ser Ser Val Leu
420 425 430
agg ctc acc gtc ttt gta ggg gcc tac acc ttc tgaggagcag gagggagcca 1407
Arg Leu Thr Val Phe Val Gly Ala Tyr Thr Phe
435 440
ccctccctgc agctacccta gctgaggagc ctgttgtgag gggcagaatg agaaaggcaa 1467
taaagggaga aag 1480
<210> 22
<211> 443
<212> PRT
<213> Squence
artificielle
<223> Description squence MBP1
de la artificielle:
humaine (squence complte)
<400> 22
Met Leu Pro Cys SerCys LeuProGly LeuLeu Trp
Ala Ser Leu Ala
1 5 10 15
Leu Leu Leu Leu LeuGly SerAlaSer GlnAsp Glu
Leu Pro Ser Glu
20 25 30
Pro Asp Ser Tyr GluCya ThrAspGly GluTrp Pro
Thr Tyr Asp Asp
35 40 45
Ser Gln His Cys AspVal AsnGluCys ThrIle Glu
Arg Leu Pro Ala
50 55 60
Cys Lys Gly Glu LysCys IleAsnHis GlyGly Leu
Met Tyr Tyr Cys
65 70 75 80
Leu Pro Arg Ser AlaVal IleAsnAsp HisGly Gly
Ala Leu Glu Pro
85 90 95
Pro Pro Pro Val Pro Pro Ala Gln His Pro Asn Pro Cys Pro Pro Gly
100 105 110
Tyr Glu Pro Asp Asp Gln Aep Ser Cys Val Asp Val Asp Glu Cys Ala
115 120 125
Gln Ala Leu His Asp Cys Arg Pro Ser Gln Asp Cys Hia Asn Leu Pro
130 135 140
Gly Ser Tyr Gln Cys Thr Cys Pro Asp Gly Tyr Arg Lys Ile Gly Pro
50 145 150 155 160
Glu Cys Val Asp Ile Asp Glu Cys Arg Tyr Arg Tyr Cys Gln His Arg
165 170 175
Cys Val Asn Leu Pro Gly Ser Phe Arg Cys Gln Cys Glu Pro Gly Phe
180 185 190
Gln Leu Gly Pro Asn Asn Arg Ser Cys Val Asp Val Asn Glu Cys Asp
195 200 205
CA 02347121 2001-04-09
WO 00/22120 PCT/FR99/02465
Met Gly Ala Pro Cys Glu Gln Arg Cys phe Asn Ser Tyr Gly Thr Phe
210 215 220
5 Leu Cys Arg Cys His Gln Gly Tyr Glu Leu His Arg Asp Gly Phe Ser
225 230 235 240
Cys Ser Asp Ile Asp Glu Cya Ser Tyr Ser Ser Tyr Leu Cys Gln Tyr
245 250 255
Arg Cys Val Asn Glu Pro Gly Arg Phe Ser Cys His Cys Pro Gln Gly
260 265 270
Tyr Gln Leu Leu Ala Thr Arg Leu Cys Gln Asp Ile Asp Glu Cys Glu
275 280 285
Ser Gly Ala His Gln Cys Ser Glu Ala Gln Thr Cys Val Asn Phe Hia
290 295 300
Gly Gly Tyr Arg Cys Val Aap Thr Asn Arg Cys VaI Glu Pro Tyr Ile
305 310 315 320
Gln Val Ser Glu Asn Arg Cys Leu Cys Pro Ala Ser Asn Pro Leu Cya
325 330 335
Arg Glu Gln Pro Ser Ser Ile Val Hia Arg Tyr Met Thr Ile Thr Ser
340 345 350
Glu Arg Ser Val Pro Ala Asp Val Phe Gln Ile Gln Ala Thr Ser Val
355 360 365
Tyr Pro Gly Ala Tyr Aan Ala Phe Gln Ile Arg Ala Gly Asn Ser Gln
370 375 380
Gly Asp Phe Tyr Ile Arg Gln Ile Asn Asn Val Ser Ala Met Leu Val
385 390 395 400
Leu Ala Arg Pro Val Thr Gly Pro Arg Glu Tyr Val Leu Asp Leu Glu
405 410 415
Met Val Thr Met Asn Ser Leu Met Ser Tyr Arg Ala Ser Ser Val Leu
420 425 430
Arg Leu Thr Val Phe Val Gly Ala Tyr Thr Phe
435 440
<210> 23
<211> 817
<212> ADN
<213> Séquence artificielle
<220>
<223> Description de la séquence artificielle: cDNA MBP1
murine (séquence partielle)
<400> 23
gctgtggcag aaacccctga cttctgccca ccacctccca gcctcaggat gctccctttt 60
CA 02347121 2001-04-09
WO 00/22120 PCT/IïR99/02465
16
gcctcctgcc tccccgggtc tttgctgctc tgggcgtttc tgctgttgct cttgggagca 120
gcgtccccac aggatcccga ggagccggac agctacacgg aatgcacaga tggctatgag 180
tgggatgcag acagccagca ctgccgggat gtcaacgagt gcctgaccat cccggaggct 240
tgcaagggtg agatgaaatg catcaaccac tacgggggtt atttgtgtct gcctcgctct 300
gctgccgtca tcagtgatct ccatggtgaa ggacctccac cgccagcggc ccatgctcaa 360
caaccaaacc cttgcccgca gggctacgag cctgatgaac aggagagctg tgtggatgtg 420
gacgagtgta cccaggcttt gcatgactgt cgccctagtc aggactgcca taaccttcct 480
ggctcctacc agtgcacctg ccctgatggt taccgaaaaa ttggacccga atgtgtggac 540
atagatgagt gtcgttaccg ctattgccag catcgatgtg tgaacctgcc gggctctttt 600
cgatgccagt gtgagccagg cttccagttg ggacctaaca accgctcttg tgtggatgtg 660
aatgagtgtg acatgggagc cccatgtgag cagcgctgct tcaactccta tgggaccttc 720
ctgtgtcgct gtaaccaggg ctatgagctg caccgggatg gcttctcctg cagcgatatc 780
gatgagtgcg gctactccag ttacctctgc cagtacc g17
<210> 24
<211> 24
<212> ADN
<213> Séquence artificielle
<220>
<223> Description de la séquence artificielle:
oligonucléotide sens-GAPDH
<400> 24
cggagtcaac ggatttggtc gtat 24
<210> 25
<211> 24
<212> ADN
<213> Séquence artificielle
<220>
<223> Description de la séquence artificielle:
oligonucléotide antisens -GAPDH
<400> 25
agccttctcc atggtggtga agac 24
<210> 26
<211> 25
<212> ADN
<213> Séquence artificielle
<220>
<223> Description de la séquence artificielle:
oligonucléotide
<400> 26
cggttggcct tggggttcag ggggg 25
<21a> 27
<211> 21
<212> ADN
<213> Séquence artificielle
CA 02347121 2001-04-09
WO 00/22120 PCT/FR99/02465
17
<220>
<223> Description de la séquence artificielle:
oligonucléotide sens MBP1
<400> 27
gccctgatgg ttaccgcaag a 21
<210> 28
<211> 21
<212> ADN
<213> Séquence artificielle
<220>
<223> Description de la séquence artificielle:
oligonucléotide antisena MBP1
<400> 28
agcccccatg gaagttgaca c 21
<210> 29
<211> 20
<212> ADN
<213> Séquence artificielle
<220>
<223> Description de la séquence artificielle:
oligonucléotide sens actine
<400> 29
gtggggcgcc ccaggcacca 20
<210> 30
<211> 1358
<212> ADN
<213> Squence artificielle
<220>
<221> CDS
<222> (1}..(885)
<220>
<223> Description de la
squence artificielle:
fragment C-terni MBP1
humaine
<400> 30
tgc acc tgc cct gat ggt aagatcgggcccgagtgtgtg gac
tac cgc 48
Cys Thr Cys Pro Asp Gly LysIleGlyProGluCysVal Asp
Tyr Arg
1 5 10 15
ata gac gag tgc cgc tac tgccagcaccgctgcgtgsac ctg
cgc tac 96
Ile Asp Glu Cys Arg Tyr CysGlnHisArgCysValAsn Leu
Arg Tyr
20 25 30
cct ggc tcc ttc cgc tgc gagccgggcttccagctgggg cct
cag tgc 144
Pro Gly Ser Phe Arg Cys GluProGlyPheGlnLeuGly Pro
Gln Cys
35 40 45
CA 02347121 2001-04-09
WO 00/22120 PCT/FR99/02465
18
aac aaccgctcctgtgttgatgtgaacgagtgtgacatgggggcccca 192
Asn AsnArgSerCysValAspValAsnGluCysAspMetGlyAlaPro
50 55 60
tgc gagcagcgctgcttcaactcctatgggaccttcctgtgtcgctgc 240
Cys GluGlnArgCysPheAsnSerTyrGlyThrPheLeuCyaArgCys
65 70 ?5 80
cac cagggctatgagctgcatcgggatggcttctcctgcagtgatatt 288
Hia GlnGlyTyrGluLeuHisArgAspGlyPheSerCysSerAspIle
85 90 95
gat gagtgtagctactccagctacctctgtcagtaccgctgcgtcaac 336
Asp GluCysSerTyrSerSerTyrLeuCysGlnTyrArgCysValAan
100 105 110
gag ccaggccgtttctcctgccactgcccacagggttaccagctgctg 384
Glu ProGlyArgPheSerCysHisCysProGlnGlyTyrGlnLeuLeu
115 120 125
gcc acacgcctctgccaagacattgatgagtgtgagtctggtgcgcac 432
Ala ThrArgLeuCysGlnAspIleAspGluCysGluSerGlyAlaHie
130 135 140
cag tgctccgaggcccaaacctgtgtcaacttccatgggggctaccgc 480
Gln CyaSerGluAlaGlnThrCysValAsnPheHiaGlyGlyTyrArg
145 150 155 160
tgc gtggacaccaaccgctgcgtggagccctacatccaggtctctgag 528
Cys ValAspThrAsnArgCysValGluProTyrIleGlnVaISerGlu
165 170 175
aac cgctgtctctgcccggcctccaaccctctatgtcgagagcagcct 576
Asn ArgCysLeuCysProAlaSerAsnProLeuCyaArgGluGlnPro
180 185 190
tca tccattgtgcaccgctacatgaccatcacctcggagcggagcgtg 624
Ser SerIleValHisArgTyrMetThrIleThrSerGluArgSerVal
4~ 195 200 205
ccc gctgacgtgttccagatccaggcgacctccgtctaccccggtgcc 672
Pro AlaAspValPheGlnIleGlnAlaThrSerValTyrProGlyAla
210 215 220
tac aatgcctttcagatccgtgctggaaactcgcagggggacttttac 720
Tyr AsnAlaPheGlnIleArgAlaGlyAsnSerGlnGlyAspPheTyr
225 230 235 240
5~ att aggcaaatcaacaacgtcagcgccatgctggtcctcgcccggccg 768
Ile ArgGlnIleAsnAsnValSerAlaMetLeuValLeuAlaArgPro
245 250 255
gtg acg ggc ccc cgg gag tac gtg ctg gac ctg gag atg gtc acc atg 816
Val Thr Gly Pro Arg Glu Tyr Val Leu Asp Leu Glu Met Val Thr Met
260 265 270
aat tcc ctc atg agc tac cgg gcc agc tct gta ctg agg ctc acc gtc 864
Asn Ser Leu Met Ser Tyr Arg Ala Ser Ser Val Leu Arg Leu Thr VaI
CA 02347121 2001-04-09
WO 00/22120 PCT/FR99/02465
19
275 280 285
ttt gta 915
ggg gcc
tac acc
ttc tgaggagcag
gagggagcca
ccctccctgc
Phe Val
Gly Ala
Tyr Thr
Phe
290 295
agctaccctagctgaggagcctgttgtgaggggcagaatgagaaaggcaataaagggaga975
aagaaagtcctggtggctgaggtgggcgggtcacactgcaggaagcctcaggctggggca1035
gggtggcacttgggggggcaggccaagttcacctaaatgggggtctctatatgttcaggc1095
ccaggggcccccattgacaggagctgggag.tctgcaccacgagcttcagtcaccccgag1155
aggagaggaggtaacgaggagggcggactccaggccccggcccagagatttggacttggc1215
tggcttgcaggggtcctaagaaactccactctggacagcgccaggaggccctgggttcca1275
ttcctaactctgcctcaaactgtacatttggataagccctagtagttccctgggcctgtt1335
tttctataaaacgaggcaactgg 1358
<210> 31
<211> 295
<212> PRT
<213> Squence
artificielle
<223> Description squence
de la artificielle:
fragment C-terni
MBP1 humaine
<400> 31
Cys Thr Cys Pro GlyTyr LysIleGlyProGluCysValAsp
Asp Arg
1 5 10 15
Ile Asp Glu Cys TyrArg CysGlnHisArgCysValAsnLeu
Arg Tyr
20 25 30
Pro Gly Ser Phe CysGln GluProGlyPheGlnLeuGlyPro
Arg Cys
35 40 45
Asn Asn Arg Ser ValAap AsnGluCysAspMetGlyAlaPro
Cys Val
50 55 60
Cys Glu Gln Arg PheAan TyrGlyThrPheLeuCysArgCys
Cys Ser
65 70 75 g0
His Gln Gly Tyr LeuHis AspGlyPheSerCysSerAspIle
Glu Arg
85 90 95
Asp Glu Cys Ser SerSer LeuCysGlnTyrArgCysValAsn
Tyr Tyr
100 105 110
Glu Pro Gly Arg SerCys CysProGlnGlyTyrGlnLeuLeu
Phe His
115 120 125
Ala Thr Arg Leu GlnAap AspGluCysGluSerGlyAlaHis
Cys Ile
130 135 140
Gln Cys Ser Glu GlnThr ValAsnPheHisGlyGlyTyrArg
Ala Cys
CA 02347121 2001-04-09
WO 00/22120 PCT/FR99/02465
145 150 155 160
Cys Val Asp Thr Asn Arg Cys Val Glu Pro Tyr Ile Gln Val Ser Glu
165 170 175
5
Asn Arg Cys Leu Cys Pro Ala Ser Asn Pro Leu Cys Arg Glu Gln Pro
180 185 190
Ser Ser Ile Val His Arg Tyr Met Thr Ile Thr Ser Glu Arg Ser Val
' 0 195 200 205
Pro Ala Asp Val Phe Gln Ile Gln Ala Thr Ser Val Tyr Pro Gly Ala
210 215 220
15 Tyr Asn Ala Phe Gln Ile Arg Ala Gly Asn Ser Gln Gly Asp Phe Tyr
225 230 235 240
Ile Arg Gln Ile Asn Asn Val Ser Ala Met Leu Val Leu Ala Arg Pro
245 250 255
Val Thr Gly Pro Arg Glu Tyr Val Leu Asp Leu Glu Met VaI Thr Met
260 265 270
Asn Ser Leu Met Ser Tyr Arg Ala Ser Ser Val Leu Arg Leu Thr Val
275 280 285
Phe Val Gly Ala Tyr Thr Phe
290 295
<210> 32
<211> 1663
<212> ADN
<213> Séquence artificielle
<220>
<221> CDS
<222> (1)..(999)
<220>
<223> Description de la séquence artificielle: fragment
c-terni fibuline 2 murine
<400> 32
gag ggc tct gaa tgt gtg gat gtg aat gag tgt gag aca ggt gtg cat 48
Glu Gly Ser Glu Cys Val Asp Val Asn Glu Cys Glu Thr Gly Val His
1 5 10 15
~JO cgc tgt ggc gag ggc caa ctg tgc tat aac ctc cct gga tcc tac cgc 96
Arg Cys Gly Glu Gly Gln Leu Cys Tyr Asn Leu Pro Gly Ser Tyr Arg
20 25 30
tgt gac tgc aag ccc ggc ttc cag agg gat gca ttc ggc agg act tgc 144
Cys Asp Cys Lys Pro Gly Phe Gln Arg Asp Ala Phe Gly Arg Thr Cys
35 40 45
att gat gtg aac gaa tgc tgg gtc tcg ccg ggc cgc ctg tgc cag cac 192
Ile Asp Val Asn Glu Cys Trp Val Ser Pro Gly Arg Leu Cys Gln His
CA 02347121 2001-04-09
WO 00/22120 PCT/FR99/02465
21
50 55 60
aca tgtgagaacacaccgggctcctaccgctgctcctgcgctgctggc 240
Thr CysGluAsnThrProGlySerTyrArgCysSerCysAlaAlaGly
65 70 75 80
ttc cttttggccgcagatggcaaacattgtgaagatgtgaacgagtgc 288
Phe LeuLeuAlaAlaAspGlyLysHisCysGluAspValAsnGluCys
85 90 95
gag actcggcgctgcagccaggaatgtgccaacatctatggctcctat 336
Glu ThrArgArgCysSerGlnGluCysAlaAsnIleTyrGlySerTyr
I00 105 110
cag tgctactgccgtcagggctaccagctggcagaggatgggcatacc 384
Gln CysTyrCysArgGlnGlyTyrGlnLeuAlaGluAspGlyHisThr
115 120 125
tgc acagacatcgatgagtgtgcacagggcgcgggcattctctgtacc 432
Cys ThrAspIleAspGluCysAlaGlnGlyAlaGlyIleLeuCysThr
130 135 140
ttc cgctgtgtcaacgtgcctgggagctaccagtgtgcatgcccagag 480
Phe ArgCysValAsnValProGlySerTyrGlnCysAlaCysProGlu
145 150 155 160
caa gggtatacaatgatggccaacgggaggtcctgcaaggacctggat 528
Gln GlyTyrThrMetMetAlaAsnGlyArgSerCysLysAspLeuAsp
165 170 175
gag tgtgcactgggcacccacaactgctctgaggctgagacctgccac 576
Glu CysAlaLeuGlyThrHisAsnCysSerGluAlaGluThrCysHis
180 185 190
aat atccaggggagtttccgctgcctgcgctttgattgtccacccaac 624
Asn IleGlnGlySerPheArgCysLeuArgPheAspCysProProAsn
195 200 205
tat gtccgtgtctcacaaacgaagtgcgagcgcaccacatgccaggat 672
Tyr ValArgValSerGlnThrLysCysGluArgThrThrCysGlnAsp
210 215 220
atc acggaatgtcaaacctcaccagctcgcatcacgcactaccagctc 720
Ile ThrGluCysGlnThrSerProAlaArgIleThrHisTyrGlnLeu
225 230 235 240
aat ttccagacaggcctactggtacctgcacatatcttccgcatcggc 768
Asn PheGlnThrGlyLeuLeuValProAlaHiaIlePheArgIleGly
245 250 255
cct gctcccgcctttgctggggacaccatctccctgaccatcacgaag 816
Pro AlaProAlaPheAlaGlyAspThrIleSerLeuThrIleThrLys
260 265 270
ggc aatgaggagggctacttcgtcacacgcagactcaatgcctacact 864
Gly AsnGluGluGlyTyrPheValThrArgArgLeuAsnAlaTyrThr
275 280 285
ggt gtggtatccctgcagcggtctgttctggagccgcgggactttgcc 912
CA 02347121 2001-04-09
WO 00122120 PCT1FR99/02465
22
Gly Val Val Ser Leu Gln Arg Ser Val Leu Glu Pro Arg Asp Phe Ala
290 295 300
cta gat gtg gag atg aag ctt tgg cgc cag ggc tct gtc act acc ttc 960
Leu Asp Val Glu Met Lys Leu Trp Arg Gln Gly Ser Val Thr Thr Phe
305 310 315 320
ctg gcc aag atg tac atc ttc ttc acc act ttt gcc cca tgaggtgaca 1009
Leu Ala Lys Met Tyr Ile Phe Phe Thr Thr Phe Ala Pro
1~ 325 330
tgtcaggcaa tccctccagg tgatgcctgg gcggtgggca gctgcgccac tcctaagtgg 1069
ctttttgctg tgactctgta acttaactta atcatgctga gctggttggt cttgagtctc 1129
taccctagag ggagggagat gcaccccagc aggcactgag tacaggccag ggtcacccga 1189
ggctagatgg tgacctgcaa actggaaaca gccatagggg gcttctgaac tccactcctc 1249
2~ aactatggct acagctgaca ttccattcct tcatccactg tgttcctcaa ttaaaaaaaa 1309
aaatcagctg tgcatggtag cacagacctt taatcctagc actggggagg cagaggtagg 1369
tagatctctg agttccaggc cagcctggtc tacactggga gttctaacca gccagagcta 1429
catagagaga ccctatctca acaaggaaaa aacgaaagaa atctctgtga gttccaggcc 1489
agcctggtct acgctgggag ttctaaccag ccagagctac atagagagat cctatctcaa 1549
caaggaaaaa tgaaagaaat cattttaaaa ggtttttttt tttgctgttg ttgtttaatg 1609
ataagagtag cacatataca ttattaaaaa tgatcaaata gcacagaaag gtta 1663
<210> 33
<211> 333
<212> PRT
<213> Séquence artificielle
<223> Description de la séquence artificielle: fragment
4Q c-terni fibuline 2 murine
<400> 33
Glu Gly Ser Glu Cys Val Asp Val Asn Glu Cys Glu Thr Gly Val His
1 5 10 15
Arg Cys Gly Glu Gly Gln Leu Cys Tyr Asn Leu Pro G1y Ser Tyr Arg
20 25 30
Cys Asp Cys Lys Pro Gly Phe Gln Arg Aap Ala Phe Gly Arg Thr Cya
35 40 45
Ile Asp Val Asn Glu Cys Trp Val Ser Pro Gly Arg Leu Cys Gln His
55 60
Thr Cya Glu Asn Thr Pro Gly Ser Tyr Arg Cys Ser Cys Ala Ala Gly
70 75 80
Phe Leu Leu Ala Ala Asp Gly Lys His Cys Glu Asp Val Asn Glu Cys
85 90 95
CA 02347121 2001-04-09
WO 00/22120 PCT/FR99/02465
23
Glu Thr Arg Arg Cys Ser Gln Glu Cys Ala Asn Ile Tyr Gly Ser Tyr
100 105 110
Gln Cys Tyr Cys Arg Gln Gly Tyr Gln Leu Ala Glu Asp Gly Hia Thr
115 120 125
Cjra Thr Asp Ile Asp Glu Cys Ala Gln Gly Ala Gly Ile Leu Cys Thr
130 135 140
Phe Arg Cys Val Asn Val Pro Gly Ser Tyr Gln Cys Ala Cys Pro Glu
145 150 155 160
Gln Gly Tyr Thr Met Met Ala Aan Gly Arg Ser Cys Lys Asp Leu Asp
165 170 17s
Glu Cys Ala Leu Gly Thr His Asn Cys Ser Glu Ala Glu Thr Cys Hia
180 185 190
2~ Asn Ile Gln Gly Ser Phe Arg Cys Leu Arg Phe Asp Cys Pro Pro Aen
195 200 205
Tyr Val Arg Val Ser Gln Thr Lys Cys Glu Arg Thr Thr Cys Gln Aap
210 215 220
Ile Thr Glu Cya Gln Thr Ser Pro Ala Arg Ile Thr His Tyr Gln Leu
225 230 235 240
Asn Phe Gln Thr Gly Leu Leu VaI Pro Ala His Ile Phe Arg Ile Gly
3~ 245 250 255
Pro Ala Pro Ala Phe Ala Gly Asp Thr Ile Ser Leu Thr Ile Thr Lya
260 265 270
Gly Asn Glu Glu Gly Tyr Phe Val Thr Arg Arg Leu Asn Ala Tyr Thr
275 280 285
Gly Val Val Ser Leu Gln Arg Ser Val Leu Glu Pro Arg Asp Phe Ala
290 295 300
Leu Asp Val Glu Met Lys Leu Trp Arg Gln Gly Ser Val Thr Thr Phe
305 3I0 315 320
Leu Ala Lys Met Tyr Ile Phe Phe Thr Thr Phe Ala Pro
325 330