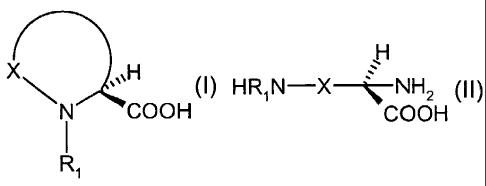Note: Descriptions are shown in the official language in which they were submitted.
CA 02449836 2003-12-04
WO 02/101003 PCT/FR02/01983
1
PREPARATION STEREOSELECTIVE DE L-ACIDES AMINES
CYCLIQUES
La présente invention concerne la préparation stéréosélective de
L-acides aminés cycliques ou de leurs sels et dérivés.
Il existe une demande grandissante d'acides aminés cycliques
notamment la L-proline, l'acide L-pipécolique ou l'acide L-pipérazine-2-
carboxylique et leurs sels en raison de leur utilisation pour la synthèse de
nouveaux médicaments. Étant donné qu'il s'agit de molécules chirales, les
synthèses en sont difficiles.
D'un point de vue biosynthétique, seule la formation du cycle de la
L-proline a été élucidée et décrite. Elle peut s'effectuer selon deux voies :
- soit par cyclisation de l'acide glutamique semialdéhyde pour
former l'acide A1-pyrroline-5-carboxylique, suivie d'une réduction du cycle
insaturé;
- soit directement à partir de la L-ornithine par cyclodéamination.
Toutefois, ces procédés n'apportent pas de solution satisfaisante
pour la synthèse des acides aminés cycliques chiraux à l'échelle industrielle.
Par ailleurs, une activité enzymatique de conversion directe de la
L-ornithine en L-proline a été décrite pour la première fois chez Clostridium
(R. N. Costilow et al., J. Biol. Chem., 246 (21), (1971), 6655-60 ; W. L. Muth
et
al., J. Biol. Chem., 249 (23), (1974), 7457-62) et une conversion totale de
20 mM de L-ornithine en L-proline a été obtenue in vitro en présence de
l'enzyme partiellement purifiée. Cette ornithine cyclodéaminase (ocd) de
Clostridium possède la particularité d'être activée par le NAD, un cofacteur
impliqué habituellement dans les réactions d'oxydation, et semble être assez
CA 02449836 2003-12-04
WO 02/101003 PCT/FR02/01983
2
est inhibée en présence de 40 mM de D-ornithine, tandis qu'en présence de
40 mM de L-lysine son activité n'est pas réduite. La possibilité que la
cyclodéaminase agisse sur la L-lysine n'a ainsi même pas été envisagée par
ces auteurs, probablement parce qu'il est très souvent admis que les enzymes
du métabolisme sont très spécifiques.
Quelques années plus tard, la corrélation entre l'expression d'un
gène d'Agrobacterium impliqué dans la dégradation de la nopaline et une
activité ocd a été établie (N. Sans et al., Eur. J. Biochem., 173, (1988), 123-
130), et le gène codant pour ocd a alors été séquence.
Un deuxième gène très homologue a par la suite été trouvé dans
une autre souche d'Agrobacterium par les mêmes auteurs (U. Schindler et al.,
J. Bacteriol., 171, (1989), 847-854). Les propriétés des deux cyclodéaminases
ont été étudiées et seule une activité sur la L-ornithine a été mise en
évidence.
Ainsi, de nouveau la possibilité que la L-Lysine soit un substrat de la
cyclodéaminase n'a pas été envisagée, seul son effet inhibiteur a été examiné
(Schindler et al., ibid.).
Depuis, le séquençage total ou partiel de nombreux organismes a
permis d'identifier plusieurs nouveaux gènes ayant une homologie forte avec ce
gène ocd d'Agrobacterium. Cependant, ni l'activité enzymatique du polypeptide
codé par chacun de ces gènes, ni son spectre d'activité n'ont été étudiées,
cette enzyme étant réputée spécifique de l'ornithine. En particulier, la
spécificité
de ces enzymes et leur niveau d'activité catalytique restent inconnus alors
que
ce sont des éléments déterminants pour la productivité d'un procédé
enzymatique.
Pour trois de ces gènes trouvés chez des Streptomyces
producteurs de métabolites dérivés de l'acide L-pipécolique, une activité
lysine
cyclodéaminase pour le polypeptide codé a été postulée (WO-A1-96/01901 ;
CA 02449836 2003-12-04
WO 02/101003 PCT/FR02/01983
3
L. E. Khaw et al., J. Bacteriol., 180, (1998), 809-814 ; I. Molnar et al.,
Gene,
169, (1996), 1-7 ; H. Motamedi et al., Eur. J. Biochem., 249, (1998), 528-
534).
Ainsi, il a été suggéré qu'une cyclodéaminase codée par le gène
rapL (Khaw et al., ibid.) ou pipA (WO-A-9601901) pourrait être impliquée dans
la voie de synthèse de la Rapamycine via une transformation de la lysine en
acide pipécolique. A ce jour, même si elle a été parfois suggérée obiter
dictum,
aucune activité de cyclodéamination de L-lysine en acide L-pipécolique n'a
toutefois été mise en évidence, exemplifiée, ou quantifiée. Cette
méconnaissance de la réaction réellement catalysée par ces gènes est
démontrée par la diversité des hypothèses sur la voie biologique menant à
l'acide pipécolique par certains de ces auteurs qui relatent également dans
ces
publications que la conversion de lysine en acide pipécolique pourrait se
faire
via la D-lysine (Khaw et al., ibid.).
D'autres études biochimiques ont décrit la formation d'acide
L-pipécolique à partir d'acide a-aminoadipique par réduction d'un cycle
insaturé
(A. J. Aspen et al., Biochemistry, 1(4), (1962), 606-612) chez Aspergillus et,
sur
la base d'expériences de marquages isotopiques, de telles voies de
biosynthèse avaient été également proposées chez un Streptomyces par les
hommes de l'art (J. W. Reed et al., J. Org. Chem., 54(5), (1989), 1161-65).
Le fait que les enzymes de biosynthèse de la L-proline, et en
particulier l'ornithine cyclodéaminase, n'aient pas été proposées pour
fabriquer
de nouveaux acides aminés cycliques constitue une preuve supplémentaire
que, de l'opinion générale des hommes de métier, il n'était pas possible de
réaliser la biosynthèse de l'acide pipécolique de cette façon. Ainsi plusieurs
procédés biotechnologiques de bioconversion conduisant aux formes
énantiomériquement pures, et plus particulièrement à la forme L de l'acide
pipécolique ou de l'acide pipérazine-2-carboxylique ont été protégés par des
brevets depuis quelques années. Les exemples figurant dans ces publications
CA 02449836 2003-12-04
WO 02/101003 PCT/FR02/01983
4
font état d'une production maximale de 15 g/L de l'acide aminé cyclique
aucune mention n'a été trouvée de l'emploi d'une cyclodéaminase.
Tous ces documents, à l'exception d'un seul, décrivent la
résolution d'un substrat racémique. Ils emploient des activités de type
N-acylase (WO 95/10604, WO 99/07873), amidase (EP-A-0 686 698),
aminoacide oxydase (JP 06030789), nitrilase (JP 06038781, JP 11127885) ou
estérase (WO 00/12745) agissant sur des substrats adéquats.
L'ensemble de ces méthodes comporte par ailleurs un certain
nombre d'inconvénients parmi lesquels on peut citer une limitation du
rendement maximal à 50% sur le mélange racémique, la nécessité d'une
séparation du produit ainsi formé et du substrat non transformable par
l'enzyme
pour récupérer la forme énantiomérique recherchée, et la nécessité du
développement éventuel d'un recyclage de l'autre forme énantiomérique. Seul
JP 06030781 décrit la conversion de la L-lysine, un substrat chiral
commercialement disponible et peu onéreux, en acide L-pipécolique par divers
isolats bactériens non-recombinants mais sans la séquence de réactions
chimiques empruntée pour effectuer cette bioconversion. Les performances, en
termes de rendement et de productivité, mentionnées dans ce document - à
savoir une production en 7 jours d'environ 4,2 g/L d'acide L-pipécolique à
partir
de 10 g/L de L-Lysine - restent tout à fait insuffisantes pour qu'un tel
procédé
soit avantageux et économiquement compétitif. Un des buts de l'invention vise
à obtenir des performances supérieures à celle connue de l'art antérieur, et
notamment des productions en acide aminés cycliques de l'ordre de 5, 10 voire
20 fois supérieures ou plus à celles divulguées dans l'état de la technique.
La demanderesse a maintenant montré qu'il est possible de
convertir avec des rendements élevés des solutions concentrées d'acides
di-aminés en des solutions d' acides a-imino-cycliques, notamment en solution
aqueuse de sels d'ammonium d'acides a-imino-cycliques, en mettant en oeuvre
CA 02449836 2003-12-04
WO 02/101003 PCT/FR02/01983
une enzyme codée par le gène de l'ornithine cyclodéaminase (EC 4.3.1.12) de
la souche d'Agrobacterium C58 ou par un gène homologue à celui-ci, ou des
microorganismes recombinants surproduisant de telles enzymes.
5 L'invention a pour objet un procédé de production d'un L-acide
aminé cyclique de formule (I) ou d'un sel ou dérivé d'un acide aminé de
formule (1):
X %%\H (I)
\N COOH
Ri
dans laquelle :
* Rl est choisi parmi l'atome d'hydrogène, un radical alkyle linéaire
ou ramifié comportant de 1 à 6 atomes de carbone et un radical acyle linéaire
ou ramifié comportant de 1 à 6 atomes de carbone ; et
* X représente une chaîne hydrocarbonée saturée, ou
partiellement ou totalement insaturée, linéaire ou ramifiée en Cl-C9, de
préférence en C2-C4, comprenant éventuellement dans la chaîne et/ou en bout
de chaîne un ou plusieurs hétéroatomes ou hétérogroupes choisis parmi O, S,
P, NR2, R2 représentant H ou un groupe alkyle ou acyle en C1-C4, la dite
chaîne
étant également éventuellement substituée par un ou plusieurs radicaux,
identiques ou différents choisis parmi -R, -OR, -SR, =0, -C(O)OR, -C(S)OR,
-C(O)NR'R", -C(S)NR'R", -CN, -NO2, -X', -MgX' (X' étant un atome d'halogène
choisi parmi fluor, chlore, brome et iode), -NR'R", -NR'C(O)R, -SiR et -SiOR,
R,
R' et R", identiques ou différents, représentant l'hydrogène ou un radical
hydrocarboné, linéaire ou ramifié, saturé, ou totalement ou partiellement
insaturé, et comportant de 2 à 20 atomes de carbone, étant entendu que R' et
R" peuvent former un cycle avec l'atome qui les porte,
caractérisé en ce que :
a) l'on fait réagir un L-acide diaminé de formule (11) :
CA 02449836 2011-09-09
6
HR1N-X-\NH2 (II)
COOH
dans laquelle X et Ri sont tels que définis ci-dessus ;
ou un sel ou dérivé de celui-ci,
ou un mélange énantiomère comprenant un L-acide diaminé de
formule (II) et un D-acide diaminé correspondant, leurs sels ou dérivés en
proportions variables, de préférence en milieu aqueux,
en présence d'une ornithine cyclodéaminase, ou d'un polypeptide
homologue à l'ornithine cyclodéaminase, l'enzyme ou le polypeptide homologue
étant obtenu à partir d'un vecteur d'expression recombinant exprimant la dite
l o enzyme ou le dit polypeptide homologue,
b) l'on récupère le L-acide aminé cyclique de formule (I) ou un sel
ou dérivé de celui-ci en un excès énantiomérique d'au moins 80 %.
L'invention a pour objet un procédé de production d'un L-acide aminé
cyclique de formule I ou d'un de ses sels ou dérivés:
X
H (I)
COOH
R,
dans laquelle:
* R, est choisi parmi l'atome d'hydrogène, un radical alkyle linéaire ou
ramifié
comportant de 1 à 6 atomes de carbone et un radical acyle linéaire ou ramifié
comportant de 1 à 6 atomes de carbone, et
20 * X représente une chaîne hydrocarbonée saturée, ou partiellement ou
totalement insaturée, linéaire ou ramifiée en C3-C5,
et dans laquelle le cycle possède 5, 6 ou 7 maillons,
CA 02449836 2011-09-09
7
caractérisé en ce que
a) l'on fait réagir un L-acide diaminé de formule Il:
,.H
HR,N X NH2 (II)
COOH
dans laquelle X et Ri sont tels que définis ci-dessus;
ou un sel ou dérivé de celui-ci,
ou un mélange énantiomère comprenant un L-acide diaminé de formule (II)
et un D-acide diaminé correspondant, leurs sels ou dérivés en proportions
variables,
en présence d'une enzyme recombinante exprimée par le gène pipA* de
séquence SEQ ID n 1 ou le gène rapL* de séquence SEQ ID 2, ou le gène
rapL** de séquence SEQ ID n 5,
b) l'on récupère le L-acide aminé cyclique de formule (I) ou un sel ou dérivé
de celui-ci en un excès énantiomérique d'au moins 80%.
Par "dérivé d'acide aminé de formule (I) ou (II)", on entend un
amide ou ester de ceux-ci.
Plus spécifiquement, la présente invention a pour objet un
procédé de production d'un L-acide aminé cyclique de formule (I) ou d'un sel
ou dérivé d'un acide aminé de formule (1):
X\ %\H (I)
N COOH
Ri
CA 02449836 2011-09-09
7a
dans laquelle RI représente H ou un groupe alkyle en Ci-Cs ou
acyle en Ci-C6 et X représente une chaîne hydrocarbonée saturée linéaire ou
ramifiée en C2-C9, de préférence en C2-C4, éventuellement interrompue par un
ou plusieurs hétéroatomes ou hétérogroupes choisis parmi O, S, NR2, R2
représentant H ou un groupe alkyle ou acyle en C1-C4, et/ou éventuellement
substituée par un ou plusieurs groupes hydroxy, amino, ou halogéné,
caractérisé en ce que
a) l'on fait réagir un L-acide diaminé de formule (II) :
_H
HR1N-X-- -NH2 (II)
COOH
dans laquelle X et Ri sont tels que définis ci-dessus ;
ou un sel ou dérivé de celui-ci,
ou un mélange énantiomère comprenant un L-acide diaminé de
formule (II) et un D-acide diaminé correspondant, leurs sels ou dérivés en
proportions variables, de préférence en milieu aqueux,
en présence d'une ornithine cyclodéaminase ou d'un polypeptide
homologue à I'ornithine cyclodéaminase, l'enzyme ou le polypeptide homologue
étant obtenu à partir d'un vecteur d'expression recombinant exprimant la dite
enzyme ou le dit polypeptide homologue, et
b) l'on récupère le L-acide aminé cyclique de formule I ou un sel
ou dérivé de celui-ci en un excès énantiomérique d'au moins 80%.
Lorsque la chaîne hydrocarbonée comporte une ou plusieurs
insaturations de nature éthylénique et/ou acétylénique, celles-ci ne sont de
préférence pas portées par les atomes de carbone situés en position a des
atomes d'azote formant les fonctions amines de l'acide diaminé de formule
(11).
CA 02449836 2011-09-09
7b
On préfère également que, lorsque la chaîne hydrocarbonée
comporte deux ou plus hétéroatomes, les dits hétéroatomes soient séparés par
au moins deux atomes de carbone.
La présente invention a pour objet un composé de formule (I) ou sel ou
dérivé du composé de formule (I):
X
.H (~)
COOH
Ri
dans laquelle:
* Ri est choisi parmi l'atome d'hydrogène, un radical alkyle linéaire ou
ramifié
comportant de 1 à 6 atomes de carbone et un radical acyle linéaire ou ramifié
comportant de 1 à 6 atomes de carbone, et
* X représente une chaîne hydrocarbonée saturée, ou partiellement ou
totalement insaturée, linéaire ou ramifiée en C5-C9 comprenant éventuellement
dans la chaîne et/ou en bout de chaîne un ou plusieurs hétéroatomes ou
hétérogroupes choisis parmi O, S, P, NR2, R2 représentant H ou un groupe
alkyle
ou acyle en C1-C4, ladite chaîne étant également éventuellement substituée par
un
ou plusieurs radicaux, identiques ou différents choisis parmi -R, -OR, -SR,
=0, -
C(O)OR, -C(S)OR, -C(O)NR'R", -C(S)NR'R", -CN, NO2, -X, -MgX, -NR'R", -
NR'C(O)R, -SiR et -SiOR, R, R' et R", identiques ou différents, représentant
l'hydrogène ou un radical hydrocarboné, linéaire ou ramifié, saturé, ou
totalement
ou partiellement instauré, et comportant de 2 à 20 atomes de carbone, étant
entendu que R' et R" peuvent former un cycle avec l'atome qui les porte.
La présente invention a pour objet un polynucléotide comprenant la
séquence SEQ ID n 1, SEQ ID n 2 ou SEQ ID n 5.
CA 02449836 2003-12-04
WO 02/101003 PCT/FR02/01983
8
Les composés de formule (I) obtenus selon le procédé de la
présente invention comportent la plus souvent un cycle à 3, 4, 5, 6 ou 7
maillons qui sont les cycles les plus fréquemment rencontrés dans le domaine
de la chimie organique. Les composés de formule (I) pour lesquels le cycle
possède 5, 6 ou 7 maillons sont préférés et sont ceux qui paraissent les plus
accessibles pour l'homme du métier. Le procédé de la présente invention ne
saurait cependant se limiter à la synthèse de ces composés à cycles de 5, 6 ou
7 maillons.
On préfère en outre le procédé pour lequel le composé de formule
(I) comprend un cycle à six maillons, X représentant une chaîne hydrocarbonée
à quatre maillons. Plus spécifiquement encore, le procédé est mis en oeuvre
pour l'obtention d'un composé de formule (I) dans lequel X est une chaîne
alkylène linéaire ou ramifiée. Dans la présente invention, il doit être
compris,
par chaîne hydrocarbonée, une chaîne comportant des atomes de carbone et
d'hydrogène.
Pour le procédé selon la présente invention, il doit être compris
que le L-acide diaminé de formule (II), tel que défini ci-dessus, est mis en
présence d'au moins une enzyme et/ou d'au moins un polypeptide homologue
à l'ornithine cyclodéaminase, obtenus à partir d'un vecteur d'expression
recombinant exprimant ces enzyme(s) ou ces polypeptide(s) homologue(s).
Par ornithine cyclodéaminase, on entend toute enzyme
susceptible de cycliser un acide diaminé, plus précisément un amino-a-
aminoacide et notamment l'ornithine et la lysine. L'ornithine cyclodéaminase à
laquelle on se réfère dans la suite du texte est de préférence l'ornithine
cyclodéaminase (EC 4.3.1.12) de la souche d'Agrobacterium C58.
L'ornithine cyclodéaminase de la souche C58 d'Agrobacterium,
codée par un gène porté par le plasmide TiC58, est décrite par N. Sans et al.,
CA 02449836 2003-12-04
WO 02/101003 PCT/FR02/01983
9
Eur. J. Biochem., 173, (1988), 123-130, et sa séquence polypeptidique est
également accessible sur Genbank (gi : 68365).
Par polypeptide homologue à l'ornithine cyclodeaminase, on
entend les polypeptides ayant une séquence d'acides aminés homologues à
celle d'une ornithine cyclodeaminase, notamment celle de la souche C58
d'Agrobacterium.
Ces séquences homologues peuvent être définies comme les
séquences similaires à au moins 25 % de la séquence d'acides aminés du
gène ocd d'Agrobacterium et ayant une activité d'ornithine cyclodeaminase
telle
que décrite par R. N. Costilow et al., J. Biol. Chem., 246 (21), (1971), 6655-
60.
Le terme "similaires" se réfère à la ressemblance parfaite ou
identité entre les acides aminés comparés mais aussi à la ressemblance non
parfaite que l'on qualifie de similitude. Cette recherche de similitudes dans
une
séquence polypeptidique prend en compte les substitutions conservatives qui
sont des substitutions d'acides aminés de même classe, telles que des
substitutions d'acides aminés aux chaînes latérales non chargées (tels que
l'asparagine, la glutamine, la sérine, la thréonine, et la tyrosine), d'acides
aminés aux chaînes latérales basiques (tels que la lysine, l'arginine, et
l'histidine), d'acides aminés aux chaînes latérales acides (tels que l'acide
aspartique et l'acide glutamique) ; d'acides aminés aux chaînes latérales
apolaires (tels que la glycine, l'alanine, la valine, la leucine,
l'isoleucine, la
proline, la phénylalanine, la méthionine, le tryptophane, et la cystéine).
Plus généralement, par "séquence d'acides aminés homologue",
on entend donc toute séquence d'acides aminés qui diffère de la séquence
d'acides aminés de l'ornithine cyclodeaminase codée par le gène ocd
d'Agrobacterium par substitution, délétion et/ou insertion d'un acide aminé ou
d'un nombre réduit d'acides aminés, notamment par substitution d'acides
aminés naturels par des acides aminés non naturels ou pseudoacides aminés
à des positions telles que ces modifications ne portent pas significativement
atteinte à l'activité biologique du polypeptide codé.
CA 02449836 2003-12-04
WO 02/101003 PCT/FR02/01983
Avantageusement, une telle séquence d'acides aminés
homologue est similaire à au moins 35 % de la séquence du polypeptide codé
par le gène ocd d'Agrobacterium, de préférence au moins 45 %.
L'homologie est généralement déterminée en utilisant un logiciel
5 d'analyse de séquence (par exemple, Séquence Analysis Software Package of
the Genetics Computer Group, University of Wisconsin Biotechnology Center,
1710 University Avenue, Madison, WI 53705, U.S.A.). Des séquences d'acides
aminés similaires sont alignées pour obtenir le maximum de degré d'homologie
(i.e. identité ou similitude, comme défini plus haut). A cette fin, il peut
être
10 nécessaire d'introduire de manière artificielle des espaces ("gaps") dans
la
séquence. Une fois l'alignement optimal réalisé, le degré d'homologie est
établi
par enregistrement de toutes les positions pour lesquelles les acides aminés
des deux séquences comparées sont identiques, par rapport au nombre total
de positions.
Ces polypeptides ont en commun de présenter une activité
d'acide oc-5- ou a-s-d!ami nés-déaminase, sans que celle-ci ait nécessairement
été établie jusqu'à présent.
Parmi de telles séquences homologues, sont comprises les
séquences des polypeptides homologues à l'ornithine cyclodeaminase
d'Agrobacterium trouvés chez les microorganismes appartenant aux genres
Agrobacterium, Aeropyrum, Archaeoglobus, Brucella, Corynebacterium,
Halobacterium, Mesorhizobium, Methanobacterium, Pseudomonas, Rhizobium,
Rhodobacter, Sinorhizobium, Schizosaccharomyces, Sulfolobus,
Thermoplasma, Staphylococcus, et Streptomyces.
On peut citer en particulier les séquences des polypeptides
homologues à l'ornithine cyclodeaminase d'Agrobacterium trouvés chez les
espèces ou souches listées dans le tableau 1 suivant :
CA 02449836 2003-12-04
WO 02/101003 PCT/FR02/01983
11
Tableau 1 : Microorganismes ayant des séquences de polypeptides
homologues à celle de l'ornithine cyclodeaminase d'Agrobacterium
Souche ou espèce Numéro Genbank (gi:) du
polypeptide homologue
- Aeropyrum pernix (strain K1) 7521229
- Agrobacterium tumefaciens 1066073
- Agrobacterium tumefaciens plasmide pAtK84b, 11269716 (fragment)
- Agrobacterium tumefaciens plasmide pTiAch5, 2498689
- Agrobacterium tumefaciens plasmide pTiC58, 68365
- Archaeoglobus fulgidus, 11499255
- Brucella melitensis biovarAbortus, 1354828
- Corynebacterium glutamicum, 4127671
- Halobacterium sp. NRC-1, 10581639 (ocd1) ; 10580873 (ocd2)
- Mesorhizobium loti, 13472795
- Mesorhizobium loti, 13472097
- Mesorhizobium loti, 13475945
- Mesorhizobium loti, 13475655
- Mesorhizobium loti, 13471680
- Methanobacterium thermoautotrophicum (strain 7482770
Delta H), ou Methanothermobacter thermautotrophicus
- Pseudomonas aeruginosa (strain PAO1), 11350019 (ocdl) ; 11350319 (ocd2)
- Y4tK Rhizobium sp. NGR234, 2182641
- Rhizobium ou Sinorhizobium meliloti, 420888
- Rhodobacter capsulatus, 7437141
- Schizosaccharomyces pombe, 12044475
- Staphylococcus aureus, 13700033
- Streptomyces hygroscopicus, 7481884
- Streptomyces hygroscopicus var ascomycetus, 9280393
- Sulfolobus solfataricus, 13813523 (ocdl) ; 13816686 (ocd2)
- Thermoplasma acidophilum, 10639650
- Thermoplasma volcanium, 13541655
CA 02449836 2003-12-04
WO 02/101003 PCT/FR02/01983
12
On peut citer en outre le polypeptide codé par le gène pipA de
Streptomyces pristinaespiralis (ATCC-25486) dont la séquence est décrite dans
WO-A1-9601901 et les séquences des polypeptides homologues à l'ornithine
cyclodeaminase d'Agrobacterium susceptibles d'être trouvés chez les espèces
ou souches productrices de rapamycine, d'ascomycine, et de FK-506, ainsi que
les Streptomyces ou autres microorganismes producteurs de streptogramines,
en particulier Streptomyces loïdensis (ATCC-1 1415), Streptomyces mitakaensis
(ATCC-15297), Streptomyces olivaceus (ATCC-12019), Streptomyces
ostréogriseus (ATCC-27455), Streptomyces virginiae (ATCC-13161).
On peut citer également les polypeptides homologues à l'ornithine
cyclodeaminase d'Agrobacterium trouvés chez des organismes eucaryotes tels
que Arabidopsis thaliana, Drosophila melanogaster, Homo sapiens, Macropus
fuliginosus, Mus musculus, et Rattus norvegicus et listées dans le tableau 2
suivant :
Tableau 2 : Organismes supérieurs ayant des séquences de polypeptides
homologues à celle de l'ornithine cyclodeaminase d'Agrobacterium
Souche ou espèce Numéro Genbank (gi :)du
polypeptide homologue
- Arabidopsis thaliana, 9950040
- Drosophila melanogaster, 7293295
- Homo sapiens, 13647115
- Macropus fuliginosus, 283930
- Mus musculus, 3913376
- Rattus norvegicus 5931745
Dans un mode de réalisation particulier de l'invention, ces
polypeptides sont codés par des gènes homologues à celui de l'ornithine
cyclodéaminase de la souche C58 d'Agrobacterium.
Par gène homologue de celui de l'ornithine cyclodéaminase de la
souche C58 d'Agrobacterium, on entend tout gène ayant :
CA 02449836 2003-12-04
WO 02/101003 PCT/FR02/01983
13
a) une séquence nucléotidique similaire à la séquence codante
du gène de I'ornithine cyclodéaminase de la souche C58 d'Agrobacterium ; ou
b) une séquence nucléotidique hybridant avec la séquence
codante du gène de l'ornithine cyclodéaminase de la souche C58
d'Agrobacterium ou sa séquence complémentaire, dans des conditions
stringentes d'hybridation ; ou
c) une séquence nucléotidique codant pour un polypeptide
homologue à l'ornithine cyclodeaminase d'Agrobacterium tel que défini ci-
dessus ou ayant une activité d'ornithine cyclodeaminase.
De préférence, une telle séquence nucléotidique homologue selon
l'invention est similaire à au moins 75% de la séquence du gène ocd
d'Agrobacterium ou du gène pipA, rapL, pipA de séquence SEQ ID n 1, rapt
de séquence SEQ ID n 2 et raapL*L* de séquence SEQ ID n 5 définis ci-après,
de préférence encore au moins 85%, ou au moins 90%.
De manière préférentielle, une telle séquence nucléotidique
homologue hybride spécifiquement à la séquence complémentaire de la
séquence du gène ocd d'Agrobacterium ou du gène pipA, rapL, pipA de
séquence SEQ ID n 1, rapt de séquence SEQ ID n 2 et rapt L de séquence
SEQ ID n 5 définis ci-après, dans des conditions stringentes. Les paramètres
définissant les conditions de stringence dépendent de la température à
laquelle
50% des brins appariés se séparent (Tm).
Pour les séquences comprenant plus de 30 bases, Tm est définie
par la relation : Tm = 81,5 + 0,41 (%G+C) + 16,6 Log(concentration en cations)
- 0,63 (%formamide) - (600/nombre de bases) (Sambrook et al., 1989).
Dans des conditions de stringence appropriées, auxquelles les
séquences aspécifiques n'hybrident pas, la température d'hybridation peut être
de préférence de 5 à 10 C en dessous de Tm, et les tampons d'hybridation
utilisés sont de préférence des solutions de force ionique élevée telle qu'une
solution 6xSSC par exemple.
CA 02449836 2003-12-04
WO 02/101003 PCT/FR02/01983
14
Le terme "séquences similaires" employé plus haut se réfère à la
ressemblance parfaite ou identité entre les nucléotides comparés mais aussi à
la ressemblance non parfaite que l'on qualifie de similitude. Cette recherche
de
similitudes dans les séquences nucléiques distingue par exemple les purines et
les pyrimidines.
Une séquence nucléotidique homologue inclut donc toute
séquence nucléotidique qui diffère de l'une des séquences identifiées, par
mutation, insertion, délétion ou substitution d'une ou plusieurs bases, ou par
la
dégénérescence du code génétique.
De telles séquences homologues peuvent être obtenues à partir
de microorganismes appartenant aux genres Agrobacterium, Aeropyrum,
Archaeoglobus, Brucella, Corynebacterium, Halobacterium, Mesorhizobium,
Methanobacterium, Pseudomonas, Rhizobium, Rhodobacter, Sinorhizobium,
Schizosaccharomyces, Sulfolobus, Thermoplasma, Staphylococcus et
Streptomyces.
On peut citer en particulier les séquences des gènes codant pour
des polypeptides homologues à l'ornithine cyclodeaminase d'Agrobacterium
trouvés chez les espèces ou souches listées dans le tableau 1 précédent.
On peut citer en outre la séquence du gène pipA de Streptomyces
pristinaespiralis (ATCC-25486) décrite dans WO-A1-9601901 et les séquences
des gènes codant pour des polypeptides homologues à l'ornithine
cyclodeaminase d'Agrobacterium susceptibles d'être trouvés chez les espèces
ou souches productrices de rapamycine, d'ascomycine, et de FK-506, ainsi que
les Streptomyces ou autres microorganismes producteurs de streptogramines,
en particulier Streptomyces ioïdensis (ATCC-11415), Streptomyces mitakaensis
(ATCC-15297), Streptomyces olivaceus (ATCC-12019), Streptomyces
ostréogriseus (ATCC-27455), Streptomyces virginiae (ATCC-13161).
On peut citer également les séquences des gènes eucaryotes de
Arabidopsis thaliana, Drosophila metanogaster, Homo sapiens, Macropus
fuliginosus, Mus musculus, et Rattus norvegicus qui sont décrits comme codant
CA 02449836 2003-12-04
WO 02/101003 PCT/FR02/01983
pour des polypeptides homologues à l'ornithine cyclodeaminase
d'Agrobacterium et listés dans le tableau 2 précédent.
Parmi les gènes convenant bien au procédé de l'invention, on
5 peut citer notamment les gènes rapL de Streptomyces hygroscopicus et pipA
de Streptomyces pristinaespirales dont la séquence est décrite respectivement
par Schwecke et al., Proc. Nati. Acad. Sci. U.S.A., 92(17), (1995), 7839-43 et
dans WO-A1-9601901. Selon un mode de réalisation de l'invention, le
composé de formule générale (II) ou le mélange énantiomère comprenant un L-
10 acide aminé de formule (II) et un D-acide aminé correspondant, leurs sels
ou
dérivés en proportions variables est mis en présence d'une suspension du
microorganisme producteur du polypeptide homologue à l'ornithine
cyclodeaminase.
Des gènes homologues préférés tels que décrits ci-dessus sont
15 des gènes synthétiques obtenus par mutagénèse dirigée tel que décrit plus
bas.
Selon un mode de réalisation de l'invention, le composé de
formule générale (II) ou le mélange énantiomère comprenant un L-acide aminé
de formule (li) et un D-acide aminé correspondant, leurs sels ou dérivés en
proportions variables est mis en présence d'une enzyme isolée du
microorganisme producteur.
Selon un autre mode de réalisation de l'invention, le composé de
formule (II) ou le mélange énantiomère comprenant un L-acide diaminé de
formule (II) et un D-acide diaminé correspondant en proportions variables est
mis en présence d'une enzyme à l'état purifié.
Selon un autre mode de réalisation de l'invention, le composé de
formule (II) ou le mélange énantiomère comprenant un L-acide diaminé de
formule (II) et un D-acide diaminé correspondant en proportions variables est
30, mis en présence d'une enzyme recombinante, ce dernier mode de réalisation
étant préféré.
CA 02449836 2003-12-04
WO 02/101003 PCT/FR02/01983
16
On a pu constater de manière surprenante que les polypeptides
tels que décrits ci-dessus permettaient dans les conditions de l'invention la
synthèse stéréospécifique des composés de formule (l) de l'invention sans
nécessiter l'addition exogène de NAD au milieu réactionnel.
Dans un mode de réalisation de l'invention, on ajoute à la
préparation enzymatique ou à la suspension cellulaire éventuellement
perméabilisée, un composé de formule (II) ou un mélange d'énantiomères du
composé de formule (II) et de D-acide diaminé correspondant en une
concentration supérieure à environ 0,05 M, de préférence supérieure à environ
01 M, cette même concentration étant inférieure à environ et 3 M, de
préférence inférieure à 2,5 M, leurs sels ou dérivés et on laisse incuber à
une
température comprise entre 10 C et 100 C, avantageusement entre 20 C et
70 C, préférentiellement entre 25 C et 45 C, pendant une durée comprise
entre quelques heures et plusieurs jours, sous agitation.
On peut de la sorte obtenir des rendements molaires en composé
de formule (I) d'au moins 20, avantageusement d'au moins 80, de préférence
d'au moins 90% avec un excès énantiomère supérieur à 80, avantageusement
supérieur 90, de préférence supérieur à 95%.
Avantageusement, le composé de formule (1) est sous la forme
d'un sel d'ammonium.
En variante, on peut également mettre à incuber les enzymes
extraites du milieu de culture ou purifiées avec le composé de formule (ll) ou
un
mélange d'énantiomères du composé de formule (II) ou de D-acide diaminé
correspondant dans un milieu tamponné ou non à un pH compris entre 6 et 11
et de préférence entre 7 et 10.
Le produit obtenu peut être recueilli par précipitation, cristallisation
ou chromatographie d'échanges d'ions.
Parmi les substrats préférés pour la préparation d'un composé de
formule (I), on peut citer en particulier la L-lysine ou un mélange de L- et
CA 02449836 2003-12-04
WO 02/101003 PCT/FR02/01983
17
D-lysine, la L-thialysine (S-2-aminoéthyl-L-cystéine), la L-ornithine, un
mélange
de L- et D- 5-(R,S)-hydroxylysine, un mélange de L- et D- azalysine (acide y-N-
2aminoéthyldiaminopropionique).
Afin de rendre le procédé plus particulièrement efficace, la
surproduction du polypeptide à activité d'ornithine cyclodéaminase est
particulièrement avantageuse.
Dans cette optique, on introduit le gène d'intérêt codant pour un
polypeptide tel que défini ci-dessus dans un microorganisme hôte, de
ro préférence surexprimant le polypeptide.
A cet effet, un vecteur contenant un acide nucléique comprenant
l'une des séquences nucléotidiques telle que définie ci-dessus ou une
séquence homologue est transféré dans une cellule hôte qui est mise en
culture dans des conditions permettant l'expression, de préférence la
surexpression du polypeptide correspondant.
La séquence d'acide nucléique d'intérêt peut être insérée dans un
vecteur d'expression, dans lequel elle est liée de manière opérante à des
éléments permettant la régulation de son expression, tels que notamment des
promoteurs, activateurs et/ou terminateurs de transcription.
Les signaux contrôlant l'expression des séquences nucléotidiques
(parmi lesquels on peut citer les promoteurs, les activateurs, et les
séquences
de terminaison) sont choisis en fonction de l'hôte cellulaire utilisé. A cet
effet,
les séquences nucléotidiques selon l'invention peuvent être insérées dans des
vecteurs à réplication autonome au sein de l'hôte choisi, ou des vecteurs
intégratifs de l'hôte choisi. De tels vecteurs seront préparés selon les
méthodes
couramment utilisées par l'homme du métier, et les clones en résultant peuvent
être introduits dans un hôte approprié par des méthodes standard.
CA 02449836 2003-12-04
WO 02/101003 PCT/FR02/01983
18
Parmi les vecteurs préférés, on peut citer les plasmides
pTZ18 (Sigma, St-Louis), pQE70 (Qiagen, Munich) et PQE60 (Qiagen,
Munich).
Lorsqu'ils ont été utilisés, les plasmides QIAGEN ont présenté
l'inconvénient majeur d'impliquer la formation de polyhistidines dans les
enzymes obtenues. Aussi, ces plasmides doivent-ils être modifiés pour
s'affranchir de cet inconvénient. Les séquences codantes doivent être ainsi
modifiées ou disruptées. Ces modifications n'ont pas entraîné de différence en
terme de niveau d'expression du gène codant pour la cyclodéaminase.
Les vecteurs d'expression sont introduits dans les cellules hôtes
et celles-ci sont mises en culture dans des conditions permettant la
réplication
et/ou l'expression de la séquence nucléotidique transfectée.
Des exemples de cellules hôtes incluent notamment E. coli, de
préférence les souches Escherichia coli DH5a, Escherichia coli 82033, et
Escherichia coli K12 (MG 1655).
Toutefois, d'autres organismes hôtes appartenant à des genres
différents conviennent également.
Des résultats particulièrement intéressants sont obtenus lorsqu'on
transfecte E. coli avec des gènes modifiés par rapport aux gènes
correspondants natifs de façon à permettre une expression optimale du gène
d'intérêt dans E. coll.
Le gène hétérologue est modifié de façon à comprendre une
proportion inférieure à 65%, avantageusement inférieure à 55% de bases
G + C, tout en codant pour le même polypeptide que le gène natif ou un
polypeptide homologue tel que défini précédemment.
Ainsi, pour un acide aminé donné codé par un codon de l'ADN
natif, on remplace le cas échéant la troisième base du codon, voire la seconde
base du codon lorsque celle-ci est G ou C par A ou T, lorsque le codon obtenu
correspond au même acide aminé que celui codé par le codon natif.
CA 02449836 2003-12-04
WO 02/101003 PCT/FR02/01983
19
Les gènes modifiés qui ont permis d'obtenir les meilleurs taux
d'expression et également les rendements les plus élevés en acides a-imino-
cycliques sont ceux dans lesquels les codons natifs ont été remplacés pour un
acide aminé donné du polypeptide ayant l'activité acide aminé cyclodéaminase
s par les codons respectifs suivants représentés au tableau 3.
Tableau 3 : Code de conversion utilisé
Acide aminé. Codon utilise Acide aminé Codon utilisé
A Ala GCA L Leu TTA
R Arg CGT K Lys AAA
N Asn AAT M Met ATG
D Asp GAT F Phe TTT
C Cys TGT P Pro CCA
Q Gln CAA S Ser TCT
E Glu GAA T Thr ACT
G Gly GGT W Trp TGG
H His CAT Y Tyr TAT
I Ile ATT V Val GTT
Les gènes modifiés ont été obtenus par assemblage de tronçons
obtenus par PCR et mutagénèse dirigée à partir des gènes natifs
correspondants.
Parmi les gènes modifiés ayant donné les meilleurs résultats, on
peut citer les gènes modifiés pipA* et rapL** ayant respectivement les
séquences SEQ ID n 1 et SEQ ID n 5 représentées en annexe de la présente
demande.
Les polynucléotides correspondant à ces gènes modifiés
constituent un autre objet de l'invention.
CA 02449836 2003-12-04
WO 02/101003 PCT/FR02/01983
Le gène pipA* a été obtenu à partir de la séquence du polypeptide
codé par le gène pipA de Streptomyces pristinaespiralis représentée en annexe
en tant que séquence SEQ ID n 3.
Le gène rapL** a été obtenu à partir de la séquence du
5 polypeptide codé par le gène rapL de Streptomyces hygroscopicus représentée
en annexe en tant que séquence SEQ ID n 4.
Les gènes modifiés sont insérés dans des vecteurs de clonage
comme décrit ci-dessus pour les gènes natifs transfectés dans des hôtes
recombinants.
10 Les microorganismes hôtes, le cas échéant, recombinants
comportant un gène existant à l'état natif ou modifié comme décrit ci-dessus
sont cultivés, éventuellement en présence d'un inducteur d'expression, par
exemple I'IPTG.
Lorsque les cellules atteignent une densité de l'ordre de au moins
15 une unité d'absorbance à 600 nm pour des cultures standards et de l'ordre
de
quelques dizaines ou plus de telles unités pour des cultures à haute densité,
elles sont séparées de leur milieu de culture et soumises à un traitement de
perméabilisation qui peut être physique (par exemple, traitements alternatifs
de
congélation et décongélation, traitements aux ultrasons ou à la presse de
20 French, broyage mécanique) ou chimique (par exemple, ajout de solvant ou
d'agents complexants tels l'EDTA), ou enzymatique (par exemple, ajout de
lysozyme).
La suspension cellulaire peut alors être utilisée pour réaliser la
préparation des produits de formule (II) comme décrit ci-dessus où la protéine
d'intérêt produite peut ensuite être récupérée et purifiée. A titre d'exemple,
en
utilisant un procédé de type "Fed-batch" ou équivalent, on obtient des
concentrations, exprimées en grammes de cellules sèches par litre de
suspension cellulaire, supérieures à environ 10 g/L, avantageusement
supérieure à 30 g/L. Ces mêmes concentrations sont, en règle générale,
inférieures à 60 g/L, ou bien encore inférieures à 50 g/L, sans toutefois que
ceci n'indique un limite infranchissable.
CA 02449836 2003-12-04
WO 02/101003 PCT/FR02/01983
21
Les procédés de purification utilisés sont connus de l'homme du
métier. Le polypeptide recombinant obtenu peut être purifié à partir de lysats
et
extraits cellulaires, du surnageant du milieu de culture, par des méthodes
utilisées individuellement ou en combinaison, telles que le fractionnement,
les
s méthodes de chromatographie, les techniques d'immuno-affinité à l'aide
d'anticorps mono- ou polyclonaux spécifiques, etc.
Toutefois, il n'est pas nécessaire de récupérer cette protéine.
Selon un mode de réalisation avantageux, les cellules hôtes
produisant le polypeptide ayant l'activité enzymatique recherchée sont
détruites
afin que leur contenu cytoplasmique soit libéré dans le milieu contenant le
substrat acide diaminé dont les deux fonctions amines sont distantes (par la
voie la plus courte lorsque plusieurs chemins sont possible) de quatre ou cinq
chaînons, sous forme L ou sous forme d'un mélange d'énantiomères et de
rs manière à permettre la mise en présence de l'enzyme et de son substrat.
Les exemples suivants illustrent l'invention.
Exemple 1 :
Amplification par PCR du gène pipA de Streptomyces pristinaespiralis à
partir de l'ADN total de la souche.
1.1.Extraction de l'ADN total.
La souche Streptomyces pristinaespiralis ATCC 25486 a été
cultivée à 28 C pendant 24 h dans le milieu TSB (DIFCO), puis 10 mL de la
culture ainsi obtenue ont été centrifugés pendant 5 min à 11000 g. Le culot de
centrifugation a été repris par 1 mL d'une solution de 10 mM Tris pH 8,0
contenant 2 mM d'EDTA et 5 mg/mL de, lysozyme. La suspension ainsi obtenue
a été laissée sous agitation pendant 30 min à 37 C, puis 100 pL d'une solution
de SDS à 20% ont été ajoutés. Après incubation à 30 C pendant quelques
minutes, 125 pL d'une solution de protéinase K à 2 mg/mL ont été encore
ajoutés. Ensuite l'extraction de l'ADN a été poursuivie en utilisant les
réactifs du
CA 02449836 2009-09-30
22
kit DNeasy*`f'issue Kit (QIAGEN, Munich) et en respectant les indications du
fournisseur. L'ADN ainsi extrait a été purifié par extraction
phénol/chloroforme,
précipité par de l'éthanol et repris dans 100 pi d'eau.
1.2.Amplification du gène pipA.
Le gène pipA a été amplifié par PCR à partir de l'ADN total ainsi
préparé en utilisant, pour la synthèse des amorces, sa séquence décrite dans
le brevet WO-A1-96/01901. La réaction a été effectuée dans un volume de
50 pL de tampon 20 mM Tris-HCI pH 8.8 contenant 10 mM KCI, 10 mM
*
(NH4)2SO4, 2 mM MgSO4, 0,1% Triton X-100, 2 pL de la solution d'ADN total,
250 pM de chacun des quatre dNTP, 1 unité de la Vent DNA Polymérase
(BioLabs) et 500 nM de chacun des deux oligodésoxynucléotides suivants
5'CCCGAATTCCGACACCACGCACGGACGAGAAG
5' CCCTGCAGGCATCTCTCTCCTCGCGAGGGC.
Le protocole de PCR appliqué a débuté par une étape de
dénaturation à 97 C pendant 4 min suivie d'une incubation à 80 C pendant
1 min, et s'est poursuivi par 5 cycles caractérisés par une séquence de 1 min
de dénaturation à 97 C, suivie de 1 min d'hybridation à 55 C, puis de
1 min 30 sec d'élongation à 72 C. Ensuite 40 cycles caractérisés par une
séquence de 1 min de dénaturation à 97 C, suivie de 1 min d'hybridation à
50 C, puis de 1 min 30 sec d'élongation à 72 C ont été effectués. Le protocole
s'est achevé par une étape d'élongation à 72 C pendant 10 min.
*
Le fragment ainsi amplifié a été purifié sur fa colonne du Qiaquick
PCR purification kit (QIAGEN), puis digéré par EcoRl et Pstl et cloné dans le
vecteur pTZ18 (SIGMA, St-Louis, MO) préalablement coupé. Deux
constructions plasmidiques (pKT 35, pKT 36) obtenues suite à des
amplifications PCR indépendantes ont été séquencées. Les séquences de ces
deux fragments ainsi clonés étaient identiques. La protéine spécifiée, de
séquence SEQ ID n 3, diffère cependant d'un acide aminé (alanine) à la
position 87 par rapport à la séquence publiée dans WO-A1-96/01901 (glycine).
* (marques de commerce)
CA 02449836 2003-12-04
WO 02/101003 PCT/FR02/01983
23
Exemple 2:
Amplification par PCR du gène rapL de Streptomyces hygroscopicus à
partir d'une suspension de cellules de la souche.
La souche Streptomyces hygroscopicus ATCC 29253 a été
cultivée à 28 C pendant 24 h dans le milieu TSB (DIFCO). 50 pL de la
suspension cellulaire ainsi obtenue ont été soumis à des étapes
d'échauffement et de refroidissement suivants : 30 sec à 65 C, 30 sec à 8 C,
90 sec à 65 C, 180 sec à 97 C, 60 sec à 8 C, 180 sec à 65 C, 60 sec à 97 C,
60 sec à 65 C, 20 min à 80 C.
L'amplification du gène rapL a été réalisée par PCR à partir d'une
suspension de cellules en utilisant pour la synthèse des amorces sa séquence
décrite (Schwecke et al., 1995). La réaction a été effectuée dans un volume de
50 pL de tampon 20 mM Tris-HCI pH 8.8 contenant 10 mM KCI, 10 mM
(NH4)2SO4, 2 mM MgSO4, 0,1 % Triton X-100, 10 pL de la suspension de
cellules préalablement traitée, 250 pM de chacun des quatre dNTP, 1 unité de
la Vent DNA Polymérase (BioLabs) et 500 nM de chacun des deux
oligodésoxynucléotides suivants :
5'CCCGAATTCGAGGTTGCATGCAGACCAAGGTTCTGTGCCAG
5'CCCAAGCTTAGATCTTTATTACAGCGAGTACGGATCGAGGACG
Le protocole PCR appliqué a débuté par une étape de
dénaturation à 97 C pendant 4 min suivie d'une incubation à 80 C pendant
1 min, et s'est poursuivi tout d'abord par 5 cycles caractérisés par une
séquence de 1 min de dénaturation à 97 C, suivie de 1 min d'hybridation à
60 C, puis de 1 min 30 sec d'élongation à 72 C, puis par 5 nouveaux cycles
caractérisés par une séquence de 1 min de dénaturation à 97 C, suivie de
1 min d'hybridation à 52 C, puis de 1 min 30 sec d'élongation à 72 C. Ensuite
cycles caractérisés par une séquence de 1 min de dénaturation à 97 C,
30 suivie de 1 min d'hybridation à 50 C puis de 1 min 30 sec d'élongation à 72
C
CA 02449836 2003-12-04
WO 02/101003 PCT/FR02/01983
24
ont été effectués. Le protocole s'est achevé par une étape d'élongation à 72 C
pendant 10 min.
Le fragment ainsi amplifié a été purifié sur la colonne du Qiaquick
PCR purification kit (QIAGEN), puis digéré par EcoRi et Hindlll et cloné dans
le
s vecteur pTZ18 préalablement coupé. Trois constructions plasmidiques
obtenues (pKT30, pKT31, pKT34) suite à des amplifications PCR
indépendantes ont été séquencées. Les séquences de ces trois fragments
ainsi clonés étaient identiques. La protéine spécifiée, de séquence SEQ ID
n 4, diffère cependant de cinq acides aminés par rapport à la séquence
publiée par Schwecke et al., à savoir que la protéine de l'invention contient
en
positions 50, 84, 102, 127 et 129 respectivement un résidu de thréonine,
glutamine, acide aspartique, alanine et alanine.
Exemple 3 :Synthèse totale par PCR du gène pipe
Le gène pipA d'une taille de 1066 pb issu de Streptomyces
pristinaespiralis et dont la séquence est décrite dans le brevet
WO-A1-96/01901, est composé d'une proportion en G + C de 69,6%. Ce gène
a été réécrit suivant le code génétique optimisé pour l'expression dans
Escherichia coli décrit dans le tableau 3. Le gène réécrit pipA*, de séquence
SEQ ID n 1 est désormais composé d'une proportion en G + C de 38%. Afin
d'introduire un site de restriction unique Kpnl, le résidu thréonine 97 a été
codé
par ACC au lieu de ACT prévu par le code du tableau 3. Deux codons de
terminaison de la traduction TAA ont été également ajoutés en position 15 et
1080 (1er nucléotide du codon), respectivement de la séquence SEQ ID n 1, et
des sites de restriction uniques EcoRl et Ncol d'une part, Hindlll d'autre
part,
ont été introduits respectivement en 5' et en 3' du gène permettant ainsi son
insertion dans un vecteur de clonage.
La synthèse du gène pipA* a été effectuée en assemblant deux
tronçons EcoRl-%l (tronçon 1 de 305 pb - nucléotide 1 à nucléotide 304) et
Kpnl - Hindlll (tronçon 2 de 798 pb - nucléotide 305 à nucléotide 1092).
CA 02449836 2003-12-04
WO 02/101003 PCT/FR02/01983
Chaque tronçon a été synthétisé par extension d'oligonucléotides
simple brin d'une longueur de 50 à 60 nucléotides se chevauchant (brins "sens"
et brins "anti-sens"), suivie d'une amplification par des oligonucléotides
d'extrémité 5' et 3'.
5
Synthèse du tronçon 1 : La réaction PCR a été effectuée dans un
volume de 100 pL de tampon 20 mM Tris-HCI pH 8.8 contenant 10 mM KCI,
10 mM (NH4)2SO4, 2 mM MgSO4, 0,1% Triton X-100, 0,01 pM des
oligonucléotides listés ci-après, 0,3 mM de chacun des quatre dNTP et 2 unités
10 de Vent DNA Polymerase (BioLabs). Le protocole PCR appliqué était composé
de 30 cycles caractérisés par une séquence de 30 sec de dénaturation à 96 C,
suivie de 30 sec d'hybridation à 55 C, puis de 30 sec d'élongation à 72 C. Au
cycle numéro 10, les oligonucléotides des extrémités 5' (PIPAUP) et 3'
(PIPADI) ont été ajoutés à une concentration finale de 0,5 pM. A la fin du
cycle
15 30, une période d'élongation de 2 minutes à 72 C a été ajoutée.
Les oligonucléotides suivants ont été utilisés pour la réaction :
PipaVO, PipaV2, PipaV4, PipaV6, PipaV8, PipaD4, PipaD5, PipaD6 et PipaD7.
Les séquences de ces oligonucléotides sont décrites au tableau 4.
Le produit PCR ainsi obtenu a été purifié sur la colonne du
20 Qiaquick PCR purification kit (QIAGEN), puis digéré successivement par les
enzymes de restrictions Kpni et EcoRI, et à nouveau purifié sur gel d'agarose.
Une bande de taille apparente de 300 pb a été extraite du gel en utilisant le
Qiaquick gel extraction kit (QIAGEN). Ce produit PCR purifié a été ensuite
ligué
au vecteur pTZ18, préalablement digéré par les enzymes EcoRI et Kpnl et
25 purifié sur gel d'agarose, par la T4 DNA ligase (BioLabs). La réaction de
ligation s'est effectuée pendant une nuit à 16 C dans le tampon de ligation
fourni avec l'enzyme. Le produit de la ligation a été ensuite transformé dans
la
souche E. coli DH5 CIP104738 (D. Hanahan, J. Mol. Biol., 166, (1983), 557-
580) par choc thermique et des clones recombinants sélectionnés sur milieu LB
(DIFCO) agar supplementé avec ampicilline (100 mg/L).
CA 02449836 2009-09-30
26
Les plasmides contenus dans les transformants résistants à
l'ampicilline ont été isolés en utilisant le système de préparation de
plasmides
QlAprep*Spin Miniprep*Kit (QIAGEN). Pour quatre de ces plasmides dont
l'analyse par des enzymes de restriction appropriés montrait un insert de
taille
attendue, les fragments EcoRI-KAnI ont été séquencés. Chacune des
séquences des quatre constructions contenait entre une et trois déviations par
rapport à la séquence attendue. Le plasmide pSP23 pour lequel la séquence
du fragment était correcte à l'exception d'une délétion de 6 pb a été soumis à
une étape de mutagenèse dirigée suivant une méthode publiée (Ansaldi et ai.,
Anal. Biochem., 234, (1996), 110-111) pour corriger cette délétion. Le
plasmide
pSP39 a été ainsi obtenu. Le séquençage du fragment EcoRI-Kpnl du plasmide
pSP39 a été réalisé et la séquence obtenue a été trouvée conforme à celle
attendue.
Synthèse du tronçon 2: La réaction PCR pour la synthèse du
tronçon Kpnl-Hindlll a été effectuée comme décrit pour le tronçon 1 à
l'exception de la période d'élongation dont la durée a été allongée à 1 minute
pour les 30 cycles et à 4 min à la fin du protocole. Au cycle numéro 10, les
oligonucléotides des extrémités 5' (PIPAV29) et 3' (PIPAD3) ont été ajoutés à
une concentration finale de 0,5 pM.
Les oligonucléotides suivants ont été utilisés pour la réaction
PipaV8, PipaV10, PipaV12, PipaV14, PipaV16, PipaV18, PipaV20, PipaV22,
PipaV24, PipaV26, PipaV28, PipaD8, PipaD9, PipaD10, PipaD11, PipaD12,
PipaD13, PipaD14, PipaD15, PipaD16 et PipaD17. Les séquences de ces
oligonucléotides sont décrites au tableau 4.
Le produit PCR a été purifié sur la colonne du Qiaquick PCR
purification kit (Q(AGEN), puis digéré par les enzymes de restriction Kpnnl et
Hindlll et ligué au vecteur pTZI8 préalablement digéré par les mêmes
enzymes. La transformation de la souche E. coli DH5a, et la sélection des
transformants et l'analyse des plasmides ont été réalisés comme décrit
ci-dessus pour le tronçon 1. Le plasmide pSP25 pour lequel la séquence du
* (marques de commerce)
CA 02449836 2003-12-04
WO 02/101003 PCT/FR02/01983
27
fragment Kpnnl-Hindlll comportait cinq déviations par rapport à la séquence
attendue a été soumis à plusieurs étapes de mutagenèse dirigée (Ansaldi et
a/., 1996, ibid.) pour corriger ces erreurs. Le plasmide pSP42 a été ainsi
obtenu.
Assemblage des 2 tronçons : Le gène entier pipA* a été assemblé par clonage
du fragment Kpnl-Hindlll extrait du plasmide pSP42 dans le plasmide pSP39
préalablement coupé par les enzymes Kpnnl et Hindlll. Un des plasmides
résultant, le plasmide pSP43 contenant un fragment EcoRi-Hindlll de taille
attendue a été séquencé et la séquence du gène ainsi vérifiée.
Tableau 4 : Synthèse du gène pipA* séquence des oligonucléotides
Fragment EcoRl-Kpnl :
Pipa VO 5' TTCCAAGGAATTTGTTTACCCAATCTTCAGTCCGTTCGAATTCGAGGTTCC
PipaV2 5' TGATGTTGCAGAAGTTGTTGCAGCAGTTGGTCGTGATGAATTAATGCGTC
PipaV4 5' CAGAAATTGGTCGTGGTGAACGTCATTTATCTCCATTACGTGGTGGTTTA
PipaV6 5' GAATGGATGCCACATCGTGAACCAGGTGATCATATTACTTTAAAAACTGT
PipaV8 5' TGGTTTACCAACTATTTTAGGTACCGTTGCACGTTATGATGATACTACTG
Pipa D4 5' ATA GTT GGT AAA CCA AAA CGA CCT GGA TTT GCT GGA GAA TAA CCA
ACA GTT TTT AAA GTA
Pipa D5 5' ATG TGG CAT CCA TTC CCA AAT ACC TGG AAC TGG TTC AGA ACG TTC
TAA ACC ACC ACG TAA 3'
Pipa D6 5' CAC GAC CAA TTT CTG CTA AAC CAC CAG TTA AAC GAT CGA TAA TAC
GAC GCA TTA ATT CAT 3'
Pipa D7 5' ACT TCT GCA ACA TCA CGA CGA CCT AAA ACC CAA GTT TCC ATG GAA
CCT CGA ATT CG 3'
Oligos externes :
PIPAUP 5' CCCGAATTCGAGGTTCCATGGAAAC
PIPADI 5' CAGTAGTATCATCATAACGTGC
CA 02449836 2003-12-04
WO 02/101003 PCT/FR02/01983
28
Fragment Kpnl-Hindlll :
PipaV8 TGGTTTACCAACTATTTTAGGTACCGTTGCACGTTATGATGATACTACTG
PipaV10 TATTAACTGCATTACGTACTGGTGCAGCATCTGCTGTTGCATCTCGTTTA
PipaV12 TTAATTGGTACTGGTGCACAAGCAGTTACTCAATTACATGCATTATCTTT
PipaV14 GGATACTGATCCAGCACATCGTGAATCTTTTGCACGTCGTGCAGCATTTA
PipaV16 CACGTATTGCAGCAGAAGCAGATGTTATTTCTACTGCAACTTCTGTTGCA
PipaV18 GGTGTTCGTGAACATTTACATATTAATGCAGTTGGTGCAGATTTAGTTGG
PipaV20 ACGTGCATTTGTTACTGCAGATCATCCAGAACAAGCATTACGTGAAGGTG
PipaV22 GTCCACAATTAGCACATTTATGTGCAGATCCAGCAGCAGCAGCAGGTCGT
PipaV24 GGTTTTGCATTTGAAGATGCATTAGCAATGGAAGTTTTTTTAGAAGCAGC
PipaV26 TATTGAACATCATCCAGGTGATGCATTAGATCCATATGCATTACAACCAT
Pipa V28 5' ATA AAG ATC TAA GCT TCC CC
Pipa D8 5' GGGG AAG CTT AGA TCT TTA TTA ATG TGC TGG TGC TGC TAA TGG
TAA TGG TAA TGG TTG TAA TGC AT 3'
Pipa D9 5' GGA TGA TGT TCA ATA CCA ACA CGA ATA CCT AAA TCA CGT TCT GCT
GCT GCT TCT AAA AAA 3'
Pipa D10 5' TTC AAA TGC AAA ACC AGT AGA ATC AAA AAC AGA TAA AGT ATC TTG
ACG ACC TGC TGC TGC 3'
Pipa D11 5' GTG CTA ATT GTG GAC CTA AAC GAT CAG CAG ATA ATT GTT GAC ATT
CAC CTT CAC GTA ATG 3'
Pipa D12 5' GTA ACA AAT GCA CGT TCT AAT AAA CCT AAT GGT AAT TCA GTT TTA
CCA ACT AAA TCT GCA 3'
Pipa D13 5' ATG TTC ACG AAC ACC AGT ATC TGG TAA AAC TGG ACC TTG ACC AAC
TGC AAC AGA AGT TGC 3'
Pipa D14 5' CTG CTG CAA TAC GTG CTG GTT CTG CAA TTT CAA CAG AAA CAC CAG
TAA ATG CTG CAC GAC 3'
CA 02449836 2003-12-04
WO 02/101003 PCT/FR02/01983
29
Pipa D15 5' GCT GGA TCA GTA TCC CAA ACT AAT GCA CGT TGT AAT GGT AAA ACT
AAA GAT AAT GCA TGC 3'
Pipa D16 5' ACC AGT ACC AAT TAA ACC TAA AGT ATG AGA ATC TGG ACG TGC TAA
TAA ACG AGA TGC AAC 3'
Pipa D17 5' GTA ATG CAG TTA ATA AAA CAC CAT CCA TTA ATG CAG TTA ATG CAC
CAG TAG TAT CAT CAT 3'
Oligos externes :
PIPA V29 5' CTA TTT TAG GTA CCG TTG CA
PIPAD3 5' CCCAAGCTTAGATCTTTATTAATGTG
Exemple 4:
Synthèse totale par ligation du gène rapL*, et obtention du variant rapL**
Le gène rapL d'une taille de 1029 pb issu de Streptomyces
hygroscopicus et dont la séquence a été publiée (Schwecke et al., 1995,
ibid.),
est composé d'une proportion en G + C de 68%. Ce gène a été réécrit en
suivant le code génétique employé pour la synthèse du gène pipA* à l'exemple
3. Le gène réécrit rapL*, de séquence SEQ ID n 2 est désormais composé
d'une proportion en G + C de 38%. Afin d'introduire un site de restriction
unique
Knl, le résidu thréonine 97 a été codé par ACC au lieu de ACT prévu par le
code représenté au tableau 3. Deux codons TAA de terminaison de la
traduction ont été également ajoutés et des sites de restriction uniques EcoRI
et Sphl d'une part, Hindlll d'autre part ont été introduits respectivement en
5' et
en 3' du gène permettant ainsi son insertion dans différents vecteurs de
clonage.
La synthèse totale du gène rapL* a été effectuée en assemblant
les trois tronçons EcoRI-Kpnl (tronçon 1 de 305 pb), Kpnni-Pstl (tronçon 2 de
316 pb), et Pstl-Hindlll (tronçon 3 de 440 pb).
CA 02449836 2003-12-04
WO 02/101003 PCT/FR02/01983
Chaque tronçon a été synthétisé par ligation d'oligonucléotides
simple brin phosphorylés en 5' d'une longueur de 50 nucléotides en moyenne,
recouvrant la totalité de la séquence à cloner (brins "sens" et brins "anti-
sens")
et se chevauchant. Les séquences et positions de ces oligonucléotides sont
5 décrites au tableau 5. Les oligonucléotides, mélangés de façon équimolaire
(1 pM) dans le tampon de chevauchement (20 mM Tris-HCI, pH 8; 0,08 M
NaCI) dans un volume final de 20 pL, ont été chauffés 10 minutes à 70 C et
maintenus dans le bloc chauffant jusqu'à retour à la température ambiante. Le
produit ainsi obtenu a été ensuite ligué par la T4 DNA ligase pendant une nuit
à
10 16 C en présence de vecteur pTZ18 (rapport de concentration molaire
vecteur/oligonucléotides 1/100) préalablement digéré par les enzymes de
restriction appropriés.
Le produit de ligation a été utilisé pour transformer par
électroporation la souche 02033 (E. coli K12, pro, thi, rpsL, hsdS, AlacZ,
15 F'(AIacZM15, lacla, traD36, proA+, proB+)) et les clones recombinants ont
été
sélectionnés sur milieu LB agar contenant 100 pg/mL ampicilline, 0,5 mM IPTG
et 30 pg/mL X-Gal.
Les plasmides contenus dans plusieurs transformants blancs
résistants à l'ampicilline ont été isolés en utilisant le système de
préparation de
20 plasmides QlAprep Spin Miniprep Kit (QIAGEN). Pour chaque tronçon, les
inserts de deux à quatre plasmides dont l'analyse par enzymes de restriction
appropriés montrait un fragment de taille attendue, ont été séquencés.
Synthèse du tronçon 1 :
25 Le plasmide pTZ18 coupé par EcoRl et Kpnl a été ligaturé avec
les oligonucléotides suivants (dont la séquence est illustrée au tableau 5) :
IcdV-1, IcdV-2, IcdV-3, lcdV-4, IcdV-5, IcdV-6, IcdVrev-16, IcdVrev-17,
lcdVrev-
18, IcdVrev-19, lcdVrev-20, IcdVrev-21, IcdVrev-22. Le plasmide pSP3 dont la
séquence de l'insert comportait seulement deux déviations par rapport à la
30 séquence attendue a été retenu. Les deux erreurs ont été corrigées par
mutagenèse dirigée suivant une méthode publiée (Ansaldi et al., 1996, ibid.)
et
CA 02449836 2003-12-04
WO 02/101003 PCT/FR02/01983
31
les corrections confirmées par séquençage de l'insert du plasmide pSP14
résultant.
Synthèse du tronçon 2 :
s Le plasmide pTZ18 coupé par Kjnl et Pstl a été ligaturé avec les
oligonucléotides suivants (dont la séquence est illustrée au tableau 5) : IcdV-
7,
IcdV-8, IcdV-9, IcdV-10, IcdV-11, lcdV-12, lcdVrev-9, IcdVrev-10, IcdVrev-11,
IcdVrev-12, IcdVrev-13, lcdVrev-14, IcdVrev-15. Le plasmide pSP8 dont la
séquence de l'insert comportait seulement une déviation par rapport à la
séquence attendue a été retenu. L'erreur a été corrigée par mutagenèse
dirigée (Ansaldi et al., 1996, ibid.) et la correction confirmée par
séquençage de
l'insert du plasmide pSP15 résultant.
Synthèse du tronçon 3 :
Le plasmide pTZ18 coupé par Pstl et Hindlll a été ligaturé avec
les oligonucléotides suivants (dont la séquence est illustrée au tableau 5) :
lcdV-13, lcdV-14, IcdV-15, IcdV-16, lcdV-17, lcdV-18, lcdV-19, lcdV-20, lcdV-
21,
IcdVrev-1, IcdVrev-2, IcdVrev-3, IcdVrev-4, IcdVrev-5, IcdVrev-6, IcdVrev-7,
lcdVrev-8. Le plasmide pSP12 dont la séquence de l'insert comportait
seulement une délétion de 25 pb et une déviation ponctuelle a été retenu. Les
deux erreurs ont été corrigées par mutagenèse dirigée (Ansaldi et al., 1996,
ibid.) et les corrections confirmées par séquençage de l'insert du plasmide
pSP26 résultant.
Assemblage des 3 tronçons : Les fragments corrigés Kpnl-Pstl,
puis Pstl-Hindlll sont sous-clonés successivement dans le plasmide pSP14
contenant déjà le fragment EcoRI-Kpnl, reconstituant ainsi le gène synthétique
rapL*. La séquence du gène rapL* du plasmide résultant pSP33 a été
confirmée par séquençage.
CA 02449836 2003-12-04
WO 02/101003 PCT/FR02/01983
32
Obtention du variant rapL** (Séquence SEQ ID n 5) : Le variant
rapL** a été obtenu à partir du gène rapL* du plasmide pSP33 en introduisant
par mutagenèse dirigée (Ansaldi et al., 1996, ibid.) les cinq changements
nécessaires pour que la protéine codée par le gène rapL** soit la même que
celle codée par le gène amplifié et séquencé décrit à l'exemple 2 et
corresponde à la séquence SEQ ID n 4, tout en respectant le code génétique
décrit au tableau 3. Un plasmide pSP36 dérivé du plasmide pTZI 8 et contenant
le gène rapL** a été ainsi obtenu.
Tableau 5 : Synthèse du gène rapL* séquence des oligonucléotides
Fragment EcoRi-Kpni :
Brin "sens" :
IcdV-1: 5'-AATTCGAGGTTGCATGCAAACTAAAGTTTTATGTCAACGTGATATTAA-3'
IcdV-2: 5'p-ACGTATTTTATCTGTTGTT GGTCGTGATGTT ATG ATG GAT
CGT TTA ATT T-3'
lcdV-3: 5' p-CTGAAGTTCAT GCA GGT TTT GCA CGT TTA GGT
CGT GGT GAAACT GATGAA-3'
IcdV-4 : 5' p-CCACCA CCA CGT CCA GGT TTT GCA CGT GGT
GGT GAT GTT CCAGGT GTTAT -3'
IcdV-5 : 5' p-T GAA TTT ATG CCA CAT CGT GCA TCT GGT
ATT GGT GTTACT ATGAAA ACTG -3'
IcdV-6 : 5' p-TT TCT TAT TCT CCA GAA AAT TTT GAA CGT
TTTAAT TTACCA ACT ATT GTT GGT AC-3'
Brin "anti-sens":
IcdVrev-16 : 5' p-CAACAATAG TTGGTAAATTAAAA-3'
IcdVrev-17 : 5' p-CGTTCAAAATTTTCTGGAGAATAAGAAACAGTTTTCATAGTAACACCAAT-
3'
IcdVrev-18 : 5' p-ACCAGATGCACGATGTGGCATAAATTCAATAACACCTGGAACATCACCA-
3'
IcdVrev-19: 5' p-
CCACGTGCAAAACCTGGACGTGGTGGTGGTTCATCAGTTTCACCACGACCT-3'
CA 02449836 2003-12-04
WO 02/101003 PCT/FR02/01983
33
IcdVrev-20: 5' p-
AAACGTGCAAAACCTGCATGAACTTCAGAAATTAAACGATCCATCATAAC-3'
IcdVrev-21 : 5' p-ATCACGACCAACAACAGATAAAATACGTTTAATATCACGTTGA-3'
IcdVrev-22: 5' p-CATAAAACTTTAGTTTGCATGCAACCTCG-3'
Fragment Kpni-Psti :
Brin "sens":
IcdV-7 : 5' p-CGTTTCT CGT TTA GGT GAT GATTCT GGTTCT
ATG GTT GCA TTA GC -3'
IcdV-8 : 5' p-A GAT GCA GCAACTATT ACT GCAATGCGTACT GGT
GCA GTT GCA TCT GTT A -3'
IcdV-9 : 5' p-CT ACT CGT TTATTAGCACGTCCAGGT TCT ACT ACT
TTA GCA TTA ATT GGT -3'
IcdV-10 : 5' p- GCA GGT GCACAA GCAGTTACT CAA GCA CAT
GCA TTA TCT CGT GTT TTA CC-3'
IcdV-11 : 5' p-A TTA GAACGT ATTTTA ATTTCTGAT ATT AAA
GCA GAA CAT GCA GAA TCT T-3'
IcdV-12 : 5' p-TTGCAGGTCGT GTT GCA TTTTTAGAA TTA CCA GTT
GAA GTT ACT GATGCAGCAACT GCA ATG GCA ACT
GCA-3'
Brin "anti-sens":
IcdVrev-9: 5' p-GTTGCCATTGCAGTTGCTGCATCAGTAACTTCAA-3'
IcdVrev-10 : 5' p-
CTGGTAATTCTAAAAATGCAACACGACCTGCAAAAGATTCTGCATGTTCTGCTTT-3'
IcdVrev-11 : 5' p-AATATCAGAAATTAAAATACGTTCTAATGGTAAAACACGAGATAATGCA-
3'
IcdVrev-12: 5' p-
TGTGCTTGAGTAACTGCTTGTGCACCTGCACCAATTAATGCTAAAGTAGTA-3'
IcdVrev-13 : 5' p-
GAACCTGGACGTGCTAATAAACGAGTAGTAACAGATGCAACTGCACCAGT-3'
(cdVrev-14 : 5' p-ACGCATTGCAGTAATAGTTGCTGCATCTGCTAATGCAACC-3'
IcdVrev-15 : 5' p-ATAGAACCAGAATCATCACCTAAACGAGAAACGGTAC-3'
CA 02449836 2003-12-04
WO 02/101003 PCT/FR02/01983
34
Fragment Psti-Hindlll
Brin "sens":
IcdV-13 : 5' p-GATGTTTTA TGT ACT GTT ACTTCTGTTCC -3'
IcdV-14 : 5' p-A GTT GGT GGT GGT CCAGTTGTTCCA GCA GAA
CCA CGTCAAGCACAT TTA C-3'
IcdV-15 : 5' p-AT GTT AAT GGT ATT GGT GCA GATGAACAAGGT
AAAACTGAA TTA CCA AAA -3'
IcdV-16 : 5' p-GCA TTA TTA GAT GAT GCA TTT ATT TGT
GTTGATCAT CCA GGT CAA GCA CG-3'
IcdV-17 : 5' p-T GCA GAA GGT GAA TTT CAACAA
TTACCAGATCGTGAA TTA GGT CCA TCT T-3'
lcdV-18: 5' p-TA GCA GAT TTA TGT GCAGCACCAGAA ATT GCA
GCACCACAT CCA GAA CGT-3'
IcdV-19: 5' p- TTA TCT GTT TTTGATTCTACT GGT TCT GCA
TTT GCA GAT C ATATTGCA TTA GAT GTTTTATTA-3'
IcdV-20 : 5' p-GGT TTT GCA GAT GAA TTA GGT TTA GGT CAT
AAAATGTCTATT GAAT-3'
IcdV-21 : 5' p-CTACT CCA GAA GAT GTT TTA GAT CCA TAT
TCT TTATAATAAAGATCTA -3'
Brin "anti-sens":
IcdVrev-1 : 5' p-
AGCTTAGATCTTTATTATAAAGAATATGGATCTAAAACATCTTCTGGAGTAGATT
CAATA GACATTTTATGAC -3'
lcdVrev-2: 5' p-
CTAAACCTAATTCATCTGCAAAACCTAATAAAACATCTAATGCAATATGA-3'
lcdVrev-3: 5' p-
TCTGCAAATGCAGAACCAGTAGAATCAAAAACAGATAAACGTTCTGGATG-3'
lcdVrev-4: 5' p-TGGTGCTGCAATTTCTGGTGCTGCACATAAATCTGCTAAAGATGGACCT-
3'
IcdVrev-5 : 5' p-
AATTCACGATCTGGTAATTGTTGAAATTCACCTTCTGCACGTGCTTGACCT-3'
lcdVrev-6: 5' p-GGATGATCAACACAAATAAATGCATCATCTAATAATGCTTTTGGTAATT-
3'
IcdVrev-7 : 5' p-
CAGTTTTACCTTGTTCATCTGCACCAATACCATTAACATGTAAATGTGCTT-3'
CA 02449836 2003-12-04
WO 02/101003 PCT/FR02/01983
IcdVrev-8: 5' p-
GACGTGGTTCTGCTGGAACAACTGGACCACCACCAACTGGAACAGAAGTAACAGT
ACATAAAACATCTGCA-3'
5
Exemple 5 : Surexpression du gène pipA* chez Escherichia coll.
Le gène pA* a été cloné dans le vecteur pQE60 (QIAGEN)
(plasmide dont les séquences codantes ont été préalablement modifiées
10 comme indiqué précédemment), entre les sites de restriction Ncol et Hindlil
à
partir du plasmide pSP43 construit à l'exemple 3. Le plasmide pSP47 résultant
a été introduit dans une souche d'E. coli K12 MG1655 (Vidal et al., 1998)
exprimant le gène lacl à partir du plasmide auxiliaire pREP4 (QIAGEN). La
souche +75 ainsi obtenue a été cultivée dans le milieu LB et l'expression du
15 gène pipA* a été induite par l'ajout d'IPTG suivant le protocole du
fournisseur.
Les protéines totales des cellules ont été séparées par électrophorèse en gel
polyacrylamide/SDS et colorées par le bleu de Coomassie. Une protéine au
poids moléculaire de 36 kD a été détectée et son taux d'expression a été
estimé à environ 5% de protéines totales.
Exemple 6 : Surexpression des gènes pipA, rapL, rapL*, et rapL** chez
Escherichia coll.
En procédant comme décrit à l'exemple 5 une souche d'E. coli
surexprimant le gène pipA (souche +353) a été obtenue en introduisant le
plasmide pKT37. Ce plasmide a été obtenu à partir du plasmide pKT36, décrit
à l'exemple 1, par clonage du gène d'intérêt entre les sites de restriction
Ncol et
Hindlll du vecteur pQE60.
De même, des souches d'E. coli surexprimant les gènes rapL
(souche +38), rapL* (souche +60), ou rapL** (souche +73) ont été obtenues en
introduisant respectivement les plasmides pSP32, pSP37, ou pSP45. Ces
plasmides pSP32, pSP37, ou pSP45 ont été obtenus respectivement à partir
des plasmides pKT30, pSP33, ou pSP36, décrits dans les exemples 2 et 4, par
CA 02449836 2003-12-04
WO 02/101003 PCT/FR02/01983
36
clonage du gène d'intérêt entre les sites de restriction Sphl et Hindlll du
vecteur
pQE70.
Le taux d'expression des ces différentes protéines à été
déterminé comme décrit à l'exemple 5. Pour chaque souche, une protéine au
poids moléculaire attendu a été détectée avec un taux d'expression d'environ
5% de protéines totales.
Exemple 7 : Amplification par PCR et surexpression du gène ocd
d'Agrobacterium tumefaciens chez Escherichia coll.
Une préparation du plasmide Ti-C58 d'Agrobactérium tumefaciens
CIP 104333 a été réalisée comme décrit par ailleurs (Hayman et al., Mol. Gen.
Genet., 223, (1990), 465-473). Le gène ocd (Sans et al., (1988), ibid.) a été
amplifié par PCR à partir de cette préparation du plasmide Ti-C58.
La réaction a été effectuée dans un volume de 50 pL de tampon
20 mM Tris-HCI pH 8.8 contenant 10 mM KCI, 10 mM (NH4)2SO4, 2 mM
MgSO4, 0,1% Triton X-100, 2 ng du plasmide Ti-C58, 200 pM de chacun des
quatre dNTP, 1 unité de la Vent DNA Polymérase (BioLabs) et 500 nM de
chacun des deux oligodésoxynucléotides suivants :
5'CCCGCATGCCTGCACTTGCCAACC
5'CCCAGATCTTAACCACCCAAACGTCGGAAA
Le protocole PCR appliqué a débuté par une étape de
dénaturation à 94 C pendant 2 min suivie par 30 cycles caractérisés par une
séquence de 30 sec de dénaturation à 94 C, suivie de 30 sec d'hybridation à
52 C, puis de 1 min 30 sec d'élongation à 72 C. Le protocole s'est achevé par
une étape d'élongation à 72 C pendant 10 min.
Le fragment ainsi amplifié a été digéré par Ncol et PU III et cloné
dans le vecteur pQE70 (QIAGEN) (plasmide dont les séquences codantes ont
été préalablement modifiées comme indiqué précédemment) préalablement
coupé. Le fragment Ncol-Bglll du plasmide pKR7 ainsi obtenu a été séquencé.
La protéine spécifiée diffère de deux acides aminés par rapport à la séquence
publiée (Sans et al., (1988), ibid.), à savoir le résidu 212GIn remplacé par
la
CA 02449836 2003-12-04
WO 02/101003 PCT/FR02/01983
37
lysine et le résidu 29711e remplacé par la leucine. Les deux déviations ont
été
confirmées par séquençage d'un produit d'amplification par PCR obtenu
indépendamment.
En procédant comme décrit à l'exemple 5 une souche d'E. coli
surexprimant le gène ocd (souche +78) a été obtenue en introduisant le
plasmide pKR7. Le taux d'expression de la protéine codée par le gène ocd a
été déterminé comme décrit à l'exemple 5. Une protéine de poids moléculaire
conforme à celui qui était attendu a été détectée avec un taux d'expression
d'environ 5% des protéines totales.
Exemple 8 :
Production d'acide L-pipécolique à partir de la L-lysine par des
suspensions cellulaires des souches d'E. coli recombinantes.
La souche +75, construite à l'exemple 5, est cultivée dans le
milieu minéral MS (Richaud et al., J. Biol. Chem., 268, (1993), 26827-35)
contenant 2 g/L glucose, 50 mg/L ampicilline et 30 mg/L kanamycine. Au bout
de 14 heures de culture, 400 mL de milieu minéral MS contenant 2 g/L glucose
sont ensemencées au 1/10 avec la préculture de la souche +75. Cette culture
est placée à 37 C sous agitation jusqu'à atteindre une DO600nrr, de 0,7. Le
gène
pipA* est alors induit pendant 4 heures à 37 C par l'ajout de 1 mM d'IPTG à
200 mL de culture. Une culture témoin de 200 mL sans ajout d'IPTG est
également réalisée. A la fin de cette étape, les deux cultures sont
centrifugées
à 13000 g et les culots cellulaires sont repris dans du milieu minéral MS de
façon à concentrer les cellules 100 fois. Les cellules sont alors
perméabilisées
par deux étapes de congélation/décongélation à -20 C favorisant l'accès du
substrat à l'enzyme. La L-lysine monohydrochloride (pH = 7) est alors ajoutée
à
une concentration finale de 1 M dans les suspensions cellulaires (volume
final = 2 mL). La réaction enzymatique se fait à 37 C sous agitation. Au bout
de
20 heures on prélève un échantillon de 300 pL de chaque culture que l'on
centrifuge à 13000 g de façon à récupérer le surnageant. Une solution diluée
CA 02449836 2003-12-04
WO 02/101003 PCT/FR02/01983
38
des surnageants est ensuite déposée sur couche mince de silice et en parallèle
dosée par CLHP.
Un composé à la migration chromatographique (Rf = 0.22 ;
éluant : butanol/acide acétique/eau 4/1/1 en CCM) et à la coloration par la
ninhydrine indiscernables de celles de l'acide L-pipécolique est détecté dans
le
cas de la culture induite à l'IPTG et absent dans le cas de la culture témoin
non
induite.
Le dosage par CLHP (conditions décrites ci-dessous) indique une
concentration de 40 g/L de L-pipécolate au bout de 20 heures. L'excès
énantiomérique est supérieur à 95%.
Des essais de production d'acide L-pipécolique à partir de la
L-lysine ont été réalisés avec les souches +38, +60, +73, +78 et +353
construites aux exemples 6 et 7, dans les mêmes conditions que celles
appliquées ci-dessus pour la souche +75. L'analyse par chromatographie des
surnageants respectifs sur couche mince de silice après coloration par la
ninhydrine n'a révélé une production d'acide L-pipécolique qu'avec les souches
+73, +78 et +353.
Description de la méthode CLHP utilisée :
- Colonne Phenomenex Synergi Polar-RP-80 4p (250 x 4,6 mm)
thermostatée à 30 C
- Détection UV à 210 nm
- Volume d'échantillon injecté : 10 pL
- Élution isocratique par une solution de 0,05% de TFA dans
l'eau pendant 5 minutes suivi d'un lavage de la colonne par 80%
d'acetonitrile et 0,05% de TFA dans l'eau.
- Temps de rétention : lysine à 2,98 min et acide pipécolique à
4,62 min.
CA 02449836 2003-12-04
WO 02/101003 PCT/FR02/01983
39
Exemple 9 :
Production d'acide L-thiomorpholine-2-carboxylique à partir de la
L-thialysine
La production d'acide L-thiomorpholine-2-carboxylique a été
réalisée à partir d'une suspension cellulaire de la souche +75 préparée comme
décrit à l'exemple 8. La L-thialysine (S-(2-aminoethyl)-L-cysteïne) (SIGMA)
est
ajoutée à une concentration finale de 1 M. Une chromatographie sur couche
mince de silice du surnageant de la suspension incubée est réalisée et elle
révèle un composé à la migration (Rf = 0,28 ; éluant butanol/acide acétique/
eau 4/1/1) et à la coloration par la ninhydrine distinctes de celles de la
L-thialysine et de l'acide L-pipécolique.
Dans les mêmes conditions, la souche +78 décrite à l'exemple 7
produit un composé identique à celui produit par la souche +75.
Exemple 10:
Production d'un mélange d'acides 5-R- et 5-S- hydroxy-L-pipécolique à
partir d'un mélange de L- et D- 5-(R,S) hydroxylysine
La production d'un mélange d'acides 5-R- et 5-S- hydroxy-
L-pipécolique a été réalisée à partir d'une suspension cellulaire de la souche
+75 préparée comme décrit à l'exemple 8. La 5-R,S-hydroxy-D,L-lysine
(SIGMA) est ajoutée à 1 M final. Une chromatographie sur couche mince de
silice du surnageant de la suspension incubée est réalisée et elle révèle deux
composés présents en proportions équivalentes, à la migration (Rf1 = 0,14 ;
Rf2 = 0,20 ; éluant butanol/acide acétique/eau 4/1/1) et à la coloration par
la
ninhydrine distinctes -de celles de la 5-R,S-hydroxy-D,L-lysine et de l'acide
L-pipécolique.
Dans les mêmes conditions, la souche +78 décrite à l'exemple 7
produit deux composés identiques à ceux produits par la souche +75.
CA 02449836 2003-12-04
WO 02/101003 PCT/FR02/01983
Exemple 11 : Préparations d'autres acides aminés cycliques
La grande polyvalence du procédé selon la présente invention peut être
illustrée grâce aux acides aminés cycliques suivant, obtenus en opérant selon
le mode opératoire décrit à l'exemple 8, en modifiant la nature de l'acide
5 diaminé de départ:
L-Acide diaminé L-Acide aminé cyclique
Acide 2,6-diaminoheptanedioïque Acide pipéridine-2,6-heptanedioïque
Acide 2,6-diamino-5-hydroxyhexanoïque Acide 5-hydroxypipéridine-2-
carboxylique
Acide 2-amino-3-(2- Acide thiomorpholine-3-carboxylique
aminoéthylsulfanyl)propanôique
Azalysine (acide j3N-2-aminoéthyl-a,
~i- Acide pipérazine-2-carboxylique
diaminopropionique)
CA 02449836 2003-12-04
WO 02/101003 PCT/FR02/01983
1/5
LISTE DE SEQUENCES
<110> Rhodia
<120> Préparation stéréosélective de L-acides aminés cycliques
<130> BFF 00/0696
<140>
<141>
<160> 5
<170> Patentln Ver. 2.1
<210> 1
<211>= 1097
<212> ADN
<213> Séquence artificielle
<220>
<223> Description de la séquence artificielle: séquence nucléique du gène
synthétique pipA*
<400> 1
gaattcgagg ttccatggaa acttgggttt taggtcgtcg tgatgttgca gaagttgttg 60
cagcagttgg tcgtgatgaa ttaatgcgtc gtattatcga tcgtttaact ggtggtttag 120
cagaaattgg tcgtggtgaa cgtcatttat ctccattacg tggtggttta gaacgttctg 180
aaccagttcc aggtatttgg gaatggatgc cacatcgtga accaggtgat catattactt 240
taaaaactgt tggttattct ccagcaaatc caggtcgttt tggtttacca actattttag 300
gtaccgttgc acgttatgat gatactactg gtgcattaac tgcattaatg gatggtgttt 360
tattaactgc attacgtact ggtgcagcat ctgctgttgc atctcgttta ttagcacgtc 420
cagattctca tactttaggt ttaattggta ctggtgcaca agcagttact caattgcatg 480
cattatcttt agttttacca ttacaacgtg cattagtttg ggatactgat ccagcacatc 540
gtgaatcttt tgcacgtcgt gcagcattta ctggtgtttc tgttgaaatt gcagaaccag 600
cacgtattgc agcagaagca gatgttattt ctactgcaac ttctgttgca gttggtcaag 660
gtccagtttt accagatact ggtgttcgtg aacatttaca tattaatgca gttggtgcag 720
atttagttgg taaaactgaa ttaccattag gtttattaga acgtgcattt gttactgcag 780
atcatccaga acaagcatta cgtgaaggtg aatgtcaaca attatctgct gatcgtttag 840
gtccacaatt agcacattta tgtgcagatc cagcagcagc agcaggtcgt caagatactt 900
tatctgtttt tgattctact ggttttgcat ttgaagatgc attagcaatg gaagtttttt 960
tagaagcagc agcagaacgt gatttaggta ttcgtgttgg tattgaacat catccaggtg 1020
atgcattaga tccatatgca ttacaaccat taccattacc attagcagca ccagcacatt 1080
aataaagatc taagctt 1097
<210> 2
<211> 1061
<212> ADN
<213> Séquence artificielle
<220>
<223> Description de la séquence artificielle: séquence nucléique du gène
synthétique rapL*
<400> 2
gaattcgagg ttgcatgcaa actaaagttt tatgtcaacg tgatattaaa cgtattttat 60
ctgttgttgg tcgtgatgtt atgatggatc gtttaatttc tgaagttcat gcaggttttg 120
cacgtttagg tcgtggtgaa actgatgaac caccaccacg tccaggtttt gcacgtggtg 180
gtgatgttcc aggtgttatt gaatttatgc cacatcgtgc atctggtatt ggtgttacta 240
tgaaaactgt ttcttattct ccagaaaatt ttgaacgttt taatttacca actattgttg 300
CA 02449836 2003-12-04
WO 02/101003 PCT/FR02/01983
2/5
gtaccgtttc tcgtttaggt gatgattctg gttctatggt tgcattagca gatgcagcaa 360
ctattactgc aatgcgtact ggtgcagttg catctgttac tactcgttta ttagcacgtc 420
caggttctac tactttagca ttaattggtg caggtgcaca agcagttact caagcacatg 480
cattatctcg tgttttacca ttagaacgta ttttaatttc tgatattaaa gcagaacatg 540
cagaatcttt tgcaggtcgt gttgcatttt tagaattacc agttgaagtt actgatgcag 600
caactgcaat ggcaactgca gatgttttat gtactgttac ttctgttcca gttggtggtg 660
gtccagttgt tccagcagaa ccacgtcaag cacatttaca tgttaatggt attggtgcag 720
atgaacaagg taaaactgaa ttaccaaaag cattattaga tgatgcattt atttgtgttg 780
atcatccagg tcaagcacgt gcagaaggtg aatttcaaca attaccagat cgtgaattag 840
gtccatcttt agcagattta tgtgcagcac cagaaattgc agcaccacat ccagaacgtt 900
tatctgtttt tgattctact ggttctgcat ttgcagatca tattgcatta gatgttttat 960
taggttttgc agatgaatta ggtttaggtc ataaaatgtc tattgaatct actccagaag 1020
atgttttaga tccatattct ttataataaa gatctaagct t 1061
<210> 3
<211> 355
<212> PRT
<213> Streptomyces pristïnaespiralis
<220>
<223> Séquence protéique du gène pipA
<400> 3
Met Glu Thr Trp Val Leu Gly Arg Arg Asp Val Ala Glu Val Val Ala
1 5 10 15
Ala Val Gly Arg Asp Glu Leu Met Arg Arg Ile Ile Asp Arg Leu Thr
20 25 30
Gly Gly Leu Ala Glu Ile Gly Arg Gly Glu Arg His Leu Ser Pro Leu
35 40 45
Arg Gly Gly Leu Glu Arg Ser Glu Pro Val Pro Gly Ile Trp Glu Trp
50 55 60
Met Pro Ris Arg Glu Pro Gly Asp Ris Ile Thr Leu Lys Thr Val Gly
65 70 75 80
Tyr Ser Pro Ala Asn Pro Ala Arg Phe Gly Leu Pro Thr Ile Leu Gly
85 90 95
Thr Val Ala Arg Tyr Asp Asp Thr Thr Gly Ala Leu Thr Ala Leu Met
100 105 110
Asp Gly Val Leu Leu Thr Ala Leu Arg Thr Gly Ala Ala Ser Ala Val
115 120 125
Ala Ser Arg Leu Leu Ala Arg Pro Asp Ser Ris Thr Leu Gly Leu Ile
130 135 140
Gly Thr Gly Ala Gin Ala Val Thr Gln Leu Ris Ala Leu Ser Leu Val
145 150 155 160
Leu Pro Leu Gln Arg Ala Leu Val Trp Asp Thr Asp Pro Ala Ris Arg
165 170 175
Glu Ser Phe Ala Arg Arg Ala Ala Phe Thr Gly Val Ser Val Glu Ile
180 185 190
Ala Glu Pro Ala Arg Ile Ala Ala Glu Ala Asp Val I1e Ser Thr Ala
195 200 205
CA 02449836 2003-12-04
WO 02/101003 PCT/FR02/01983
3/5
Thr Ser Val Ala Val Gly Gln Gly Pro Val Leu Pro Asp Thr Gly Val
210 215 220
Arg Glu His Leu His Ile Asn Ala Val Gly Ala Asp Leu Val Gly Lys
225 230 235 240
Thr Glu Leu Pro Leu Gly Leu Leu Glu Arg Ala Phe Val Thr Ala Asp
245 250 255
His Pro Glu Gln Ala Leu Arg Glu Gly Glu Cys Gln Gln Leu Ser Ala
260 265 270
Asp Arg Leu Gly Pro Gln Leu Ala His Leu Cys Ala Asp Pro Ala Ala
275 280 285
Ala Ala Gly Arg Gln Asp Thr Leu Ser Val Phe Asp Ser Thr Gly Phe
290 295 300
Ala Phe Glu Asp Ala Leu Ala Met Glu Val Phe Leu Glu Ala Ala Ala
305 310 315 320
Glu Arg Asp Leu Gly Ile Arg Val Gly Ile Glu His His Pro Gly Asp
325 330 335
Ala Leu Asp Pro Tyr Ala Leu Gln Pro Leu Pro Leu Pro Leu Ala Ala
340 345 350
Pro Ala His
355
<210> 4
<211> 343
<212> PRT
<213> Streptomyces hygroscopicus
<220>
<223> Séquence protéique du gène rapL
<400> 4
Met Gln Thr Lys Val Leu Cys Gln Arg Asp Ile Lys Arg Ile Leu Ser
1 5 10 15
Val Val Gly Arg Asp Val Met Met Asp Arg Leu Ile Ser Glu Val His
20 25 30
Ala Gly Phe Ala Arg Leu Gly Arg Gly Glu Thr Asp Glu Pro Pro Pro
35 40 45
Arg Thr Gly Phe Ala Arg Gly Gly Asp Val Pro Gly Val Ile Glu Phe
50 55 60
Met Pro His Arg Ala Ser G1y Ile Gly Val Thr Met Lys Thr Val Ser
65 70 75 80
Tyr Ser Pro Gln Asn Phe Glu Arg Phe Asn Leu Pro Thr Ile Val Gly
85 90 95
Thr Val Ser Arg Leu Asp Asp Asp Ser Gly Ser Met Val Ala Leu Ala
100 105 110
CA 02449836 2003-12-04
WO 02/101003 PCT/FR02/01983
4/5
Asp Ala Ala Thr Ile Thr Ala Met Arg Thr Gly Ala Val Ala Ala Val
115 120 125
Ala Thr Arg Leu Leu Ala Arg Pro Gly Ser Thr Thr Leu Ala Leu Ile
130 135 140
Gly Ala Gly Ala Gln Ala Val Thr Gln Ala His Ala Leu Ser Arg Val
145 150 155 160
Leu Pro Leu Glu Arg Ile Leu Ife Ser Asp Ile Lys Ala Glu His Ala
165 170 175
Glu Ser Phe Ala Gly Arg Val Ala Phe Leu Glu Leu Pro Val Glu Val
180 185 190
Thr Asp Ala Ala Thr Ala Met Ala Thr Ala Asp Val Leu Cys Thr Val
195 200 205
Thr Ser Val Pro Val Gly Gly Gly Pro Val Val Pro Ala Glu Pro Arg
210 215 220
Gln Ala His Leu His Val Asn Gly Ile Gly Ala Asp Glu Gln Gly Lys
225 230 235 240
Thr Glu Leu Pro Lys Ala Leu Leu Asp Asp Ala Phe Ile Cys Val Asp
245 250 255
His Pro Gly Gln Ala Arg Ala Glu Gly Glu Phe Gln Gln Leu Pro Asp
260 265 270
Arg Glu Leu Gly Pro Ser Leu Ala Asp Leu Cys Ala Ala Pro Glu Ile
275 280 285
Ala Ala Pro His Pro Glu Arg Leu Ser Val Phe Asp Ser Thr Gly Ser
290 295 300
Ala Phe Ala Asp His Ile Ala Leu Asp Val Leu Leu Gly Phe Ala Asp
305 310 315 320
Glu Leu Gly Leu Gly His Lys Met Ser Ile Glu Ser Thr Pro Glu Asp
325 330 335
Val Leu Asp Pro Tyr Ser Leu
340
<210> 5
<211> 1061
<212> ADN
<213> Séquence artificielle
<220>
<223> Description de la séquence artificielle: séquence nucléique du gène
synthétique rapL**
<400> 5
gaattcgagg ttgcatgcaa actaaagttt tatgtcaacg tgatattaaa cgtattttat 60
ctgttgttgg tcgtgatgtt atgatggatc gtttaatttc tgaagttcat gcaggttttg 120
cacgtttagg tcgtggtgaa actgatgaac caccaccacg tactggtttt gcacgtggtg 180
gtgatgttcc aggtgttatt gaatttatgc cacatcgtgc atctggtatt ggtgttacta 240
CA 02449836 2003-12-04
WO 02/101003 PCT/FR02/01983
5/5
tgaaaactgt ttcttattct ccacaaaatt ttgaacgttt taatttacca actattgttg 300
gtaccgtttc tcgtttagat gatgattctg gttctatggt tgcattagca gatgcagcaa 360
ctattactgc aatgcgtact ggtgcagttg cagcagttgc aactcgttta ttagcacgtc 420
caggttctac tactttagca ttaattggtg caggtgcaca agcagttact caagcacatg 480
cattatctcg tgttttacca ttagaacgta ttttaatttc tgatattaaa gcagaacatg 540
cagaatcttt tgcaggtcgt gttgcatttt tagaattacc agttgaagtt actgatgcag 600
caactgcaat ggcaactgca gatgttttat gtactgttac ttctgttcca gttggtggtg 660
gtccagttgt tccagcagaa ccacgtcaag cacatttaca tgttaatggt attggtgcag 720
atgaacaagg taaaactgaa ttaccaaaag cattattaga tgatgcattt atttgtgttg 780
atcatccagg tcaagcacgt gcagaaggtg aatttcaaca attaccagat cgtgaattag 840
gtccatcttt agcagattta tgtgcagcac cagaaattgc agcaccacat ccagaacgtt 900
tatctgtttt tgattctact ggttctgcat ttgcagatca tattgcatta gatgttttat 960
taggttttgc agatgaatta ggtttaggtc ataaaatgtc tattgaatct actccagaag 1020
atgttttaga tccatattct ttataataaa gatctaagct t 1061