Note: Descriptions are shown in the official language in which they were submitted.
CA 03128119 2021-07-20
WO 2020/188216 PCT/FR2020/050571
1
Description
Titre : Méthode ultrasensible de détection de la mort cellulaire
Domaine technique
[1] L'invention concerne une méthode de détection de la mort cellulaire par
l'utilisation de techniques de PCR (Polymerase Chain Reaction) : qPCR, i.e.
quantitative PCR ; ddPCR i.e. digital droplet PCR,...) ou toute autre
technique de
détection de faible quantité d'ADN (nanostring, etc...).
Technique antérieure
[2] La mort des cellules est l'arrêt irréversible des fonctions vitales
avec
modification des structures au niveau cellulaire et moléculaire. Ce processus
de
mort cellulaire peut survenir de diverses façons relativement différentes
(Gallouzzi
et al., Cell Death Differ, 2018).
[3] On va principalement distinguer la mort cellulaire par apoptose, ou
mort
cellulaire régulée, qui se produit selon une séquence d'évènements finement
régulés et initiée en réponse à un signal de mort (stress oxydatif, stress
exogènes,
dommages à l'ADN, infection virale,...) ou la mort cellulaire programmée
intervenant dans le maintien de l'homéostasie tissulaire où chaque cellule
possède une durée de vie plus ou moins déterminée.
[4] La mort par nécrose résulte quant à elle de processus cellulaires en
général déclenchés accidentellement comme par exemple le gel, la chaleur,
une lésion mécanique,... Le choix de la réponse par l'apoptose ou la nécrose
peut
également être dépendante de l'amplitude de l'agression.
[5] En terme mécanistique, lors de la mort par apoptose, on observe des
altérations de la membrane, des signalisations enzymatiques (comme
l'activation
de protéines caspases), la condensation du noyau et du cytoplasme, une
agrégation de la chromatine, des dommages mitochondriaux et la fragmentation
de l'ADN nucléaire en fragments inter-nucléosomaux. De façon intéressante, la
plupart des agents anti-tumoraux peuvent induire un phénomène d'apoptose.
CA 03128119 2021-07-20
WO 2020/188216 PCT/FR2020/050571
2
[6] A l'inverse, les critères morphologiques des cellules nécrotiques sont
différents. Une déstructuration de la membrane cellulaire va provoquer une
entrée
massive d'eau dans la cellule, une destruction des organites intracellulaires,
ce qui
conduit à la libération des enzymes lytiques des lysosomes et des peroxysomes
entraînant la digestion de la cellule, une dégradation de l'ADN puis sa mort.
[7] Si les voies menant à la mort cellulaire par apoptose ou par nécrose,
sont
mécanistiquement très différentes, il a été observé dans chacune de ces voies,
une dégradation de l'ADN, précédée par des cassures simple et double brin, la
génération de fragments d'ADN de haut poids moléculaire, dégradés
progressivement en fragments internucléosomaux (apoptose) ou de façon
aléatoire (nécrose).
[8] Les méthodes de détection de l'apoptose peuvent être catégorisées selon
4 grands principes, selon qu'elles détectent i) l'altération de la membrane
plasmique, ii) l'activation des caspases, iii) les dommages mitochondriaux,
iv) la
fragmentation de l'ADN.
[9] Concernant l'activation des caspases, il est possible de mesurer
l'activité
caspase, par la fusion d'un site tétrapeptidique de clivage par les caspases à
un
rapporteur fluorométrique ou luminescent (on citera le kit Caspase-Glo de la
société Promega). Cette méthode a pour avantage d'être rapide, simple
d'utilisation et distribuée par plusieurs fournisseurs. Cependant, cette
méthode est
couteuse, nécessite un spectrofluorimètre ou luminomètre et ne permet de
détecter que l'apoptose Caspase-dépendante.
[10] Pour la détection de l'altération de la membrane plasmique, la
détection
des phosphatidylsérines exposées dans la couche externe de la membrane
plasmique est également proposée par un grand nombre de fournisseurs (on
citera Pacific BlueTM Annexin V de BioLegend ). Le principe est l'utilisation
d'annexine V (protéine se liant spécifiquement aux phosphatidylsérines de
façon
calcium dépendante), couplée à différents rapporteurs (majoritairement
fluorescents) permettant sa détection. L'analyse est réalisée en cytométrie en
flux
(FACS) ou en microscopie. Cette méthode, largement proposée, offre un choix
important de fluorochromes et permet de détecter les cellules apoptotiques et
nécrotiques. Cependant, cette méthode nécessite un cytomètre en flux et la
CA 03128119 2021-07-20
WO 2020/188216 PCT/FR2020/050571
3
quantification de l'apoptose de cellules adhérentes en cytométrie en flux est
très
délicate. L'utilisation de la microscopie peut permettre de pallier cette
problématique. La quantification est cependant plus laborieuse. Cette méthode
est
donc peu adaptée aux cellules adhérentes.
[11] Parmi les
méthodes détectant la fragmentation de l'ADN, on peut tout
d'abord citer une méthode de visualisation de l'ADN fragmenté après
électrophorèse sur gel suivie d'une révélation par un agent intercalant. Le
principe
consiste en l'extraction de l'ADN génomique puis la séparation des fragments
par
électrophorèse sur gel d'agarose (Apoptotic DNA ladder kit). Si cette méthode
a
l'avantage d'avoir un coût faible et de ne pas nécessiter d'appareils
spécifiques et
coûteux, cette méthode est extrêmement peu sensible.
[12] On citera également une méthode de quantification de l'ADN
cytoplasmique couplé à des histones, par ELISA et l'utilisation d'anticorps
contre
l'ADN et des histones (kit Oeil Death Detection ELISA, de la société Roche).
Cette
méthode est par exemple décrite dans le brevet US5637465. Bien qu'assez
simple à mettre en oeuvre, cette méthode est relativement laborieuse. Sa
sensibilité, ainsi que la gamme de détection sont limitées bien que son coût
soit
élevé.
[13] Hooker et aL, 2012 (Nucl Acids Res, 40(15)e113) ; Hooker et aL, 2009
(J
Cell Mol Med, 13(5):948-958), et Staley et aL, 1997 (Cell Death Differ, 4:66-
75)
décrivent une méthode de détection de l'ADN fragmenté en utilisant une
"quantitative ligation-mediated PCR" qui permet d'amplifier les fragments à
bords
francs (blunt ends) formés suite à l'activation des nucléases. Cette méthode
laborieuse nécessite de multiples étapes.
[14] Enfin,
Botezatu et aL, 2000 (Clin Chem 46(8) :1078-1084) ; Umetani et aL,
2006 (Clin Chem 52(6) :1062-1069) ; Lou et al., Int J Mol Med 35 : 72-80) ;
Fawzy
et aL, 2016 (J Egypt Natl Canc lnst, 28 : 235-242) décrivent des méthodes de
détection/quantification de l'ADN génomique circulant (ou libre), et plus
particulièrement la détection d'ADN génomique issu de la mort cellulaire. Ces
méthodes impliquent l'amplification de séquences répétées Alu par PCR
quantitative. Dans cette méthode, les échantillons d'ADN sont des échantillons
de
sérum et de plasma.
CA 03128119 2021-07-20
WO 2020/188216 PCT/FR2020/050571
4
[15] Il reste ainsi nécessaire de mettre au point une méthode de détection
de
la mort cellulaire sensible, simple et rapide, ayant un coût modéré et
fonctionnant
sur des cellules adhérentes ou en suspension et même sur des tissus.
Exposé de l'invention
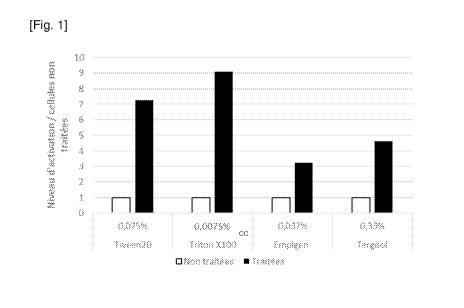
[16] La présente invention concerne une méthode de détection de la mort
cellulaire basée sur la détection de fragments d'ADN génomique d'origine
nucléaire présents dans le cytoplasme cellulaire. En effet, les inventeurs ont
mis
au point une méthode très sensible permettant de détecter des fragments d'ADN
génomique d'origine nucléaire dans le cytoplasme.
[17] Contrairement aux méthodes de détection de fragments d'ADN par PCR
de l'état de la technique selon lesquelles les fragments d'ADN sont détectés
et
quantifiés dans un extrait cellulaire total (noyau + cytoplasme) ou bien dans
des
fluides biologiques comme le plasma, la détection se fait sur un extrait
cytoplasmique. Cet extrait cytoplasmique est obtenu par incubation de
l'échantillon
cellulaire avec un tampon de lyse ou un tampon hypotonique afin de lyser ou
perméabiliser la membrane plasmique sans perméabilisation de la membrane
nucléaire.
[18] La détection à partir d'extraits cytoplasmiques permet d'obtenir une
méthode plus rapide, plus simple à mettre en oeuvre et moins couteuse,
comparativement aux méthodes de l'état de la technique.
[19] Les extraits cytoplasmiques correspondent à tout ou partie du contenu
cytoplasmique cellulaire. Lorsque le processus de mort cellulaire est
déclenché,
l'extrait cytoplasmique est enrichi en ADN génomique d'origine nucléaire, par
rapport à des cellules n'ayant pas enclenché le processus de mort.
[20] La détection des fragments d'ADN génomique d'origine nucléaire
présents
dans le cytoplasme est mesurée par amplification / détection d'une zone
présente
en au moins une copie dans le génome. Ainsi, ces fragments d'ADN génomique
peuvent être avantageusement détectés et quantifiés dans un échantillon. La
quantité de fragments d'ADN récupérés est proportionnelle à la quantité de
cellules ayant enclenché le processus de mort dans l'échantillon, permettant
ainsi
de détecter et quantifier ce processus.
CA 03128119 2021-07-20
WO 2020/188216 PCT/FR2020/050571
[21] La détection de fragments d'ADN génomique dans le cytoplasme, en
utilisant des amorces spécifiques ciblant des séquences répétées présentes
dans
le génome, confère une sensibilité accrue à la méthode selon l'invention. Les
résultats comparatifs obtenus confirment que la sensibilité est supérieure à
celle
5 obtenue avec des méthodes classiques existantes et déjà commercialisées.
Brève description des dessins
[22] D'autres caractéristiques, détails et avantages de l'invention
apparaîtront
à la lecture de la description détaillée ci-après, et à l'analyse des dessins
annexés,
sur lesquels :
Fig. 1
[23] [Fig. 1] représente le niveau d'activation de la mort cellulaire
réalisé sur
des extraits cytoplasmiques de cellules HepG2 obtenus en utilisant différents
détergents dans la méthode selon l'invention.
Fig. 2
[24] [Fig. 2] représente le niveau d'activation de la mort cellulaire
réalisé sur
des extraits cytoplasmiques de cellules OCI-AML3 obtenus en utilisant
différents
détergents dans la méthode selon l'invention.
Fig. 3
[25] [Fig. 3] représente le niveau d'activation de la mort cellulaire
réalisé sur
des extraits cytoplasmiques de cellules OCI-AML3 obtenus en utilisant
différents
détergents dans la méthode selon l'invention.
Fig. 4
[26] [Fig. 4] représente le niveau d'activation de la mort cellulaire
réalisé sur
des extraits cytoplasmiques de cellules MDA-MB-231 obtenus en utilisant
différents détergents dans la méthode selon l'invention.
Fig. 5
[27] [Fig. 5] représente la comparaison de la sensibilité de la méthode de
détection de mort cellulaire par qPCR selon la présente invention par rapport
à
une méthode de l'état de la technique Caspase Glo 3/7.
CA 03128119 2021-07-20
WO 2020/188216 PCT/FR2020/050571
6
Fig. 6
[28] [Fig. 6] représente la comparaison de la sensibilité de la méthode de
détection de mort cellulaire par qPCR selon la présente invention par rapport
à
une méthode de l'état de la technique la cytométrie en flux (marquage Annexine
V,
Iodure de propidium).
Fig. 7
[29] [Fig. 7] représente la comparaison de la sensibilité de la méthode de
détection de mort cellulaire par qPCR (lyse directe) selon la présente
invention par
rapport à la cytométrie en flux (marquage Annexine V, Iodure de propidium).
Fig. 8
[30] [Fig. 8] représente la comparaison de la sensibilité de la méthode de
détection de mort cellulaire par qPCR selon la présente invention par rapport
à la
cytométrie en flux (marquage Annexine V, Iodure de propidium).
Fig. 9
[31] [Fig. 9] représente la comparaison de la sensibilité de la méthode de
détection de mort cellulaire par qPCR (lyse directe) selon la présente
invention par
rapport à la cytométrie en flux (marquage Annexine V, Iodure de propidium).
Fig. 10
[32] [Fig. 10] représente la comparaison de la sensibilité ainsi que du
seuil de
saturation de la méthode de détection de mort cellulaire par qPCR selon la
présente invention par rapport à la cytométrie en flux (marquage Annexine V,
Iodure de propidium.
Fig.11
[33] [Fig. 11] représente la comparaison de la sensibilité de la méthode de
détection de mort cellulaire par qPCR selon la présente invention par rapport
à la
cytométrie en flux (marquage Annexine V, Iodure de propidium).
Fig. 12
CA 03128119 2021-07-20
WO 2020/188216 PCT/FR2020/050571
7
[34] [Fig. 12] représente la comparaison de la sensibilité de la méthode de
détection de mort cellulaire par ddPCR selon la présente invention sur
cellules
isolées.
Fig. 13
[35] [Fig. 13] représente le niveau d'activation de la mort cellulaire
réalisé sur
des extraits cytoplasmiques de différentes lignées cellulaires à l'aide d'un
couple
d'amorces ciblant une séquence présente en une copie par génome.
Fig. 14
[36] [Fig. 14] représente le niveau d'activation de la mort cellulaire
réalisé sur
des extraits cytoplasmiques de différentes lignées cellulaires à l'aide d'un
couple
d'amorces ciblant une séquence présente en deux copies par génome.
Fig. 15
[37] [Fig. 15] représente le niveau d'activation de la mort cellulaire
réalisé sur
des extraits cytoplasmiques provenant de la lignée cellulaire OCI-AML3 à
l'aide de
deux couple d'amorces chacun ciblant une séquence présente en une copie par
génome
Fig. 16
[38] [Fig. 16] démontre une augmentation de la quantité des fragments d'ADN
dans la fraction cytoplasmique obtenus après extraction avec un tampon de lyse
décrit dans la méthode selon l'invention, et détectés par électrophorèse
capillaire,
et ceci exclusivement dans des cellules traitées par une drogue induisant leur
mort
(staurosporine) (MDA 50 et MDA 100) comparativement à des cellules non
traitées par la staurosporine (MDA NT).
Description détaillée
[39] Les dessins et la description ci-après contiennent, pour l'essentiel,
des
éléments de caractère certain. Ils pourront donc non seulement servir à mieux
faire comprendre la présente invention, mais aussi contribuer à sa définition,
le
cas échéant.
CA 03128119 2021-07-20
WO 2020/188216 PCT/FR2020/050571
8
[40] La présente invention concerne donc une méthode de quantification de
la
mort cellulaire dans un échantillon cellulaire caractérisée en ce qu'au moins
une
séquence présente sur des fragments d'ADN génomique d'origine nucléaire est
amplifiée à partir de l'extrait cytoplasmique dudit échantillon.
[41] Les inventeurs ont en effet avantageusement exploité la présence
anormale des fragments d'ADN génomique d'origine nucléaire localisés dans le
cytoplasme des cellules lors du processus de mort cellulaire. L'ADN génomique
fragmenté d'origine nucléaire peut ainsi être détecté à partir d'extraits
cytoplasmiques des cellules.
[42] On entend par mort cellulaire au sens de la présente invention, la
mort
cellulaire par apoptose et/ou la mort cellulaire par nécrose.
[43] On entend par fragments d'ADN génomique ou ADN génomique
fragmenté , les fragments d'ADN d'origine nucléaire générés lors des
processus
de mort cellulaire.
[44] L'échantillon cellulaire peut être un échantillon de cellules en
culture in
vitro, telles que des cellules adhérentes, des cellules en suspension, un
échantillon comprenant des cellules tumorales circulantes, un échantillon
comprenant des cellules tumorales circulantes purifiées, un échantillon
sanguin
contenant des cellules circulantes ou tout autre échantillon tel qu'un
échantillon de
plasma, un échantillon d'urine, un échantillon de salive.
[45] Selon un mode de réalisation, la méthode de quantification de la mort
cellulaire dans un échantillon cellulaire comprend :
- l'obtention d'un extrait cytoplasmique à partir d'un échantillon
cellulaire ;
- l'amplification d'au moins une séquence dans ledit extrait cytoplasmique
;
- la quantification de l'ADN génomique détecté dans ledit extrait
cytoplasmique.
[46] On entend par extrait cytoplasmique , la partie soluble du
cytoplasme
cellulaire, encore appelée cytosol, qui est récupéré après la perméabilisation
spécifique de la membrane plasmique sans altération de la membrane nucléaire,
suivie d'une centrifugation. L'extrait cytoplasmique et ses méthodes
d'obtention
sont connus de l'homme du métier et sont décrits dans l'état de la technique
par
exemple dans Suzuki, Keiko et al. "REAP: A two minute cell fractionation
method."
CA 03128119 2021-07-20
WO 2020/188216 PCT/FR2020/050571
9
BMC research notes vol. 3 294. 10 Nov. 2010, doi:10.1186/1756-0500-3-294 ou
dans Gary Zieve and Sheldon Penman, Small RNA species of the HeLa cell:
Metabolism and subcellular localization Ce!!, May 1976, Pages 19-31.
[47] Typiquement, l'extrait cytoplasmique est obtenu par incubation de
l'échantillon cellulaire avec un tampon de lyse ou un tampon hypotonique.
[48] L'incubation de l'échantillon avec le tampon de lyse permet
avantageusement de lyser ou perméabiliser la membrane plasmique sans
perméabilisation de la membrane nucléaire.
[49] Ainsi, on entend par tampon de lyse au sens de la présente
invention,
tout tampon permettant de lyser ou perméabiliser la membrane plasmique sans
perméabilisation de la membrane nucléaire.
[50] L'homme du métier pourra choisir le tampon de lyse adéquat.
Typiquement, un détergent non-ionique pourra être utilisé, tel que décrit dans
le
brevet US5637465.
[51] L'homme du métier sait également comment choisir la concentration
adéquate du tampon pour permettre la lyse ou la perméabilisation de la
membrane plasmique sans perméabilisation de la membrane nucléaire.
[52] Typiquement, un anticorps anti-ADN pourra être utilisé par l'homme du
métier pour déterminer la concentration adéquate du tampon pour permettre la
lyse ou la perméabilisation de la membrane plasmique sans perméabilisation de
la
membrane nucléaire.
[53] Après traitement des cellules n'ayant pas enclenché le processus de
mort
avec des concentrations croissantes de détergents, celles-ci sont mises en
présence d'un anticorps anti-ADN. L'exclusion nucléaire de cet anticorps
confirme
l'intégrité de la membrane nucléaire et permet ainsi de déterminer la
concentration
optimale de détergent. Une trop forte concentration en détergent
perméabilisera la
membrane nucléaire permettant à l'anticorps de pénétrer dans le noyau et ainsi
de
détecter l'ADN nucléaire. Cette méthode est calquée sur une méthode de
détection d'une protéine à localisation nucléaire (Postfixation detergent
treatment
for immunofluorescence suppresses localization of some integral membrane
CA 03128119 2021-07-20
WO 2020/188216 PCT/FR2020/050571
proteins. Goldenthal KL, Hedman K, Chen JW, August JT, Willingham MC. J
Histochem Cytochem. 1985 Aug ; 33(8):813-20).
[54] A titre illustratif, le tampon de lyse pourra être choisi parmi
l'acide 4-(2-
hydroxyéthyl)-1-pipérazine éthane sulfonique),
l'acide
5 éthylènediaminetétraacétique, le chlorure de sodium, les saponines telles la
digitonine ou la saponine, le Tween-20, NP40, le tergitol, le Triton X-100,
l'Igepal
CA630, l'Empigen, ou une combinaison.
[55] Selon un mode de réalisation, le tampon de lyse est un mélange d'acide
4-(2-hydroxyéthyl)-1-pi pérazi ne éthane sulfonique,
d'acide
10 éthylènediaminetétraacétique, de chlorure de sodium, de digitonine.
[56] Ces tampons permettent de lyser ou perméabiliser la membrane
plasmique sans perméabilisation de la membrane nucléaire.
[57] Dans un mode de réalisation, le tampon de lyse est un mélange d'acide
4-
(2-hydroxyéthyl)-1-pipérazine éthane sulfonique à 50mM,
d'acide
éthylènediaminetétraacétique à 5mM, de chlorure de sodium à 150mM, et de
digitonine à 50 pg/m1..
[58] Selon un mode de réalisation, l'extrait cytoplasmique pourra être
obtenu
par les étapes suivantes :
- incubation de l'échantillon cellulaire avec un tampon de lyse ou un
tampon
hypotonique.
- centrifugation ou filtration de l'échantillon cellulaire lysé.
[59] L'étape de centrifugation ou de filtration permet de séparer le
surnageant
du reste des débris cellulaires.
[60] La fraction de l'extrait cytoplasmique correspond à une fraction
aliquote de l'extrait cytoplasmique.
[61] Selon un mode de réalisation, la fraction de l'extrait cytoplasmique
pourra
être obtenue par les étapes suivantes :
- incubation de l'échantillon cellulaire avec un tampon de lyse ou un
tampon
hypotonique ;
- centrifugation ou filtration de l'échantillon cellulaire lysé ;
CA 03128119 2021-07-20
WO 2020/188216 PCT/FR2020/050571
11
- prélèvement d'une fraction aliquote de l'extrait cytoplasmique dans le
surnageant
obtenu après l'étape de centrifugation ou filtration.
[62] Selon un mode de réalisation, la méthode d'obtention de la fraction de
l'extrait cytoplasmique pourra comprendre en outre une étape de dilution de
ladite
fraction aliquote prélevée, au moins 5 fois dans l'eau, préférentiellement 10
fois
dans l'eau.
[63] Typiquement, l'étape de dilution pourra être appliquée lorsque la
méthode
de quantification est une méthode de quantification par PCR.
[64] De manière avantageuse, les inventeurs ont démontré qu'il était
possible
de détecter la mort cellulaire sur des extraits cytoplasmiques à l'aide
d'amorces
ciblant une séquence présente en une copie sur le génome nucléaire.
[65] Ainsi, et selon un mode de réalisation, la méthode de quantification
de la
mort cellulaire selon la présente invention est caractérisée en ce que ladite
au
moins une séquence est une séquence d'ADN présente en une copie sur lesdits
fragments d'ADN génomique.
[66] La méthode de quantification de la mort cellulaire selon la présente
invention permet également de détecter la mort cellulaire sur des extraits
cytoplasmiques à l'aide d'amorces ciblant une séquence présente en plus d'une
copie dans le génome nucléaire.
[67] A titre illustratif, la méthode selon la présente invention permet de
détecter
la mort cellulaire sur des extraits cytoplasmiques à l'aide d'amorces ciblant
une
séquence présente en deux copies dans le génome nucléaire.
[68] Ainsi, selon une mode de réalisation, la méthode de quantification de
la
mort cellulaire selon la présente invention est caractérisée en ce que ladite
au
moins une séquence est une séquence d'ADN présente en au moins deux copies
sur lesdits fragments d'ADN génomique.
[69] La sensibilité de la méthode selon l'invention est accrue
proportionnellement au nombre de séquences d'ADN répétées lorsque celles-ci
sont utilisées comme marqueurs pour détecter et quantifier la quantité d'ADN
génomique cytoplasmique.
CA 03128119 2021-07-20
WO 2020/188216 PCT/FR2020/050571
12
[70] Selon un mode de réalisation, la méthode de quantification de la mort
cellulaire selon la présente invention est caractérisée en ce que ladite au
moins
une séquence est une séquence d'ADN répétée.
[71] Typiquement, l'ADN génomique inclut des séquences répétées qui
pourront être des petits éléments nucléaires intercalés (SINE pour Short
lnterspersed Nuclear Elements) ou des longs éléments nucléaires intercalés
(LINE
pour Long lnterspersed Nuclear Elements).
[72] Parmi les SINE, les séquences Alu, MIR, MI R3 pourront être choisies.
[73] Parmi les LIN E, les séquences LINE1 pourront être choisies.
[74] Ainsi, et selon un mode de réalisation, la méthode de quantification
de la
mort cellulaire selon la présente invention est caractérisée en ce que ladite
au
moins une séquence d'ADN répétée est choisie parmi les SINE tels que Alu, MIR,
MIR3 et les LINE tels que LINE1.
[75] Les inventeurs ont également démontré qu'il est possible de détecter
la
mort cellulaire sur des extraits cytoplasmiques à l'aide de plusieurs couples
d'amorces, chaque couple d'amorces ciblant une séquence d'ADN présente dans
le génome nucléaire.
[76] Ainsi, selon un mode de réalisation, la méthode de quantification de
la
mort cellulaire selon la présente invention est caractérisée en ce que
plusieurs
séquences différentes sont amplifiées simultanément.
[77] L'homme du métier pourra utiliser tout type de technique permettant de
détecter et quantifier les séquences de l'ADN génomique.
[78] La détection/quantification des fragments d'ADN génomique pourra être
réalisée par des techniques de PCR quantitative ou toute autre technique de
détection de faible quantité d'ADN connues de l'Homme du métier.
[79] Selon un mode de réalisation, la quantification et la détection d'ADN
génomique fragmenté est réalisée par PCR quantitative (qPCR). Le principe de
la
qPCR repose sur la possibilité de déterminer la quantité de matrice d'ADN
présente dans un échantillon en temps réel à l'aide d'un agent intercalant ou
d'une
CA 03128119 2021-07-20
WO 2020/188216 PCT/FR2020/050571
13
sonde (Taqman,0). La fluorescence émise est directement propositionnelle à la
quantité d'amplicons générés pendant la réaction PCR.
[80] L'homme du métier pourra utiliser tout appareil pour mettre en oeuvre
la
technique de qPCR. A titre illustratif, le lecteur de PCR en temps réel Light
Cycler
480 system de la société Roche pourra être utilisé.
[81] Ainsi, la méthode de quantification de la mort cellulaire selon la
présente
invention est caractérisée en ce que la détection/quantification est réalisée
par
qPCR.
[82] Selon un mode de réalisation, la quantification des fragments d'ADN
génomique est réalisée par digital droplet PCR (ddPCR). La ddPCR est une PCR
par micro-fluidique basée sur le partitionnement de chaque échantillon dans
20000 gouttelettes de 1 nL.
[83] Selon un mode de réalisation, la quantification des fragments d'ADN
génomique est réalisée par la technologie Nanostring. Le principe du
Nanostring
repose sur deux étapes clés. En amont, deux sondes sont conçues
spécifiquement pour chaque cible d'intérêt. L'une des sondes, appelée sonde de
capture, est couplée à une biotine, qui sera utilisée pour immobiliser les
molécules
d'intérêt sur un support dédié au comptage. La deuxième sonde, appelée
rapporteur , est spécifique de la molécule d'intérêt. Elle contient une
succession
de 6 fluorochromes de 4 couleurs différentes, dont l'arrangement définit un
code-
barres qui sera propre à chaque molécule d'intérêt. C'est ce code-barres qui
va
permettre l'ultra-sensibilité de cette technique et donc la possibilité
d'analyser du
matériel biologique en faible quantité (Plateforme Nanostring LABEX DEEP).
[84] Selon un mode de réalisation, la quantification des fragments d'ADN
génomique est réalisée par PCR multiplexe ou multiplexage. Pour la mise en
oeuvre d'une PCR multiplexe, un set de plusieurs couples d'amorces sera
utilisé
de manière à amplifier simultanément plusieurs séquences présentes dans le
génome.
[85] Selon un mode de réalisation, une séquence unique est détectée et
quantifiée en utilisant par exemple l'amorce sens 5'
CGCCTGGATCATGTCAAGTCA 3' (SEQ ID NO: 1) et l'amorce anti-sens 5'
CA 03128119 2021-07-20
WO 2020/188216 PCT/FR2020/050571
14
AGGCTAAGTTAGGGCCTCTGC 3' (SEQ ID NO: 2) ou l'amorce sens 5'
AACATAAGCTGAGGCCAGCCT 3' (SEQ ID NO: 3) et l'amorce anti-sens 5'
GTGTCTACTGCCAACCTGTGC 3' (SEQ ID NO : 4).
[86] Selon un mode de réalisation, une séquence présente en deux copies est
détectée et quantifiée en utilisant l'amorce sens 5'
TCTCCACAACACTTAGTGGACAGT 3' (SEQ ID NO: 5) et l'amorce anti-sens 5'
AGAGGAGGTGGTAGCTGGAGA 3' (SEQ ID NO : 6).
[87] Selon un mode de réalisation, un multiplexage peut-être réalisé en
utilisant simultanément par exemple le couple d'amorces SEQ ID NO : 1 et SEQ
ID NO : 2 avec le couple d'amorces SEQ ID NO :3 et SEQ ID NO :4..
[88] Selon un mode de réalisation, la séquence Alu est détectée et
quantifiée
en utilisant l'amorce sens 5' AGGTGAAACCCCGTCTCTACT 3' (SEQ ID NO: 7)
et l'amorce anti-sens 5' CCATTCTCCTGCCTCAGCCT 3' (SEQ ID NO : 8).
[89] Selon un mode de réalisation, la séquence LINE1 est détectée et
quantifiée en utilisant l'amorce sens 5' GTCAGTGTGGCGATTCCTCAG 3' (SEQ
ID NO: 9) et l'amorce anti-sens 5' AGTAATGGGATGGCTGGGTCAA 3' (SEQ ID
NO: 10) ou en utilisant l'amorce sens 5' AACAACAGGTGCTGGAGAGGA 3'
(SEQ ID NO: 11) et l'amorce anti-sens 5' ATCGCCACACTGACTTCCACA 3'
(SEQ ID NO :12).
[90] Selon un mode de réalisation, la quantité d'acide nucléique amplifié
dans
ledit échantillon d'acide nucléique sera comparée à la quantité d'acide
nucléique
amplifié d'un échantillon contrôle.
[91] On entend par échantillon contrôle , un échantillon cellulaire
dans lequel
le processus de mort cellulaire n'est pas enclenché.
[92] Typiquement, une augmentation de la quantité d'acide nucléique
amplifié
dans ledit échantillon par comparaison à la quantité d'acide nucléique
amplifié
de l'échantillon contrôle est indicatif de la mort cellulaire.
[93] La quantification de la mort cellulaire par la détection de
fragments d'ADN
dans le cytoplasme de cellules selon la présente invention permet de
diagnostiquer une pathologie, de suivre les effets d'un traitement sur la mort
CA 03128119 2021-07-20
WO 2020/188216 PCT/FR2020/050571
cellulaire, d'obtenir un pronostic de la pathologie, de réaliser un criblage
de
composés, d'optimiser des conditions de culture cellulaire.
[94] Ainsi, la présente invention concerne également une méthode de suivi
de
l'efficacité et/ou de l'effet d'un traitement sur la mort cellulaire
comprenant la
5 détection de la mort cellulaire dans un échantillon cellulaire par la
méthode selon
la présente invention.
[95] La méthode s'applique à des conditions in vitro, in vivo et ex vivo.
[96] Typiquement, la présente invention peut permettre le suivi de la
réponse
d'un patient au traitement. La détection et la quantification de la mort
cellulaire
10 sont indicatives de l'efficacité ou non du traitement. La quantité d'acide
nucléique
amplifié de l'échantillon provenant du patient peut être comparée à la
quantité
d'acide nucléique amplifié d'un échantillon contrôle, ledit échantillon
contrôle
pouvant être un échantillon du patient obtenu avant administration du
traitement
ou un échantillon provenant d'un sujet non atteint de la pathologie.
Généralement,
15 une augmentation de la quantité d'acide nucléique amplifié est synonyme
d'efficacité de traitement, tandis que l'absence de variation significative
peut être
synonyme d'échec du traitement.
[97] La présente invention concerne également une méthode de diagnostic
d'une pathologie impliquant un processus de mort cellulaire comprenant la
détection de la mort cellulaire dans un échantillon cellulaire par la méthode
selon
la présente invention.
[98] Typiquement, le niveau d'activation de la mort cellulaire dans un
échantillon cellulaire serait indicatif d'une pathologie impliquant un
processus de
mort cellulaire. La quantité d'acide nucléique amplifié de l'échantillon
provenant du
patient peut être comparée à la quantité d'acide nucléique amplifié d'un
échantillon contrôle, ledit échantillon contrôle pouvant être un échantillon
provenant d'un sujet non atteint de la pathologie.
[99] La présente invention concerne également une méthode de pronostic
d'une pathologie impliquant un processus de mort cellulaire comprenant la
détection de la mort cellulaire dans un échantillon cellulaire par la méthode
selon
la présente invention.
CA 03128119 2021-07-20
WO 2020/188216 PCT/FR2020/050571
16
[100] La présente invention concerne également une méthode de criblage de
composés comprenant :
- le traitement d'un échantillon cellulaire avec un ou plusieurs composés ;
- la détection de la mort cellulaire dans ledit échantillon par la méthode
selon la
présente invention.
[101] La méthode selon l'invention permettra de déterminer le ou les composés
permettant de déclencher la mort cellulaire.
[102] La présente invention a également pour objet un kit pour la détection de
la
mort cellulaire dans un échantillon cellulaire comprenant :
- un tampon de lyse ou un tampon hypotonique apte à spécifiquement lyser ou
perméabiliser la membrane plasmique ;
- au moins une paire d'amorces amplifiant de l'ADN génomique.
[103] Le tampon de lyse permet avantageusement de lyser ou perméabiliser la
membrane plasmique sans perméabilisation de la membrane nucléaire.
[104] Le tampon de lyse et les amorces sont tels que ci-avant décrits.
[105] L'échantillon cellulaire pourra être choisi parmi un échantillon de
cellules
en culture in vitro, telles que des cellules adhérentes, des cellules en
suspension,
un échantillon comprenant des cellules tumorales circulantes, un échantillon
comprenant des cellules tumorales circulantes purifiées, un échantillon
sanguin
contenant des cellules circulantes ou tout échantillon comprenant des cellules
ayant enclenché un processus de mort cellulaire tel qu'un échantillon de
plasma,
un échantillon d'urine, un échantillon de salive.
[106] Avantageusement également, les amorces seront spécifiques de l'espèce
étudiée.
Exemples
[107] Dans les exemples qui suivent, les matériels et méthodes ci-après
détaillés ont été utilisés :
[108] Méthode
Afin de mesurer la mort cellulaire dans un échantillon donné, un protocole a
été
développé.
CA 03128119 2021-07-20
WO 2020/188216 PCT/FR2020/050571
17
[109] A partir de cellules traitées ou non par différentes drogues déclenchant
leur mort, les cellules sont lysées à l'aide d'un tampon afin de libérer dans
le
milieu les petits fragments d'ADN résultant de sa dégradation.
[110] Une étape de centrifugation ou de filtration permet de séparer le
surnageant contenant les fragments d'ADN issus de sa dégradation du reste des
débris cellulaires. Une fraction aliquote du surnageant est prélevée, i.e., la
fraction
d'extrait cytoplasmique, qui sera éventuellement diluée en fonction de la
méthode
de quantification de la mort cellulaire utilisée. Ensuite, une PCR est
réalisée sur
les échantillons à l'aide d'amorces amplifiant spécifiquement des séquences
répétées dispersées tout au long du génome, ou à l'aide d'amorces ciblant une
séquence en une copie ou deux copies sur le génome.
[111] Alternativement, la méthode de quantification de la mort cellulaire
pourra
être réalisée sur un extrait cytoplasmique issu de cellules lysées directement
dans
le milieu de culture.
[112] A l'issue de la méthode de quantification de la mort cellulaire, les
résultats
sont analysés de manière relative à une condition contrôle prédéterminée. Le
résultat final peut-être obtenu en deux à trois heures.
[113] Amorces
[114] Amorces permettant la détection et la quantification d'une séquence
unique:
5' CGCCTGGATCATGTCAAGTCA 3' (SEQ ID NO :1) et
5' AGGCTAAGTTAGGGCCTCTGC 3' (SEQ ID NO :2)
ou
5' AACATAAGCTGAGGCCAGCCT 3' (SEQ ID NO :3) et
5' GTGTCTACTGCCAACCTGTGC 3' (SEQ ID NO : 4).
[115] Amorces permettant la détection et la quantification d'une séquence
présente en deux copies par génome :
5' TCTCCACAACACTTAGTGGACAGT 3' (SEQ ID NO :5) et
5' AGAGGAGGTGGTAGCTGGAGA 3' (SEQ ID NO : 6).
[116] Amorces permettant la détection et la quantification de la séquence
répétée Alu :
CA 03128119 2021-07-20
WO 2020/188216 PCT/FR2020/050571
18
5' AGGTGAAACCCCGTCTCTACT 3' (SEQ ID NO :7)
5' CCATTCTCCTGCCTCAGCCT 3' (SEQ ID NO : 8).
[117] Amorces permettant la détection et la quantification de la séquence
répétée LINE1 :
5' GTCAGTGTGGCGATTCCTCAG 3' (SEQ ID NO :9) et
5' AGTAATGGGATGGCTGGGTCAA 3' (SEQ ID NO :10)
ou
5' AACAACAGGTGCTGGAGAGGA 3' (SEQ ID NO :11) et
5' ATCGCCACACTGACTTCCACA 3' (SEQ ID No :12).
[118] Culture cellulaire
[119] Les cellules OCI-AML3 (en suspension) sont issues de leucémie aiguë
myéloïde. Elles sont cultivées dans du milieu RPMI-1640 (Sigma-Aldrich)
supplémenté en Pénicilline-Streptomycine (Sigma-Aldrich) et 10% Sérum de Veau
Foetal (SVF - Gibco) à 37 C avec 5 % de CO2.
[120] Les cellules HepG2 (adhérentes) sont issues de carcinome de foie
humain. Elles sont cultivées dans du milieu Dulbecco's Modified Eagle Medium ¨
High Glucose (DMEM ¨ Sigma-Aldrich) supplémenté en Pénicilline-Streptomycine
(Sigma-Aldrich) et 10% Sérum de Veau Foetal (SVF - Gibco) à 37 C avec 5 % de
CO2.
[121] Les cellules MDA-MB-231 (adhérentes) sont des cellules épithéliales de
tumeur mammaire. Elles sont cultivées dans du milieu Dulbecco's Modified Eagle
Medium ¨ High Glucose (DMEM ¨ Sigma-Aldrich) supplémenté en Pénicilline-
Streptomycine (Sigma-Aldrich) et 10% Sérum de Veau Foetal (SVF - Gibco) à
37 C avec 5 % de CO2.
[122] Les cellules MOLM14 (en suspension) sont issues de leucémie aiguë
myéloïde. Elles sont cultivées dans du milieu RPMI-1640 (Sigma-Aldrich)
supplémenté en Pénicilline-Streptomycine (Sigma-Aldrich) et 10% Sérum de Veau
Foetal (SVF - Gibco) à 37 C avec 5 % de CO2.
[123] Les cellules HeLa ont été cultivées dans du milieu Dulbecco's Modified
Eagle Medium ¨ Low Glucose (DMEM ¨ Sigma-Aldrich) supplémenté en
CA 03128119 2021-07-20
WO 2020/188216
PCT/FR2020/050571
19
Pénicilline-Streptomycine (Sigma-Aldrich) et 10% Sérum de Veau Foetal (SVF -
Gibco) à 37 C avec 5 % de CO2.
[124] Ensemencement et traitement
[125] Les cellules en suspension (ex : OCI-AML3) sont ensemencées à 200 000
cellules/mL dans une plaque 6 puits. Les cellules sont ensuite traitées ou non
avec différentes drogues, à différentes concentrations et incubées pendant 16
heures.
[126] Les cellules adhérentes sont ensemencées entre 20 000 et 30 000
cellules/puits (ex: MDA-MB-231, HepG2) en plaque 48 puits dans 250 pt de
milieu. Les cellules sont ensuite traitées ou non avec différentes drogues, à
différentes concentrations et incubées pendant 24 heures.
[127] qPCR
[128] Pour la mise en oeuvre de la qPCR, la fraction cytoplasmique obtenue
sera diluée 10 fois dans l'eau.
Le kit utilisé est le SYBR qPCR Premix Ex Taq II (Takara). Les échantillons
ont
été préalablement dilués au 1/10e dans de l'eau. Une fois le Master Mix
préparé
(tel que décrit tableau ci-dessous), 6 L sont distribués dans chaque puits de
la
plaque PCR. Ensuite 4pL d'échantillon sont ajoutés. La PCR est réalisée dans
le
lecteur de PCR en temps réel Light Cycler 480 system (Roche). Le programme
d'amplification est le suivant : 95 C pendant 30 secondes, suivi de 40 cycles
décomposés en deux étapes de 95 C pendant 5 secondes et 60 C pendant 20
secondes.
[129] [Tableau 1]
Préparation du Master Mix (MM)
Volume
Réactifs Concentration Concentration pour
Sauce-mère Finale 1
Réaction
( L)
Ex taq II 2x mix 2 x 1 x 5,00
Primer F-R 10 300 0,30
ROX 50X 0,12
H20 0,58
Volume total (pl) 6,00
CA 03128119 2021-07-20
WO 2020/188216 PCT/FR2020/050571
[130] Caspase Glo
[131] Des cellules non traitées (NT) ou ayant subi un traitement (TTT) afin
d'induire la mort cellulaire ont été utilisées pour ces essais. Dans chacune
des
5 conditions NT et TTT, 100 000 ¨ 10 000 ¨ 1 000 ¨ 100 ou 10 cellules sont
transférées dans une plaque 96 puits dans 50 .1_ de milieu (en triplicats).
Ensuite
50 pL de réactif Caspase Glo 3/7 sont ajoutés. Après 1 heure d'incubation,
l'émission lumineuse issue du clivage du substrat par les caspases 3 et 7 est
déterminée à l'aide d'un luminomètre.
10 [132] Marquage Annexin V / Iodure de Propidium (IF) - Biolegend
[133] Des cellules non traitées (NT) et ayant subi un traitement (TTT) afin
d'induire la mort cellulaire ont été utilisées pour ces essais.
[134] Les cellules sont lavées avec du PBS puis resuspendues dans du Binding
Buffer 1X à la concentration de 1.106ce11u1e5/mL. 10 .1_ Annexin V Pacific
Blue et
15 10 pL d'Iodure de propidium (Kit Biolegend) sont rajoutés à 200pL de cette
suspension cellulaire qui est ensuite incubée pendant 15 min à l'obscurité et
à
température ambiante. Après une centrifugation à 300 g pendant 5 min, le
surnageant est aspiré délicatement et 500pt de Binding Buffer 1X sont ajoutés.
[135] Ces cellules sont ensuite utilisées en cytométrie en flux, pour
déterminer
20 l'effet du traitement et le nombre de cellules en apoptose. Ces cellules
sont triées
en fonction de leurs statuts en Annexine V et Iodure de propidium afin de
réaliser
ensuite une PCR.
[136] ddPCR (pour digital droplet PCR)
[137] Chaque réaction de ddPCR est effectuée de façon optimale dans environ
20 000 gouttelettes de lnL de volume.
[138] Echantillons et préparation du mix ddPCR.
Des extraits cytoplasmiques issus de cellules non-traitées et traitées afin
d'induire
la mort cellulaire ont été utilisés comme échantillons.
CA 03128119 2021-07-20
WO 2020/188216 PCT/FR2020/050571
21
[139] Un mix réactionnel de ddPCR (24pL), nécessite 11pL de 2X EvaGreen
ddPCR Supermix (Bio-Rad), 0,22pL d'amorces (sens et anti-sens chacun à
200nM final), 4pL d'échantillon et 6,78pL d'eau.
[140] Génération des gouttelettes
Les gouttelettes sont générées par le 0X200 DropletGenerator (Bio-Rad) en
émulsionnant 20pL de mix ddPCR et 20pL d'huile dans les puits de cartouches
DG8 (Bio-Rad). Puis le mélange gouttelettes/huile est transféré dans une
plaque
96 puits qui est scellée par PX1 PCR Plate Sealer (Bio-Rad).
[141] Amplification
L'amplification est réalisée dans un thermocycleur T100 (Bio-Rad) en suivant
le
programme : Activation de l'enzyme : 95 C pendant 5 min ; 40 cycles :
dénaturation à 95 C pendant 30 s puis extension à 60 C pendant 1 min.
Stabilisation du signal : 4 C pendant 5 min puis 90 C pendant 5 min.
[142] Lecture des gouttelettes
La plaque est ensuite lue par le 0X200 Droplet Reader (Bio-Rad). Les résultats
sont ensuite exportés et les données analysées avec le logiciel QuantaSoft
(Bio-
Rad).
[143] Exemple 1 : Détection de la mort cellulaire sur des cellules
adhérentes en culture (HepG2 et MDA-MB-231) ou des cellules en
suspension (OCI-AML3)
[144] Cellules adhérentes en culture HepG2
[145] La Figure 1 représente le niveau d'activation de la mort cellulaire
réalisé
sur des extraits cytoplasmiques de cellules HepG2 obtenus en utilisant des
tampons de lyse différents dans la méthode selon la présente invention.
[146] 30000 cellules sont ensemencées, puis traitées ou non durant 18h avec
lp.M de doxorubicine. Les cellules sont ensuite lysées dans un tampon
contenant
différents détergents : Tween-20 0,075%, Triton X-100 0,0075%, Empigen 0,037%
et Tergitol 0,33%. Les extraits cytoplasmiques sont centrifugés, puis une
fraction
du surnageant est prélevée et diluée (10x) dans de l'eau. La qPCR est ensuite
réalisée sur ladite fraction.
CA 03128119 2021-07-20
WO 2020/188216 PCT/FR2020/050571
22
[147] Le couple d'amorces de SEQ ID NO :9 et SEQ ID NO : 10 a été utilisé sur
les fractions d'extraits cytoplasmiques obtenues après lyse des cellules avec
le
Tergitol 0,33%. Le couple d'amorces de SEQ ID NO: 7 et SEQ ID NO :8 a été
utilisé sur les fractions d'extraits cytoplasmiques obtenues après lyse des
cellules
avec le Tween-20 0,075%, le Triton X-100 0,0075% et Empigen 0,037%.
[148] Cellules en suspension OCI-AML3
[149] La Figure 2 représente le niveau d'activation de la mort cellulaire
réalisé
sur des extraits cytoplasmiques de cellules OCI-AML3 obtenus en utilisant
différents détergents dans la méthode selon la présente invention.
[150] Les cellules sont traitées ou non durant 18h avec 1 M de doxorubicine.
Après centrifugation de 5 000 cellules celles-ci sont lysées dans un tampon
contenant différents détergents : Tergitol 0,1%, Empigen 0,1%, Digitonine
1504/ml, NP40 0,1%. La qPCR est ensuite réalisée sur des fractions d'extraits
cytoplasmiques en utilisant le couple d'amorces de SEQ ID NO : 9 et SEQ ID NO:
10.
[151] La Figure 3 représente le niveau d'activation de la mort cellulaire
réalisé
sur des extraits cytoplasmiques de cellules OCI-AML3 obtenus en utilisant un
tampon de lyse réalisé avec différents détergents dans la méthode selon la
présente invention.
[152] 5000 cellules sont traitées ou non durant 18h avec 1 M de doxorubicine
puis directement lysées dans un tampon contenant différents détergents :
Tergitol
0,1%, NP40 0,5%, Triton X-100 0,01%, Digitonine 50 g/mL. La qPCR est ensuite
réalisée sur des fractions d'extraits cytoplasmiques en utilisant le couple
d'amorces de SEQ ID NO : 7 et SEQ ID NO : 8. Les résultats de l'expérience
présentée sont des moyennes de trois expériences indépendantes.
[153] Cellules adhérentes en culture MDA-MB-231
[154] La Figure 4 représente le niveau d'activation de la mort cellulaire
réalisé
sur des extraits cytoplasmiques de cellules MDA-MB-231 obtenus en utilisant un
tampon de lyse réalisé avec différents détergents dans la méthode selon la
présente invention.
CA 03128119 2021-07-20
WO 2020/188216 PCT/FR2020/050571
23
[155] 20000 cellules sont traitées ou non durant 18h avec luM de doxorubicine
puis directement lysées dans un tampon contenant différents détergents :
Tergitol
0,1%, lgepal CA630 0,5%, Triton X-100 0,01% et Tween-20 0,5%. La qPCR est
ensuite réalisée sur des fractions d'extraits cytoplasmiques en utilisant le
couple
d'amorces de SEQ ID NO : 7 et SEQ ID NO : 8.
[156] La méthode selon la présente invention permet avantageusement de
détecter la mort cellulaire et ainsi de mesurer son niveau d'activation dans
des
échantillons de cellules adhérentes et de cellules en suspension.
[157] Exemple 2: Détermination de la sensibilité de la méthode de
détection de mort cellulaire par PCR selon la présente invention par rapport
à la méthode Caspase qlo 3/7 de l'état de la technique
[158] Des cellules MOLM14 sont traitées durant 16h avec 1pM d'étoposide. La
mort cellulaire est déterminée sur un échantillon de cellules allant de 10 à
10 000
cellules en utilisant la technique du Caspase Glo. En parallèle, une qPCR est
réalisée sur un extrait cytoplasmique provenant d'un nombre identique de
cellules
(de 10 à 10 000 cellules) avec le couple d'amorces de SEQ ID NO : 7 et SEQ ID
NO : 8. Les résultats de l'expérience présentée dans la Figure 5 sont des
moyennes de trois expériences indépendantes.
[159] La comparaison de la méthode selon la présente invention avec une
méthode de l'état de la technique (Caspase Glo), permet de mettre
avantageusement en évidence la meilleure sensibilité et un seuil de détection
inférieur de la méthode selon la présente invention.
[160] Exemple 3: Détermination de la sensibilité de la méthode de
détection de mort cellulaire selon la présente invention par rapport à la
cvtométrie en flux (marquage Annexine V. Iodure de propidium) de l'état de
la technique
[161] Cellules OCI-AML3 traitées ou non avec des concentrations
croissances d'étoposide
[162] qPCR sur un extrait cytoplasmique issu de cellules lysées après
centrifugation.
CA 03128119 2021-07-20
WO 2020/188216 PCT/FR2020/050571
24
[163] Des cellules OCI-AML3 sont traitées ou non (NT) durant 16h avec des
concentrations croissantes d'étoposide (0 - 7,5 - 15 ou 30 M).
[164] Les cellules sont marquées à l'aide du Kit Pacific BlueTM Annexin V
(Biolegend) selon les recommandations du fournisseur, puis analysées par
cytométrie en flux (MACSQuant VYB - MiltenyiBiotec). Le pourcentage de
cellules
apoptotiques (annexine V) positives est déterminé.
[165] En parallèle, une qPCR est réalisée sur un extrait cytoplasmique issu de
10000 cellules lysées après centrifugation avec le couple d'amorces de SEQ ID
NO :9 et SEQ ID NO : 10. Les résultats sont présentés dans la Figure 6.
[166] qPCR réalisée sur un extrait cytoplasmique issu de cellules lysées
directement dans le milieu de culture.
[167] Des cellules OCI-AML3 sont traitées ou non (NT) durant 16h avec des
concentrations croissantes d'étoposide (0 - 7,5 - 15 ou 30 M).
[168] Les cellules sont marquées à l'aide du Kit Pacific BlueTM Annexin V
(Biolegend) selon les recommandations du fournisseur, puis analysées par
cytométrie en flux (MACSQuant VYB ¨ Milteny iBiotec). Le pourcentage de
cellules apoptotiques (Annexine V) positives est déterminé.
[169] En parallèle, une PCR est réalisée sur un extrait cytoplasmique issu de
10000 cellules lysées directement dans le milieu de culture après
centrifugation,
avec le couple d'amorces de SEQ ID NO :9 et SEQ ID NO : 10. Les résultats sont
présentés dans la Figure 7.
[170] Cellules OCI-AML3 traitées ou non avec des concentrations
croissances de Bortezomib
[171] qPCR sur un extrait cytoplasmique issu de cellules lysées après
centrifugation.
[172] Des cellules OCI-AML3 sont traitées ou non (NT) durant 16h avec des
concentrations croissantes de Bortezomib (0- 0,125 ou 0,25 M).
[173] Les cellules sont marquées à l'aide du Kit Pacific BlueTM Annexin V
(Biolegend) selon les recommandations du fournisseur, puis analysées par
CA 03128119 2021-07-20
WO 2020/188216 PCT/FR2020/050571
cytométrie en flux (MACSQuant VYB - MiltenyiBiotec). Le pourcentage de
cellules
apoptotiques (Annexine V) positives est déterminé.
[174] En parallèle, une PCR est réalisée sur un extrait cytoplasmique issu de
10000 cellules lysées après centrifugation avec le couple d'amorces de SEQ ID
5 NO :9 et SEQ ID NO : 10. Les résultats sont présentés dans la Figure 8.
[175] qPCR réalisée sur un extrait cytoplasmique issu de cellules lysées
directement dans le milieu de culture
[176] Des cellules OCI-AML3 sont traitées ou non (NT) durant 16h avec des
concentrations croissantes de Bortezomib (0- 0,125 ou 0,25 M).
10 [177] Les cellules sont marquées à l'aide du Kit Pacific BlueTM Annexin V
(Biolegend) selon les recommandations du fournisseur, puis analysées par
cytométrie en flux (MACSQuant VYB - MiltenyiBiotec). Le pourcentage de
cellules
apoptotiques (Annexine V) positives est déterminé.
[178] En parallèle, une PCR est réalisée sur un extrait cytoplasmique issu de
15 10000 cellules lysées directement dans le milieu de culture après
centrifugation,
avec le couple d'amorces de SEQ ID NO :9 et SEQ ID NO : 10. Les résultats sont
présentés dans la Figure 9.
[179] Cellules MOLM14 traitées ou non avec des concentrations
croissances d'Etoposide
20 [180] qPCR sur un extrait cytoplasmique issu de cellules lysées après
centrifugation
[181] Des cellules MOLM14 sont traitées ou non (NT) durant 16h avec des
concentrations croissantes d'étoposide (0 ¨ 0,62 - 1,25 ¨ 2,5 ¨ 5 ou 10 M).
[182] Les cellules sont marquées à l'aide du Kit Pacific BlueTM Annexin V
25 (Biolegend) selon les recommandations du fournisseur, puis analysées par
cytométrie en flux (MACSQuant VYB - MiltenyiBiotec). Le pourcentage de
cellules
apoptotiques (annexine V) positives est déterminé.
[183] En parallèle, une PCR est réalisée sur un extrait cytoplasmique issu de
10000 cellules lysées après centrifugation, avec le couple d'amorces de SEQ ID
NO : 7 et SEQ ID NO : 8.
CA 03128119 2021-07-20
WO 2020/188216 PCT/FR2020/050571
26
[184] La sensibilité de la méthode par PCR selon la présente invention est
beaucoup plus importante que la cytométrie en flux. De plus, la méthode par
cytométrie en flux arrive à un palier de détection à partir de la
concentration de 2,5
pM d'étoposide, ce qui n'est pas le cas pour la méthode PCR. Les résultats
sont
présentés dans la Figure 10.
[185] La comparaison de la méthode selon la présente invention avec une
méthode de l'état de la technique (marquage Annexine V, Iodure de propidium),
permet de mettre avantageusement en évidence la meilleure sensibilité et une
moindre saturabilité de la méthode selon la présente invention.
[186] La méthode permet également de détecter la mort cellulaire directement
après lyse dans le milieu de culture, et ce de manière beaucoup plus sensible
qu'avec les techniques de l'art antérieur.
[187] Cellules OCI-AML3 traitées avec 1 M de doxorubicine
[188] Des cellules OCI-AML3 sont traitées ou non durant 16h avec 1pM de
doxorubicine.
[189] Les cellules sont marquées à l'aide du Kit Pacific BlueTM Annexin V
(Biolegend). 1000 cellules doublement négatives (population dn ¨ Annexine V
négatives ¨ Iodure de propidium négatives) traitées ou non-traitées sont
triées.
Ces résultats sont représentés dans la Figure 11A.
[190] Une PCR est réalisée sur un extrait cytoplasmique provenant de ces 1000
cellules (AV-/1F-) traitées (TTT) ou non traitées (NT), en utilisant le couple
d'amorces de SEQ ID NO : 9 et SEQ ID NO : 10. Les résultats de l'expérience
présentée sont des moyennes de trois expériences indépendantes. Les résultats
sont présentés dans la Figure 11B.
[191] II apparait que la méthode selon la présente invention permet de
détecter
l'apparition de la mort cellulaire sur des cellules considérées comme
doublement
négative au marquage Annexine V / Iodure de Propidium par le test de l'état de
la
technique au vu du seuil de sensibilité en faveur de la présente invention. La
présente invention permet une détection plus précoce de la mort cellulaire que
par
le test standard de l'état de la technique.
CA 03128119 2021-07-20
WO 2020/188216 PCT/FR2020/050571
27
[192] Cellules isolées
[193] Des cellules MOLM14 sont traitées ou non durant 16h avec 2,5 M
d'étoposide.
[194] Les cellules sont marquées à l'aide du Kit Pacific BlueTM Annexin V
(Biolegend). Pour chaque sous-population 1 cellule est triée et une ddPCR
(digital
droplet) est réalisée sur un extrait cytoplasmique en utilisant le couple
d'amorces
de SEQ ID NO : 9 et SEQ ID NO : 10. Le niveau d'activation de la mort
cellulaire
est déterminé et comparé avec les signaux obtenus à partir de l'extrait
cytoplasmique d'une cellule non-traitée négative au marquage Iodure de
propidium/ Annexine V.
[195] Les résultats tels que présentés dans la Figure 12 représentent la
moyenne des résultats obtenus avec 5-6 cellules.
[196] La méthode selon l'invention permet de détecter la mort cellulaire sur
un
échantillon d'une cellule et offre ainsi une sensibilité optimisée par rapport
aux
méthodes de l'état de la technique.
[197] Exemple 4: Détection de la mort cellulaire réalisée sur des extraits
cytoplasmiques de différentes lignées cellulaires à l'aide d'amorces ciblant
une séquence présente en une copie ou en deux copies sur le génome.
[198] La Figure 13 représente le niveau d'activation de la mort cellulaire
réalisé
sur des extraits cytoplasmiques des lignées cellulaires indiquées sur l'axe
des
abscisses (MDA-MB-231, Molm14, HeLa, OCI-AML3).
[199] Les cellules MDA-MB-231 sont traitées (TTT) ou non (NT) durant 24h
avec 200nM de staurosporine. Les cellules Molm14 sont traitées (TTT) ou non
(NT) durant 16h avec 0,5 M de doxorubicine. Les cellules HeLa sont traitées
(TTT) ou non (NT) durant 24h avec 400nM de staurosporine. Les cellules OCI-
AML3 sont traitées (TTT) ou non (NT) durant 16h avec 11.1.M de doxorubicine.
Après action du tampon de lyse, la qPCR est réalisée sur les extraits
cytoplasmiques, dilués 10 fois, en utilisant le couple d'amorces de SEQ ID NO
:1
et SEQ ID NO : 2.
CA 03128119 2021-07-20
WO 2020/188216 PCT/FR2020/050571
28
[200] La Figure 14 représente le niveau d'activation de la mort cellulaire
réalisé
sur des extraits cytoplasmiques des lignées cellulaires indiquées sur l'axe
des
abscisses (MDA-MB-231, Molm14, HeLa, OCI-AML3).
[201] Les cellules MDA-MB-231 sont traitées (TTT) ou non (NT) durant 24h
avec 200nM de staurosporine. Les cellules Molm14 sont traitées (TTT) ou non
(NT) durant 16h avec 0,5 M de doxorubicine. Les cellules HeLa sont traitées
(TTT) ou non (NT) durant 24h avec 400nM de staurosporine. Les cellules OCI-
AML3 sont traitées (TTT) ou non (NT) durant 16h avec 11.1.M de doxorubicine.
Après action du tampon de lyse et la centrifugation, la qPCR est réalisée sur
les
extraits cytoplasmiques, dilués 10 fois, en utilisant le couple d'amorces de
SEQ ID
NO :5 et SEQ ID NO :6
[202] Avantageusement, la méthode selon l'invention permet de détecter la mort
cellulaire par la détection et l'amplification d'une séquence d'ADN présente
en une
copie sur le génome.
[203] Exemple 5: Détection de la mort cellulaire réalisée sur des extraits
cytoplasmiques par multiplex
[204] La Figure 15 représente le niveau d'activation de la mort cellulaire
réalisé
sur des extraits cytoplasmiques de lignée cellulaire OCI-AML3.
[205] Les cellules sont traitées (TTT) ou non (NT) durant 24h avec 10 M
d'aracytine. Après centrifugation les cellules sont lysées. La qPCR est
ensuite
réalisée sur les extraits cytoplasmiques, dilués 10 fois, en utilisant
simultanément
le couple d'amorces de SEQ ID : NO : 1 et SEQ ID NO : 2 avec le couple
d'amorces de SEQ ID : NO : 3 et SEQ ID NO :4.
[206] La méthode de détection de la mort cellulaire selon la présente
invention
peut avantageusement être mise en oeuvre par une technique de multiplexage.
[207] Ainsi, et de manière avantageuse, les inventeurs ont mis au point une
méthode de détection de la mort cellulaire sensible, simple et rapide, ayant
un
coût modéré et fonctionnant sur des cellules adhérentes ou en suspension.
Cette
méthode possède une sensibilité accrue, un seuil de détection très inférieur,
et
une moindre saturabilité par rapport aux méthodes de l'état de la technique.
CA 03128119 2021-07-20
WO 2020/188216 PCT/FR2020/050571
29
[208] Exemple 6: détection de la présence de fragments d'ADN dans la
fraction cytoplasmique obtenus après extraction avec un tampon de lyse
décrit dans la méthode selon l'invention, et détectés par électrophorèse
capillaire dans des cellules traitées par une drogue induisant leur mort
(staurosporine) ou non (NT)
[209] Les cellules MDA-MB-231 ont été ensemencées dans 6 T175 à raison de
3 millions de cellules pour 35mL de milieu DMEM high glucose (10% SVF, PIS).
Le lendemain les MDA-MB-231 ont été traitées par 50nM et 100nM de
staurosporine. Le milieu des 2 T175 non traitées a été changé. Après 24h de
traitement, les cellules MDA-MB-231 ont été lysées avec 2mL de Tampon de lyse
ajoutés dans les T175 pendant 15 min à température ambiante, puis, les 2 mL
ont
été transférés dans un tube et centrifugés à 2000g pendant 5 min. Ensuite, le
surnageant a été traité à la RNaseA (20 ilg/mL) puis à la Proteinase K
(utilisation
à 10Oug/mL), et placé 15 min à 70 C. Le tampon de lyse est un mélange d'acide
4-(2-hydroxyéthyl)-1-pi pérazi ne éthane sulfonique à
50mM, d'acide
éthylènediaminetétraacétique à 5mM, de chlorure de sodium à 150mM, et de
digitonine à 50 pg/ml.
[210] Ensuite, 1/10e de volume d'acétate de sodium (NaAc) (3M, pH5.2) ont été
ajoutés. L'ADN a été précipité avec 1 volume d'isopropanol. Les échantillons
ont
été laissés 15 min à température ambiante puis centrifugés à 20 000g pendant
20
min à +4 C. Le surnageant a été aspiré et le culot a été lavé avec de
l'éthanol à
75%. Le culot a été séché puis resuspendu dans 30 I de TE à pH8 et analysés au
fragment analyzer.
[211] La présence d'ADN dans la fraction cytoplasmique des cellules MDA-MB
231 non-traitées ou traitées 24h avec 50 ou 100 nM de staurosporine a été
analysée et ce tel que représenté à la Figure 16.
Les fragments d'ADN, marqueur de la mort cellulaire, sont détectés
dans la fraction cytoplasmique des échantillons cellulaire traités par
une drogue induisant leur mort (staurosporine). Il n'y a pas ou très peu
d'ADN fragmenté dans la fraction cytoplasmique de l'échantillon
cellulaire n'ayant pas subi de traitement induisant la mort cellulaire..