Note : Les descriptions sont présentées dans la langue officielle dans laquelle elles ont été soumises.
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
1
Variants de kallikrein-2 et kallikrein-3 humaines et leurs utilisations
La présente invention concerne le domaine de la biologie, de la génétique et
de
la médecine. Elle concerne notamment de nouvelles séquences nucléotidiques
associées à des événements d'épissage alternatif des gènes correspondant à
l'antigène PSA (antigène spécifique de la prostate ou KLK3) et à la
kallikréine- 2
(KLK2). L'invention concerne également des méthodes pour la détection de la
présence ou pour la détermination du niveau d'expression de ces acides
nucléiques ou des protéines correspondantes dans des échantillons
biologiques, ainsi que des méthodes de sélection de molécules capables de
moduler leur activité ou leur expression.
L'invention est particulièrement adaptée au dépistage, au prognostique, à la
classification ou au suivi de cancers, notamment de la prostate et notamment
dans la différentiation entre le cancer de la prostate et l'hyperplasie
bénigne de
la prostate (BPH), ainsi qu'au développement de nouvelles approches
thérapeutiques de ces maladies.
Les kallikréines correspondent à un groupe de protéines dont l'activité
protéasique permet la modification post-traductionnelle de précurseurs
protéiques vers des formes biologiquement actives. Certains membres de cette
famille, la kallikréine-3, aussi connue sous la dénomination de PSA («
prostate-
specific antigen » ou antigène spécifique de la prostate), et plus récemment,
la
kallikréine-2 sont considérés comme les meilleurs marqueurs disponibles
utilisables dans la détection, le diagnostique et le suivi du cancer de la
prostate.
L'utilisation de tests mesurant la quantité de PSA dans le sang a permis de
diagnostiquer un nombre croissant de patients atteints de cancer de la
prostate
(Pca). Cependant, comme la PSA est également produite par les cellules
épithéliales prostatiques non cancéreuses, il est souvent difficile de
différencier
les patients atteints de cancers de la prostate de ceux présentant un symptôme
bénin hyperplasique (BPH). Dans le sérum, la PSA existe sous forme libre, non
complexée et sous forme complexée, notamment à l'alpha-antichymotrypsine.
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
2
La mesure de ces différentes formes et de leurs rapports permet d'améliorer le
facteur différentiateur entre Pca et BPH.
L'épissage alternatif constitue un mécanisme de régulation de l'expression des
gènes permettant de générer de la diversité fonctionnelle à partir d'une
information génétique limitée. Ce mécanisme, fortement régulé, peut subir des
altérations au cours du développement de pathologies humaines. Ainsi, la
dérégulation de la machinerie d'épissage dans les cancers peut permettre
l'expression d'isoformes ou de variants spécifiquement exprimés dans certaines
tumeurs humaines. Ces isoformes pourront avoir un rôle fonctionnel déterminant
dans le développement ou le maintien de l'état pathologique. L'expression
spécifique de telles isoformes constitue un événement de choix pour une
approche rationnelle et ciblée de développement de médicaments et/ou de
méthodes de diagnostique. Une technologie de profilage de l'expression
génétique (DATAS) a été récemment développée pour identifier de façon
systématique les gènes et, à l'intérieur de ces gènes, les domaines
susceptibles
d'étre altérés par épissage alternatif (W099/46403).
La présente invention décrit à présent de nouveaux événements génétiques liés
à des épissages alternatifs des gènes PSA et KLK2 dans des tissus
prostatiques. La présente invention découle notamment de la construction d'un
répertoire des altérations d'épissage associées à des tissus tumoraux de la
prostate, et de l'identifications d'altérations structurales dans les gènes
PSA et
KLKZ ou dans les ARNm correspondants. La présente invention fournit ainsi de
nouvelles approches thérapeutiques et diagnostiques des cancers, notamment
du cancer de la prostate.
Plus particulièrement, une analyse qualitative différentielle a été effectuée
à
partir d'ARN extraits d'échantillons de tissus prostatiques provenant de zones
tumorale et non tumorale de patients atteints de carcinomes de la prostate.
Cette analyse a été effectuée par criblage différentiel qualitatif grâce à la
mise
en oeuvre de la technique DATAS (décrite dans la demande n° W099/46403)
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
3
qui présente des avantages inégalés. L'application de la technologie DATAS sur
des ARNs issus de tissus prostatiques tumoraux et non-tumoraux a permis
d'isoler divers fragments d'ADNc dérivés des ARNm de la kalikreine 2 et de la
kalikreine 3 (PSA) humaines. Ces résultats ont ensuite permis d'identifier un
certain nombre d'ADNc révélateurs d'événements liés à des épissages
alternatifs.
La présente invention décrit donc des événements moléculaires originaux qui
peuvent aboutir à l'expression spécifique d'isoformes ou de variants de KLK3
(PSA) et de KLK2 dans les tissus prostatiques et plus spécifiquement dans les
tissus cancéreux ou les tissus associés à une hyperplasie bénigne (BPH). La
présente invention fournit des données moléculaires qui justifient
l'utilisation
d'un ou de plusieurs de ces variants comme cibles thérapeutiques et
diagnostiques nouvelles et utilisables avantageusement dans le diagnostic et
le
traitement des cancers et notamment du cancer de la prostate.
Un premier aspect de la présente demande concerne donc des variants de PSA
et KLK2 humains, notamment des variants d'épissage. L'invention concerne les
acides nucléiques correspondant à ces variants ou aux altérations spécifiques
qu'ils présentent, ainsi que les protéines (ou polypeptides ou domaines
protéiques) codés.
Un autre aspect de la présente demande concerne des méthodes et outils pour
détecter la présence de ces variants ou altérations dans des échantillons
biologiques (sang, plasma, urine, sérum, salive, biopsies ou cultures
cellulaires,
etc.), ou pour déterminer leurs) quantités) ou proportions) respective(s). De
tels outils comprennent notamment des sondes ou amorces nucléiques, des
anticorps ou autres ligands spécifiques, des kits, supports, puces, etc. Les
méthodes de détection peuvent inclure les méthodes d'hybridation, de PCR, de
chromatographie, d'immunologie, etc. Ces méthodes sont particulièrement
adaptées à la détection, la caractérisation, le suivi de la progression ou de
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
4
l'efficacité d'un traitement de cancers, notamment du cancer de la prostate,
ou à
la détermination d'une prédisposition.
Un autre aspect de la présente demande concerne des outils et méthodes pour
la production de composés actifs sur les variants décrits, c'est-à-dire
capables
de moduler leur expression ou leur activité. Ces outils et méthodes incluent
notamment des acides nucléiques, des vecteurs, des cellules recombinantes
(ou préparations dérivées de telles cellules), des tests de liaison, etc.
L'invention
vise également les composés ainsi identifiés ou produits, des compositions
pharmaceutiques les contenant, ainsi que leurs utilisations thérapeutiques.
La présente invention est ainsi applicable au diagnostic et au développement
de
stratégies thérapeutiques de cancers, notamment de la prostate.
Variants de KLK2 et KLK3
Un premier aspect de la présente demande concerne donc des variants de KLK-
2 et KLK-3 (PSA), ou des altérations génétiques particulières affectant ces
gènes (ou les ARN ou protéines correspondants). Un objet plus particulier de
l'invention concerne des acides nucléiques correspondant à ces variants de
PSA et KLK2 humains ou aux altérations spécifiques qu'ils présentent, ainsi
que
les protéines (ou polypeptides ou domaines protéiques) codées.
Un certain nombre d'isoformes des gènes KLK2 et KLK3 ont été décrites dans
l'art antérieur.
K-LM correspond à la rétention totale de l'intron 1 de KLK2 (numéro d'accés
Genbank : AF336106) (David et.al (2002)). David et.al précisent que
l'expression de l'ARN messager de K-LM est restreinte à l'épithelium
prostatique
et que la protéine K-LM peut étre détéctée par immunohistochimie dans les
cellules épithéliales sécrétrices (malgré aucune donnée indiquant la
spécificité
de l'anticorps utilisé). Aucune donnée indique la présence de K-LM dans des
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
sérums humains. K-LM semble ëtre détécté dans deux échantillons de fluides
seminaux et dans un échantillon de tissus correspondants à des hyperplasies
bénignes de la prostate. La forme endogène de K-LM n'a pu être détéctée dans
des lignées prostatiques (avec ou sans stimulation androgénique). Aucun
5 résultat n'est montré concernant une expression préférentielle ou
différentielle
de K-LM dans des tissus ou sérums issus de patients atteints d'un cancer de la
prostate.
Un variant de KLK2 utilisant un site alternatif entre l'exon 4 et l'exon 5
correspondant ainsi à une phase ouverte de lecture de 669 paires de bases au
lieu de 783 a été décrit (numéro d'accés Genbank : S39329) (Riegman et.al
(1991 )).
Trois variants possédant des régions 3'UTR plus importantes ont été décrites
(Liu et.al (1999)) (numéro d'accés Genbank : AF188745-7). Un de ces variants
aurait une phase ouverte de lecture équivalente à KLK2 sauvage, un deuxième
variant aurait une phase ouverte de lecture correspondant à celle du variant
décrit précédemment (Riegman et.al (1991 )). Un de ces variants présente une
délétion de 13 nucléotides entre l'exon 3 et l'exon 4 codant ainsi pour une
protéine tronquée de 97 acides aminés dans sa partie carboxy-terminale. Les
auteurs présentent quelques données d'expression par RT-PCR mais n'offrent
aucun résultat concernant la ou les protéines correspondantes.
PSA-LM correspond à la rétention totale de l'intron 1 de PSA (David et.al
(2002)) (numéro d'accés Genbank : AF335477, AF335478, AJ459784). David
et.al précisent que l'expression de l'ARN messager de PSA-LM est restreinte à
l'épithelium prostatique et que la protéine PSA-LM peut ëtre détéctée par
immunohistochimie dans les cellules épithéliales sécrétrices. Aucune donnée
indique la présence de PSA-LM dans des sérums humains, fluides seminaux ou
tissus correspondants à des hyperplasies bénignes de la prostate. La forme
endogène de PSA-LM n'a pu être détéctée dans des lignées prostatiques (avec
ou sans stimulation androgénique). Aucun résultat n'est montré concernant une
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
6
expression préférentielle ou différentielle de PSA-LM dans des tissus ou
sérums
issus de patients atteints d'un cancer de la prostate.
Un variant de PSA possédant une délétion de 129 nucléotide dans l'exon 3 a
été décrit (Tanaka et.al (2000)), appelé également PSA-RP3 (Heuzé-Vourc'h
et.al (2003)). Tanaka et.al présente des données qualitatives d'expression de
ce
variant par RT-PCR dans des tissus prostatiques malins et bénins. L'expression
de la protéine correspondante n'a pas été caractérisée.
Deux variants de PSA, correspondant à une rétention totale de l'intron 3 (PA
424) et à une rétention partielle des derniers 442 nucléotides de l'intron 4
(PA
525) ont été décrits (numéros d'accés Genbank : M21896, M21897) (Riegman
et.al (1988)). PA 424 pourrait se traduire en une protéine mature de 156
acides
aminés. Les 16 derniers acides aminés seraient différents de PSA sauvage. PA
525 pourrait se traduire en une protéine mature de 214 acides aminés. Les 28
derniers acides aminés seraient différents de PSA sauvage. Riegman et.al
présentent aucune donnée complémentaire d'expression différentiatrice des
ARNs messagers ou des protéines.
PA 424 et PA 525 décrits ci-dessus sont très proches de PSA-RP1 et PSA-RP2
qui ont été isolés ultérieurement (numéros d'accés Genbank : AJ310937,
AJ310938) (Heuzé et.al (1999) ; Heuzé-Vourc'h et.al (2001 )). Bien que des
lignées cellulaires de type COS transfectées avec les cDNAs de PSA-RP1 et de
PSA-RP2 peuvent exprimer et sécréter les protéines correspondantes, Heuzé
et.al ne montrent aucun résultat démontrant l'expression des protéines PSA-
RP1 et PSA-RP2 endogènes dans des tissus prostatiques.
Un autre groupe (Meng et.al (2002)) a caractérisé l'expression de l'ARN
messager de PSA-RP1 par Northern et hybridization in situ. Aucune différence
d'expression n'a pu étre observée entre des tissus microdissectés sains et
tumoraux. Un anticorps spécifique de PSA-RP1 a permis de détecter par
immunohistochimie l'expression de la protéine PSA-RP1 dans le cytoplasme
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
7
des cellules épithélilales à partir de coupes de tissus prostatiques sains et
tumoraux.
Un variant de PSA correspondant à une rétention de la partie 5' de l'intron 4,
PSA-RPS, a été soumis à Genbank (numéro d'accés : AJ512346)
Un variant de PSA comportant une délétion dans l'exon 3, PSA-RP4, a été
soumis à Genbank ( numéro d'accés : AJ459782).
La présente demande décrit maintenant l'existence de formes différentes des
gènes PSA et KLK2, et leur corrélation à des situations pathologiques. Ces
isoformes ont été identifiées à partir d'échantillons tumoraux. La description
des
ADNc et des protéineslpolypeptides codés par ces ADNc est indiquée ci-
dessous. Les séquences complètes sont fournies dans la Liste de Séquences
annexée à la présente demande. Les caractéristiques principales des variants
spécifiques selon l'invention sont décrites dans les exemples.
Un premier objet de l'invention concerne des acides nucléiques comprenant la
séquence des variants de PSA et KLK2 décrits dans la présente demande, ou
une partie spécifique de celle-ci.
Un autre objet de l'invention concerne des acides nucléiques spécifiques des
altérations génétiques portées par les variants de PSA et KLKZ décrits dans la
présente demande. De tels acides nucléiques peuvent être notamment
complémentaires de régions mutées, de domaines introniques retenus ou de
jonctions nouvellement créées par des délétions.
Un autre objet de l'invention concerne un acide nucléique comprenant tout ou
partie d'une séquence dérivée des ARNs messagers (ou des ADNc) de KLK2-
EHT002 à KLK2-EHT011 et de PSA-EHT001 à PSA-EHT027 ou de KLK2-EHTb
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
8
à KLK2-EHTI et de PSA-EHTa à PSA-EHTu ou toute combinaison de ces
variants ainsi que leurs utilisations pour la mise en oeuvre d'une méthode de
diagnostic, de détection ou de suivi de cancers, notamment du cancer de la
prostate et plus particulièrement de la forme bénigne de ce dernier, BPH.
Un autre objet de l'invention réside dans tout acide nucléique caractérisé en
ce
qu'il comprend une séquence choisie parmi
a) les séquences SEQ ID NOs : 1 à 49,
b) un variant des séquences SEQ ID NOs : 1 à 49 résultant de la
dégénérescence du code génétique,
c) le brin complémentaire des séquences SEQ ID NOs : 1 à 49, et
d) un fragment spécifique des séquences a) à c).
Le terme fragment ou partie « spécifique » désigne un fragment caractéristique
des variants considérés, typiquement un fragment portant au moins une
altération génétique caractéristique des variants considérés. De tels
fragments
spécifiques se distinguent donc de la séquence sauvage par la présence d'une
caractéristique structurale propre (e.g., mutation, nouvelle jonction,
rétention
d'intron, délétion d'une séquence, codon stop, nouvelle séquence résultant
d'un
décalage du cadre de lecture, etc.) résultant d'un événement d'altération mis
en
évidence par les demandeurs chez des patients. Cette caractéristique
structurale propre est également désignée par l'expression « séquence cible ».
Des fragments spécifiques selon l'invention comprennent donc au moins une
séquence cible telle que définie ci-avant. Des fragments préférés comportent
au
moins 5 nucléotides consécutifs de la séquence considérée, de préférence au
moins 8, plus préférentiellement au moins 12. Des fragments peuvent
comprendre jusqu'à 50, 75 ou 100 nucléotides, voire plus.
Au sens de l'invention, les acides nucléiques peuvent étre des ADN, choisis de
préférence parmi les ADNc et les ADNg, ou des ARN. II peut s'agir d'acides
nucléiques synthétiques ou semi-synthétiques, de fragments PCR,
d'oligonucléotides, de régions double- ou simple-brin, etc. Les acides
nucléiques
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
9
peuvent être produits par synthèse, par voie recombinante, par clonage, par
assemblages) génétique(s), mutagénèse, etc., ou une combinaison de ces
techniques.
Les acides nucléiques peuvent être utilisés pour produire un variant de PSA ou
KLK-2 de l'invention in vitro, ex vivo, in vivo ou dans un système de
transcription
acellulaire. Ils peuvent également être utilisés pour la fabrication de
molécules
antisens ou interférentes (RNAi), capables de réduire l'expression ou la
traduction des ARNm correspondants dans une cellule. Ils peuvent aussi servir
à la production de sondes, notamment marquées, permettant par des réactions
d'hybridation de mettre en évidence de manière spécifique la présence d'une
forme mutée de PSA ou KLK-2 de l'invention dans un échantillon. Ils peuvent
encore servir à la production d'amorces nucléiques, utiles à l'amplification
d'un
variant de PSA ou KLK-2 (ou d'une séquence cible d'un tel variant) dans un
échantillon, notamment dans un but de dépistage ou de diagnostic.
A cet égard, un autre objet de l'invention concerne une sonde nucléique
caractérisée en ce qu'elle permet la détection d'un acide nucléique tel que
défini
ci-avant, typiquement par hybridation sélective à partir d'une population
d'acides
nucléiques test. Généralement, la sonde comprend la séquence d'un acide
nucléique tel que défini ci-avant ou une partie (spécifique) de la séquence
d'un
tel acide nucléique. La partie spécifique est préférentiellement
caractéristique
d'un variant tel que décrit ci-avant, notamment une partie contenant une
altération associée au cancer de la prostate. Elle comprend typiquement de 10
à
1000 nucléotides, de préférence de 50 à 800, et est généralement simple-brin.
Un exemple particulier de sonde est représenté par un oligonucléotide
spécifique et complémentaire d'une région au moins d'un acide nucléique tel
que défini ci-avant. L'oligonucléotide est typiquement simple-brin, et
comporte
généralement de 10 à 100 bases. Des exemples spécifiques d'oligonucléotides
de l'invention sont fournis dans le Tableau 1. Les oligonucléotides et/ou les
sondes nucléiques selon l'invention peuvent ëtre marquées, par exemple au
moyen de marqueurs radioactifs, enzymatiques, fluorescents, luminescents, etc.
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
Un autre objet de l'invention concerne une amorce nucléique, permettant
l'amplification (sélective) d'un acide nucléique tel que défini ci-avant ou
d'une
partie (spécifique) d'un tel acide nucléique. La partie amplifiée comporte
5 préférentiellement une altération caractéristique d'un des variants décrits
cl-
avant, notamment d'une altération associée au cancer de la prostate. Une
amorce selon l'invention est typiquement simple-brin, et avantageusement
composée de 3 à 50 bases, de préférence de 3 à 40 et encore plus
préférentiellement de 3 à 35 bases. Une amorce particulière est complémentaire
10 d'une région au moins du gène PSA ou KLK-2, ou de l'ARN correspondant.
Un mode de réalisation préféré réside dans une amorce constituée d'un acide
nucléique simple-brin comprenant de 3 à 50 nucléotides complémentaires d'une
partie au moins de l'une des séquences SEQ ID NOs : 1 à 49 ou de leur brin
complémentaire. Des exemples de telles amorces nucléiques sont fournies dans
la section expérimentale.
L'invention concerne également un couple d'amorces comprenant une
séquence sens et une séquence inverse, caractérisé en ce que les amorces de
ladite paire s'hybrident avec une région d'un acide nucléique tel que défini
cl-
avant et permettent l'amplification d'au moins une portion de cet acide
nucléique.
Des couples d'amorces particuliers selon l'invention sont fournis dans le
Tableau 2.
Un autre objet de la présente demande concerne tout vecteur comprenant un
acide nucléique tel que défini ci-avant. II peut s'agir de plasmides,
cosmides,
épisomes, chromosomes artificiels, virus, phages, etc. On peut citer divers
plasmides commerciaux tels que pUC, pcDNA, pBR, etc. Parmi les vecteurs
viraux, on peut citer les rétrovirus, adénovirus, AAV, herpès virus, etc.
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
11
Un autre objet de l'invention concerne les cellules recombinantes comprenant
un acide nucléique ou un vecteur tels que définis ci-avant. Les cellules
peuvent
être procaryotes ou eucaryotes. Parmi les cellules procaryotes on peut citer
notamment les bactéries telles que E. coli. Parmi les cellules eucaryotes, on
peut mentionner les cellules de levure ou les cellules de mammifères,
d'insectes
ou de plantes. II peut s'agir de cultures primaires ou de lignées. On peut
citer les
cellules COS, CHO, 3T3, HeLa, etc.
Un autre objet de l'invention concerne une composition comprenant un acide
nucléique tel que défini ci-avant immobilisé sur un support. L'invention
concerne
notamment des compositions comprenant une pluralité d'acides nucléiques en
mélange, sous forme soluble ou immobilisée à un support, la composition
comprenant au moins un acide nucléique tel que défini ci-avant.
Un autre objet de l'invention concerne un (produit comprenant un) support sur
lequel un ou plusieurs acides nucléiques tels que définis ci-avant sont
immobilisés. Le support peut âtre solide, plan ou non, régulier ou non, comme
par exemple du nylon, verre, plastique, métal, fibre, céramique, silice,
polymère,
etc., ou tout autre matériau compatible. Les acides nucléique sont
préférentiellement immobilisés par une extrémité, dans des conditions laissant
la molécule accessible pour une réaction d'hybridation. Les acides nucléiques
peuvent être arrangés de manière précise sur le support, et déposés en
plusieurs exemplaires.
Dans une variante particulière, on utilise un ou plusieurs oligonucléotides
spécifiques pour caractériser chaque évènement d'épissage alternatif (voir
figure 9). On peut notamment utiliser un oligonucléotide spécifique d'un exon
éliminé, permettant la quantification de la forme longue, et/ou un
oligonucléotide
spécifique de l'un des exons adjacents non impliqué dans l'épissage,
permettant
de quantifier les formes longues et courtes de l'ARN, et/ou un ou plusieurs
(e.g.
trois) oligonucléotides spécifiques des jonctions, dont l'un est spécifique de
la
nouvelle séquence générée après épissage, permettant de quantifier la forme
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
12
épissée. II est entendu que d'autres combinaisons d'oligonucléotides peuvent
étre envisagées, notamment l'emploi d'un seul ou de deux oligonucléotides
seulement. Concernant plus précisément les oligonucléotides de jonction, ils
sont idéalement centrés sur les jonctions, même si des oligonucléotides
décalés
sur la jonction peuvent également étre mis en oeuvre. Avantageusement, on
utilise des oligonucléotides ne présentant pas de structures secondaires qui
pourraient interférer avec leur capacité d'hybridation. D'une manière
générale, il
est préférable d'avoir sur la puce un profil thermodynamique homogène pour
l'ensemble des oligos générés, à savoir le Tm (65°C) et la longueur (24-
25
mers). Par ailleurs, au cours de leur synthèse, les oligonucléotides peuvent
étre
modifiés en 5' par un groupement NH2-C6 favorisant leur flexibilité et
permettant d'établir une liaison covalente avec le polymère constituant le
revétement du support.
Un autre objet de l'invention concerne un (produit comprenant un) support sur
lequel une ou plusieurs cellules recombinantes telles que définies ci-avant
sont
immobilisées ou cultivées. Le support peut étre solide, plan ou non, régulier
ou
non, comme par exemple du nylon, verre, plastique, métal, fibre, céramique,
silice, polymère, etc., ou tout autre matériau compatible. Les cellules sont
par
exemple réparties dans des puits d'une microplaque ou immobilisées dans un
gel ou sur un support adapté.
L'invention concerne également les peptides et séquences protéiques codés par
tout ou partie des isoformes KLK2-EHT002 à KLK2-EHT011 et PSA-EHT001 à
PSA-EHT027 , ou KLK2-EHTb à KLK2-EHTI et PSA-EHTa à PSA-EHTu
notamment celles décrites parmi les séquences SEQ ID NO : 50 à 167 ainsi que
leurs utilisations pour la mise en oeuvre d'une méthode de diagnostic, de
détection ou de suivi de cancers, notamment du cancer de la prostate et plus
particulièrement de la forme bénigne de ce dernier, BPH.
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
13
Un objet particulier de la présente demande concerne un polypeptide
comprenant tout ou une partie spécifique d'une séquence choisie parmi SEQ ID
NOs : 50 à 167. Des polypeptides particuliers sont composés ou comprennent
une séquence ou une partie de séquence créée par l'altération du gène ou du
messager correspondant. Au sens de l'invention, le terme « partie » désigne
préférentiellement au moins 5 résidus contigus, de préférence au moins 8, plus
préférentiellement au moins 10, encore plus préférentiellement au moins 15.
Comme expliqué ci-avant, les altération d'épissage du gène PSA ou KLK-2
conduisent à la production de protéines mutées, comprenant des séquences
nouvellement créées (séquences cibles). II peut s'agir de séquences nouvelles
(e.g., traduction décalée, insertions), de jonctions nouvelles, etc. Des
peptides
particuliers de l'invention correspondent ou comprennent tout ou une partie
spécifique des séquences SEQ ID Nos : 53, 56, 59, 62, 65, 67 (résidus 146-
150), 70, 71, 73, 76, 79, 81, 93, 95, 98, 106, 108, 110, 112, 117, 119
(résidus
66-70 ou 74-79), 121 (résidus 117-121 ), 123 (résidus 25-29, 51-55 ou 105-111
),
126, 131, 133, 134, 135 (résidus 64-68) et 155.
Un autre objet de l'invention concerne un (produit comprenant un) support sur
lequel un ou plusieurs polypeptides tels que définis ci-avant sont
immobilisés.
Le support peut étre solide, plan ou non, régulier ou non, comme par exemple
du nylon, verre, plastique, métal, fibre, céramique, silice, polymère, etc.,
ou tout
autre matériau compatible. Les polypeptides sont préférentiellement
immobilisés
par une extrémité, dans des conditions laissant la molécule accessible pour
une
réaction d'interaction avec un ligand spécifique, tel qu'un anticorps. Les
polypeptides peuvent étre arrangés de manière précise sur le support, et
déposés en plusieurs exemplaires.
Des techniques d'immobilisation de matériels (tels que acides nucléiques,
polypeptides, anticorps, etc.) sur des supports ont été décrites dans la
littérature, et notamment dans les demandes ou brevets n° EP619 321,
W091108307, US4,925,785 et GB2,197,720.
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
14
Ligands saécifiaues
L'invention concerne par ailleurs des ligands spécifiques, de préférence
peptidiques, en particulier des anticorps (polyclonaux, monoclonaux) et leurs
fragments, spécifiques des régions peptidiques caractéristiques des protéines
codées par KLK2-EHT002-011 et de PSA-EHT001-027 ou par KLK2-EHTb à
KLK2-EHTI et PSA-EHTa à PSA-EHTu (codées par les domaines introniques
retenus ou par les jonctions spécifiquement créées) et leurs utilisations pour
la
détection, le diagnostique ou le suivi de cancers et notamment du cancer de la
prostate. En particulier, il pourra s'agir de diagnostiquer la forme BPH et de
la
différencier du carcinome de la prostate.
A cet égard, un autre objet de l'invention concerne tout anticorps capable de
se
lier, de préférence de manière sélective, à un polypeptide tel que défini ci-
dessus. L'anticorps peut ëtre polyclonal ou monoclonal. II peut également
s'agir
de fragments et dérivés d'anticorps présentant substantiellement la méme
spécificité antigénique, en particulier des fragments d'anticorps (e.g., Fab,
Fab'2, CDRs), d'anticorps humanisés, polyfonctionnels, monocaténaires (ScFv),
etc. Les anticorps peuvent être produits à l'aide de méthodes
conventionnelles,
comprenant l'immunisation d'un animal et la récupération de son sérum
(polyclonal) ou de cellules spléniques (de manière à produire des hybridomes
par fusion avec des lignées cellulaires appropriées).
Des méthodes de production d'anticorps polyclonaux à partir d'espèces variées
sont décrites dans l'art antérieur. Typiquement, l'antigène est combiné avec
un
adjuvant (e. g., Freund's adjuvant) et administré à un animal, typiquement par
injection sous-cutanée. Des injections répétées peuvent étre réalisées. Les
échantillons sanguins sont collectés et l'immunoglobuline ou le sérum sont
séparés. Les méthodes classiques de production d'anticorps monoclonaux
comprennent l'immunisation d'un animal avec un antigène, suivie de la
récupération des cellules spléniques qui sont ensuite fusionnées avec des
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
cellules immortalisées, telles que des cellules de myélome. Les hybridomes
résultant produisent des anticorps monoclonaux et peuvent être sélectionnés
par dilutions limites de manière à isoler les clones individuels. Les
fragments
Fab ou F(ab')2 peuvent être produits par digestion à l'aide d'une protéase
selon
5 les techniques conventionnelles.
L'invention concerne également une méthode de production d'anticorps,
comprenant l'injection d'un polypeptide tel que défini ci-avant ou d'un
fragment
immunogène de ce dernier à un animal non humain et la récupération des
10 anticorps ou des cellules productrices d'anticorps. Les anticorps préférés
sont
des anticorps spécifiques des isoformes de PSA et KLK-2 décrites dans la
présente demande, et essentiellement non-spécifique des formes sauvages.
L'invention concerne des hybridomes produisant les anticorps monoclonaux
15 décrits ci-dessus et leur utilisation pour produire lesdits anticorps.
Les anticorps peuvent être couplés à des fragments hétérologues telles que des
toxines, des marqueurs, des médicaments ou tout autre agent thérapeutique, de
façon covalente ou non, soit directement, soit par l'intermédiaire d'agents de
couplage. Les marqueurs peuvent être choisis parmi les radio-marqueurs, des
enzymes, des agents fluorescents, des particules magnétiques, etc.
Les anticorps de l'invention peuvent être utilisés comme agents de criblage ou
pour détecter ou quantifier la présence ou la quantité d'isoformes de PSA ou
KLK-2 dans des échantillons collectés à partir d'un sujet, typiquement, un
fluide
biologique provenant d'un mammifère, par exemple d'un être humain.
Un autre objet de l'invention concerne un (produit comprenant un) support sur
lequel un ou plusieurs anticorps (ou fragments ou dérivés) tels que définis ci-
avant sont immobilisés. Le support peut être solide, plan ou non, régulier ou
non, comme par exemple du nylon, verre, plastique, métal, fibre, céramique,
silice, polymère, etc., ou tout autre matériau compatible. Les anticorps sont
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
16
préférentiellement immobilisés par une extrémité, dans des conditions laissant
la molécule accessible pour une réaction d'interaction avec un antigène
spécifique. Les anticorps peuvent étre arrangés de manière précise sur le
support, et déposés en plusieurs exemplaires.
Méthodes de détection/diaetnostic
La présente demande décrit également de nouveaux procédés de détection
d'une pathologie ou d'une prédisposition à une pathologie chez un sujet,
comprenant la détermination de la présence, dans un échantillon dudit sujet,
d'un acide nucléique, ou d'une altération génétique ou d'une protéine ou
polypeptide tels que définis ci-avant.
La détermination peut être réalisée par différentes techniques, telles que
séquençage, hybridation et/ou amplification sélectives. Des méthodes
utilisables
pour déterminer la présence de protéines sont basées par exemple sur des
réactions immuno-enzymatiques, telles que ELISA, RIA, EIA, etc. Des
techniques utilisables pour déterminer la présence de gènes ou d'ARN altérés
sont par exemple la PCR, la RT-PCR, la réaction de ligation en chaîne (LCR),
la
technique de PCE ou TMA (« Transcriptional Mediated Amplification »), la
migration sur gel, l'électrophorèse, notamment la DGGE (« denaturing gel
gradient electrophoresis »), etc.
Dans le cas où une étape d'amplification est réalisée, celle-ci met
préférentiellement en oeuvre une amorce ou un couple d'amorces tel que défini
ci-avant.
Un objet particulier de l'invention concerne l'utilisation d'acides nucléiques
complémentaires et spécifiques de fragments des gènes ou des messagers de
KLK2-EHT002-011 et de PSA-EHT001-027 ou de KLK2-EHTb à KLK2-EHTI et
de PSA-EHTa à PSA-EHTu (e.g., domaines introniques retenus, jonctions
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
17
spécifiquement crées, mutations particulières, etc.) pour la détection de
cancers,
notamment du cancer de la prostate, et plus particulièrement de sa forme
bénigne, BPH. Cette détection pourra en particulier s'effectuer grâce à des
puces à ADN ou à la mise en oeuvre d'une PCR à partir de fluides biologiques
tels que du sang (le sérum notamment ou a partir de cellules épithéliales
circulantes purifiées), des urines, du fluide séminal, etc.
L'invention réside également dans la mise au point et l'utilisation de tests
immunologiques contenant un ou plusieurs anticorps tels que décrits ci-dessus
ou fragments de ces derniers. Ces tests permettent de détecter et/ou mesurer
individuellement un variant à l'aide d'un anticorps spécifique, plusieurs
variants,
en parallèle, grâce aux anticorps spécifiques appropriés, ou un ou plusieurs
rapports entre les isoformes telles que décrites ci-dessus ou entre lesdites
isoformes et d'autres formes décrites pour kallikreine-2 et PSA.
Une méthode particulière comprend la mise en contact d'un échantillon
provenant d'un sujet avec une sonde telle que définie ci-avant, et la mise en
évidence d'une hybridation.
Une autre méthode particulière comprend la mise en contact d'un échantillon
provenant d'un sujet avec une amorce ou une paire d'amorces telles que
définies ci-avant, et la mise en évidence d'un produit d'amplification.
Une autre méthode particulière comprend la mise en contact d'un échantillon
provenant d'un sujet avec un anticorps tel que défini ci-avant, et la mise en
évidence d'un complexe antigène-anticorps.
Typiquement, des plusieurs tests sont réalisés en parallèle, à partir de
plusieurs
échantillons etlou en utilisant plusieurs sondes, amorces et/ou anticorps.
Ainsi,
dans un mode particulier, le procédé de l'invention comprend la détermination,
en parallèle, de la présence de plusieurs variants ou altérations génétiques
tels
que décrits ci-avant dans un échantillon d'un patient. Les procédés selon
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
18
l'invention peuvent étre mis en oeuvre à partir d'échantillons biologiques
variés,
notamment de fluides biologiques (e.g., sang, plasma, urine, sérum, salive,
etc.), de biopsies de tissus ou de cultures cellulaires, par exemple et, plus
généralement, à partir de tout échantillon susceptible de contenir des acides
nucléiques ou des protéines (ou polypeptides). L'échantillon biologique peut
étre
traité avant la mise en oeuvre du procédé, pour faciliter ou permettre
l'accessibilité des polypeptides ou acides nucléiques qu'il contient.
L'échantillon
peut également étre purifié, centrifugé, fixé, etc., éventuellement congelé ou
conservé avant usage.
Dans un mode de réalisation particulier, l'invention concerne un procédé de
détection de la présence d'une forme altérée de KLK-2 ou KLK-3 chez un sujet,
comprenant la mise en contact, in vitro ou ex vivo, d'un échantillon dudit
sujet
avec une sonde, une amorce ou un ligand spécifique tels que définis ci-avant
et
la détermination de la formation d'un hybride, d'un produit d'amplification ou
d'un complexe, respectivement, la dite formation étant indicative de la
présence
d'une forme altérée.
Un autre objet de l'invention réside dans un kit utilisable pour la mise en
oeuvre
d'un procédé tel que défini ci-avant comprenant
i. un couple d'amorces ou une sonde ou un anticorps tels que
définis ci-avant, et
ü. les réactifs nécessaires à l'amplification ou à une réaction
d'hybridation ou immunologique.
L'invention réside également dans la mise au point d'une méthode permettant
de détecter et/ou de mesurer des partenaires spécifiques d'un ou de plusieurs
de ces variants par ajout de l'un ou de plusieurs de ces variants ou de leurs
fragments dans des fluides biologiques à tester, tels que du sang (sérum
notamment), des urines ou du fluide séminal.
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
19
Criblage de composés actifs
Les variants spécifiques de KLK2 et de KLK3 selon l'invention ont été
identifiés
et isolés à partir de sujets pathologiques et représentent donc des cibles
thérapeutiques particulièrement intéressantes pour le traitement des cancers
et
notamment du cancer de la prostate.
A cet égard, un objet particulier de l'invention réside dans des méthodes de
sélection, d'identification, de caractérisation, d'optimisation ou de
production de
composés actifs, comprenant une étape de détermination de la capacité d'un
composé test à moduler l'expression ou l'activité d'un polypeptide tel que
défini
ci-avant.
Les composés sont plus particulièrement sélectionnés sur la base de leur
capacité à moduler la synthèse d'un polypeptide tel que défini ci-avant (c'est-
à-
dire notamment la production ou la maturation des ARNs correspondants, ou
leur traduction) ou l'activité d'un tel polypeptide (c'est-à-dire notamment
leur
maturation, transport, ou leur interaction avec des cibles intra- ou extra-
cellulaires).
Dans une variante de réalisation, la méthode comprend la mise en contact in
vitro ou ex vivo d'un composé test avec un polypeptide tel que défini ci-avant
ou
un acide nucléique codant un tel polypeptide (e.g., gène, ADNc, ARN), et la
sélection des composés se liant audit polypeptide ou acide nucléique. La
liaison
au polypeptide au gène ou à l'ARN correspondant peut être mesurée par
différentes techniques, telles que le déplacement d'un ligand marqué, la
migration sur gel, l'électrophorèse, etc. Elle peut être réalisée in vitro,
par
exemple en utilisant le polypeptide ou l'acide nucléique immobilisé sur un
support.
Dans une autre variante de réalisation, la méthode comprend la mise en contact
in vitro ou ex vivo d'un composé test avec une cellule exprimant un
polypeptide
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
tel que défini ci-avant, et la sélection ou l'identification de composés
modulant
l'expression ou l'activité dudit polypeptide. La modulation de l'expression
peut
étre déterminée par dosage des ARN ou des protéines, ou au moyen d'un
système rapporteur.
5
Les cellules utilisées peuvent être toute cellule compatible, notamment des
cellules eucaryotes ou procaryotes telles que mentionnées plus haut. On
utilise
typiquement une cellule modifiée pour exprimer ladite molécule, notamment des
cellules recombinantes. De telles cellules recombinantes peuvent être
10 préparées par introduction d'un acide nucléique recombinant exprimant le
polypeptide, ou d'un vecteur comprenant celui-ci. De telles cellules
recombinantes constituent des objets particuliers de l'invention.
Le procédé peut être mis en oeuvre pour sélectionner ou identifier un
activateur
15 ou un inhibiteur de l'expression ou de l'activité de l'antigène spécifique
de la
PSA ou de KLK-2. Les procédés de sélection peuvent être réalisés en différents
formats, comme par exemple en plaques multi-puits, en testant en parallèle de
multiples composés candidats.
20 Dans un mode de réalisation particulier, le composé est un acide nucléique
anti-
sens, capable d'inhiber l'expression des variants décrits. L'acide nucléique
anti-
sens peut comprendre tout ou partie de séquences spécifiques des variants
décrits. L'anti-sens peut notamment comprendre une région complémentaire de
la forme d'épissage identifiée (e.g., d'une séquence cible), et inhiber (ou
réduire)
sa traduction en protéine.
Selon un autre mode de réalisation, le composé est un composé chimique,
d'origine naturelle ou synthétique, notamment une molécule organique ou
inorganique, d'origine végétale, bactérienne, virale, animale, eucaryote,
synthétique ou semi-synthétique, capable de moduler l'expression ou l'activité
de l'un ou de plusieurs des variants décrits ci-dessus.
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
21
On préfère des composés spécifiques, c'est-à-dire capables de moduler
l'expression ou l'activité des variants, sans affecter significativement
l'expression ou l'activité des formes sauvages.
Les composés ainsi identifiés sont utilisables pour la préparation d'une
composition destinée au traitement du cancer de la prostate.
Un autre objet de l'invention réside dans l'utilisation d'un composé capable
de
moduler, i.e. de stimuler, d'inhiber ou de réduire, l'expression d'un ou de
plusieurs variants tels que décrits ci-dessus, pour la préparation d'une
composition destinée au traitement des cancers et notamment du cancer de la
prostate.
Dans le contexte de l'invention, le terme « traitement » désigne le traitement
préventif, curatif ou palliatif, ainsi que la prise en charge des patients
(réduction
de la souffrance, amélioration de la durée de vie, ralentissement de la
progression de la maladie), etc. Le traitement peut en outre être réalisé en
combinaison avec d'autres agents actifs.
Un autre objet de l'invention concernent des méthodes de sélection,
d'identification, ou de caractérisation de composés actifs utilisables dans le
cadre de la préparation de compositions destinées au traitement de pathologies
cancéreuses, comprenant la mise en contact d'un ou de plusieurs composés
tests avec des extraits cellulaires exprimant les protéines décrites dans la
présente invention, ou avec lesdites protéines purifiées.
L'invention porte également sur un procédé de production d'un médicament
pour le traitement de cancers, notamment du cancer de la prostate, comprenant
(i) la sélection de composés actifs selon les procédés ci-dessus et (ü) le
conditionnement dudit composé ou d'un analogue fonctionnel de celui-ci en
présence d'un véhicule acceptable sur le plan pharmaceutique. L'analogue
fonctionnel est typiquement un composé dérivé du composé actif identifié, par
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
22
modification chimique, notamment dans le but d'améliorer son activité ou sa
pharmaco-cinétique, ou dans le but de reauire sa zoxicne. ~ anaw~uc
fonctionnel peut étre une « pro-drogue » du composé identifié. Des techniques
de préparation d'analogues fonctionnels sont bien connues de l'homme du
métier, par exemple la modélisation moléculaire, le couplage de groupes NO,
etc. Le procédé peut à cet égard comprendre une étape intermédiaire de
synthèse du composé sélectionné ou de l'analogue fonctionnel de celui-ci.
Le véhicule ou excipient acceptable sur le plan pharmaceutique peut étre
choisi
parmi des solutés tampons, solvants, liants, stabilisants, émulsifiants, etc.
Des
solutés tampons ou diluant sont notamment le dicalcium phosphate, sulfate de
calcium, lactose, cellulose, kaolin, mannitol, chlorure de sodium, amidon,
sucre
en poudre et hydroxy propyl methyl cellulose (HPMC) (pour libération retard).
Des liants sont par exemple l'amidon, la gélatine et des solutés de
remplissage
comme le sucrose, glucose, dextrose, lactose, etc. Des gommes naturelles ou
synthétiques peuvent aussi être utilisées, comme notamment l'alginate, la
carboxymethylcellulose, la méthylcellulose, la polyvinyl pyrrolidone, etc.
D'autres
excipients sont par exemple la cellulose et du stéarate de magnésium. Des
agents stabilisants peuvent étre incorporés aux formulations, comme par
exemple des polysaccharides (acacia, agar, acide alginique, gomme guar et
tragacanth, la chitine ou ses dérivés et des éthers de cellulose. Des solvants
ou
solutés sont par exemple la solution Ringer, l'eau, l'eau distillée, des
tampons
phosphates, des solutions salines phosphatées, et autres fluides
conventionnels.
Un autre objet de l'invention concerne l'utilisation de ligands cytotoxiques
spécifiques d'un ou de plusieurs variants tels que décrits ci-dessus localisés
à la
surFace des cellules cancéreuses et en particulier des cellules cancéreuses
prostatiques.
D'autres aspects et avantages de la présente invention apparaîtront à la
lecture
des exemples qui suivent, qui doivent ëtre considérés comme illustratifs et
non
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
23
limitatifs. Ces exemples indiquent clairement que les isoformes identifiées
peuvent s'exprimer dans des systèmes biologiques tant au niveau de l'ARN
qu'au niveau protéique dans les tissus et sérums.
LEGENDE DES TABLEAUX ET FIGURES
Tableau 1 : Séquence des oligonucléotides spécifiques (SEQ ID NOs : 168-
220). Colonne 1 : Nom de l'oligonucléotide. Colonne 2: Séquence de
l'oligonucléotide. Colonne 3 : SEQ ID NO des oligonucléotides revendiqués.
Tableau 2 : Couples d'amorces utilisés pour l'amplification des isoformes de
PSA et de KLK2.
Tableau 3 : Valeurs des signaux de fluorescence obtenues par hybridation à
partir de tissus humains (Clontech) d'un micro-array regroupant des
oligonucléotides dont les oligonucléotides SEQ ID NOs : 168-220. Colonne 1
Nom de l'oligonucléotide. Colonne 2: SEQ ID NO. Colonne 3-4: Valeurs
correspondant à prostate/coeur. Colonne 5-6: Valeurs correspondant à
prostate/rein. Colonne 7-8 : Valeurs correspondant à prostatelprostate.
Colonne
9-10 : Valeurs correspondant à prostate/petit intestin. Le signe #N/A indique
que
la valeur était inférieure à deux fois le bruits de fonds.
Tableau 4 : Valeurs des signaux de fluorescence obtenues par hybridation à
partir de lignées cellulaires d'un micro-array regroupant des oligonucléotides
dont les oligonucléotides SEQ ID NOs : 168-220. Colonne 1 : Nom de
l'oligonucléotide. Colonne 2 : SEQ ID NO. Colonne 3-4: Valeurs correspondant
à Mda2b/BT549. Colonne 5-6 : Valeurs correspondant à Mda2b/MCF7. Colonne
7-8: Valeurs correspondant à Mda2b/Mda231. Colonne 9-10: Valeurs
correspondant à Mda2b/T47D. Le signe #N/A indique que la valeur était
inférieure à deux fois le bruits de fonds.
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
24
Tableau 5 : Valeurs des signaux de fluorescence obtenues par hybridation à
partir de tissus bénins et tumoraux issus de patients atteints de cancer de la
prostate d'un micro-array regroupant des oligonucléotides dont les
oligonucléotides SEQ ID NOs : 168-220. Colonne 1 : Nom de l'oligonucléotide.
Colonne 2: SEQ ID NO. Colonne 3-4: Valeurs correspondant au tissu
tumoral/tissu bénin du patient 15068. Colonne 5-6 : Valeurs correspondant au
tissu tumoral/tissu bénin du patient 9648. Colonne 7-8 : Valeurs correspondant
au tissu tumoral/tissu bénin du patient 8827. Colonne 9-10: Valeurs
correspondant au tissu tumoral/tissu bénin du patient 10063. Le signe #N/A
indique que la valeur était inférieure à deux fois le bruits de fonds.
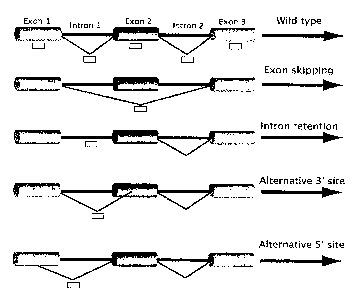
Figure 1 : Disposition des oligonucléotides spécifiques. Des oligonucléotides
matérialisés par des rectangles sont conçus pour s'hybrider de façon
spécifique
à des événements d'épissage : rétention d'intron, délétion d'exon, utilisation
de
sites cryptiques 3' et 5'.
Figure 2: Disposition d'oligonucléotides spécifiques. Cinq oligonucléotides
matérialisés par un trait peuvent être conçus pour analyser l'expression d'une
forme longue matérialisée par 3 exons et d'une forme courte matérialisée par 2
exons.
Figure 3 : Marquage des formes longues et courtes synthétiques. Des ARNs
synthétiques sont produits à partir de plasmides linéarisés exprimant les
cDNAs
correspondants. Les ARNs de la forme longue sont marqués à l'aide de cyanine
3, les ARNs de la forme courte sont marqués à l'aide de cyanine 5.
Figure 4 : Démonstration de la spécificité d'hybridation des oligonucléotides.
Cinq oligonucléotides ont été utilisés pour différencier les formes longues
des
formes courtes mélangées à quantités égales. Deux exemples sont présentés,
Bene A et gene B.
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
Figure 5 : Mesures quantitatives de rapports entre formes longues et formes
courtes. Le pourcentage de forme longue (wt) a été établi à : 0, 20, 40, 60,
80
et 100 % (3 exemples sont présentés, gene A, B et C).
5 Figure 6: Spécificité du micro-array à oligonucléotides PSA et KLK2. Les
oligonucléotides spécifiques de PSA sont révélés par les isoformes de PSA
marquées à la cyanine 3. Les oligonucléotides spécifiques de KLK2 sont révélés
par les isoformes de KLK2 marquées à la cyanine 5.
10 Figure 7 : Schéma de l'amplification linéaire d'ARN.
Figure 8 : Exemple d'hybridation de la lame PSA / KLK2 à l'aide de sondes
provenant de tissus tumoraux et sains d'un méme patient.
15 Figure 9 : Mesure de l'expression différentielle de certaines isoformes de
PSA et
de KLK2 via l'analyse des oligonucléotides discriminants correspondants entre
tissus tumoraux et tissus sains au sein de mémes patients. Colonne 1 : nature
de l'isoforme, colonne 2 : oligonucléotide discriminant correspondant, colonne
3
à 6 : log2 (rapport expression tumorale l expression normale).
Figure 10 : Mesure de l'expression différentielle de certaines isoformes de
PSA
et de KLK2 via l'analyse des oligonucléotides discriminants correspondants
entre lignée cellulaire de cancer de la prostate (Mda-2b et LNCap) et lignée
cellulaire de cancer du sein (T47D). Colonne 1 : nature de l'isoforme, colonne
2: oligonucléotide discriminant correspondant, colonne 3,4: log2 (rapport
expression lignée prostate / expression lignée sein). Les isoformes
surexprimées de façon relative dans la lignée prostate sont indiquées en
orange. Les isoformes surexprimées de façon relative dans la lignée sein sont
indiquées en bleu.
Figure 11 : Mesure de l'expression différentielle de certaines isoformes de
PSA
et de KLK2 via l'analyse des oligonucléotides discriminants correspondants
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
26
entre différents tissus humains. Colonne 1 : nature de l'isoforme, colonne 2
oligonucléotide discriminant correspondant, colonne 3, 4, 5 et 6 : log2
(rapport
expression tissu prostate / tissus coeur, rein, petit intestin et prostate
respectivement). Les isoformes surexprimées de façon relative dans le tissu
prostate sont indiquées en orange. Les isoformes surexprimées de façon
relative dans les autres tissus sont indiquées en bleu.
Figure 12 : Illustration graphique des signaux de fluorescence obtenus pour
certaines isoformes avec des tissus normaux.
Figure 13: Amplifications par PCR à l'aide d'oligonucléotides amorces
spécifiques de trois isoformes de PSA . A)PSA-EHT003 B) PSA-EHT023 et C)
PSA-EHT012
Figure 14: Annotations de trois anticorps polyclonaux produits. Cette figure
regroupe les informations concernant les anticorps SE3962, SE3963, SE4101
produits, les épitopes choisis, les peptides synthétisés, le couplage à la KLH
utilisé et les isoformes susceptibles d'être reconnues par ces anticorps.
Figure 15 : Titres des trois anticorps par tests ELISA.
Détermination des titres de SE3962 en A), SE3963 en B) et SE4101 en C).
PPI : sérums préimmuns
PP : sérums issus de la première récolte
GP : sérums issus de la deuxième récolte
Figure 16 : Résultats de « western blots » avec l'anticorps EHT-SE3962 à
partir
de sérums à faible taux de PSA totale en A), à taux moyens de PSA totale en
B), à taux élevés de PSA total en C). Deux bandes correspondant à des poids
moléculaires attendus pour KLK2-EHT004 et KLK2-EHT006 sont observées. Le
déplacement des signaux avec des doses croissantes de l'épitope synthétique
spécifique (de 1 à 50 ~.g) non observé avec une forte dose de peptide non
spécifique à 250 ~,g démontre la spécificité de cet anticorps.
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
27
Figure 17 : Résultats de « western blots » avec l'anticorps EHT-SE3963 à
partir
de tissus prostatiques. Une bande correspondant à un poids moléculaire
attendu pour PSA-EHT021 est observée. Deux autres bandes de plus haut
poids moléculaires sont également révélées.
EXEMPLES
A - ISOLEMENT DES VARIANTS DE PSA ET DE KLK2
L'analyse qualitative différentielle a été effectuée à partir d'ARN poly
adénylés
(poly A+) extraits d'échantillons tumoraux et normaux prostatiques. Les ARN
poly A+ sont préparés selon des techniques connues de l'homme de métier. II
peut s'agir en particulier d'un traitement au moyen d'agents chaotropiques
tels
que le thiocyanate de guanidium suivi d'une extraction des ARN totaux au
moyen de solvants (phénol, chloroforme par exemple). De telles méthodes sont
bien connues de l'homme du métier (voir Maniatis et al., Chomczynsli et al.,
Anal. Biochem. 162 (1987) 156), et peuvent être aisément mises en oeuvre en
utilisant des kits disponibles dans le commerce. A partir de ces ARN totaux,
les
ARN poly A+ sont préparés selon des méthodes classiques connues de
l'homme de métier et décrites dans les kits commerciaux.
Ces ARN poly A+ servent de matrice à des réactions de transcription inverse à
l'aide de transcriptases inverses. Des transcriptases inverses dépourvues
d'activité RNase H sont avantageusement utilisées. Elles permettent d'obtenir
des brins d'ADN complémentaire de tailles supérieures à ceux obtenus à l'aide
de transcriptases inverses classiques. De telles préparations de
transcriptases
inverses sans activité RNase H sont disponibles commercialement.
Conformément à la technique DATAS, pour chaque point de la cinétique des
hybridations d'ARNm (C) avec des ADNc (T) et des hybridations réciproques
d'ARNm (T) avec des ADNc (C) sont réalisées.
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
28
Ces hétéroduplexes ARNm/ADNc sont ensuite purifiés selon les protocoles
prévus par la technique DATAS.
Les séquences d'ARN non appariées avec un ADN complémentaire sont
libérées de ces hétéroduplex sous l'action de la RNase H, cette enzyme
dégradant les séquences d'ARN appariées. Ces séquences non appariées
représentent les différences qualitatives qui existent entre des ARN par
ailleurs
homologues entre eux. Ces différences qualitatives peuvent être localisées
n'importe où sur la séquence des ARN, aussi bien en 5' ou en 3' qu'à
l'intérieur
de la séquence et notamment à l'intérieur de la séquence codante. Selon leur
localisation, ces séquences peuvent étre non seulement des modifications
d'épissage mais également des conséquences de translocations ou de
délétions.
Les séquences d'ARN représentant les différences qualitatives sont ensuite
clonées selon les techniques connues de l'homme de métier et notamment
celles décrites dans le brevet relatif à la technologie DATAS.
Ces séquences sont regroupées au sein de banques de ADNc qui constituent
des banques qualitatives différentielles. Une de ces banques contient les
exons
et les introns spécifiques de la situation saine ; les autres banques
contiennent
les évènements d'épissage caractéristiques des conditions pathologiques.
Les fragments issus des gènes humains de KLK2 et de KLK3 proviennent de
ces banques.
Quatre échantillons tumoraux ont été mélangés pour former un « pool » tumoral.
Ce pool d'ARN a été traité à la Dnase à l'aide du kit « DNA free » de la
société
Ambion (n° cat. 1906).
Cet ARN est ensuite reverse-transcrit à l'aide de la transcriptase inverse du
kit
« High capacity cDNA Archive » de la société Aplied Biosystems (n° cat.
4322171 ).
L'ADNc ainsi produit sert de matrice pour des réactions de PCR afin
d'amplifier
spécifiquement différentes régions d'ARNs messagers dérivés de la kallikreine-
2
et de la kallikreine-3 humaines selon le protocole suivant
Tampon Invitrogen 10X : 2pl
DNTPs 2mM: 2pL
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
29
MgCl2 50mM: 0.6pL
Primer amont 10pM: 0.4pL
Primer aval 10pM: 0.4pL
Taq polymerase : 0.2NL
H20: 13.4pL
cDNA 1 pL
Volume final : 19pL
Selon le programme de cycles suivant:
94C 3min
94C 30sec )
55C 1 min ) 35 cycles
72C 3min )
72C 6min
Les oligonucléotides utilisés comme amorces de PCR sont les suivants
Pour KLK2
163 KLK2-1-S GGTTCTCTCCATCGCCTTG
164 KLK2-1-AS CTCCTTTAGTCTGAAGCCTCACC
165 KLK2-2-S TGTATTTCACCACGACTATATCTCCC
166 KLK2-2-AS GCTCTAGCACACATGTCATTGGA
167 KLK2-3-S CAGTCATGGATGGGCACACT
168 KLK2-3-AS CTCAGACCCAGGCATCTGG
169 KLK2-4-S GCCAGATGGTGTAGCTGGG
170 KLK2-4-AS CATGATGTGATACCTTGAAGCACC
171 KLK2-5-S CCCTATCCAATTCTTTTGGGT
172 KLK2-5-AS GCTTTGATGCTTCAGAAGGC
173 KLK2-6-S CCTGCCAAGATCACAGATGTTG
174 KLK2-6-AS TGGTTAGCTTTCAGATTGCAGC
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
212 KLK2-7-S tgggaagaagaacaacgagca
213 KLK2-7-AS tttagggaatcagagaactggcc
214 KLK2-8-S agctcaatgtgtgtgcatgtgag
215 KLK2-8-AS aaaggatgcgggaagtcaga
5 216 KLK2-9-S cagcataattcacccattc
217 KLK2-9-AS tctacctgttcactgctgcttcc
218 KLK2-10-S ggagtgacgatgaggatgacc
219 KLK2-10-AS
gtcagttcagtgatcagaatgac
220 KLK2-11-AS
gctacagctgaaaccagcc
10 221 KLK2-12-S ccactacagagccctcactcca
222 KLK2-13-AS
aatgcttctcacactcccagc
249 klk2 startCCTGTGTCAGCATGTGGGACC
250 klk2 stop TGGGACAGGGGCACTCAGGG
251 klk2e spe cctgggggtatagttgccactat
Pour PSA
175 PSA-1-S CGTGACGTGGATTGGTGAGA
176 PSA-1-AS GCTGGCCTTAGAGGTTATCCTG
177 PSA-2-S GGCCTGAACTGTGTCTTCCC
178 PSA-2-AS GTGAACTTGCGCACACACG
179 PSA-3-S TGGCAGGTGCTTGTGGC
180 PSA-3-AS CTCCTCCCTCAGACCCAGG
181 PSA-4-S GTCCAGCCCACAACAGTG
182 PSA-4-AS CCTTGAAGCACACCATTACAGAC
183 PSA-5-S CCTAAATCCATCTCCTATCCGAGTC
184 PSA-5-AS CAGGATGAAACAGGCTGTGC
185 PSA-6-S TGCTGTGAAGGTCATGGACC
186 PSA-6-AS GACGCCTTGTTGGCTTCTAGAC
200 PSA-7-S tcccagagaccttgatgctt
201 PSA-7-AS gtttgcaggttggtggctg
202 PSA-8-S gtcccggttgtcttcctcac
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
31
203 PSA-8-AS gacccatttgttgtctcaggc
204 PSA-9-S ctgaacacacgcacgggat
205 PSA-9-AS ccaaagcccttccttttctca
206 PSA-10-S ttggaaacccacgccaaa
207 PSA-10-AS cctcagagtggctcagctgtag
208 PSA-11-S tgactccctcaaggcaataggtta
209 PSA-11-AS tgtttgctcactcccaccttct
210 PSA-12-S tgctggacagaagcaggaca
211 PSA-12-AS atcatcactccctccacatcc
247 PSA start GGAGAGCTGTGTCACCATGTGG
248 PSA stop ATAGGGGTGCTCAGGGGTTGG
Les produits amplifiés sont ensuite clonés dans le système « Topo » de la
société Invitrogen (n° cat. K4600) selon le protocole fourni. Les
produits de
ligation sont transformés dans des cellules « Top 10 » compétentes. Les
colonies sont identifiées sur milieu agar/LB supplémenté d'ampicilline.
Les ADNc présents dans ces colonies sont amplifiés de façon individuelle par
amplification PCR à l'aide d'amorces Sp6 et T7 selon le protocole suivant
Primer T7 1 OpM : 2pL
Primer Sp6 10pM 2pL
MgCl2 50mM: 1.2pL
DNTPs 2mM: 4pL
Tampon 10x 4pL
Taq polymerase: 0.2pL
H20: 25.6pL
Colonie : 1 pL
vol final 40pL
selon le programme de cycles suivant:
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
32
94°C 5min
94°C 30sec)
55°C 30sec) 30cycles
72°C 1 min)
72°C 5min
Les produits d'amplification sont ensuite purifiés par P100 pour ëtre
séquencés
à l'aide du kit « Big Dye Terminator » de la société Applied Biosystems selon
le
protocole fourni par ce fournisseur. Les réactions de séquence sont analysées
à
l'aide d'un séquenceur 3100 d'Applied Biosystems. Le tableau 2 représente les
différents ADNc ainsi que les couples d'oligonucléotides amorces utilisés pour
leur obtention et permettant leur amplification dans un échantillon.
B - IDENTIFICATION ET DESCRIPTION DES VARIANTS
Variants de KLK2
La numérotation des nucléotides se réfère au numéro d'accession de Genbank
M18157 sauf autre précision. La protéine de référence est la KLK2 munie de
son peptide signal.
Les séquences KLK2-EHT002 à KLK2-EHT011 (SEQ ID NO: 1 à 7)
correspondent à des séquences comportant une phase ouverte de lecture avec
un codon d'initiation et de terminaison de la traduction.
Les séquences KLK2-EHTb à KLK2-EHTI (SEQ ID NO : 8 à 15 ) correspondent
à des séquences exprimées de type « ESTs » pouvant comporter une, deux ou
trois phases de lecture avec ou non un codon d'initiation ou de terminaison de
la
traduction.
KLK2-EHT002 (SEQ ID NO : 1 )
Cette isoforme présente i) une rétention partielle d'une partie 5' de l'intron
2 (nt
1935-2020) et ü) une utilisation de deux sites cryptiques d'épissage dans la
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
33
partie 3' de l'exon 3 (nt 3728) et la partie 5' de l'exon4 (nt 3937). Ces deux
événements correspondent à des sites d'épissage consensuels. Cette isoforme
KLK2-EHT002 possède un codon stop après l'exon 2 et code ainsi pour une
protéine tronquée après le résidu n° 69 (KLK2-EHT002prota / SEQ ID NO :
50 ).
54 acides aminés peuvent être clivés pour former la séquence KLK2-
EHT002protb / SEQ ID N0:51 . On peut remarquer que les nucléotides
correspondant aux positions Genbank (M18157) 1821 et 3581 dans SEQ ID
NO :1 correspondent à C et A alors que la référence Genbank indique T et G
respectivement. Ces différences peuvent s'expliquer par l'existence d'un
polymorphisme à ces positions, par des erreurs dans la séquence référencée,
bien que l'on ne peut exclure des mutations introduites par la polymerase. Ces
deux changements n'affectent pas la séquence protéique traduite.
KLK2-EHT003 (SEQ ID NO : 2)
Cette isoforme présente i) une délétion totale de l'exon 2 et ü) une rétention
d'une partie 5' de l'intron 4 (nt 4061-4097). Ces deux événements
correspondent à des sites d'épissage consensuels. Cette isoforme KLK2-
EHT003 code pour une protéine comprenant 34 acides aminés supplémentaires
au-delà du résidu thréonine numéro 15 (KLK2-EHT003prota / SEQ ID NO : 52 ).
Ces 34 acides aminés peuvent être clivés pour former la séquence KLK2-
EHT003protb / SEQ ID NO: 53 . On peut remarquer que les nucléotides
correspondant aux positions Genbank (M18157) 3774 et 5486 dans SEQ ID
NO :2 correspondent à C et T alors que la référence Genbank indique T et G
respectivement. Ces différences peuvent s'expliquer par l'existence d'un
polymorphisme à ces positions, par des erreurs dans la séquence référencée,
bien que l'on ne peut exclure des mutations introduites par la polymerase. Ces
deux changements n'affectent pas la séquence protéique traduite.
KLK2-EHT004 (SEQ ID NO : 3)
Cette isoforme présente une délétion totale de l'exon 3. Cette isoforme KLK2-
EHT004 code pour une protéine comprenant 70 acides aminés supplémentaires
au-delà du résidu thréonine numéro 15 (KLK2-EHT004prota / SEQ ID NO : 54 ).
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
34
Ces 70 acides aminés peuvent étre clivés pour former la séquence KLK2-
EHT003protb l SEQ ID NO : 55. Les 16 derniers acides aminés sont nouveaux
et pourraient présenter un ou des épitopes spécifiques de cette isoforme, KLK2-
EHT004protc / SEQ ID NO: 56 . On peut remarquer que le nucléotide
correspondant à la position Genbank (M18157) 4097 dans SEQ ID N0:3
correspont à A alors que la référence Genbank indique G. Cette différence peut
s'expliquer par l'existence d'un polymorphisme à cette position, par des
erreurs
dans la séquence référencée, bien que l'on ne peut exclure des mutations
introduites par la polymerase. Ce changement n'affecte pas la séquence
protéique traduite.
KLK2-EHT006 (SEQ ID NO :4 )
Cette isoforme présente une utilisation de deux sites cryptiques d'épissage
dans
la partie 3' de l'exon 3 (nt 3728) et la partie 5' de l'exon4 (nt 3937). Cet
événement correspond à des sites d'épissage consensuels. Cette isoforme
KLK2-EHT006 code pour une protéine de 149 acides aminés (KLK2-
EHT006prota / SEQ ID NO : 57 ). 134 acides aminés peuvent être clivés pour
former la séquence KLK2-EHT006protb / SEQ ID NO 58. Les 12 derniers
acides aminés sont nouveaux et pourraient présenter un ou des épitopes
spécifiques de cette isoforme, KLK2-EHT006protc / SEQ ID NO : 59 . On peut
remarquer que le nucléotide correspondant à la position Genbank (M18157)
3689 dans SEQ ID NO :4 correspond à T alors que la référence Genbank
indique C. Cette différence peut s'expliquer par l'existence d'un
polymorphisme
à cette position, par des erreurs dans la séquence référencée, bien que l'on
ne
peut exclure des mutations introduites par la polymerase. Ce changement
n'affecte pas la séquence protéique traduite.
KLK2-EHT007 (SEQ ID NO : 5)
KLK2-EHT007 présente une rétention de la partie 5' de l'intron 4. Cette
isoforme KLK2-EHT007 code pour une protéine de 224 acides aminés (KLK2-
EHT007prota / SEQ ID NO : 60). 209 acides aminés peuvent être clivés pour
former la séquence KLK2-EHT007protb / SEQ ID NO : 61 . Les 14 derniers
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
acides aminés sont nouveaux et pourraient présenter un ou des épitopes
spécifiques de cette isoforme, KLK2-EHT007protc / SEQ ID NO : 62
KLK2-EHT009 (SEQ ID NO : 6)
5 KLK2-EHT009 présente i) une délétion d'une séquence dans l'exon 3 (nt 3671-
3793) et ü) l'utilisation d'un site cryptique d'épissage dans la partie 5' de
l'exon 4
(nt 3937) (site d'épissage consensuel). Cette isoforme KLK2-EHT009 code
pour une protéine de 123 acides aminés (KLK2-EHT009prota / SEQ ID NO : 63
). 108 acides aminés peuvent étre clivés pour former la séquence KLK2-
10 EHT009protb / SEQ ID NO : 64 . Les 5 derniers acides aminés sont nouveaux
et pourraient participer à un ou des épitopes spécifiques de cette isoforme,
KLK2-EHT009protc / SEQ ID NO : 65 .
KLK2-EHT011 (SEQ ID NO : 7)
15 Cette isoforme présente une utilisation d'un site cryptique d'épissage dans
la
partie 5' de l'exon4 (nt 4041 ). Cet événement correspond à des sites
d'épissage
consensuels. Cette isoforme KLK2-EHT011 code pour une protéine de 165
acides aminés (KLK2-EHT011 prota / SEQ ID NO : 66 ). 150 acides aminés
peuvent étre clivés pour former la séquence KLK2-EHT011 protb / SEQ ID NO
20 67 . Le dernier acide aminé remplace un résidu phenylalanine en résidu
tryptophane et pourrait participer à un épitope spécifique de cette isoforme.
KLK2-EHTb (SEQ ID NO : 8)
Cette isoforme présente une rétention d'une partie 5' de l'intron 1 suivi
d'une
25 délétion comprise entre les positions 701 et 1058 inclues. Cette isoforme
KLK2
EHTb code pour une protéine comprenant 104 acides aminés supplémentaires
au-delà du résidu thréonine numéro 15 (KLK2-EHTb1, SEQ ID NO :68). Ces
104 acides aminés peuvent être clivés pour former la séquence KLK2-EHTb2,
SEQ ID NO :69. Les 59 derniers acides aminés (KLK2-EHTb3, SEQ ID NO :70)
30 représentent une nouvelle séquence par rapport à une isoforme déjà décrite,
K-
LM (David et.al, (2002)). On peut remarquer que les nucléotides en position
97,
214, 249 de SEQ ID NO :8 correspondent à G, C, T alors que la référence
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
36
genbank indique C, T, C respectivement. Ces différences peuvent s'expliquer
par l'existence d'un polymorphisme à ces positions, par des erreurs dans la
séquence référencée, bien que l'on ne peut exclure des mutations introduites
par la polymerase. Les mutation 97, 214 n'affectent pas la séquence protéique
traduite. La mutation 249 transforme un résidu sérine en résidu phenylalanine.
On peut également remarquer que les nucléotides 1192-1199, GAAGAACA de
la référence genbank sont remplacés par les nucléotides 303-306, AAAC dans
SEQ ID NO :8. Les quinze derniers acides aminés de KLK2-EHTb1 remplacent
ainsi une séquence ouverte comprenant les 17 acides aminés composant
KLK2-EHTb4, SEQ ID NO :71.
KLK2-EHTc (SEQ ID NO : 9)
Cette isoforme utilise un site cryptique dans l'intron 1 qui occupe la
position
1157. Cette isoforme KLK2-EHTc code pour une protéine comprenant en outre
6 acides aminés au-delà du résidu thréonine numéro 15 (KLK2-EHTc1, SEQ ID
NO :72). Ces 6 acides aminés peuvent être clivés pour former la séquence
KLK2-EHTc2, SEQ ID NO :73. On peut remarquer que les nucléotides 1192-
1199, GAAGAACA de la référence genbank sont remplacés par les nucléotides
71-74, AAAC dans SEQ ID NO :9. Ce changement intervient après un codon
stop.
KLK2-EHTd (SEQ ID NO : 10)
Cette isoforme présente une rétention d'une partie 5' de l'intron 1 suivi
d'une
délétion comprise entre les positions 657 et 1209 inclues. Cette isoforme KLK2-
EHTd code pour une protéine comprenant au moins 41 acides aminés
supplémentaires au-delà du résidu thréonine numéro 15 (KLK2-EHTd1, SEQ ID
NO :74). Ces 41 acides aminés supplémentaires peuvent étre clivés pour former
la séquence KLK2-EHTd2, SEQ ID N0:75. Les 11 derniers acides aminés
supplémentaires (KLK2-EHTd3, SEQ ID N0:76) représentent une nouvelle
séquence par rapport à une isoforme déjà décrite, K-LM (David et.al, 2002). ).
La séquence prédite par continuation de la traduction dans l'intron 1 produit
une
protéine de 83 acides aminés après clivage : KLK2-EHTd4, SEQ ID NO :77.
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
37
KLK2-EHTe (SEQ ID NO : 11 )
KLK2-EHTe présente une séquence inconnue de 140 nucléotides comprise
entre un exon 2 tronqué en 3' et l'exon 3. Cette isoforme KLK2-EHTe code pour
une protéine comprenant en outre 19 acides aminés au-delà du résidu glycine
occupant la position numéro 52 (KLK2-EHTe1, SEQ ID NO :78). Ces 19 acides
aminés représentent la séquence KLK2-EHTe2, SEQ ID NO :79.
KLK2-EHTf (SEQ ID NO : 12)
Cette isoforme utilise deux sites cryptiques, le premier dans la partie 3' de
l'exon
2 (position 1876), le second dans l'intron 2 (position 3349). Cette isoforme
KLK2-EHTf code pour une protéine comprenant en outre 57 acides aminés
compris entre les résidus histidine en position 49 et asparagine en position
70
(KLK2-EHTf1, SEQ ID NO :80). Ces 57 acides aminés représentent la séquence
KLK2-EHTf2, SEQ ID NO :81. On peut remarquer que le nucléotide en position
269 de SEQ ID NO :12 correspond à C, alors que la référence genbank indique
T. Cette différence peut s'expliquer par l'existence d'un polymorphisme à
cette
position , par une erreur dans la séquence référencée, bien que l'on ne peut
exclure une mutation introduite par la polymerase. La mutation 269 transforme
un résidu phenylalanine en résidu leucine.
KLK2-EHTj (SEQ ID NO :13)
Cette isoforme présente une délétion dans l'intron 2 entre les positions 2473
et
3001. KLK2-EHTj code pour une protéine comprenant l'une des deux phases de
lectures correspondant à la KLK2-EHTj1 (SEQ ID N0:82), ou KLK2-EHTj2
(SEQ ID NO :83).
KLK2-EHTk (SEQ ID NO : 14)
Cette isoforme utilise deux sites cryptiques, le premier situé dans l'intron 4
en
position 5049 et le deuxième dans l'exon 5 en position 5469. KLK2-EHTk code
pour une protéine comprenant l'une des deux phases de lecture correspondant
à KLK2-EHTk1 (SEQ ID NO :84) ou KLK2-EHTk2 (SEQ ID NO :85).
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
38
KLK2-EHTI (SEQ ID NO :15)
Cette isoforme utilise un site cryptique dans l'intron 2 en position 2991.
Cette
isoforme code pour une protéine comprenant l'une des trois phases de lecture
correspondant à KLK2-EHTI1 (SEQ ID NO :86), KLK2-EHTI2 (SEQ ID NO :87)
ou KLK2-EHTI3 (SEQ ID NO :88).
Variants de PSA (ou KLK3)
La numérotation des nucléotides se réfère au numéro d'accession de Genbank
M27274 sauf autre précision. La protéine de référence est la PSA munie de son
peptide signal.
Les séquences PSA-EHT001 à PSA-EHT027 (SEQ ID NO: 16 à 34 )
correspondent à des séquences comportant une phase ouverte de lecture avec
un codon d'initiation et de terminaison de la traduction.
Les séquences PSA-EHTa à PSA-EHTu (SEQ ID NO : 35 à 49) correspondent
à des séquences exprimées de type « ESTs » pouvant comporter une, deux ou
trois phases de lecture avec ou non un codon d'initiation ou de terminaison de
la
traduction.
PSA-EHT001 (SEQ ID NO :16)
Cette isoforme présente une rétention d'un fragment délété de l'intron 1 (nt
721
811 puis 971-1272). Cette isoforme PSA-EHT001 code pour une protéine de 51
acides aminés (PSA-EHT001 prota / SEQ ID NO : 89 ). 36 acides aminés
peuvent être clivés pour former la séquence PSA-EHT001 protb / SEQ ID NO
90 . On peut remarquer que le nucléotide correspondant à la position Genbank
(M27274) 738 dans SEQ ID N0:16 correspond à G alors que la référence
Genbank indique T. Cette différence peut s'expliquer par l'existence d'un
polymorphisme à cette position, par des erreurs dans la séquence référencée,
bien que l'on ne peut exclure des mutations introduites par la polymerase. Ce
changement remplace un résidu tryptophane par un résidu glycine.
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
39
PSA-EHT003 (SEQ ID NO :17)
Cette isoforme présente une rétention d'un fragment délété de l'intron 1 (nt
721-
874 puis 920-1272). Cette isoforme PSA-EHT003 code pour une protéine de 89
acides aminés (PSA-EHT003prota / SEQ ID NO : 91 ). 74 acides aminés
peuvent ëtre clivés pour former la séquence PSA-EHT003protb / SEQ ID NO
92 . Les 20 derniers acides (PSA-EHT003protc / SEQ ID NO : 93) représentent
une information nouvelle par rapport à une isoforme déjà décrite comprenant
une rétention totale de l'intron 1
PSA-EHT004 (SEQ ID NO : 18)
Cette isoforme utilise un site cryptique d'épisage 3' situé dans l'intron 1 en
position 1142 (site consensuel) Cette isoforme PSA-EHT004 code pour une
protéine de 47 acides aminés (PSA-EHT004prota / SEQ ID NO : 94 ). 32 acides
aminés peuvent étre clivés pour former la séquence PSA-EHT004protb / SEQ
IDN0:95 .
PSA-EHT005 (SEQ ID NO : 19)
Cette isoforme présente une rétention d'un fragment délété de l'intron 1 (nt
721-
792 puis 1149-1272). Cette isoforme PSA-EHT005 code pour une protéine de
68 acides aminés (PSA-EHT005prota / SEQ ID NO : 96). 53 acides aminés
peuvent ëtre clivés pour former la séquence PSA-EHT005protb / SEQ ID NO
97. Les 28 derniers acides (PSA-EHT005protc / SEQ ID NO : 98 ) représentent
une information nouvelle par rapport à une isoforme déjà décrite comprenant
une rétention totale de l'intron 1
PSA-EHT007 (SEQ ID NO : 20)
Cette isoforme utilise un site cryptique d'épisage 5' situé dans l'exon 1 en
position 693 et un site cryptique 3' situé dans l'intron 1 en position 1149
Cette
isoforme PSA-EHT007 code pour une protéine de 23 acides aminés (PSA-
EHT007prota / SEQ ID NO : 99 ).
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
PSA-EHT008 (SEQ ID NO :21 )
Cette isoforme utilise un site cryptique d'épisage 3' situé dans l'intron 1 en
position 1202 (site consensuel) Cette isoforme PSA-EHT008 code pour une
protéine de 27 acides aminés (PSA-EHT008prota / SEQ ID NO : 100 ). 12
5 acides aminés peuvent étre clivés pour former la séquence PSA-EHT008protb l
SEQ ID NO : 101 . On peut remarquer que le nucléotide correspondant à la
position Genbank (M27274) 679 dans SEQ ID NO :21 correspond à T alors que
la référence Genbank indique G. Cette différence peut s'expliquer par
l'existence d'un polymorphisme à cette position, par des erreurs dans la
10 séquence référencée, bien que l'on ne peut exclure des mutations
introduites
par la polymerase. Ce changement remplace un résidu tryptophane par un
résidu leucine.
PSA-EHT009 (SEQ ID NO : 22)
15 Cette isoforme présente une rétention d'un fragment délété de l'intron 2
(nt
2119-2447 puis 2988-3226). Cette isoforme PSA-EHT009 code pour une
protéine de 69 acides aminés (PSA-EHT009prota / SEQ ID NO: 102). 54
acides aminés peuvent être clivés pour former la séquence PSA-EHT009protb /
SEQ ID NO : 103 . On peut remarquer que le nucléotide correspondant à la
20 position Genbank (M27274) 1966 dans SEQ ID NO :22 correspond à A alors
que la référence Genbank indique G. Cette différence peut s'expliquer par
l'existence d'un polymorphisme à cette position, par des erreurs dans la
séquence référencée, bien que l'on ne peut exclure des mutations introduites
par la polymerase. Ce changement ne modifie pas la séquence protéique.
25 D'autres mutations ponctuelles sont identifiées après le codon stop.
PSA-EHT012 (SEQ ID NO : 23)
Cette isoforme utilise un site cryptique d'épisage 3' situé dans l'intron 2 en
position 2426 (site consensuel) Cette isoforme PSA-EHT012 code pour une
30 protéine de 83 acides aminés (PSA-EHT012prota / SEQ ID NO: 104). 68
acides aminés peuvent étre clivés pour former la séquence PSA-EHT004protb /
SEQ ID NO : 105. Les 14 derniers acides aminés (PSA-EHT012protc l SEQ ID
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
41
NO : 106) représentent une information nouvelle par rapport à PSA de type
sauvage et sont ainsi susceptibles d'inclure un ou des épitoges spécifiques de
cette isoforme. On peut remarquer que les nucléotides correspondant aux
positions Genbank (M27274) 1966 et 2459 dans SEQ ID NO :23 correspondent
à A et A alors que la référence Genbank indique G et G respectivement. Ces
différences peuvent s'expliquer par l'existence d'un polymorphisme à ces
positions, par des erreurs dans la séquence référencée, bien que l'on ne peut
exclure des mutations introduites par la polymerase. Ces deux changements
n'affectent pas la séquence protéique traduite.
PSA-EHT013 (SEQ ID NO : 24)
Cette isoforme utilise un site cryptique d'épisage 3' situé dans l'intron 1 en
position 1945 (site consensuel) Cette isoforme PSA-EHT013 code pour une
protéine de 75 acides aminés (PSA-EHT013prota / SEQ ID NO: 107). 60
acides aminés peuvent étre clivés pour former la séquence PSA-EHT013protb /
SEQ ID NO : 103 . Ces 60 acides aminés représentent une information nouvelle
par rapport à PSA de type sauvage ef sont ainsi susceptibles d'inclure un ou
des épitoges spécifiques de cette isoforme.
PSA-EHT015 (SEQ ID NO :25)
Cette isoforme utilise un site cryptique d'épisage 5' situé dans l'exon 1 en
position 703 et un site cryptique 3' situé dans l'exon 2 en position 2030
Cette
isoforme PSA-EHT015 code pour une protéine de 41 acides aminés (PSA-
EHT015prota / SEQ ID NO: 109). Les 30 derniers acides aminés (PSA-
EHT015protb / SEQ ID NO : 110) représentent une information nouvelle par
rapport à PSA de type sauvage et sont ainsi susceptibles d'inclure un ou des
épitoges spécifiques de cette isoforme. On peut remarquer que le nucléotide
correspondant à la position Genbank (M27274) 2094 dans SEQ ID N0:25
correspont à C alors que la référence Genbank indique T. Cette différence peut
s'expliquer par l'existence d'un polymorphisme à cette position, par des
erreurs
dans la séquence référencée, bien que l'on ne peut exclure des mutations
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
42
introduites par la polymerase. Ce changement remplace un résidu sérine par
un résidu proline.
PSA-EHT016 (SEQ ID NO : 26)
Cette isoforme utilise un site cryptique d'épisage 3' situé dans l'exon 2 en
position 2053 (site consensuel) Cette isoforme PSA-EHT016 code pour une
protéine de 39 acides aminés (PSA-EHT016prota / SEQ ID NO : 111 ). 24
acides aminés peuvent être clivés pour former la séquence PSA-EHT016protb /
SEQ ID NO : 112 . Ces 24 acides aminés représentent une information nouvelle
par rapport à PSA de type sauvage et sont ainsi susceptibles d'inclure un ou
des épitopes spécifiques de cette isoforme.
PSA-EHT018 (SEQ ID NO : 27)
Cette isoforme présente une rétention d'un fragment délété de l'intron 2 (nt
2119-2588 puis 3114-3226). Cette isoforme PSA-EHT018 code pour une
protéine de 69 acides aminés (PSA-EHT018prota / SEQ ID NO: 113). 54
acides aminés peuvent être clivés pour former la séquence PSA-EHT018protb /
SEQ ID NO : 114 . On peut remarquer que le nucléotide correspondant à la
position Genbank (M27274) 2545 dans SEQ ID NO :27 correspond à T alors
que la référence Genbank indique A. Cette différence peut s'expliquer par
l'existence d'un polymorphisme à cette position, par des erreurs dans la
séquence référencée, bien que l'on ne peut exclure des mutations introduites
par la polymerase. Ce changement ne modifie pas la séquence protéique.
PSA-EHT019 (SEQ ID NO : 28)
Cette isoforme présente une délétion d'un fragment situé dans l'exon 3
(nucléotide 3828-3933). Cette isoforme PSA-EHT019 code pour une protéine de
100 acides aminés (PSA-EHT019prota / SEQ ID NO : 115). 85 acides aminés
peuvent être clivés pour former la séquence PSA-EHT019protb / SEQ ID NO
116. Les 6 derniers acides aminés (PSA-EHT019protc / SEQ ID NO : 117)
représentent une information nouvelle par rapport à PSA de type sauvage et
sont ainsi susceptibles d'inclure un ou des épitopes spécifiques de cette
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
43
isoforme. On peut remarquer que les nucléotides correspondant aux positions
Genbank (M27274) 3786 et 3943 dans SEQ ID NO :28 correspondent à T et à A
alors que la référence Genbank indiquent C et C respectivement. Ces
différences peuvent s'expliquer par l'existence de polymorphismes à ces
positions, par des erreurs dans la séquence référencée, bien que l'on ne peut
exclure des mutations introduites par la polymerase. Le premier changement
ne modifie pas la séquence protéique. Le deuxième remplace un résidu sérine
par un résidu arginine.
PSA-EHT021 (SEQ ID NO : 29)
Cette isoforme utilise un site cryptique d'épisage 3' situé dans l'exon 3 en
position 3885 (site consensuel) et présente également une délétion dans la
partie 3' de l'exon 3 (nucléotides 3903-4025). Cette isoforme PSA-EHT021 code
donc pour une protéine de 177 acides aminés (PSA-EHT021 prota / SEQ ID
NO : 118). 162 acides aminés peuvent être clivés pour former la séquence
PSA-EHT021 protb / SEQ ID NO : 119 . Les nouvelles jonctions créées autour
des résidus 69 et 76 représentent une information nouvelle par rapport à PSA
de type sauvage et sont ainsi susceptibles d'inclure un ou des épitopes
spécifiques de cette isoforme. On peut remarquer que le nucléotide
correspondant à la position Genbank (M27274) 1966 dans SEQ ID NO :29
correspond à A alors que la référence Genbank indique G. Cette différence peut
s'expliquer par l'existence d'un polymorphisme à cette position, par des
erreurs
dans la séquence référencée, bien que l'on ne peut exclure des mutations
introduites par la polymerase. Ce changement ne modifie pas la séquence
protéique.
PSA-EHT022 (SEQ ID NO : 30)
Cette isoforme présente une délétion dans la partie 3' de l'exon 3
(nucléotides
3903-4025). Cette isoforme PSA-EHT022 code donc pour une protéine de 220
acides aminés (PSA-EHT022prota / SEQ ID NO : 120). 205 acides aminés
peuvent étre clivés pour former la séquence PSA-EHT022protb / SEQ ID NO
121 . La nouvelle jonction créée autour du résidu 119 représente une
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
44
information nouvelle par rapport à PSA de type sauvage et est ainsi
susceptible
d'inclure un ou des épitopes spécifiques de cette isoforme. On peut remarquer
que le nucléotide correspondant à la position Genbank (M27274) 1966 dans
SEQ ID NO :30 correspond à A alors que la référence Genbank indique G.
Cette différence peut s'expliquer par l'existence d'un polymorphisme à cette
position, par des erreurs dans la séquence référencée, bien que l'on ne peut
exclure des mutations introduites par la polymerase. Ce changement ne
modifie pas la séquence protéique.
PSA-EHT022 (SEQ ID NO : 30) correspond à un variant de PSA soumis à
Genbank le 24 Octobre 2002 (numéro d'accés : AJ459782).
PSA-EHT023 (SEQ ID NO :31 )
Cette isoforme comprend une délétion d'un fragment de l'exon 2 (nucléotides
1990-2040), l'utilisation d'un site cryptique en 3' de l'exon 3 en position
3885
(site consensuel) et une rétention d'un fragment 5' de l'intron 3 (nucléotides
4043-4060) (site consensuel). Cette isoforme code pour une protéine de 207
acides aminés (PSA-EHT023prota /SEQ ID NO: 122). 192 acides aminés
peuvent être clivés pour former la séquence PSA-EHT023protb / SEQ ID NO
123. Les nouvelles jonctions créées autour des résidus 27, 53 et dans la
région
105-111 représentent une information nouvelle par rapport à PSA de type
sauvage et sont ainsi susceptibles d'inclure un ou des épitopes spécifiques de
cette isoforme. On peut remarquer que les nucléotides correspondant aux
positions Genbank (M27274) 2060 et 5731 dans SEQ ID NO :31 correspondent
à G et à G alors que la référence Genbank indiquent T et T respectivement. Ces
différences peuvent s'expliquer par l'existence de polymorphismes à ces
positions, par des erreurs dans la séquence référencée, bien que l'on ne peut
exclure des mutations introduites par la polymerase. Le premier changement
remplace un résidu cystéine par un résidu glycine. Le deuxième ne modifie pas
la séquence protéique..
PSA-EHT025 (SEQ ID NO : 32)
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
Cette isoforme correspond à la délétion de l'exon 3. Cette isoforme code pour
une protéine de 85 acides aminés (PSA-EHT025prota / SEQ ID NO : 124). 70
acides aminés peuvent être clivés pour former la séquence PSA-EHT025protb /
SEQ ID NO : 125. Les 16 derniers acides aminés (PSA-EHT025protc / SEQ ID
5 NO : 126) représentent une information nouvelle par rapport à PSA de type
sauvage et sont ainsi susceptibles d'inclure un ou des épitopes spécifiques de
cette isoforme. On peut remarquer que les nucléotides correspondant aux
positions Genbank (M27274) 2118-4186 et 5791 dans SEQ ID N0:32
correspondent à G et à G alors que la référence Genbank indiquent AT et C
10 respectivement. Ces différences peuvent s'expliquer par l'existence de
polymorphismes à ces positions, par des erreurs dans la séquence référencée,
bien que l'on ne peut exclure des mutations introduites par la polymerase. La
concordance des sites 3' de l'exon 2 et 5' de l'exon 4 suggère une mutation
introduite par la polymerase dans cette région. Le dernier changement ne
15 modifie pas la séquence protéique.
PSA-EHT026 (SEQ ID NO : 33)
Cette isoforme présente une délétion d'un fragment situé dans l'exon 3
(nucléotide 3781-4025). Cette isoforme PSA-EHT026 code pour une protéine de
20 78 acides aminés (PSA-EHT026prota / SEQ ID NO : 127). 63 acides aminés
peuvent étre clivés pour former la séquence PSA-EHT026protb / SEQ ID NO
128 .
PSA-EHT027 (SEQ ID NO : 34)
25 Cette isoforme utilise un site cryptique d'épissage situé en 5' dans l'exon
3 en
position 3780 et une délétion de l'exon 4 Cette isoforme PSA-EHT027 code
pour une protéine de 144 acides aminés (PSA-EHT027prota / SEQ ID NO : 129
). 129 acides aminés peuvent être clivés pour former la séquence PSA-
EHT027protb / SEQ ID NO: 130 . Les 67 derniers acides aminés (PSA-
30 EHT027protc / SEQ ID NO : 131 ) représentent une information nouvelle par
rapport à PSA de type sauvage et sont ainsi susceptibles d'inclure un ou des
épitopes spécifiques de cette isoforme. On peut remarquer que le nucléotide
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
46
correspondant à la position Genbank (M27274) 1966 dans SEQ ID N0:34
correspond à A alors que la référence Genbank indique G. Cette différence peut
s'expliquer par l'existence d'un polymorphisme à cette position, par des
erreurs
dans la séquence référencée, bien que l'on ne peut exclure des mutations
introduites par la polymerase. Ce changement ne modifie pas la séquence
protéique.
PSA-EHTa (SEQ ID NO :35)
Cette isoforme présente une rétention de 91 nucléotides de la partie 5' de
l'intron 1 suivie d'une délétion des 152 nucléotides suivants (pour retrouver
ensuite l'intron 1 ). Cette isoforme PSA-EHTa code pour une protéine de 90
acides aminés (PSA-EHTa1, SEQ ID N0:132) dont les 75 derniers acides
aminés peuvent être clivés (PSA-EHTa2, SEQ ID NO :133), représentant une
information différente par rapport à PSA et dont les 44 derniers acides aminés
(PSA-EHTa3, SEQ ID N0:134) représentent une information nouvelle par
rapport à une rétention totale de l'intron 1 déjà décrite (David et.al,
(2002)). Q
remplace P en position 26 des 74 derniers acides aminés. On peut remarquer
que les nucléotides en position 90 et 234 de SEQ ID NO :35 correspondent à A,
C, alors que la référence genbank indique C et T. D'autre part, les
nucléotides
G et C en position 243 et 293 différent également par rapport à la référence
genbank . Cependant, ces deux nucléotides correspondent effectivement à une
séquence génomique _publiée ( numéro d'accés Genbank : NT 011190 ). Ces
différences peuvent s'expliquer par l'existence d'un polymorphisme à ces
positions, par des erreurs dans la séquence référencée , bien que l'on ne peut
exclure des mutations introduite par la polymerase. Ainsi, un résidu glutamine
a
remplacé le résidu proline (mutation 90), et un résidu thréonine a remplacé un
résidu isoleucine (mutation 234).
PSA-EHTd (SEQ ID NO : 36)
Cette isoforme présente une délétion des 9 derniers nucléotides de l'exon 2 et
des 243 premiers nucléotides de l'exon 3. Cette isoforme PSA-EHTd code pour
une protéine présentant une délétion de 84 acides aminés (PSA-EHTd1, SEQ
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
47
ID NO :135). Un nouveau domaine est crée entre le résidu cystéine 66 et le
résidu thréonine 151.
PSA-EHTf (SEQ ID NO : 37)
Cette isoforme présente une rétention de l'intron 3 délété de 105 nucléotides
(2420-2526). Cette isoforme PSA-EHTf code pour une protéine tronquée après
le résidu asparagine numéro 69 lequel est substitué par résidu lysine (PSA-
EHTf1, SEQ ID NO :136). On peut remarquer que le nucléotide en position 56
de SEQ ID NO :37 correspond à G, alors que la référence genbank indique A.
Cette différence peut s'expliquer par l'existence d'un polymorphisme à cette
position, par une erreur dans la séquence référencée, bien que l'on ne peut
exclure une mutation introduite par la polymerase. Cette mutation remplace un
résidu histidine en résidu arginine.
PSA-EHTh (SEQ ID NO : 38)
Cette isoforme résulte de l'utilisation d'un site cryptique d'épissage au sein
de
l'intron 4 (en position 5472). Cette isoforme PSA-EHTh code pour une protéine
comprenant l'une des deux phases de lecture correspondant à PSA-EHTh1
(SEQ ID NO :137) ou PSA-EHTh2 (SEQ ID NO :138). On peut remarquer que
les nucléotides en position 79, 199 et 258 de SEQ ID NO :38 correspondent à
C, C et G, alors que la référence genbank indique T, T et A. Ces différences
peuvent s'expliquer par l'existence d'un polymorphisme à ces positions, par
des
erreurs dans la séquence référencée, bien que l'on ne peut exclure une
mutation introduite par la polymerase.
PSA-EHTj (SEQ ID NO : 39)
Cette isoforme résulte de l'utilisation d'un site cryptique d'épissage au sein
de
l'intron 4 (en position 5257). Cette isoforme PSA-EHTj code pour une protéine
comprenant l'une des trois phases de lecture correspondant à PSA-EHTj1 (SEQ
ID NO :139), PSA-EHTj2 (SEQ ID NO :140) ou PSA-EHTj3 (SEQ ID NO :141 )
PSA-EHTk (SEQ ID NO : 40)
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
48
Cette isoforme présente une rétention de la partie 3' de l'intron 3, puis une
rétention d'un intron 4 tronqué (entre positions 4337 et 5516). Cette isoforme
code pour une protéine comprenant l'une des trois phases de lecture
correspondant à PSA-EHTk1 (SEQ ID N0:142), PSA-EHTk2 (SEQ ID
NO :143), ou PSA-EHTk3 (SEQ ID NO :144).
PSA-EHTI (SEQ ID NO : 41 )
Cette isoforme utilise un site cryptique dans l'exon 4 en position 4274 et un
autre site cryptique dans l'intron 4 en position 4538. On peut remarquer que
le
nucléotide en position 79 de SEQ ID NO :41 correspond à C, alors que la
référence genbank indique T. Cette différence peut s'expliquer par l'existence
d'un polymorphisme à cette position, par une erreur dans la séquence
référencée, bien que l'on ne peut exclure une mutation introduite par la
polymerase. PSA-EHTI code pour une proteine comprenant l'une des trois
phases de lecture correspondant à PSA-EHTI1 (SEQ ID NO :145), PSA-EHTI2
(SEQ ID NO :146) ou PSA-EHTI3 (SEQ ID NO :147). Dans PSA-EHTI3, cette
mutation remplace un résidu isoleucine en résidu threonine.
PSA-EHTm (SEQ ID NO : 42)
Cette isoforme présente une rétention d'un intron 1 tronqué (entre 1214 et
1755). PSA-EHTm code pour une protéine comprenant l'une des trois phases
de lecture correspondant à PSA-EHTm1 (SEQ ID NO :148), PSA-EHTm2 (SEQ
ID NO :149) ou PSA-EHTm3 (SEQ ID NO :150).
PSA-EHTn (SEQ ID NO : 43)
Cette isoforme présente une rétention d'un intron 1 tronqué (entre 1366 et
1736). PSA-EHTn code pour une protéine comprenant l'une des trois phases
de lecture correspondant à PSA-EHTn1 (SEQ ID NO :151 ), PSA-EHTn2 (SEQ
ID NO :152) ou PSA-EHTn3 (SEQ ID NO :153).
PSA-EHTp (SEQ ID NO : 44)
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
49
Cette isoforme résulte de l'utilisation d'un site cryptique d'épissage dans
l'intron
1 (en position 1240). PSA-EHTp peut coder pour une protéine comprenant 27
acides aminés supplémentaires au-delà du résidu isoleucine occupant la
position 15 (PSA-EHTp1, SEQ ID NO :154). Ces 27 acides aminés représentant
la séquence PSA-EHTp2 (SEQ ID NO :155) peuvent étre libérés après clivage.
PSA-EHTq (SEQ ID NO : 45)
Cette isoforme présente une rétention d'un intron 2 tronqué (entre positions
2740 et 3167). PSA-EHTq code pour une protéine comprenant l'une des deux
phases de lecture correspondant à PSA-EHTq1 (SEQ ID NO :156), ou PSA-
EHTq2 (SEQ ID NO :157).
PSA-EHTr (SEQ ID NO : 46)
Cette isoforme présente une rétention d'un intron 2 tronqué (entre positions
2589 et 3199). PSA-EHTr code pour une protéine comprenant l'une des trois
phases de lecture correspondant à PSA-EHTr1 (SEQ ID NO :158), PSA-EHTr2
(SEQ ID NO :159) ou PSA-EHTr3 (SEQ ID NO :160).
PSA-EHTs (SEQ ID NO :47 )
Cette isoforme présente une rétention d'un intron 4 tronqué (entre positions
4516 et 4889). On peut remarquer que les nucléotides en position 54, 93 et 201-
208 de SEQ ID NO :47 correspondent à C, A et TGCCGCTG, alors que la
référence genbank indique T, G et AG--GTGT. Ces différences peuvent
s'expliquer par l'existence d'un polymorphisme à ces positions, par des
erreurs
dans la séquence référencée, bien que l'on ne peut exclure une mutation
introduite par la polymerase. Cette isoforme code pour une protéine comprenant
l'une des deux phases de lecture correspondant à PSA-EHTs1 (SEQ ID
NO :161 ) ou PSA-EHTs2 (SEQ ID NO :162). La mutation en position 54
remplace dans PSA-EHTs1 un résidu leucine en résidu proline.
PSA-EHTt (SEQ ID NO : 48)
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
Cette isoforme présente une rétention d'un intron 4 tronqué (entre positions
4727 et 5111 ). On peut remarquer que les nucléotides en position 137 et 239
de
SEQ ID NO :48 correspondent à G et A, alors que la référence genbank indique
A et G. Ces différences peuvent s'expliquer par l'existence d'un polymorphisme
5 à ces positions, par des erreurs dans la séquence référencée, bien que l'on
ne
peut exclure une mutation introduite par la polymerase. Cette isoforme code
pour une protéine comprenant l'une des deux phases de lecture correspondant
à PSA-EHTt1 (SEQ ID NO :163) ou PSA-EHTt2 (SEQ ID NO :164).
10 PSA-EHTu (SEQ ID NO : 49)
Cette isoforme résulte de l'utilisation d'un site cryptique dans l'intron 4
(en
position 5056). On peut remarquer que le nucléotide en position 48 de SEQ ID
NO:49 correspond à T, alors que la référence genbank indique C. Cette
différence peut s'expliquer par l'existence d'un polymorphisme à cette
position,
15 par une erreur dans la séquence référencée, bien que l'on ne peut exclure
une
mutation introduite par la polymérase. PSA-EHTu code pour une protéine
comprenant l'une des trois phases de lecture correspondant à PSA-EHTu1
(SEQ ID N0:165), PSA-EHTu2 (SEQ ID N0:166) ou PSA-EHTu3 (SEQ ID
NO :167). La mutation 48 remplace le résidu alanine en résidu valine dans
20 PSA-EHTu2.
C - VALIDATION DE L'EXPRESSION DES ISOFORMES DE PSA ET DE KLK2
PAR L'UTILISATION D'UN MICRO-ARRAY COMPORTANT DES
25 OLIGONUCLEOTIDES DE JONCTION
L'expression des variants de PSA et de KLK2 décrits dans cette invention a été
établie à l'aide d'un micro-array comportant des oligonucléotides capables de
s'hybrider de façon spécifique avec ces variants. Sur la base de leurs
30 séquences, les variants d'épissage de PSA et klk2 proviennent d'évènements
de natures difFérentes (Figure 1 ).
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
51
~ « exon skipping » : l'oligonucléotide spécifique (e.g., discriminant) est
dessiné pour étre complémentaire de la séquence créée par la jonction
exon1-exon3
~ rétention d'introns : l'oligonucléotide spécifique est localisé dans la
séquence de l'intron.
~ dans le cas d'une utilisation alternative de sites d'épissage en 5' ou 3',
l'oligonucléotide discriminant est dessiné pour étre complémentaire de
(i.e., placé sur) l'une ou l'autre de ces nouvelles jonctions.
~ des oligonucléotides dans les exons et sur les jonctions des formes
sauvages de klk2 et PSA sont aussi générés.
C1 - Description du micro-array à oligonucléotides de jonction.
Cette étude a consisté à générer 149 oligonucléotides de 24 à 25 mers. Leurs
séquences sont jointes en documents annexe (Tableau 1 ). Les oligonucléotides
« discriminants », complémentaires des jonctions spécifiques créeés par les
variants décrits sont revendiqués (SEQ ID NO : 168 à SEQ ID NO : 220).
5 oligonucléotides ont été utilisés pour caractériser chaque évènement
d'épissage alternatif (voir figure 2). Un oligonucléotide est spécifique de
l'exon
éliminé et permet la quantification de la forme longue. Un second
oligonucléotide est spécifique de l'un des exons adjacents non impliqué dans
l'épissage et permet de quantifier les formes longues et courtes de l'ARN.
Enfin,
trois oligonucléotides sont spécifiques des jonctions ; parmi eux, l'un est
spécifique de la nouvelle séquence générée après épissage et permet de
quantifier la forme épissée. II est entendu que d'autres combinaisons
d'oligonucléotides peuvent être envisagées, notamment l'emploi d'un seul ou de
deux oligonucléotides seulement.
Concernant le design des oligonucléotides, étant donné que les sondes o sont
plus courtes que les sondes produits PCR classiquement utilisées, il est
nécessaire de vérifier que ces sondes n'hybrident pas de manière aspécifique
d'autres gènes que celui pour lequels elles ont été dessinées. De plus, il est
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
52
indispensable d'assurer que les oligonuclèotides ne présentent pas de
structures secondaires qui pourraient interférer avec leur capacité
d'hybridation.
D'un manière générale, il est préférable d'avoir sur la puce un profil
thermodynamique homogène pour l'ensemble des oligos générés, à savoir le
Tm (65°C) et la longueur (24-25 mers). Au cours de leur synthèse,
les
oligonucléotides sont par ailleurs modifiés en 5' par un groupement NHZ-C6
favorisant leur flexibilité et permettant d'établir une liaison covalente avec
le
polymère constituant le revêtement de la lame de verre.
Concernant plus précisément les oligonucléotides de jonction, ils sont
idéalement centrés sur les jonctions, mais nous avons considéré également les
possibilités d'oligonucléotides décalés sur la jonction.
En terme de logiciel de design d'oligonucléotides, nous avons sélectionné le
logiciel Primer Finder.
Les critères que nous avons sélectionnés sont les suivants
- %GC : 40% à 60% pour les 24-mers et les 30-mers, 30% à 60% pour les
40 mers.
- Concentrations en oligonucléotides : 50 nM
- Concentrations en sels : 50 mM
- Ignorer les oligonucléotides ayant des tendances à former des structures
secondaires en « épingles à cheveux » ou ayant des possibilités de
s'autodimériser.
Dans le but de valider notre technologie, nous avons dans un premier temps
travaillé avec des isoformes clonées (cf. figure 3). Les plasmides contenant
les
isoformes longues et courtes ont été linéarisés avant transcription in vitro.
Le
milieu réactionnel contient de l'aminoallyl-UTP qui interagit chimiquement
avec
le fluorochrome. Nous avons choisi de marquer les isoformes longues en Cy3 et
les isoformes courtes en CyS. L'ARN présentant de nombreuses structures
secondaires qui géneraient l'hybridation, il a été imaginé d'introduire dans
le
protocole une étape de fragmentation chimique. Après purification les
isoformes
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
53
ont été mélangées puis hybridées sur lame de verre (3D Link, Mororola ou
Codelink, Amersham).
La figure 4 représente les résultats obtenus sur deux des clones correspondant
à des gènes différents. Cette expérience a été réalisée dans le but vérifier
la
spécificité d'hybridation de nos oligonucléotides. Pour cela, nous avons
amplifié
de l'ARN de drosophile par transcription in vitro dans lequel nous avons
introduit
un exogène control d'Arabidopsis thaliana qui nous servira à calibrer le
scanner
au moment de la lecture des lames.
Les isoformes marquées (5ng par isoformes) sont ensuite diluées dans le cRNA
de drosophile qui crée un environnement complexe nous permettant de vérifier
la spécificité de l'hybridation sachant que l'ARN de drosophile ne contient
pas la
séquence permettant l'hybridation de la cible (analyse bioinformatique). Après
hybridation, la lame a été lue dans les 2 canaux, celui de la Cy3 et de la
CyS.
L'intensité de fluorescence est mesuré sur chaque spot et les valeurs sont
normalisées par le calcul de la médiane.
Chaque oligonucléotide est spotté en quadruplet. Les oligonucléotides
correspondant à l'exon 2, aux jonctions 1-2 et 2-3 ont été dessinés pour
n'hybrider que la forme longue, c'est pourquoi ils apparaissent en rouge sur
l'image générée par QuantArray. Les oligonucléotides spécifiques de la
jonction
1-3 sont supposés n'hybrider que les formes courtes, ils apparaissent
effectivement en vert. Puisqu'on a utilisé un mélange équimolaire d'isoformes
longues et courtes, la superposition des deux images donne des spots oranges.
(Des expériences similaires ont été réalisées pour déterminer la sensibilité
de
notre puce en diluant les isoformes jusque 26 pg).
De cette expérience nous pouvons voir que après normalisation 50% de
l'hybridation est due à la forme longue si l'on se base sur l'exon commun.
Entre
90% et 100% de l'hybridation est due à la forme longue sur l'exon 2, les
jonctions 1-2 et 2-3. Moins de 7% de l'hybridation est due à la forme longue
sur
la jonction 1-3.
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
54
Ces expériences ont permis de valider le design des oligos et la spécificité
des
hybridations. Ce fort degré de spécificité est crucial si l'on veut utiliser
cet outil
pour quantifier les isoformes.
Les résultats précédents ont été obtenus en utilisant un mélange équimolaire
d'isoformes longues et courtes. L'étape suivante a donc été de montrer que
l'outil est quantitatif (figure 5) ; pour cela nous avons fait varier dans nos
échantillons la quantité de formes longues de 0% à 100% par tranches de 20%
(axe des abscisses) en marquant les formes longues et les formes courtes avec
des fluorochromes différents.
Nous avons après normalisation des intensités de fluorescence, mesuré le % de
formes longues sur la base des valeurs obtenues sur l'exon commun que nous
avons reportées sur l'axe des ordonnées. Comme en témoigne le graphe, les
valeurs mesurées sont très proches des valeurs théoriques.
De l'ensemble de ces études, nous pouvons prévoir d'utiliser l'outil qu'est
l'oligoarray dans le but de définir qualitativement et quantitativement
l'expression d'exons épissés ou de rétention d'introns.
149 oligonucléotides de 24 et e 25 mers ont été conçus pour réaliser le micro-
array. Ces oligonucléotides ont été repris à une concentration de 25 uM dans
un
tampon Phosphate de Sodium 150 mM. Les oligonucléotides ont ensuite été
déposés sur lames de verre (Codelink , Amersham), puis ces lames sont
incubées dans une chambre saturante en NaCI pendant 16h. Les sites réactifs
non utilisés seront ensuite bloqués en utilisant une solution d'éthanolamine
50
mM, Tris 0.1 M, SDS 0.1 % à pH 9, elles sont ensuite lavées dans une solution
à
4xSSC/0.1 %SDS.
Les cibles sont hybridées dans le tampon 5X SSC, 0.1 % SDS, 0.1 mg/ml ADN
de sperme de saumon, à une température de 50°C pendant 16h. Elles sont
ensuite lavées en augmentant la stringence des bains
4X SSC pour éliminer le cover slip
2X SSC / 0.1 % SDS pendant 5min à 50°C
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
0.2X SSC pendant 5 minutes à température ambiante
0.1X SSC pendant 5 minutes à température ambiante
C2 - Détermination de la capacité d'hybridation des oligonucléotides
5
La capacité d'hybridation et la spécificité d'hybridation d'oligonucléotides
discriminants entre PSA et klk2 ont été vérifiées. Pour cela, nous avons poolé
plusieurs isoformes correspondant à klk2 que l'on a marqué à la cyanine 5 et
plusieurs isoformes de PSA qui ont été marquées à la cyanine 3 (figure 6). Les
10 cRNA ont ensuite été cohybridés sur une même lame qui est lue dans les 2
canaux et lorsque l'on superpose les 2 images, il apparaît qu'il n'y a pas
d'hybridation croisée entre les oligonucléotides PSA et klk2 à l'exception
d'un
oligo déposé en quadruplet qui a été redessiné.
15 C3 - Etudes d'échantillons tumoraux et sains de patients
Puisque la quantité de matériel biologique dont nous disposons le plus souvent
est insuffisante, nous avons eu recours à une amplification d'ARN (figure 7).
La
première étape consiste à reverser de l'ARNm en présence d'oligodT grâce à la
20 Superscript II. La RnaseH va dégrader l'ARN ayant servi de matrice et
laisser
des amorces qui seront utilisées par la DNA polymérase I pour la synthèse du
second brin d'ADNc. Les fragments synthétisés seront assemblés par la DNA
ligase. A l'issue de cette étape , on se retrouve avec une structure d'ADN
double brin qui sera reconnue par la T7 DNA polymérase, elle va amplifier le
25 brin correspondant à la séquence du messager et synthétiser des molécules
de
cRNA qui s'hybrideront aux sondes de séquence complémentaire (sens du
mRNA).
Pour chaque patient, 8ug de cibles correspondant aux échantillons tumoraux et
sains et marqués à l'aide de 2 fluorochromes différents ont été cohybridés sur
30 une même lame. Les intensités de fluorescence ont été mesurées dans les 2
canaux et normalisées selon la méthode de l'intensité globale dans un logiciel
d'analyse de fluorescence sur lame de verre (GeneTrafFic).
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
56
La figure 8 représente la superposition des deux images obtenues pour l'un des
patients.
Pour les 3 autres patients analysés nous pouvons faire des remarques
similaires : la qualité du signal de fluorescence est bonne, les intensités
sont
supérieures à 1500 en général.
Concernant les échantillons tumoraux et bénins provenant des 4 patients,
l'analyse des signaux a démontré que certaines isoformes étaient exprimées
différentiellement parmi plusieurs patients (Figure 9).
C4 - Etudes de lignées de cancer de la prostate et de cancer du sein
Des expériences ont consisté à comparer les profils d'expression d'isoformes
PSA et de KLK22 entre des lignées de cancer de la prostate et de cancer du
sein.
Pour cela, nous avons amplifié de l'ARN de deux lignées de cancer de la
prostate (Mda2b et LnCAP) et de 4 lignées de cancer du sein (Mda231, T47D,
Mcf7 et BT549).
Nous avons ensuite cohybridé chaque lignée de cancer de la prostate avec les
différentes lignées de cancer du sein après avoir marqué les lignées Mda2b et
LnCAP à la Cyanine 3 et les lignées de cancer du sein à la Cyanine 5.
Les lames ont été lues dans les 2 canaux et les intensités de fluorescence ont
été normalisées dans GeneTraffic selon la méthode d'intensité globale. Nous
avons scindé l'étude en deux groupes d'hybridations en fonction de la lignée
prostatique.
Nous avons ensuite identifié une liste d'oligonucléotides discriminants dont
l'expression est dérégulée dans au moins une des hybridations au sein d'un
méme groupe d'hybridation. Nous avons sélectionné les oligos pour lesquels un
ratio inférieur à 0.66 ou supérieur à 1.5 a été calculé (soit -0.58<Mean log2
ratio>0.58). Nous avons choisi de présenter les résultats des l'analyses Mda2b
vs T47D et LnCAP vs T47D pour lesquels nous avons observé l'expression
différentielle la plus importante et affectant le plus grand nombre
d'oligonucléotides discriminants.
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
57
Une expression différentielle de 15 isoformes parmi lesquelles 3 sont
surexprimées dans les lignées issues de cancer de la prostate par rapport à
une
lignée issue de cancer du sein à savoir PSA-EHT019, PSA-EHTj et PSA-EHTI a
pu être observée. Les 12 autres sont sous-exprimées dans le cancer de
prostate (Figure 10).
C5 - Etudes de tissus
Des expériences ont consisté à vérifier la spécificité tissulaire de
l'expression
des isoformes de PSA et de KLK2. Pour cela nous avons choisi 4 tissus : la
prostate, le coeur, le rein, et l'intestin. Nous avons amplifié les ARN de ces
4
tissus et nous avons co-hybridé l'ARNc de la prostate marqué à la Cyanine 3
avec les ARNc des autres tissus marqués à la Cyanine 5. Nous avons aussi co
hybridé l'ARNc de prostate marqué à la Cyanine 3 avec de ARNc de prostate
marqué à la Cyanine 5.
Les lames ont été lues dans les 2 canaux et les intensités de fluorescence ont
été normalisées dans GeneTraffic selon la méthode d'intensité globale.
Nous avons ensuite identifié une liste d'oligonucléotides discriminant dont
l'expression est dérégulée dans au moins une des hybridations au sein du
groupe d'hybridation regroupant ces 4 hybridations. Nous avons sélectionné les
oligonucléotides pour lesquels un ratio inférieur à 0.66 ou supérieur à 1.5 a
été
calculé (soit -0.58<Mean log2 ratio>0.58).
II a ainsi été mis en évidence une expression dérégulée de plusieurs isoformes
de PSA et de KLK2 en fonction du tissu sain testé ( prostate, coeur, petit
intestin,
rein). Ce sont : PSA-EHT003, PSA-EHT005, PSA-EHT013, klk2-EHTb, klk2
EHTd, klk2-EHTj klk2-EHTf et PSA-EHTI, PSA-EHT019 et klk2-EHTe (figure 11
et 12).
C6 - Récapitulation des signaux d'hybridation obtenus
Les tableaux 3, 4 et 5 regroupent les signaux d'hybridation obtenus sur le
micro-
array à oligonucléotides à partir de tissus sains (tableau 3), de lignées
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
58
cellulaires (tableau 4) et de tissus provenant de patients atteints de cancer
de la
prostate (tableau 5). Les valeurs supérieures à deux fois la valeur du bruit
de
fonds représentant une hybridation significative sont indiquées. Les valeurs
inférieures à deux fois le bruits de fonds sont représentées par #NA. II
apparaît
alors que tous les oligonucléotides discriminants sauf les oligonucléotides
SEQ
ID NO : 184, 215 et 220 produisent des signaux significatifs dans au moins un
des systèmes étudiés. L'expression des isoformes décrites dans la présente
invention est donc confirmée par cette approche. II convient également de
noter
que l'isoforme PSA-EHT 023 qui est associée à l'oligonucléotide SEQ ID NO
184 a pu être detectée par une approche PCR plus sensible (voir chapître D
suivant).
En conclusion, il apparaît que la majorité des isoformes décrites dans cette
invention soient effectivement exprimées dans un des modèles étudiés. Des
expressions tissulaires spécifiques et tumorales spécifiques ont également été
mises en évidence.
D - VALIDATION DE L'EXPRESSION DES ISOFORMES
DE PSA ET DE KLK2 PAR PCR.
Une technique de PCR de jonction a été utilisée afin de révéler l'existence de
certaines isoformes. Le principe repose sur l'amplification spécifique des
isoformes grâce à des oligonucléotides désignés spécifiquement sur la nouvelle
jonction résultant de fépissage alternatif déjà décrit. Ces amplifications
sont
réalisées sur des ARN issus des zones bénignes et des zones tumorales des
prostates des mêmes patients, ainsi que sur des contrôles plasmidiques.
Les résultats des amplifications par PCR sont présentés en figure 13. La
flèche
représente la bande de la taille attendue. On recherche une amplification
spécifique des isoformes dans les pools T et N , avec un contrôle wt négatif,
c'est à dire aucune amplification spécifique de la taille de l'isoforme sur le
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
59
plasmide sauvage. Le plasmide avec l'isoforme clonée sert de contrôle positif
d'amplification.
isoformes Fi ure conclusions
#
PSA-EHT003 A Amplicon de taille et de squence attendues
pour le
plasmide PSA-003.
Amplification aspcifique sur le plasmide
wt, ne
correspondant pas la taille de l'isoforme
recherche.
Amplification positive sur les 2 pools,
uniquement la
taille attendue de l'isoforme.
PSA-EHT023 B Amplicon de taille et de squence attendues
pour le
plasmide PSA-023.
Amplification aspcifique sur le plasmide
wt, ne
correspondant pas la taille de l'isoforme
recherche.
Amplification positive sur les 2 pools,
la taille
attendue de l'isoforme mais aussi la taille
obtenue
avec le plasmide wt.
PSA-EHT012 C Amplification aspcifique sur le plasmide
wt, ne
correspondant pas la taille de l'isoforme
recherche.
Amplification positive sur les 2 pools,
la taille
attendue de l'isoforme.
10
En conclusion, cette technique permet également de mettre en évidence la
présence de certaines isoformes dans les tissus prostatiques. D'une
sensibilité
accrue par rapport au micro-array, l'approche PCR a notamment révélé
l'expression de PSA-EHT012.
E - PRODUCTION D'ANTICORPS ET EXPRESSION DES PROTEINES
Afin de déterminer l'existence de protéines encodées par certains des variants
décrits dans la présente invention, des anticorps polyclonaux spécifiques de
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
certaines des isoformes ont été produits. Ces anticorps ont été utilisés dans
des
« western-blots » afin de détecter l'expression des protéines correspondantes.
Production d'anticorps et expression des protéines.
5 Tous les peptides et les anticorps ont été produits par la société
Eurogentec
(Belgique). 20-30 milligrammes des peptides correspondant aux séquences
décrites dans la figure 14 ont été synthétisés par chimie Fmoc avec une pureté
supérieure à 70%. Pour provoquer une réponse immune, la KLH a été couplée
à 5 milligrammes de chaque peptide par l'utilisation de MBS (m-
10 maleimidobenzoyl-N-hydroxysuccinimide ester) ou de glutaraldehyde.
Deux lapins (SPF New Zealand white rabbits) ont été immunisés avec 200
microgrammes de peptide couplé. La première injection a été effectuée avec de
l'adjuvant de Freunds complet alors que les injections successives ont été
effectuées en adjuvant de Freunds incomplet. Un protocole standard a été
15 utilisé comportant des injections aux jours 0, 14, 28 et 56 et des
collections de
sérums aux jours 0, 38 et 66. Le saignement final a eu lieu au jour 87.
L'évolution du titre dans les sérums a été effectuée par test ELISA (figure
15).
Les antigènes, peptides synthétiques ou KLH ont été appliqués dans les puits
des plaques ELISA (100 nanogramme dans du PBS à 4°C pendant 16 heures).
20 Après saturation (BSA 1 mg/ml à 25°C pendant 2 heures), des
dilutions
successives de sérums (préimmuns : PPI, de la première récolte : PP et de la
deuxième récolte : GP) ont été incubées à 25°C pendant 2 heures. Le
système
HRP / OPD a été utilisé pour révéler la fixation des anticorps par mesure de
la
densité optique à 492 nm. Les titres obtenus vis à vis des épitopes
selectionnés
25 sont satisfaisants.
Analyse par "Western Blots".
Les extraits protéiques ont été préparé à partir de tissus et de lignées
cellulaires
à l'aide d'un tampon de lyse (Tris 50 mM pH=7.5, EGTA 5 mM, NaCI 150 mM,
30 Triton 1 %, NaF 50 mM, inhibiteurs de protéases (Roche)). Les extraits ont
été
quantifiés par la méthode de Bradford. Pour les tissus, 20 microgrammes
d'extraits ont été déposés sur gel de poly-acrylamide-SDS, Pour les sérums, 15
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
61
microlitres d'une dilution de sérum non purifié au cinquantième ou d'une
dilution
de sérum purifié (kit Aurum BioRad n° 732-6701 ) au huitième ont été
utilisés.
Après electrophorése dénaturante, les protéines séparées sont transférrées sur
membrane PVDF. Les variants de PSA et de KLK2 sont ensuite détectés par
incubation de la membrane avec un anticorps polyclonal produit spécifiquement
(cf chapitre précédent). Après lavage, la membrane est incubée avec un
anticorps secondaire anti-immunoglobulin marqué à la peroxidase HRP (dilution
1/5000). Les bandes sont ensuite visualisées par détection ECL (Amersham).
Anticorps EHT- SE3962
Cet anticorps a été généré à parir d'un épitope commun aux variants KLK2-
EHT004, et KLK2-EHT006. Les tailles attendues pour ces deux variants sont de
17 kD (KLK2-EHT006) et de10 kD (KLK2-EHT004). Deux bandes migrant aux
tailles attendues peuvent être observées à partir de sérums (Figure
16).L'anticorps semble reconnaitre ces bandes spécifiquement car il peut étre
déplacé par des doses croissantes de peptides synthétiques correspondant à
l'épitope choisi (figure 16D). On peut observer une hétérogénéité selon les
échantillons de sérums. On n'observe pas de corrélation évidente avec le taux
de PSA total (figure 16 A), B) et C))
Anticorps EHT-SE3963
Cet anticorps a été généré contre un épitope de jonction correspondant à PSA-
EHT021 (taille attendue 20 kD). Trois bandes de poids moléculaires d'environ
22, 25 et 40 kD sont observées à partir de tissus prostatiques (figure 17). La
bande de plus faible poids moléculaire pourrait correspondre à PSA-EHT021. La
bande à 25 kD pourrait correspondre à un variant déjà décrit et présentant un
des deux événement d'épissage associé à PSA-EHT021 (Tanaka et al, 2000).
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
62
REFERENCES
David et.ai, (2002) J. Biol. Chem
Riegman et al (1988) Biochem. Biophys. Res. Commun. 155, 181-188.
Riegman et.al (1991) Mol. Cell. Endicronol. 76, 181-190.
Liu et.al (1999) Biochem. Biophys. Res. Commun. 264, 833-839
Heuzé et.al (1999) Cancer Res. 59, 2820-2824.
Heuzé-Vourc'h et.al (2001 ) Eur J Biochem. 268, 4408-4413.
Heuzé-Vourc'h et al (2003) Eur J Biochem. 270, 706-714
Meng et.al (2002) Cancer Epidemiology, Biomarkers and Prevention 11, 305-
309.
Tanaka et.al (2000) Cancer Res. 60, 56-59.
Young et.al (1992) Biochemistry 31, 818-824.
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
63
TABLEAU 1
Nom S uence 5' - 3' SEQ fD NO:
PSA-exon1- wt GTTGTCTTCCTCACCCTGTCCGTG
PSA-exon2- wt AGTGCGAGAAGCATTCCCAACCCT
PSA-exon2bis- wt AGGTGCTTGTGGCCTCTCGTGGCA
PSA-exon3- wt ACGATATGAGCCTCCTGAAGAATC
PSA-exon4- wt CTTGACCCCAAAGAAACTTCAGTG
PSA-exon5- wt AATGGTGTGCTTCAAGGTATCACG
PSA-'ctex1-2-wt TGACGTGGATTGGCGCTGCGCCCC
PSA-ctex2-3-wt CTGCATCAGGAACAAAAGCGTGAT
PSA-'ctex3-4- wt AACCAGAGGAGTTCTTGACCCCAA
PSA-'ctex4-5-wt AGCACCTGCTCGGGTGATTCTGGG
PSA-ct-ex-int1wt TGACGTGGATTGGTGAGAGGGGCC
PSA-'ct-ex-int2wt CCCCCTCTGCAGGCGCTGCGCCCC
PSA-'ct-ex-int3wt CTGCATCAGGAAGTGAGTAGGGGC
PSA-ct-ex-int4wt CTTCCTCCCCAGCAAAAGCGTGAT
PSA-ct-ex-int5wt AACCAGAGGAGTGTACGCCTGGGC
PSA-ct-ex-int6wt CCTGGCCCGTAGTCTTGACCCCAA
PSA-'ct-ex-int7wt AGCACCTGCTCGGTGAGTCATCCC
PSA-'ct-ex-int8wt TTTTACCCTTAGGGTGATTCTGGG
PSA-intron 1 CTCTTTTCTGTCTCTCCCAGCCCC
PSA-intron 2 AGAGAGGGAAAGTTCTGGTTCAGG
PSA-intron 2bis GGGAGCGAAGTGGAGGATACAACC
PSA-intron 4 CCGTGTCTCATCTCATTCCCTCCT
PSA-001-int-int CCAGCACCCCAGCTCCCAGCTGCT 168
PSA-001-int3' CCAACCCTATCCCAGAGACCT T
GA
PSA-001-int3'bis AGGATACCCAGATGCCAACCAGAC
PSA-003-int-int CCATACCCCCAGCCCCTCCCACTT 169
PSA-003-int3' GCCCCTCAATCCTATCACAGTCTA
PSA-004-'ctex1-intGTGACGTGGATTGCTGTGAGTGTC 170
PSA-004intron1 GACACCTCCTTCTTCCTAGCCAGG
PSA-005-'ct-int1-int1AGGCTCTTTCCCCCCAACCCTATC 171
PSA-008-'ctex1-intGTGACGTGGATTGGATACCCAGAT 172
PSA-009-'ct-fnt2-int2TCCGCCTCTTATTCCATTCTTTCT 173
PSA-009-int3' GAGGCGCAGAGAAGGAGTGGTTCC
PSA-009-int3'bis GAGACACAGAGAAGGGCTGGTTCC
PSA-010=xt-ex1-ex2TGACGTGGATTGGTGCTGCACCCC
PSA-0012=ctex2-int2GCATCAGGAATCTCCATATCCCCC 174
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
64
PSA-012-int3' TCACCTGTGCCTTCTCCCTACTGA
PSA-013 ct-intl-ex1TGACGTGGATTGCACCCCCTCTGC 175
PSA-013-int3' GGCATTTTCCCCAGGATAACCTCT
PSA-014-int3' GGACTGGGGGAGAGAGGGAAAGTT
PSA-015-ex1-ex2 GTCTTCCTCACCCTGAGCTTGTGG 176
PSA-015-ex1-ex2bisCTTCCTCACCCTGAGCTTGTGGCC 177
PSA-016-ex1-ex2 TGACGTGGATTGGGCAGTCTGCGG 178
PSA-018-'ct-int2-int2GAGAAAAGAAAGGACCCTGGGGAG 179
PSA-018-'ct-ex1-ex2TGACGTGGATTGGAGCTGCGCCCC
PSA-018-int3' GAAGTGGAGGATACAACCTTGGGC
PSA-019-ex3 CAGTCTGTTTCATCCTGAAGACAC
PSA-019-'ct-ex4-5 AGCACCTGCTGGGGTGATTCTGGG
PSA-019-'ct-ex3 ATTTCAGGTCAGCCTGCCGAGATC 180
PSA-020=ct-ex3 CTGCATCAGGAAGCCAGGTGATGA
PSA-020-'xt-ex4-ex5AGCACCTGCTAGGGTGATTCTGGG
PSA-020-ex3 GTGATGACTCCAGCCACGACCTCA
PSA-021=ct-ex3 CTGCATCAGGAAGCCAGGTGATGA 181
PSA-021-'ct-ex3-2 GTGATGACTCCAGCATTGAACCAG 182
PSA-022-ex3 TGATGACTCCAGCATTGAACCAGA
PSA-023-'ct-ex2 GTTCGGATTGTCTCTCGTGGCAG 183
PSA-023-'ct-ex5 AATGGGGTGCTTCAAGGTATCACG
PSA-023-'ct-in3-ex4CTGGGCCAGATGTCTTGACCCCAA 184
PSA-025-'ct-ex2-ex4TGCATCAGGAATCTTGACCCCAAAG 185
PSA-026-'ct-ex3 TTGCTGGGTCAGCATTGAACCAGA 186
PSA-027-'ct-ex3-ex5ATCTTGCTGGGTCGGGTGATTCTG 187
PSA-027-'ct-ex3-ex5bisCTTGCTGGGTCGGGTGATTCTGGG 188
PSA-001= ct-int1 CCAGCACCCCAGCTCCCTGCTCCC
PSA-d=ct-ex2-ex3 CTGCCCACTGCACCTGCTACGCCT 189
PSA-d-exon3 GGGGCAGCATTGAACCAGAGGAGT
PSA-f-'ct-int5' TTGGTAACTGGCTTCGGTTGTGTC
PSA-f 'ct-int2 CCCTCTCTTCTCTGTCTCACCTGTG 190
PSA- -ct-ex2-int2 CTGCATCAGGAATCTCCATATCTC
PSA- -ct-ex2-int2bisGCATCAGGAATCTCCATATCTCCC
PSA-h=ct-ex4-int4 AGACCTGCTCGGAGCTGGACGCT 191
PSA-h-'ct3' GGAACTGCTATCTGTTATCTGCCTG
PSA-h-exon5bis TGTCTGTAATGGTGTGCTTCAAGG
PSA-'=ct-ex4-int4 AAGCACCTGCTCGTGGGTCATTCT 192
PSA-k=ct-ex4-int4 CACCTGCTCGGTGAGTCATCCCTA 193
PSA-k= ct-int4 GAGTCATCCCTACCCCTCTGTTGG
PSA-I-'ct-ex4-int4AGAAGGTGACCAAGTTCAGCACAC 194
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
PSA-I-'ct-int3' AGGAACAGGGACCACAACACAGAA
PSA-m-int1-5' GATGCTTGGCCTCCCAATCTTGCC
PSA-m-'ct-int1 ACCCAGATGCCACCAGCCACCAAG 195
PSA-n-int1-5' GCCAACCAGACACCTCCTTCTTCC
PSA-n= ct-i nt1 C C T TAGGAAAAACAT GAAG 196
CG T C T
PSA- -'ct-ex1-int1GTGACGTGGATTGCCAGGCTATCT 197
PSA- -ct-int5' CCAACTGGTGAAACCCCATCTCTA
PSA- -'ct-int2 AAAATTAGCCAGGCTACCTACCCA 198
PSA-r-'ct-int2 CCCTGAGAAAAGCCGCATCTACAG 199
PSA-r-'ct-int3' CATCTACAGCTGAGCCACTCTGAG
PSA-s-'ct-int4 GGTTATTCTTACAGCAGAGAGGAGG 200
PSA-s= ct-int3' GAGTCAGGAACTGTGGATGGTGCT
PSA-t=ct-int5' TGGGACATAGCAGTGAACAGACAG
PSA-t-'ct-int4 GCTCTCAGGGAGGGCAGCAGGGAT 201
PSA-u=ct-int4-ex5 GGCCTGGCTCAGGGTGATTCTGGG 202
KLK-2-exon1-wt GTTCTCTCCATCGCCTTGTCTGTG
KLK-2-exon2-wt AGTGTGAGAAGCATTCCCAACCCT
KLK-2-exon2bis-wt GTACAGTCATGGATGGGCACACTG
KLK-2-exon3-wt CTGAAGCATCAAAGCCTTAGACCAG
KLK-2-exon4-wt CCAGGAGTCTTCAGTGTGTGAGCC
KLK-2-exon5-wt CACTTGTCTGTAATGGGGTGCTTC
KLK2-'ctex1-2-wt TGGGGTGCACTGGTGCCGTGCCCC
KLK2-'ctex2-3- ATTGCCTAAAGAAGAATAGCCAGG
wt
KLK2-'ctex3-4- AACCAGAGGAGTTCTTGCGCCCCA
wt
KLK2-ctex4-5-wt AGACACTTGTGGGGGTGATTCTGG
KLK2-intron1-wt ACAGTTCAGCCCAGACAATGTGCC
KLK2-intron2-wt AGACACAGGGAGGGCTGGTTTCAG
KLK2-intron3-wt AGCCCAGTTTTTCTCTGACCCATA
KLK2-intron4-wt GGGAAGCAGCAGTGAACAGGTAGA
KLK2-'ct-ex-int1wtTGGGGTGCACTGGTGAGATTGGGG
KLK2-ct-ex-int2wt CCCCCTCCGCAGGTGCCGTGCCCC
KLK2-'ct-ex-int3wtTTGCCTAAAGAAGTAAGTAGGACC
KLK2-ct-ex-int4wt CTTCCTCCCCAGGAATAGCCAGGT
KLK2-'ct-ex-int6wtTCTGACCCATAGTCTTGCGCCCCA
KLK2-'ct-ex-int7wtGACACTTGTGGGGTGAGTCATCCC
KLK2-ct-ex-int8wt CTTTACCCTTAGGGTGATTCTGGG
KLK2-002-'ct-int2-ex3TCACTTCTCAGGAATAGCCAGGTC 203
KLK2-002=ct-ex3-ex4GATGTTGTGAAGGAGTCTTCAGTG 204
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
66
KLK2-002-ex4 AGCCTCCATCTCCTGTCCAATGAC
KLK2-003-exon5 CACTTGTCTGTAATGGTGTGCTTC
KLK2-003= ct-ex1-ex3TGGGGTGCACTGGAATAGCCAGGT 205
KLK2-003= ct-int4-ex5CTGGAGGGGAAAGGGTGATTCTGG 206
KLK2-004=ct-ex2-ex4TTGCCTAAAGAATCTTGCGCCCCA 207
KLK2-004-int4 AACATCTGGAGGGGAAAAGTGAGT
KLK2-005-int4 AACATCTGGAGGGGAAAGGTGAGT
KLK2-008-ex4 ATCCTCCATCTCCTGTCCAATGAC
KLK2-008-'ct-ex3-ex4GAACCAGAGGAGTGAGTCTTCAGC
KLK2-009-'ct-ex3-ex4GAACCAGAGGAGTGAGTCTTCAGT 208
KLK2-009=ct-ex3 TGAAGACTCCAGCATCGAACCAGA 209
KLK2-009-ex4 CTTCAGTGTGTGAGCCTCCATCTC
KLK2-011-'ct-ex3-ex4AACCAGAGGAGTGGTAAAGACACT 210
KLK2-011=ct-ex4-int4AGACACTTGTGGGGTGAGTCATCC
KLK2-a-exon3 ATGAGCCTTCTGAAGCATCAAAGC
KLK2-a-exon3bis CCCACACCCGCTCTACAATATGAG
KLK2-b-'ct-int1 CTGACTCTTCCCCGCG~1GGCTATCT 211
KLK2-b-'ct-int3' ACTCTTTGCCCCAGACCCGTCATT
KLK2-c= ct-int1 '~GGGGTGCACTGACCCGTCATTCA 212
KLK2-d=ct-int5' GCGGGTTCTGACTCTTATGCTGAA
KLK2-d= ct-int1 CAGCCTCGTCCCCCCAACCACAAC 213
KLK2-e-ex2 CAGTCATGGATGGGCACACTGTGG
KLK2-e-ex2-140nt? '~AGTGGAACCCTGCTATCTGCCGA 214
KLK2-e=ct-140nt?-ex3TTTTCTCAGGAATAGCCAGGTCTG 215
KLK2-f-'ct-ex2-int2GA2'GGGCACACTCCTGTTTTCTAA 216
KLK2-f=ct3' CCTTTCCCCATTTTCTCTCTCCTC
KLK2- -ex5 CACTTGTCTGTAATGGGTGCTTCA
KLK2- -int4 AGTCATCCCTACTCCCAACATCTG
KLK2-h= ct3' GAGTCTTCAGTGTGTGAGCCTCCA
KLK2-h= ct3'bis GTCCAATGACATGTGTGCTAGAGC
KLK2-i-ex4 ACAGGTGGTAAAGACACTTGTGGG
KLK2- -'ct-int3' CTGCTACT CCACACT CCT CAGATG
KLK2-= ct-int2 ACATCCCTCCACCCTCATGCCTCT 217
KLK2-k-'ct-int5' AGTCTCTCCCCTCCACTCCATTCT 218
KLK2-k-'ct-int5'-6nt-ex5CCTGCCGATGGCCCACTTGTCTGT 219
KLK2-I=ct-int2-ex3CCCCAGCTGCAGGAATAGCCAGGT 220
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
67
TABLEAU 2
Isoformes Couple d'oligonuclotides
KLK2-EHT002 249 / 166
KLK2-EHT003 249 / 170
KLK2-EHT004 249 / 174
KLK2-EHT006 249 / 166
KLK2-EHT007 249 / 174
KLK2-EHT009 249 / 166
KLK2-EHT011 249 / 174
KLK2-EHTb 163/213
KLK2-EHTc 163/213
KLK2-EHTd 163/213
KLK2-EHTe 167/172
KLK2-EHTf 167/172
KLK2-EHT' 214/215
KLK2-EHTk 218/170
KLK2-EHTI 221 /172
PSA-EHT001 247 / 203
PSA-EHT003 247 / 203
PSA-EHT004 247 / 203
PSA-EHT005 247 / 203
PSA-EHT007 247 / 203
PSA-EHT008 247 / 203
PSA-EHT009 247 / 207
PSA-EHT012 247 / 205
PSA-EHT013 247 / 176
PSA-EHT015 247 / 176
PSA-EHT016 247 / 176
PSA-EHT018 247 / 207
PSA-EHT019 247 / 248
PSA-EHT021 247 / 248
PSA-EHT022 247 / 182
PSA-EHT023 247 / 182
PSA-EHT025 247 / 248
PSA-EHT026 247 / 248
PSA-EHT027 247 / 182
PSA-EHTa 175/203
PSA-EHTd 179/178
PSA-EHTf 179/205
PSA-EHTh 181/182
PSA-EHT' 181 /182
PSA-EHTk 181 /182
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
68
PSA-EHTI 181 /209
PSA-EHTm 200/201
PSA-EHTn 200/201
PSA-EHT 2021203
PSA-EHT 204/207
PSA-EHTr 204/207
PSA-EHTs 208/211
PSA-EHTt 208/211
PSA-EHTu 210/182
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
69
Ç
O O N 00 00 N lp Q Q tn M a Q CO Q a Q a a N a a a M a a
Z O I~ M O N f~ w w 00 O w w N w w w w w M w w w O w w
~ ~ M ~ cO N co Z Z ~ r Z Z O Z Z Z Z Z ~ Z Z Z ~ Z Z
~ O 'd' d' d' N ~ ~ ~ M f~ ~ ~k N *k ~k ~k ~k ~k ~ ~k *k ~k r ~ ~k
N
J
w
N
Ö ~ ~ ~ dN' Q Q fpO dp' Q Q M Q Q Q Q Q ~ Q Q Q N Q Q
U LLJ d' M r '~' O N Z Z M ~ Z Z r Z Z Z Z Z d' Z Z Z N Z Z
00 ~ tn ~ O N ~ ~k N ~ ~k ~k r *k ~k ~ ~k ~k N ~k *k ~ r ~ ~k
J
f' N N N ~ N a Q O M Q a M a Q O O M Q Q a a a a
ô O co O N O ao Z Z ~ ~ Z Z O Z Z °~ M ° Z Z Z Z Z Z O
p J Z I~ r M N M r ~ ~k O M ~ ~ M ~ ~k ~ 00 00 ~
â
ôW~ ~,~~,~N~~~~N~ÖQ~~a~o~M~~QQ~~~c~o
m I' N Cfl d' O M Z N M O O O r M Z Z ~ N p O
OD ~ f~ N O ~ ~- f~ N ~ ~ r r d' M N ~ ~ r r
J
r O 00 M O 00 00 d' Ln M M Q _Q ~_' N Q
Ö Ö~ÖNOONOzz~~C'O~o00Ö~z000MÖ~ZZNNZO
J Z f' r M N M r ~ ~k O M r r M r ~k ~' 00 00 p ~k ~k r CV.~ p
O
LLJ '~' N °~ ~ N N a Q M ~ d- ~ ~. ~ Q ° p dM" ~ Q Q O ty.
Q d
OLj~'~~zZM~Nf~.MOZO000COZZOh-ZN
LLl m ~ ~ M ~ 1' N ~k ~k ~ ~ 0 0 r r ~ r M r M
J
L
~~~-MrQ~QaQM~aMr~Qaaa~aaa~a~
w
CO
Vif'
w
Ln
r
w
w
w
w
w
w
w
Ln
w
I~
00
Z
Vit'
CO
N
O
w
w
w
d-
M
ao
w
Z
ao
Z
Z
Z
~
~
Z
~
~
Z
Z
Z
Z
~
Z
Z
Z
~
Z
~
ONNM~kr*k~k~kd'r~kMr~k*k~k*k~~k~k~:t-~kr
J
N
m
~
M~OQ_a__aa_d~"~'Q_MM_aa__aa_~Qa_a_Na_
d' LL!
r
~
d'
N
Z
O
Z
Z
Z
~
O
Z
~
~
Z
Z
Z
Z
O
Z
Z
Z
O
Z
<'''
, X
O
~
~7
M
~k
N
~k
~k
~k
~
r
*k
r
r
~k
~k
~k
~k
CO
~
~k
~k
N
~
c-
W
J
r
~ ~ ~ ~ ~ N M d ~ ~ ~ N Q
O ' .
N M d'Lf~C C C C C C Cr N N
i i ~ ~ ' ' n 'T
~ ~ ~ ; ; ; ; C ~ .C
r N N M d'lf~c-N M ~'X ~(>(X X X X XO O
C ~ N N N ~ ~ N N N ~ N NOLO O O ~
7 O O O O O O~ .,.~.,r.,..~.,r.,~.~+'~.~..,..~...'...,r.,.r+-..,r.+~p
O O
O X X X X X X U U U U U U U U UC C C =
U U U O O O
a a a a a aa a a a a Q a Q a a a aa a a a a a a
a n.a.a a.~~ a a a.a a a a.a a a a.a.a a.~ a.a ~
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
~M_apNa_~_aa_a_are_a~~p_aa_~_aa_M_a_ap~_a_a_a_a
N ~ Z M h Z p Z Z Z Z °~ ~' Z ~''m' ~ Z Z ~ Z Z ~ Z Z r r- Z Z Z Z
N ~ Cfl l.t~ ~k N *k ~ ~ ~k ~ N ~ N N N ~k ~ c- ~ *k N ~k ~ M M ~ ~ ~ ~k
~°Q; ~Q~QaaaÖ~Q°r°od'o~.~'nQa~QQ~,°,~QQ~MaQaQ
O N Z ~ 'd' Z M Z Z Z Z ~ ~ Z N.r c4 Z Z ~ Z Z M Z Z N O Z Z Z 'Z
O ~ ~ N N ~ ~ ~k ~ *k ~ N CO ~ c- N r ~ ~k N ~k ~k lI~ ~ ~k ~ O ~k ~ ~k ~k
Q N Q Q Q O) Q Q ~ N Q Q ~ ~ d r. d p Q Q ~ CO d' ~ N d' a a ~y-
Z d' Z p Z Z Z O Z Z ~" N Z Z O M Z N Z ~' Z Z r N ~' cfl 0 ~'' Z Z r
~ r ~ ~ ~ ~k ~, M ~k ~k (~/ ~ ~ ~k d' ly ~ I~ ~ Iw. ~ ~ N fer. lw, c- r ~ ~ ~k
pp
~ N ~W N p ô Q ~ ~ Q ~ ~ ~ ~:~ ~ a ~ a N due., ~ agio ~ ô
CO M r' O d' ~" M Z p CO Z.: O O ~ N Ln O N Z 'd' Z r- M Lt2' r N N r N Cfl M
r M N N ~ O ~ ~ N ~: M h ~ r N (V . r ~ M ~ CO CO c-' r (p ~ r r 0 0
~ due' ~ O O Z ~ O ~ Z ~ N O z t o~o ô N oOO ~ Z z
N c- O r r ~ ~ M ~- ~k r ~ ~ ~~ (~ N M f' f' fw, ~ ~ N h I~' P ~ I~ ~ ~k M
N p O ~ a ~ p r. M N M ~ 'dy O ~- d" O
mMN~rc'~.,,~--o°o~Qro°oc~oW.nOCCaNaiNQQ~~'mn~r.coQQ~r
ln M d' OQ Z CO O0 Z t0 Z LO d' d' ~'O In O Z Z ICI d' r O N r z Z N
M r M d. ~ O ~ f~ O ~ r N N QQ Ci? ~ ~k ~k M c- r d- 00 r- ~ ~ M
.r r. r r
~c'~oQcNO°oQ~QQQ~~:~Q~~~QQQQa~aa~~Q4QQ
~ Z Z Z Z Z ~- p Z d' o~ N Z Z Z Z Z ~' Z Z O ~ Z Z Z Z
M '- ~k N N ~ ~ ~ ~k ~k ~y N ~. ~k ~~ cy N ~k ~ *k ~k ~k M ~k ~k N N ~k ~k ~k
~
~NZrm.Nn~~ZZZ~~c'~ozo~o iNQaQQQ~.°~QQNNQQQQ
zzzzz~ozz~ zzzz
~ N ~k CO I~ ~ rte'." ~ *k ~k ~ ~ ~ *k ~- M M ~ ~k ~k ~k ~k op ~k ~k tp ~ ~k
*k *k ~k
O O r N M Vif' Ln C~f~00O O r CV M
O ~ ~ ~ ~ ~ ~ ~ ~ ~ ~ M M M M
t- r r r c- c- r c-r ~ ~ ~ c-c- c-
c- N N ~ r fn N N L(7
C ~ ~ ~ N , ~ y N -O .~~ tn N C
V
+-' r r ~ r N ~ '-X r X ~ r d'MM d' M M N
O X O . X . -OX N a ~ ~ ~ ~ X X XX X X X X
C ~. .,,
C C
'TC~N O ' N 'TM M ~ ~--'M 'M M r r r . O O OO O O O O
i ; T , M M , M M
.,-.L..,-..,-..~..,-..+~.,...U r .Y+~.,r ~, , , , ,
CC U U U U C C X ' C UC C ~ ~ ~ C ~ X
'T'T",~ , , , 'i'T',, 'i','i'i, , , ' 'ii '' ' ~ '
i N ~
, , , ,, , , , , ,
MM 'd'd'Lf)00O O O O c-N MM d'1.nLnCO000000O O OO O O r e-CVM
OO O O O O O O O r O r rc-r r r r r ~ r ~ r ~-N N N N N N N
OO O O O O O O O O O O OO O O O O O O O O O OO O O O O O O
QQ Q d Q Q Q d Q d Q Q Q Q Q Q
a.a.a a.~ a.a a a a ~ a a~ a.a d a a a a a.a a.a.a.a.a a.a ~
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
71
r Cfl M M lt) N ILn M O N Cfl O Cp0
~. a a Q a ~ o~ N O~ Wn Q Q C Q ~ ~ Q ~,,~ Q Q N ~ r, Q Q M Q Q Q
M Z Z Z Z ~ '~' ~ M Z M Z Z Z Z d' u~ Z c~ Z Z ~ ~ Z Z r. Z Z Z
M~k.-~~~ M0~ll~M~kM~k*kWk~NN~Cfl W~Nrd'~k~kO~~k~k
r
~a_a_a_a Mr'd'COQ_O_Q_Q_Q_QMpQ_p_Q_Q~OOQQdO.'QQQ
~ZZZZ t'-~~'-~Zd'ZZZZpMZNZZ'-rr O
M ~ ~ ~ ~ M 'd' lt~ ~ ~k r ~ ~ ~ ~k N lf~ *k M ~ ~k '~ c- N W ~ M ~k ~k ~k
r r r r Lp
QOd- ~Q~NQQQQQd-~QQQOQ~OQ,QQ~QQQQ
~ Z Z ~ ~ ~ Z ~ M Z Z Z Z Z ~ ~''~ Z Z Z Q Z ~ Z Z Z Z Z Z Z Z
r ~ ~ M ~ OO r ~ ~ ~ ~ ~ r ~ ~ ~~ ~~ O ~
Iln rCflM~ NQQN_~Nr Mr LnN_d'da.N'~pQ
M o ~ N ~ N N ~ p M N Z'Z d' 0~0 ~ ~ M MN N M N O z z O z m z
r r M d' d' Cfl ~ ~ r *k ~k r CV N ln M M l.n ~ 07 r N ~
ô ~ yn M z ~o Z Z Z ~ M O Z '~ o °° c~ co ~,°,' Q C ~ a Q
a
r ~k ~k p p M: O CO r ~k N ~ ~ ~k p r d' ~k ~ ~, ~ O N p ~ y ~ ~k ~ ~k
~ Q a ~ ~ r co r oo Q r. Q Q Q '~' M O Q M ~' r' oo ~ Q d ô Q Q Q
fer. w, r 0 r r N O w (' w w CO N w Lp N O f~ r Cfl w a w ~~ w
O Z Z N r I~.. N lfy O Z CO Z Z Z, It~ M M Z d' p ~ CO O N Z ,Z p Z Z Z
LU ~ ~ r N N I~ CO Cfl ~ r ~k *k ~ (V ~ M ~k ~ d. Cfl N M ~k ~ ~ ~k ~ ~k
~;aaaa ~,°N~MQ j QQ tQ~°~Qô CQ~~MQ C~QQQ
~zzzz N Mrz~:zzzza~MZ zz ~o zz zzz
N*k~k~~~ In~/'(~/~,r~k~~k~~r'~'~N~~k~NN~k~N~~~k
Q Q Q Q ô T ~ ô Q N a a Q Q ô ~ Q ~ a a ~ ~ ô d 4 ~ Q Q Q
Z Z Z Z ~ M Z M Z Z Z Z Z ~ Z Z ~ N Z Z Z Z Z
N ~k ~k ~k ~k M oi ~ N *k N *k ~k ~ ~k N o ~k t' ~ ~ ~ N tf~ ~k ~k ~ *k ~k ~k
d'LnCflf'OC O O r N M d' Ln CO1~ 00O
0000a000CO o0 O O W O O O O O O O
c-r r r r r r r r r r r r r c-r
N
X
N
U ~ i M N N d~ ~.~' d'
tn'C~M M M r x C C C ( ''r~. '*' C
n .
~
c'~7c .,..,U . _ . _ ~ , C
.
~
U ~ N N ~ N N d' '~' 'd'd'' . r r LfC N M
T M ~ N ~ ~ d' d"M ~~ y
~ , , , .,-. +.. .,-.y"
.~..,r+...,.~.,...,...~.X ~ .,r+.X X ~ X ~C ~ X
U UU U U U U N C CU N N X N . X C r'T' U ~ ~ C C
U O Ui
' '' ' ' ' ' ~ 'T'T.sue.,r.~.~ ~ ~ ~-'T~.'''...,r.~.~.~.'W'T
~ ii ~ ~ i ~ r
M ~ Cflfer.I~. r U M U U U U ~ . . .U C U U U
N U U V
N NN N N N O 'n, . ., ',',',, . , ~,. . . ',',',',
, T
O OO O O O O 'U'W,'_.,! .C.C~ .'.~C.Y' ~ EE C C L L
~
Q Q d 4~ d Q QQ d ~ Q Q Q Q Q Q
c~c~c~c~c~c~ c~v~c~c~cnc~c~v~c~cepc~c~c~c~c~c~c~c~cOc~c~c~c~c~
X
a.a.a a.a aa~ a.a.a a a.a.a.a.a a a a ~ ~ ~ aa a.n.a a.a a ~
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
72
~~ ô~aMaa~Maôaa~~raaaaQda~
zz zozzpoz~zzr~r~zzzzzzzo
c- ~k ~ N N N d' c- ~- ~ c- *k ~ N N ~ N ~k ~ c- ~ ~k ~ ~k ~ ~ ~k ~k Co
cfl Q Q M M_ ~'') ~ ~ N Q '~~ Q Q N ~ Q '- Q Q ~ ~ Q a a a a Q a
°°
O Z Z d~ O ~ O M M ~ LO w w O O w 00 w w O w w w w w ~ w r'
Z N Z Z ~ Z Z Z Z Z Z Z Z Z Z p
r ~ ~ .r- N N T M O ~ p ~ ~k ~ M ~ c- ~ ~k O c- ~ ~k ~k ~k ~k ~ ~ f~.
Q IO M N Ö ~ t"N~ O N ~ f' Q Q ~ f' a a Q a ~ Q Q Q oo Q p Q ô Q
d' ~ ~ h ~ ~ Z Z ~ ~ Z Z Z Z ~ Z Z Z ~ Z ~ Z ~ Z
N r ~ c- ~ ~k ~ ~k ~ ~k ~ r- ~k ~k ~k
GO ~- Ln ~ 00 ~ O p c- Cfl N O ~ M I' ~ ~ O O 'd' O
O~O M O~ CO d~ ~
M O~ M I' c- d' 0~0 Lf~ ~k c- p c- ~ c- ~ ~ O c- M ~ -~k Iw,
t~ ln M N Ö ~ M O N tn I~ a a ~ r~ Q ~ Q i° ~ ô Q ~ ~ Q p Q
NoNOC~ON~ ~~o~O~Mo~OZZ'-~ZOZIf~oOMZ~-~Z~ZtON
~ ~k ~ ~k c- ~k c- c- N ~ c-
Cfl !~- d' c- N N CO I~ Cfl Q Q N O Q p Q M '- Q N r,
~ O c- O 00 CO 00 M M w w O w O w ln ~ d' w p N Q ~ Q 'd' op
N O M d- M ~ O O o0 M N Z Z O O Z ~- Z O r' d' Z p d~ Z 1' Z- T O)
~ CO CO r, N N O N O N N ~ ~ ~ c- ~ c- ~ c- O c- ~ ~ O *k CV ~ p C4
CNOQQÖ~ ~~QQQ~QQQ~QNQ~Mo°oQ~QQQ LQ~
N Z Z r- ~ ~ ~ Z Z Z ~ Z Z Z ~ Z r- Z ~ ~ M Z ~ Z Z Z Z Z N
N ~ ~ c'~ ~ c- M ~ ~ ~k c- ~k ~ ~k c- ~k m *k ~- N N ~ ~- *k ~ *k ~ ~ p~
m ~~QQQhQQQ~Q~QNC°~c'ooQôaQQQQr°~
N Z Z ~ ~ ~ ~ Z Z Z d~ Z Z Z ~ Z ~ Z M r' O z d' Z Z Z Z Z ~
e- ~k ~k r- N M N ~k ~k *k ~ ~k ~ *k d- *k ~ ~k c- ~- N ~ c- ~ *k ~ ~k ~k o
N N N N N
X X
N M~ ~ ~ ~ N X
' ' d' -~ ~ ~ VM d ~ ~ .~..'-..,~~.,...~..~-~
d (Lf~~ .'-. ~ N N M d'lnc' ' ' r N M d y y y~ ~ ~~~ -NX
~-.~, '
-'~
C C, ~ C C C C C C C r'N M d C C C C X X 7CX X X 7C.
~ xx x x "
~
0 0 0 0 0 o o 0 0 o u~o 00 0 0 0 ' ' '
x x x x x x o0 o a~L L L L ' 1 1I 1 1 1 N N N
U UU U U '-~.~.,-.'-~-~-s-.~-..,.r".r.'r~~-..,r.'...+.
N N N N N N UC~U U C C = C U U O O O
~ tn.~..~~ N N N N N N ~'~'~'' T T _ 'T' ' UU U U U O,O,O
T '' ' ' '
Q QQ Q d ~ ~ ~ ~ ~ ~ ~~ ~ ~ ~ ~ ~ ~ ~ ~ ~~ ~
d ~ d ~ ~ ~ ~ ~ ~ ~~ ~ ~
~ ~ ~ ~ ~ ~ ~~ ~ ~ ~ ~ ~ ~
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
73
~a~aaa~aQaaaaaM~~Qar~a aa°°'~NNQa
CO ~. CO w w w N w w w w w w w Cfl O r w w CO. Ln -~ w O c- CO O w w
~r'z~ozzz~zzzzzzz~p~zzp~-z zz~~~~zz
N ~k c- ~ ~k ~ N *k ~k ~ ~k ~k ~ ~k f' N r ~k ~ 00 r ~k ~ ~k N M r r ~ *k
N _<C °° _Q a _Q ~'' Q_ _Q a _Q Q_ Q_ _Q w r~ M Q a N o0 ,Q Q
Q N Ö M ~ Q Q
Z t~ Z Z Z O Z Z Z Z Z Z Z N O w w °O r ~, w' r ~ t~
r ~- O Z Z ~ r Z Z Z °~ ~ p Z Z
r ~ ~k ~ LO ~ ~ ~ ~ ~k ~ ~ N ~ r ~ ~ M M ~ ~ *k O ~ N CO ~k
Q Q ô a Q Q a a M Q d' ~ Q Q Q 000 ~ O Q CO Q ~ C~O Ö Q Q
r ~k N ~k ~k *k N ~k ~k ~ ~ ~k ~' ~ ~ Cfl ~ ~ ~k ,M~, r 1' *k O ~k r N r ~k ~k
O r 1~ a Q O O Q I' 00 ~ _f~- O Q a N O d' N 00 O I' r
pO~NpÖ~zzNMzNcN-~NÖzzL~NT 000~'prMO~ON~
OO r I~ f~ r ,~, ~ ' ~ r r ~ (V CO N r r ~k ~k M Vif' ~ r l~' f~ ~ N fer. 00 M
OOOOrQpN~_aa_a__aaMa_~N_co~.Q_opo~_'''-~ <t~oo~.r~aoo~a_a
O t' O N
~ ~ o~O z r 'd' Ö Z Z Z Z Z ~' Z p O aO d' Z '- r M z ~ O CO tO O z Z
r ~- CV ~k M O N *k ~ ~k ~ ~k ~' ~ ~ CO N O ~ ~ r ~ ~k O M r N r ~k ~k
_ r- N o0 cfl _
O N,Q ~_Qa__Q-_aaMa~~° QN°~. _Q _QQ
Ln N (fl M (fl p
N O p Z ~ r r Z Z Z Z Z '.c~ Z O ~ d' N Z ~' p ~ Z O O O r ~''~ Z Z
Op O r ~ ~' ~ M *k ~ ~ *k ~ r ~k ~ N r S9 ~ lf? N ~ ~ O. r M N ILt~
~''~°~<Ca~°~QQaaQQQ~rrQQ.~cflQ QQ~~~QQa
00 Ln 'd' ~ w lf~ 1' w w w w. w w ('~ O ~. w N ~ w w d' 00 Cfl w w w
r~ 00 N Z Z p N Z Z Z Z Z Z Z ~ ~ '~' Z Z N' ~ Z Z Z ~ ~ ~''~ Z Z Z
r r M ~ ~ N M ~ ~k ~ ~ ~ ~k ~ ~ d' h, ~ ~ N r' ~ ~ ~k 'd' N M ~
~ppaa~~aaaaaaa~NraaMra aa°~°'MaQa
~'~° ôz'zM~zzzzzzz°°ôMzzô~z z'zô~"'~zzz
r r ~ ~ r d' ~ ~ ~ ~ ~ ~ ~k ~ CO N ~ ~ 00 M ~ ~ ~ N ~ M ~k
00O O ~- N M d'~ CO
OO O O O r r ~' r r r r
NN N N N N N N N N N N
X X X X
XX
rd N C'~M M M d' (n
'
N_ N N UXN N ~ 'U C' Lf 'd N tn
~
1 ~ ~ ~ I ? 1 ~ I 1 M Mr ~ r ~ r r
1 1-~i-~.1-~-i-~-I~
1-ir+r-Ir.irX ,~.1.-i.Ir~(i~-1-r~ ~~ ~ ~ ~ ~ 1
.FJ
r
U UU U C ~ ~ .UU U ~ .UU U U. . , . . N N I ~ Lnd'M C'~
' ~ ~
i i1 . , T 1 1 i 1 I i i X X.L.~.1-n.N-Fr~ X X 1-~
1 U U U U U ~ U U X
' '
M MM d d Lf~0000O O O r r ~ ~. . U . ~ ~ ~ U U U ~ _ N
' i ~ 1 ~
O OO O O O O O O O O r'r 1 I1 1 1 'UI 1 ~ ~ ~ 1 '
Q ~ U 'DN . !_
' !
O OO O O O O O O O O O O c0I. 1 1 1 1 1 I 1 , , 1 1 . -
1I - 1 1 1
1
1 I1 I 1 1 I 1 1 1 1 1 1 1 ~~ ~ ~ ~ ~ ~ ~ ~M ~ ~ ~ ~ '~Q ~
~ ~~ ~ ~ ~ ~ ~ ~ ~ ~ ~ ~ ~
J JJ J J J J J J J J J J J JJ J J J -JJ J J J J J J J J J
X
Y YY Y Y Y Y Y Y Y Y Y Y Y YY Y Y Y Y Y Y Y Y Y Y Y Y Y Y
~
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
74
N r-
a~ a
N
a~~- ~a
mn M
Z ~ d' r- Z
M M d
Mea ~a
N -
c-~ w
W ~
Z Z
r ~
I'
~
N
c
0
0o o
M Z
M
M M
M c~0 M : ~t a
CNO ~
~ c0 ~-- ~k
Ö.
°~ tn tn N ~k
r
~~ ° ~z
M M ~ 00. ~k
oQ
~~'c~o i z
° r co ~
~N
N N N N
C
M N .;n .;n
C_ C_ .Ç C
~ ~
~ ~ . --
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
L
> O ~ O d' N O ~ M M ~ ~ r Q a a ~ M O f~ O CO Q a a
w Z (p d' tn r O O d' r lI~ w w w d' M O 00 M CO w w w O f' O w
y- M r d~ O r i' N cfl cfl ~ Z Z Z d' p r N ~ r Z Z Z ~ M Z
~ Cfl O ~ lt~ O r d' tf~ ~ N ~ ~k ~ r r ~- N r r ~ *k ~ N r
Q L!J
J
(B
O
~~~NO~o~~~~~~~QQQ~~°~,°~~~QQQ~~~Q
%) L1J ~ M [w d' d' 00 r tL~ ~ N N Z Z Z ~ I~ ~ M O M Z Z Z O N CO Z
1. ~ ~ cM- I' M ~ N r- cN- N ~ r ~ ~k ~ r M r N M r ~ ~ ~k N r
J
r
N r oC d' r ~ a0 ~ 00 O d' M
aaa a rraaaaaQCOON
:V ~ O O N r r t~ d' O M Lt~ Cfl w w w 00 w M O CO w w w w w w N
~ 'p N CO N O O d' r- r M o0 N Z Z Z M Z ~ M M Z Z Z Z Z Z ô ~
J Z O lf~ 00 00 N 00 r 'd' d' 00 N ~ ~ ~ r ~k r d' ~ ~ ~ ~k ~ ~ r M
a ~ _
LIJ U, ~ ~ ~ trO r ~ M ~ ~ N ~ Q Q Q ~ Q ppp Ö M Q a a d a a oMO dM.
Q ~ m O O 1~ ~ ~ Z Z Z ~ Z N o ~ Z Z Z Z Z Z ~ ~'
1~ ~ CO N ~ N N O N ~ N ~ ~ ~ r ~ N N N ~ ~ ~ ~ ~ ~k M d'
Op CO 'd' Ln tC~ N O f~ M 00 N Q a M CO Ln O r r In a r 00 Q M N N
U X L N 1~~ r l(7 I' N CO 'Cf' t~ 00 CO ~ w f~ I~~ N ~' r r 00 w O 00 w ~ d' O
O O r M r O d' O N N CO M Z Z r O ln O OO d' M Z O O Z O CQ Ln
J Z IW' [w I' N f~ r M M 00 r ~ ~ r r r r r N r ~ r r ~ r Op Ln
N
(~
.O
i _
~ 1' ~ O t~ ~ ~ M O p Q Q tn ~ l'p~ ~ N M ~ Q M O Q ~ Ö oM0
O r' f~ N 'd' ~ O d' Ln CO ~ Z Z ~ M f~ r O ~ Ln Z ~ N Z O Cfl O
0- LLI p ~ L(7 N ~ N r O ~ ~ ~ ~ r r r r M ~ M ~ r N r
J
d0" ~ ~ 'd' 00 CO 1~~ N ln Gfl d' r CO ln N
Ln ln O M r 0p r a r ln I' Q M a a a N Q Q Q ,~ a a a a a ~ Q
X O r ~ 00 cfl N ~ Z r d' oO Z ~ Z Z Z r Z Z Z ~- Z Z Z Z Z n1 Z
J Z d' r N N r N ~ r r N ~ ~ ~~ ~ r ~ ~ ~~ r ~ ~ ~ ~ ~ ~j'
N
O
O M O) I' Cfl O) Q O 00 N Q M a Q Q r a a a a a a a a a
N 'd' N r N 1' w M f~ 1~. w O w w w Lf~ w w w p w v s. w w ~ w
~ x m M r ti' ici I~~ ~- Z d' O I~ Z '~ Z Z Z M Z Z Z ~ Z Z Z Z Z ~ Z
p_ J N M r ln M I~~ ~ N Cfl M ~ r ~k ~ ~k r ~k ~k ~k ~k ~k *k ~ ~k ~
Z
r r
a
w
N M ~ ~~ ~ ~ tp
i ~ i i
:,_, N M d'LnC C C C CC C _Cr N N d'-rj.+~.+~+~..M
O ~ N N C~d tn~ N M d'j~~~<>CX X>CX 7C~ C C C
'C N N N ~ ~ ~ ~ N ~N ~ ~ OLO O O
~ X X X OX X ~ .'-'.''"'_'''_''".'_'.,r.,r.".~+-..~.~.~+~O O O O O
X
7 O O O OO O U U U .U,UU _UU .U.UU .U.C~ ,C p O O O O
. . , ~ i ~ i ~ ~i ~ ~ T T T T i i i i i
a a a aa a a ~ i a a a a a aa a a a a a a a a a a a
a a
gi ~ ~ ~ ~ gi ~ ~ ~
~ . ~ ~ .. .. . . . .
a o a
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
76
NQQr_QQ_NQQOMMONQNOMQOOOQQd_'QOQ_Q_Q_QM_Q
Z Z ~ Z r Z Z N Z ~ ~' N M ~ Z mn o Z mn Z Z o o Z Z Z Z d' Z
~~ ~~ ~. ~k ~ *k ~ Cfl ~ ln M r M Cfl ~ r r M ~ N CO ~ ~ d' I' ~ ~ ~ ~k O i~
Q Q 'n Q N Q Q ~ Q ao co M O»r Q d' o~ o Q d' co Q Q r ~ Q Q Q Q ~ Q
w w Ln W.f~ w w Cfl w M I~. O M Iln w c- 00 M w CO ln w W~. O w w w yp ~~
Z Z t~ Z O Z Z ~ Z r M O W r' Z M N r Z ~ M Z Z 'd' Z Z Z Z Z
~k r ~k M ~~ ~k fer. ~k N Cfl r r M ~ r r M ~ 00 I~ ~ ~ Ln
Qaooo~oQQ~Q~ÖQQ~c~°MQm.~°n°r'°~~QQ~~QQQQMQ
Z Z o T o Z Z cet' Z M °° Z Z N h o Z N C~ M O Z Z M ~.c~ Z
Z Z Z N Z
~ ~ ~' r O *k *k tn ~ ~ rte'., *k ~k ~ M r *k d- r N lf~ ~k ~k M. d- ~k ~ ~k ~
(' ~k
Q Q o0 0 cet' Q Q r Q o d- Q Q e'> N o d N C~ r~ ~f' Q Q o oNO Q Q Q d M d
wwLt~l~.NwwOw.MOwwQO0000wCflMCflM~,w1'Ow,w,ww,~,~s
Z Z co c~ d' Z Z M Z o' o Z Z d' o ~"' Z N ~''~ oo ai Z Z d' Z Z Z Z Z
~ ~k r CO M ~ ~k Cp ~k N 'd' ~ ~ N r N ~ ~, M tO Cfl ~ ~ d. Or ~ ~
I~. ~t t~ Q ~ N Q M N ~ ~ ~ M O d' fer. N N a0 Cfl M O C0 N ln
r 0 Z O CNO Ö tc~ 000; due" z OO ~ ~ tf~ O l~ lU ~ Z Z N
r r M ~ ~ r ~k N CHI ~ ~, ~ CV N CV r r M ~ M t~ O r N M r O ~ ~ CO M
oo~Q~oQ~~oooQooormoco coi co~o.efl
O (~ yp w y-- M f' w N O M lue. oO M Q f~ O M OO N N GO '~h Q Q N O
[w., M N Z O CO Z Ln d' 00 N Z d' d' Q O O N Z O Cfl M r O M c- ~? Z' Z M ~'
r,.Oc-~kNO~MNrd"~Nrc-rM.M~l~.O)OrMI'c-00~~~0)
v co m o o d~ cfl M o r M ,d Q o o Q 4 Q Q co d'
Q c- LC1 N_ C~ a C_O r Ip Q M O Q ~ O w w M O ~., w w..~ 0 l.f~
M ~ r ~ M ~ ~ ~ o 'd' ~i' ~ r N r ~ M r ~ M ~ ~ ~ O O' ~ ~ ~k ~ ~ r
°~'' Q c~ Q o Q Q ~ aMO ~ r Q ~ ,.n M Q ,~ ~ Q Ö Ö Q Q ~ ° Q Q Q
Q N ~
~ Z pp Z N Z Z ~ o r. cy Z o~ t!) O Z ~p r Z c~ ~ Z Z O M Z Z Z Z o cet'
~ ~k ~' ~k M ~k ~k r N ~- r *k r O M ~ r r ~ r- ~ ~k ~ r N ~ ~k ~ ~k ~ r
O r N M 'd' ~ COI~QOO O r N M d'
N ~ N N M M M M M
r r r r r ~ r r r r r r r r
r N N .Nf.. r !n ~ N ~ cj-
r +~ X C X X X
~ ~ p . p -U ~ N ln N N p
' r N N r r N ~ N N r d'M M d' M M N Lf~M
X X
~ _C _ C C ~ N ~ C N ~ N N ~ ~ N
C X . M M ~ N M ' M Mr r r ' , M M , , , , M, , C'~~ i
''-'
p p p ; +~.,r.~U .,r; .~.NiGX X ~ .r--,....X +-I..r.er.~Xr-..i-.X .a.i.~-.,r
+.L+..~-..+.. ' r-r U U U U X U U U U U
U ~U U U C C X i C U C Cp p p ,U .Cp , . , p p
- ;, ~,i ' ' W N ' 'i' '~ ~ i i i i ~ W i ~ i ~ i i ~ i
i.n' O O O O r N M M d'LnLnCOCOa000O O O O O Or r N M M M
Op
O OO O O O O r O r r r r~ r r ~-r r r ~ r N N NN N N N N N
O OO O O O O O O O O O OO O O O O O O O O O O OO O O O O O
Q Q Q Q Q Q Q Q Q Q
a.~a a.a a.a.~ a a.a.~ ~a a.a a.a a.a a.a.~ a a.a.a a.a ~ a
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
77
Q a ~_''~ r N ~ N a a _a _a Q r ~ r _Q M m ~ o a M m Q ~ ch d- a ~ ~
Z Z ~ ~ o~o ~ Ö z Z Z Z Z mn ~ Z o~ M agio M z ~ ~ ~ ao o~ O w ~ r~
z N tf7 z t~ d-
M M f~ i.n 00 ~ ~k ~k ~k ~k ~- O M ~ d' r c- ~.f~ ~ ~ M ~k ~ c- r ~ N r.
QQp COOC'O~o00gaaQQy.Nn.,~ Q~yd NQo°o~Q~~~Q°~.°~t
ZZO N.I'OOZZZZZON~Zr-oOCflOzrOZ tn~Zd'M
d- N N M N ~k ~ ~ *k ~k N N ~ ~ N M c- r ~k r M ~ N' ~ N ~ r ~
CV Nt~MCflaoaaaorMa~~a~o~Qa~Maaaa
Cfl O O O O r 00 w c- w w w 00 O d' w d' Iln w 00 ~ 00 w w O M w w w w
00 0o r. oo N u~ co Z co Z Z Z N O d- Z r~. r z O ao N Z z M z Z Z Z
N r N M 00 M 'd' ~ c- ~ *k ~ c- 00 c- ~ M r ~ Cfl r ~- ~k ~ ~ ~ ~k ~k ~ ~k
MMd' d''Ntn,~apaQa~-~rQ a ~ O
c- O N - c-: O h. 00 w r w w. d' ~ r- s ~ d0 ' N .~ LN Q Q ~ -C~'O Q Q Q Q
~ t~ co . d- o0 0 Z ~ Z Z Z' 'd' tn Z O OO z cfl 'N r' z Z O . Z Z Z Z
'~t'.NN NNMd'~kN~~k~kNNM~NN~NM-r.~k~~N~~
QQN p~~ONOQMOQd'~~M~OMONMÖa~Ö~My~Q~r
ZZN ~o00OZd'OZONr-N'ONr'd'i'Zc-NZMZrZ
~k N N d' M Ln ~ r N ~ r c- f~ M O M ~- r- CIWk c- r ~ ~ ~k ç- W r r
a Q Ö ~ ~ oo N Q M ~ Q ~''~ N M ~ ~ ~ o'ro M ~ Q m ~ Q c'to Q ~ Q N ~t
ZZ~ tl~(OM Z~~Z~d' I~~C~LnoONZd'.OZd'Z~.nZd.CO
~ N t- c- N ~ ~ O d' ~ O CV ~ tC) O c- N c- ~- ~k O ~,r~ ~ ~ ~ r ~ W oQ
a a ~° O a ao r _a a ~ Q M M a ~ r~ oo ~' r-
Z Z'un ,b z .~ ~ Z Z ~ agio 2 ~ M Ö z M ~ ° z Z Z c~o ~o M:4 ~ a ~
Q
Z ~ Z Z
~T ~~kr~-~~r ~ ~-Pic- P~~,~kMc-N,~c-~O,~k
Q a ~ ~ Q ~ ~ a d N ~ a ~ ~ ~°. Q O CO M a a a ~ ~ ~ Q N Q ~ g
Z Z o0 O Z ~ M Z Z O ~ Z ~ I~ c- Z ~ ~ O Z Z Z m!? cf' O Z C4 Z r Z
r ~- ~ ~ ~k ~ ~- c- ~ d' s- ~ r ~~ ~ ~ c- N M ~ M *k r
LnCOt'00 O O ~- N M d' Ln Cflf~ 00O O
COCOo000 00 O O O O O O OO ~ O O
r r r ~ c- c-e- ~ ~- r~- c-c- N
X~XX X r C CC U ~ ~ ~ C
N
X
.~.N N Nd' -U _ ' 'T
X M ~ ~ X XX ~ cet'1 d~'d'M ~ ~ tn~~ ~ ,N.N M
, X
'
. .,....,...,+..,.r
U UU U .a-N ~ C ~ N N~ ~ X N ~ X ~ r ~ 1 C~ C ~ C C
U
1 11 _I _ ~ X ' ' .f~.r~-1r~ C1di-F~s. ~ ~ F~.1-.i~ .1~ . .F~" ' 1 ~
I U ~ .1r j .!r 1
~
U tiV .UUU U V .UU V V ~ V .CUU U U V V
N NN N O ' 1 . . 1 '' ' 1 .
I 11 I
O OO O O 'a'pl,:y! .~L t . Y Y ~ ~ E E C C s'.L I 1
I I1 tn l ~ ~
1 1 I I 1 1 I 11 1 1 1 1 1 ' 1 1 1 1 11 I 1 1 1
a aa a~ a a a a a a a a a a a 1 a a a a aa a a a a a a
fO(n(nV7 (n(!~fn(!Jfn(n(nU~U7CnfnU a
X ~ fOU7(!~fl~f/~fnt~(l~!OfI~fnfnfn(n
a aa aa~ a a a a a a
. . . . . a an. a.n.cL~ a.a a a.a a.a a ~ n.~ a.a
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
7ô
N Q N f~ ~ ~ M N M d tn Cp d' d Q N M Q d d Q d M N Q d ~ M I~
O w r O O f~ Ln M w Ln 00 M w w CO O w w w w w ~ O w. w r M f~
O Z C4 ~ O c0 M M O Z r M O Z Z M O Z Z Z Z Z N N Z Z N r M
r ~ M M N ~- N N CO ~ ~- M N ~ ~ r N ~ ~ ~ ~ ~ r N ~ ~ d' 00 N
r
r d O r L~j O N r M a r M O d d ~ N d d d d d o ~ d d ~' N °°
N W h 00 00 f~ O w lO N w w Cfl 00 w w w w w Cfl N w w I' N
d- Z M o o N ~ c"~ o Z o ~' M Z Z N M Z Z Z Z Z ~ o Z Z ~ N N
r ~k N M N N 00 M T ~ N ~ M ~ ~k c- r ~~ ~ ~k ~ ~ r N ~ ~ d' ~ N
d N CO 1' N Ln r O O d M O N d I' d d a 'n d d d d r ~''~ d o ° d-
w r M Cfl 00 Cfl d' N N w r 00 d' w 00 w w w O w w w w O f~ w c-
z d- N ~t M M ~ cfl r z M cfl o~ Z o Z Z Z o Z Z Z Z N d' Z o o M
N N N CO r r r ~' ~k r N r ~ N ~k ~ ~ N ~k *k *k ~k r c- ~ M d' r
d h. t~ o ô o ~ ~r m Q ao r M d ao d d d ~° d d d d ° ° d
N o~o 00
w O 'd" O M I' Ln d' M w O CO 00 w O w w w r w w w w Ln CO w M CO
Z M t~. M O co ~t d~ Z 'd' N ~' Z o Z Z Z ~ Z Z Z Z ~ t~ Z N ~' ~
~ N r N ~ N ~ N a0 ~ N O N ~ r ~ ~ ~k r ~ ~ ~ ~ r M ~ M
M d tfl O N ~- N 00 ln N tf~ N N d Cfl d' 00 d d d Cfl a V7 ln Ln d CO O r
N w N d' O O CO N N ~ O CO O w N O t~ w w w M w N Cfl o0 w O f~ a0
d- Z r O I' N It~ O r r O 00 I' Z r O M Z Z Z M Z O N O Z CO 00 tn
r ~ C~/ r Cp r r N d' r r r r ~ N r r ~ ~ ~ r ~ r r ~ ~ N M r
co ~ o ~ M o°°o Q Wn coo Q Q Q ~ Q ~ c''no M Q M ô
~ Z w cfl o co ~ ~ ~n o 0o Z o o ~- Z Z Z ~ Z r M t- Z r
r ~ 'c'-' r ~ r Cp M o r fw r ~ r r r ~ ~ ~ r ~ r r N ~ M o c-
Maa p~OOONj aaoôQQQ~QQQ~QQOCOQÖN~-'
O 00 ~ ~ 000 ~ ~ ~k O ~ ~ ~k ~k O ~k ~k *k M ~k ~ O ~ ~ r ~ O
N d d oo N M ~t d. ~ d d r~ o d Q d r d d d ° d d N 'n d cfl M o0
r .~ .~ O O N N O w w Cfl d' w w w O w w w O w WO , w d' CO M
M Z Z N ~ O O O O Z Z d' o Z Z Z ~ Z Z Z M Z Z M N Z o N N
r- *k ~ r M c- N O r ~k ~k N r ~ ~ ~ r ~~ ~k ~ M ~ ~ ~ N ~ r- M r
rN
N N N
NN
M d' M
X X X
~ ~ ~ ~ ~ ~ ~ ~N c'~ N
X ~ ~ ~ ~~ ~ ~ ~ r N M ~
N N '~d'O , , , 'r,N M d'. . . .~. . ~,+,,X X .~',,
N M d'~ ~~+-.
.
O C C ( C l r N M 'd'C C C C X X X X X X X.U,UO p U
C C
p ~' C X X X Xp O O O N N O O N N O
_i O O O O O O N O N NL,s-s-L , , , , , , ,N N N M M
. ,~.,r-,-..~...~.-,-.~.,r.,rr .,~.,-~.,~.,-~O O O O O
V ~ ~ ~ ~ N ~ -~+ . UC_C_C_C_U U U U U U U0 0 0 0 0
U U U '
~ ~
' ' N N N N N N . ' ' '' ' ' ' ' ' ' ' ' , ', , , ' ,
'~i'~ , , , , , , ' N N N N N N N N
Q dd Y Y Y Y Y Y ~ Y Y ~~ ~ ~ Y ~ ~ ~ Y ~ Y Y~ ~ ~ Y Y
(n(nfn J J J J J J J J J JJ J J J J J J J J J JJ J J J J
a.~a Y Y Y Y Y Y Y Y Y YY Y Y Y Y Y Y Y Y Y YY Y Y Y Y
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
79
Naa~~a~aaa~-~~a~Qaa°~ Q~~~N~~-~-aN
(fl w w 1~. f' w r w w w Iw N CO w C~ w w ~ N 00 w M M O N ln M r w O
° Z Z ° M Z M Z Z Z c'~ co d' Z c~ Z Z Z ~ o Z t~ ~ d' ~ N C~ Z
'~r
N ~ ~ M r ~k c- ~k ~k *k c- N Ln ~ N ~ ~ ~ r N ~ N N ~ c- N N N ~ d'
N a a ° ° a °~ 4 Q a °~ ~ ~ a ° a Q Q
~''~ °~ a o ~.n ~ M cfl r N a d'
ln w w- Op w 00 w w. M w M w w w M M w r CO M Cfl d' Ln w N
M Z Z N O Z r Z Z Z f~ M ~ Z N Z Z Z M ~' Z f~ d' O O f' 'd' f' Z d'
r ~ ~ N r ~ r ~ ~ ~' ~ O O ~~ r ~~ ~k ~ (V r ~k M ~ N N ln 00 d' ~ r~
~ a Q Q d' O a a Q Q M oo M Q o~ Q a a °° a a Q Q ~"~ ao m N ~ Q
cfl
CO w w Cfl O w w w w f' I' r w c- w w a M w a w w CO d' O N O~ w r
p Z Z Z d' O Z Z Z' Z ~- d' O Z M Z Z Z M Z Z Z Z O r N O ~''~ Z d'
r c-. ~ ~ ~ ~ c- r 'd' ~ r ~ ~k ~ c- ~k ~ ~ ~ t0 r N c- r ~ d-
N Q Q Q r' M.Q a Q Q N ~ ~ Q.p Q Q Q N a 4 4 Q i ° ~ d' ~ Q
O?w,wwOLI~w,wwNl'Mwd'.~.wwMw. ~~~swMNd'OI'wM
d' Z Z Z ~ ~''~ Z Z Z Z o ° Z ao Z Z Z M Z Z Z Z ° ° o ~
Z ~
r ~ ~ ~ N N .~ ~ ~ ~ ~ ~ ~ ~, ~ ~ ~ ~~ (fil ~ ~ ~ ~ o r cj' I~. N ~ r
N tue.' O O fer. tue', Q Q ~ O ~ ~( O N Q lO CO N, a Q CO o0 CO cfl d' 00 N
'CO
N c-. '~h Cfl O N' ~ O w. O f~ CO ~ N Cfl w M c- Cfl w w O O lO O r d' ln
'~.5~
CO O Ln N d' O N M Z Z ~- 'tel, f~ Z r N Z r lln- N Z Z d' CO r 00 M d' d' 00
r CO N N r O O? r ~ ~ ~ r N ~ N c- *k N c- c- ~ ~ N tn r r N r r N
M cet' ~ ~ r~ ~ Q Q ~. ~ ~ Q co ~d- Q o' cfl Q Q M ô '.n N co ~- 00
N~-aOCOOpoOMw.~NM w.OMwM0C0 w.w~ MMNCfl~.d'
O d' N f~ d' M ~ ~c- Z z M oO N Z oO O Z 'd" I~. O Z Z O ~ ~ ~- ~ fer. O . Q
r t50 c- c- N M O) M ~ ~ r d' T ~ N r- *k O. ~ N ~ ~ N ~ r cf' C~ N M r
Qaaa~QagQa~~~aQQQ~M~ dQQ~~~~QQ~
Z Z Z Z ~ Z Z Z Z Z ~ ~ ° Z Z Z Z M ~ M Z~ Z~ Z~ N ~ ~ ~ Z~ ~Z ~
~k ~k ~k ~k ~k ~k ~k *k *k N ~ *k ~:k ~k c- c-
aaaa~aa.aaQ M~aaaa~ ~ aaar N~QQ~.
a a w w O v ~. w w p M O w w w w d' d' O w w w Cfl O lit N w w-
Z Z Z Z N Z Z Z Z Z '~ d' ao Z Z Z Z r- d" ° Z Z Z °
°~ ~''» Z Z °
~ ~k ~ ~ r ~ ~ ~kk ~k ~k ~ r 1' ~~ ~k ~k ~k r M M ~k ~k ~k N p ~- r ~ ~k r
C~f~ 00O O r- N M d'Ln (p 1'
OO O O r' ~- r ~ ~ ' ~ r
~
NN N N N N N N N N N N
XX X X X
C ~
NN N U U . C_r
-
dN M c~M c'~d' ~ C ~%
'
_N ~ ~ ~ N N 'Cr M e-Lf~r d" ~ tO
C d'd'd' d' MM N c'
'
Ti ~ ~ ~ ~ ~ s-.i...~+-..f-.+. ~ C
.....,..~.+r+~X .+~,t...,.rX .~..~-.CC r d' X .p
C C C C C ~
UU C C U U U U U XX r . . . . X X r ~ M X ~ M ~ d' '
' ! ' : ~
-,+ .,+ + .I '-' .
r . ~ .
M~ld'Lf~0000O O O r c-~U ,UU U U U U ~ .U f' U ,CU U X V V
' ' ' ~ ' '
OO O O O O O O O 'c-~ ii ~ ~ ',i ~ ~ , i . . ~ T i i N .
.
OO O O O O O O O O O (~(~.a~ U 'a~ N ~ N .,!.,'_ .C.C._.' '
' ' '
~i ~ ~ i i i i ~ ~ i ~i ~ i ~ ~ ~ ~ ~ i ~ i W ~ i i ~ i i
~~ ~ ~ ~ ~ ~ ~ ~ ~ ~ ~~ ~ ~ ~ ~ ~ ~ ~ ~ ~ ~ ~ ~ ~ ~
M
~ ~ ~ ~ ~ ~~ ~ ~ ~ ~ ~ ~ ~ ~ ~ ~ ~ ~ ~ ~ ~
~ ~ ~ ~ ~ ~ ~ ~ ~
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
~a
N
N OQ w
M d' z
N CO ~
M N Q
N w
d Z
d' M ~
o,
~z
N
N N z.
'M N
N r Z
M *k
Q ~a
z ~z
a ~a
Z MZ
c-
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
81
L
O
~~~~~~NÖQNÖ~Qaa~a~Q~NaaQa
~k N ~ 000 ~k ~ ~ ~ ~k N ~ N ~ *k ~k ~
O
J
N
Q~~(~tt~~~MMMQ~M~aQQNQ~Q~~QQaQ
~ C~ ~ Ö ~ ~ N dO' ~ c j o~O Ö ~ ~k ~k ~ ~k ~ ~ N ~ ~k ~
X ~ r ~ ~ M ~- c-
W
J
L
O
~ E ~ ~ ~ a'~o ~ ~ a ~ ~ ~t° Q ~ due- Q Q Q ô Q N Q Q Q c~
z M N O o M ~ ~ N ~ M ~ ~ ~ ~k ~ ~k N ~k r-
N [~
M J
M
1
O C
°-~C~ôô~ô~d c'toQôc~o~Q~~QQQNQ~QQQ~_
m N r M d0' ~ N ~k ~ N ~ ~k ~ ~ ~ *k ~ ~ ~ ~ ~ *k ~ O
W
J
L
O
O~NM~N_QCOM00_Q_QoON_Q_Qd'd'a0_QQ_
~
7
~N
~
M
~
~
~h ZZZ
'p ZZ
~
C
p~rNC
O~Zd
"
'~~ZZd
C4
~
~k
~
r-
~k
~
M
c-
N
~k
N
d'
~
~
N
d' ~
X ~
Z r,
OJ
N
CO
o0
CO
c-
f~
O
Q
CO
Cfl
N
Q
Q
d'
Q
Q
~O
~
O
Q
Q
Q
W"
M
O C~ w
w-
O
O
~
w
N
I'
O
00
~-
M
~
O_
O
M
~
~
r-
~
~
~
M
Xi r'
m ~k
*k
~k
~
~
'
N
~
~
~
~
M
~k
~k
~
M
N
~
d
W
J
L
O M
ao
~ ~ M
~'
r.
~
Q
ao
O
r~
Q
oo
O
Q
Q
ao
m-
ao
Q
Q
Q
~.n
f~
lO
o0
O
O
~
w
N
O
M
w
tn
O)
~
~
cfl
o0
d'
O
~
w
Wp
M O
~
M
T
C~O
N
~
M
~
~
~k
~
~
~
~k
~
M
N
M
~k
*k
~k
r'
X Z
~ LIJ
,rte,
J
C C
O .-
~ ~
~ M
~
0
m ~ ZZZ
'O d' 'MNMZNOOZN~ZZMN
1 I
~.d
ln
N
~
l'
M
~
M
~
~
*k
~
~
~
~k
~
M
N
N
J
~ Z co
N ~ ~
O C
U~ 1 1 I 1 .1--,.i..n.1-i.V.Y.I--1.ir.1--I
1 1 .Q1 1 1 CVM d'ln.CC C C CC C _C~-N N
. .. .
c- N NM d'~ ~ N M d'j~X X X XX X X ~
C C CC C G X X X O O
U O O OO O O X N N UiO O O O OO O O O O L L r
Ui . 1 1 1 1 I1 1 1 L L .Ir
. ~ ~ r .,r.,.r.....,.~.,...,..
~.I~ X X XX X X +-~r .±,..,...+U UU U U C C ~ C O
V V V V V V U
N N ON O N . . . . 1 , 1 , .. . . . . . 1
1 1 11 1 1 1 1 1 1 Q i Q 1 1I 1 1 Q Q
Q Q QQ Q Q Q Q Q Q Q Q QQ a Q Q Q Q
a ~ a~ a.a.a.a a.a a.~ a.a an.a.a.a.a.a a.a.
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
82
o~ d~ o~ _Q Q M O~ _ r Q Q N Q O O a Q O Q O M Q Q cfl r r Q O r Q Q
~ N ~ Z Z ~ r Z ~ Z Z M Z ~ N Z Z od'o Z ~ ~ Z Z c~ N 'd Z c~o ~ Z Z
r r CO ~ ~ ~' r ~ 'd' ~ ~ r ~ CO CO ~ ~ r ~ M M ~ ~ d' ~ r ~ M CO ~ ~
~°. ~ ~ Q Q ~ o Q ~ Q Q ~ Q ~ ~ Q Q ~ Q ~ N Q Q o ô _~ _Q o o _Q Q_
r CW.c~ Z Z I~ O Z M Z Z d- Z O CO Z Z O Z ~ M Z Z ~ N N Z ~ ~ Z Z
r r ~ ~k ~k r M ~ N *k ~k r ~ ~ ~ ~k *k N ~ ~ ~ ~k ~k d' r r ~ M l~ ~k ~
r O r Q Q d' N Q N Q Q Q Q d' lCS Q CU C~) a Q a a a ~ ~ a a a o Q _a
Ln d' M ~ ~ O~ 'd' ~ N ~ w w w 00 r w 00 f' r ~~ w w
r r O Z Z CO Ch Z C4 Z Z Z Z M OO Z O ~. Z Z Z Z Z M O Z Z Z ~ Z Z
N N tt~ ~k ~ r e- ~ N ~ ~k ~ ~k M d' ~k r ~' ~k ~k ~k ~ ~k r M ~k ~ ~ ~f7 ~ ~k
cfl tf?QQd'.NQNQQQ~C~r'Q~' QQQQQ M_Q_Q_QO_QQ_
~ M O ' ~ w w w w r O ~ 00 0 w w w w w 0
O N CQ Z Z r r Z O Z Z Z Z- 00 00 Z N N Z Z Z Z Z M Ö > Z Z Z ~ Z Z
r- M d' ~ ~k r r ~* r ~ ~ ~k ~ N N *k r ~ ~k ~k ~k ~k ~ r' N ~ *k ~k N ~ ~
~c°~o~aQa°oN~d°.QQQQ~d~.,QQ~.~Q CQQ~~QQN~QQ
Z Z O CO Z ~ Z Z Z Z r ~ Z Z N M Z Z Z Z M cfl Z Z r d' Z Z
'd' r ~ ~ . ~k r CV ~$ f~ ~k ~ ~k ~ tD tO ~k ~k c- O ~ ~k *k ~k N cet' ~k *k N
l' ~k *k
a'no N ~ Q Q ~ ~ Q ,d Q Q Q Q Ö h Q Q ~ o Q Q Q Q r ,~ Q Q ~ ~ C Q
r- ~n N Z Z oo' co Z i.n Z Z Z Z O N Z Z r °° Z Z Z Z r' N Z. Z
°~ O Z Z
M r 00 ~k ~k r r ~ M ~ ~k ~ ~ d' d' ~k *k ~- O ~k ~k ~ ~k O N ~ ~ O M ~k ~
d' O O d' Q N r G~ o a a a Q ~ t~ Q Q ~- Q Q aC_ Q Q o c~ cn. Q c~ N Q Q
Cfl r 00 00 ~~.' M O '~. M ~ w w ~ O Ln ~ ~ ~ '~ w, w y' O N: ~ p N w W
O tp 4) III Z I' tp Z I~ Z Z Z Z r M Z Z C0 2 Z Z Z Z r M N Z o~ ô Z Z
N N O r ~~ N N ~ M ~ ~k ~ ~k Cfl O ~k ~ r ~~ ~$ ~* ~$ ~~ ~ (p M:~ M
c~ co o~ oo Q ~ aa Q M a a Q a M ~ Q Q ~ Q a a a a o M ~ Q ~- ~- Q Q
O O O '~ ' 00 Cfl ~ O ~ w w w M (~ w w ~ w w w w w ('~ Ln O ~ O Cfl w,
CU d- N d- Z 'dw O Z I~ Z Z Z Z O O Z Z r Z Z Z Z Z O O N Z o0 0 Z Z
N N I~. r ~~ N r ~$ N ~ ~k ~ ~k Ln IIO *k ~ r ~ ~$ ~$ ~k ~k M f~ N ~$ M OO ~$
*k
O O r N M d- ln CO~ 00O O r N
O ~ ~ N ~ h ~ ~ ~ ~ ~ O M M
r r r r c- r r r r r r r r r
r N N -Nf.' r tn N N ln
+ C
C . ~ ' N ~ N ' ~ ln N N
C
'~
-t-. r r';rr ~ ~ r X r X N N T r d'M M d' M M
N X X ~
' c . '
M M'TM ~ O '~N 'TM M ~ ~ M'~M M r r r '~~ M M~ ~ ~ ~ M
..+.,,r+~U '~-..N.~.~..X X X -~-'''--'.'-.X.~.+~.~'~.~X "'.+~
C C~ C U U U U C C X ' CU C C
. . . . . i , . . O O O .U.U. OU .U.U_XO U U
'T'T' ' ' '
T T ~ ~ i ~ T T ~ N Tr T T ~ i i ~ i , .
r rM M d'd'~ 00O O O O r NM M 'd'Lnln(fl00CO00OO O O O O r r
O OO O O O O O O O O r O rr r r r r r r r r rr r N N N N N
O OO O O O O O O O O O O OO O O O O O O O O OO O O O O O O
Q Q Q Q Q Q Q Q Q QQ Q Q
a aa a a a a.~ a a a.a a aa ~ a.a a a'a.a.a a~ a a a c~a a
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
83
a~Maaa~Q~~NQaQaaô~~a~ya~aQaQ~aQ
zoo.zzza,zr~~-~ozzzzz~r~ozoo,z~-zzzz~zz
~kr~P.~k~k~kr"~~~~~k*k~~k~r- jN~kr-w~~f~~k~k~~~~k~
Q Q M Q O O cfl Q Q Q Q Q ~ ~ N Q cfl o0 Q cfl a a a a °° Q
Q
O 1' f' w w w w yp O 00 w Cfl M w M w w ~ w M w w
Z ~ ~ Z Z Z ~- Z N ~ M Z Z Z Z Z m ~ ao Z O N Z eo Z Z Z Z ~''~ Z Z
~k ~' ~ ~k ~ ~k N *k ~- r- I~ ~k ~ ~k ~k ~ r- N r- *k ~ r- y.n ~ ~k *k ~k N ~k
~
aa~aaaQ ~~~oQQaQa~~Q~Q_~aQaa°°aa
w, O w w w w O d' M 'd' M ~ w w, yp M w _(p w O [w w w w w ~ w,
~k ~k o0 ~ ~ ~ ~ 0 ~ ~ ~ N ~k *k ~k ~ ~ ~ M ~ ~- ~k O N ~k ~k ~k ~k ~ ~ ~k
Q Q N Q Q Q : Q pp M ~- M I~ Q Q Q Q Q
~k ~k due' ~k ~k ~ ~ oo ~ ~ N ~ ~ *k ~k ~k ~k N ~ ~k °o ~ ~ ~ ~ ~k ~k
~k ~ ~k ~
QQMQQQQ~~~~c~°omQQQ~~.° 4~,~Q~Q~ CQ~~d
Z Z Z Z Z Z oo co d' o~ ao Z Z Z N N Z O N Z N Zw.n Z Z °~ N Z
~k N M N M ~ ~ ~ ~ ~ r O~ d' ~k N N *k M
Q Q ô Q ~ Q Q ~ ~ ô ~ ~ ~ Q Q Q ~- ~ ~ Q O w~ Q N Q N Q Q ~ oo Q
Z Z N Z Z Z Z o ~t i.c~ t~. O M Z Z Z o t~ O Z c~ r- Z N Z N Z~' Z
°~ °~ Z
~ *k C0 ~k *k ~ ~k ~ N ~ M c- ~'' *k ~k ~k ~' d' N ~ c- r- *k N ~ ~' ~k ~k N ~
~
_QQ~_Q4_a_cfloo~ooQoaQQ~~~_<CcoM_Q~QoQdMQ4
Z Z M Z Z Z .~ ~ M y Z ~ Z Z Z M O d~° Z'~ Ö Z ~ ~ N ~ 1 1
Z o Z Z °~ Z Z
~-- r- M ('~ ~ ~ r ~k ~ ~k N N d' ~ N M ~ M ~k M ~ ~ M
QQMaaa~M.~°c~o'~o~Q~aQQ~~~~~ ôadM'a~Qa~aa
zz~zzzoc~o~N~zozzzM~~-zNO~z~ozozz~zz
~k ~ N ~ ~k ~ ~- ~ N M ~ ~k ~- ~k ~k ~k N N d' ~ N N ~ N ~k M ~k ~k
M d'~ COf~00 O) O c- N M d' ~ COf' 00O
M M M M M M . M O O O O O O O O O O
U
U
<-
~ N ~ ~ C C C ~' ~
NLn M C ~-~ ln' C C
XX X X X X ';' -; _ ~ ~
~ ~ N N N ~ , M ~ NN N d' ~ .d.d'~ ,~.~ ~~-. ~ sr.Lf~
~ : . N ~ ~
X .r.+..~.,.~.-~.~..,+ .~..X .,...+..X X X X ~C X
UU U U U U U ~ ~ X X , O
O . . , , U N O C CN O N c'~O N O ~ O c-. c-' N .
w . ., , , .,.... '
.
, ,, , , , , , , -c-. +.~.,-..i-.~.. , , .r-.~, . .,..~-.-,!..~.,r.,r
N MM M LOCpi'f'~ U X ~-'-~-'U U U U X .,...U .V C U U U U
U
. V V . . j
N NN N N N N N O ',~ V .U.~.~.~.~~ .
O OO O O O O O O ~ 'Oy!a,! L .~~ . ~ ~ ~ ~_EE C C ~ i".
, - ' . ~
~
,, , , , , m , , , , ,, , , , , , , , T , ,, , , , , , ,
a aa a a a a a a a a a aa a a a a a a a a a aa a a a a a a
~ a.a.a a a a.a.a a a a n_a.a a a.a a a.a a ~ aa.~ n.~ a.a a.
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
~4
Q Q Q Q ~' ~ N M ~ O OO Q Cfl I' Q Q Q Q Q Q Q Q Q M a Q
w w. w Ln N ~ d' ~1' f~ O ~ I~ CO w w w w w w w w w p O w.
Z Z Z Z Z Z d' c0 M tn o0 0~ '~1' Z f~ r Z Z Z Z Z Z Z Z Z ~ 00 Z Z
~k ~ *k ~ ~k ~k ~ CO M N N ~ ~ *k tp d' *k ~ ~k ~k ~ ~ ~k ~ ~k O ~ ~k ~k
aaQQaQ ~~~N~~PaWQaaQaQaQaap~QQ
Z Z Z Z Z Z d- ~ N M N oo 'r Z M N Z Z Z Z Z Z Z Z Z ~ oo Z Z
~k ~k ~ *k ~k ~k c- CO M M M ln ~ ~k I' d' ~ ~k ~ ~k *k *k *k *k ~ O ~ ~k ~k
aaaQaQ a~gQ~~QQ~~aaQaMQaaaar~~ja
~k ~ ~k ~k ~ ~k ~k ~ ~k ~ ~ M ~ ~ N ~ *k ~k ~k ~ ~ *k ~k ~ ~k *k °0 ~
~k
Qa'aQQa a~QQ~~QQ~~aQQaMQQQQa~,~Q
~ ~k ~k ~k ~k ~ ~ N ~k ~k COO ~ ~k ~k O ~ ~k ~k ~k ~k ~ ~k ~k ~k *k *k ~ "NV.'
~
Q p O Q Q ~ d' M O d' Cfl '~' Q Q M N Q a Q Q a a a Q a Q ~ ~ Q
c~ co d- o~ o~ 00 00 ~ ~ r. o~
Z ~ r- Z Z O M ~ ~t N N ~ Z Z oo m Z Z Z Z Z Z Z Z Z Z M Z Z
~k ~- *k ~k ~ c- CO ~- N r- N ~k ~ ~ M ~k ~k ~k *k ~ ~k ~k ~k ~k ~k r- ~k ~k
QQ 00 f~ OOQ_QCfld'Q__Q_Q_QQ_Q_Q__Q_Q_QNQ_Q_
ZÖ~ZZ~ NoMO~N'~oMOZZ~ÖZZZZZZZZZZÖZZ
~k ~ O ~k ~k M M N O c- ~' ~ ~k ~k N N ~ ~k ~k ~k ~k ~ *k ~ ~k *k ~ ~k ~k
'N f~f~V7CflN_OQ_QO~NQ_Q_QQ_Q_QQ_Q~~Q~Q
Z Z Z Z Z ~ dM- ~ ~ o co o°o Z Z M ~ i~ Z Z Z Z Z Z Z Z ~ N Z Z
*k ~ ~ ~k ~ r' N O N d' N CO ~ ~k r d' N ~k ~k ~k *k ~ ~ ~k ~k ~- M '~ ~
p c-OLnMONQ_Q~~MQ_QQQQQQQM~"~Q_Q_
ZZZZZc~- opON~~r-aOZZMMNZZZZZZZZd'NZZ
~ N ~ 00 N M M o0 ~k ~ ~ Cfl ~ ~ ~k ~k ~k ~k ~k ~k ~k r- M
N N N N N
M d'
X X
~ ~ ~ ~ ~~ ~ ~
X ~~ ~ ~ N X
U U c-N M d'Cfl1'00
ii ~ i
C'~d.+~Lnd'd. c-N N i ~ ~ NM d'Lni ~ ~ ~ C C C G C C '.T..~,
M d'lf~i~ i ~ c-N M d'' ' ' '' 'T' U U
; ; ; ii
~ ~ ~ C C C C C C ~N M d' C C C X X X XX X X , y
+'. . "' X X X X X X UN ~ ~ ~ O O O N ~ ~ N~ ~ ~ N N
! r ~ ..~.r ~ +'~.~.+...~.
.~
. , +..+..~+...~.~. . . .,..- O O
U ,U,, U ~ U U N U N N UU U U C C C C U U U UU U U O O
UU V . . . . . .. .
. ~. , , , , , , .,.,.,.,.~.~~ ~ , , , ,, , , , ,
s..tncO-~...~~ N N N N N N
Q Q QQ Q Q ~ ~ ~ ~ ~ ~ ~~ ~ ~ ~ ~ ~ ~ ~ ~ ~ ~~ ~ ~ ~ ~
~ ~ ~ ~ ~ ~ ~~ ~ ~ ~ ~ ~ ~ ~ ~ ~ ~~ ~ ~ ~ ~
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
cO cet' M Q a Q ~ M a Q d' N Q o ~' ~ d. Q a a ~ ~ a a a a ~ cO r' o ao
M O c- w, w w (fl i.n w w M Ln '~ N O w w w d- w w w w 07 [w, c- ['
f~ N ~ Z Z Z O N Z Z O I' Z ~ 00 ~ r' Z Z Z ~ cO Z Z Z Z ~' C~ I~ d' M
N O c- *k ~ ~ M M ~$ ~ d' 00 *k r- d' ~ p *k ~ ~ M r- ~k ~ ~ ~k ~ N M d' M
d d d ~ Q Q ~ Q p ~ 't a a d ~ Q Q ~C Q °~ C~ N N
[w,~Mwww~~,wwONwOOM'MwwW'pwwwwMc-d'ON
N~~~k~k~k~C'~~*k~~~.~k~~N~~k~k~~~~k~k~k~~Nd~'~M
d d d ~ r' d d O d N d' O d a d d N 'd' M d d d N d d Cfl lf~
r- (w. w w w ~ pp w w p w ~ LO w w w w ~ N, w. w w [w w w (O M
~- N ~ ~ ~~ ~k ~k r- ~ ~ ~ N ~k M ~ ~ ~ ~k ~k ~ ~ N d0.. ~ ~ ~ N ~ ~ r r
~~,~aQaa~~aa~:a'môMaQaQ~.~~aaa~aa~p
~oozzzzoozzoz~r~zzzz~MM.zzz~nzz~~
oOr,O~~k'~~r-r-~~kN~k~~N~k~k~k~-~c-r-~k~kW~*k~kMOO
co N d <C Q Q Q o cO Q Q M ~ r- ~ cm Q Q Q Q N N ~' Q d Q ~ r- ter' M O
OOswwwwO~~ww~~-f'O w.ww(OI~~~.wwMLnLn~Ln
M ~ ~k *k ~k ~k ~k M ~ ~k ~ ~ ~ O C~i p ~: ~k ~k ~ ~ ~ ~ ~k ~k ~k ~ ~ M i' i N
~ ~ a: d d Q Q M cet' Q d cet' d. ~ r~. N 4 Q Q Q ~ M m at Q Q c~ ao o r- M
d' O '~ w w w w O O w -w O N f' ~ w a y, M r w, w w- d' O M d'
~0 ~ ~ ~ ~ ~ ~k N ~ ~ ~ M M p r dN' ~ *k ~k ~ ~ O ~ Z Z Z Ln d' cO l~ l(~
M c- c- c- ~-
d'NQddQQ~rQ~,Q~oaop~~,pQQ~,~dddQ°cor~O~r-
(W, w. w w ~ wJ- . w i' I~ 00 O w Ln w w [w. ~ w. w w Lf~ O N CO
O °~ Z Z Z Z Z M Z ao Z t' ai ~ M ~ Z d- Z Z o~ oo Z Z Z Z ~ c~ oo
~ ~
~ ~k ~ 'd' ~ c- ~$ (fl d' ~ 00 ~ ~ ~- ~k ~ c- M ~$ ~~ ~k ~k ~ N 'd' I' M
~ ~ d d d d Q ~ Q ~ d ~- M N O ~ Q ao Q Q o M Q d d Q ~ ~ r- M O
L(7 w w w w w '~' w c- w CO 00 I~ lf~ ~ d' ~ ~ N O w w. W' M Cfl O
d' N ~k ~k ~k ~k ~k ti» ~ ~k ~ ~ N M ~ ~k ~ ~ ~ ~ N *k ~k ~k ~k ~ N d~' ~ lti
lf~COfer. OCO O ~ N M d'L(7Cfl
O O O O O r' r' r' ~ r r'
N N N N N N N N N N N N
X X X X X X
N
.
N ~ ~ N N N N ~ 'U- M ~ d0 ~ tn
d'O, , d'd'd', , , d', , MM c .,rc-.r~
.,-. ,r, +-. '
-~+.+r+r+~ .~+~+. r .v.. d .Q
X X X X ~~ ~ O
N NU U U C C N U U U N U U OO ' . ~ . N CV~ N ln~'M M
. . . . . . . . . . M '
, ,, , , , , , , , , , XX .r-..,.r.,.r.~+.X X .+J, VX .f...,-..rd
N MM M d'~'~ QO0007O O ~ ~ OO U U U U U O O U O C U U X
O OO O O O O O O O O O ~ r-,, . ',',',',, , . . ., . ',',O
, ,
O OO O O O O O O O O O O O (afB~ .QU 'O'DN O O y.y!
'
, ,, , , , , , , , , , , , ,, , , , , , , , , , ,, n , , ,
~ ~~ ~ ~ ~ ~ ~ ~ ~ ~ ~ ~ ~ ~~ ~ ~ ~ ~ ~ ~ ~
~ ~~ ~ ~ ~ ~ ~ ~ ~ ~ ~ ~ ~ ~~ ~ ~ ~ ~ ~ ~ ~ ~ ~ ~~ ~ ~ ~ ~
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
86
Z Z ~ N Z
# ~ ~ d. ~.
Q Q N ~ Q
w w M O w
Z Z O O Z
*k *k ~- dWk
Q ~ N N Q
~k ~ O N ~
Q CV d~ N Q
Z~d~MZ
~ ~ N d'
Mt
z~~.r~oZ
~~~N~
z°iM~z,
z°;c~~z
~~N~o~
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
1
SÉQUENCE LISTING
<110> Exonhit Therapeutics
<120> Variants de kallikrein-2 et kallikrein-3 humaines et leurs
utilisations
<130> B0132W0
<150> FR0203186
<151> 2002-03-14
<150> FR0210975
<151> 2002-09-05
<160> 220
<170> PatentIn version 3.1
<210> 1
<211> 550
<212> DNA
<213> Homo sapiens
<400> 1
cctgtgtcag catgtgggac ctggttctct ccatcgcctt gtctgtgggg tgcactggtg 60
ccgtgcccct catccagtct cggattgtgg gaggctggga gtgcgagaag cattcccaac 120
cctggcaggt ggctgtgtac agtcatggat gggcacactg tgggggtgtc ctggtgcacc 180
cccagtgggt gctcacagct gcccattgcc taaagaagta agtaggaccc tgggatctgg 240
ggagggaatg gctgtgtccc acaggaataa cagcgggatg cttcccccag ggtcacttct 300
caggaatagc caggtctggc tgggtcggca caacctgttt gagcctgaag acacaggcca 360
aagggtccct gtcagccaca gcttcccaca cccgctctac aatatgagcc ttctgaagca 420
tcaaagcctt agaccagatg aagactccag ccatgacctc atgctgcttc gcctgtcaga 480
gcctgccaag atcacagatg ttgtgaagga gtcttcagtg tgtgagcctc catctcctgt 540
ccaatgacat 550
<2l0> 2
<211> 688
<212> DNA
<213> Homo sapiens
<400> 2
cctgtgtcag catgtgggac ctggttctct ccatcgcctt gtctgtgggg tgcactggaa 60
tagccaggtc tggctgggtc gccacaacct gtttgagcct gaagacacag gccagagggt 120
ccctgtcagc cacagcttcc cacacccgct ctacaatatg agccttctga agcatcaaag 180
ccttagacca gatgaagact ccagccatga cctcatgctg ctccgcctgt cagagcotgc 240
caagatcaca gatgttgtga aggtcctggg cctgcccacc caggagccag cactggggac 300
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
2
cacctgccacgcctcaggctggggcagcatcgaaccagaggagttcttgcgccccaggag 360
tcttcagtgtgtgagcctccatctcctgtccaatgacatgt~gtgctagagcttactctga 420
gaaggtgacagagttcatgttgtgtgctgggctctggacaggtggtaaagacacttgtgg 480
ggtgagtcatccctactcccaacatctggaggggaaagggtgattctgggggtccacttg 540
tctgtaatggtgtgcttcaaggtatcacatcatggggccctgagccatgtgccctgcctg 600
aaaagcctgctgtgtacaccaaggtggtgcattaccggaagtggatcaaggacaccatcg 660
cagccaacccctgagtgcccctgtccca 688
<210> 3
<211> 436
<212> DNA
<213> Homo sapiens
<400> 3
cctgtgtcag catgtgggac ctggttctct ccatcgcctt gtctgtgggg tgcactggtg 60
ccgtgcccct catccagtct cggattgtgg gaggctggga gtgtgagaag cattcccaac 120
cctggcaggt ggctgtgtac agtcatggat gggcacactg tgggggtgtc ctggtgcacc 180
cccagtgggt gctcacagct gcccattgcc taaagaatct tgcgccccag gagtcttcag 240
tgtgtgagcc tccatctcct gtccaatgac atgtgtgcta gagcttactc tgagaaggtg 300
acagagttca tgttgtgtgc tgggctctgg acaggtggta aagacacttg tggggtgagt 360
catccctact cccaacatct ggaggggaaa agtgagtgaa gaccctaatt ctgggctgca 420
atctgaaagc taacca 436
<210> 4
<211> 476
<212> DNA
<213> Homo sapiens
<400>
4
cctgtgtcagcatgtgggacctggttctctccatcgccttgtctgtggggtgcactggtg 60
ccgtgcccctcatccagtctcggattgtgggaggctgggagtgtgagaagcattcccaac 120
cctggcaggtggctgtgtacagtcatggatgggcacactgtgggggtgtcctggtgcacc 180
cccagtgggtgctcacagctgcccattgcctaaagaagaatagccaggtctggctgggtc 240
ggcacaacctgtttgagcctgaagacacaggccagagggtccctgtcagccacagcttcc 300
cacacccgctctacaatatgagccttctgaagcatcaaagccttagaccagatgaagact 360
ccagccatgacctcatgctgcttcgcctgtcagagcctgccaagatcacagatgttgtga 420
aggagtcttcagtgtgtgagcctccatctcctgtccaatgacatgtgtgctagagc 476
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
3
<210>
<211>
723
<212>
DNA
<213> sapiens
Homo
<400>
5
cctgtgtcagcatgtgggacctggttctctccatcgccttgtctgtggggtgcactggtg 60
ccgtgcccctcatccagtctcggattgtgggaggctgggagtgtgagaagcattcccaac 120
cctggcaggtggctgtgtacagtcatggatgggcacactgtgggggtgtcctggtgcacc 180
cccagtgggtgctcacagctgcccattgcctaaagaagaatagccaggtctggctgggtc 240
ggcacaacctgtttgagcctgaagacacaggccagagggtccctgtcagccacagcttcc 300
cacacccgctctacaatatgagccttctgaagcatcaaagccttagaccagatgaagact 360
ccagccatgacctcatgctgctccgcctgtcagagcctgccaagatcacagatgttgtga 420
aggtcctgggcctgcccacccaggagccagcactggggaccacctgctacgcctcaggct 480
ggggcagcatcgaaccagaggagttcttgcgccccaggagtcttcagtgtgtgagcctcc 540
atctcctgtccaatgacatgtgtgctagagcttactctgagaaggtgacagagttcatgt 600
tgtgtgctgggctctggacaggtggtaaagacacttgtggggtgagtcatccctactccc 660
aacatctggaggggaaaggtgagtgaagaccctaattctgggctgcaatctgaaagctaa 720
cca 723
<210> 6
<211> 409
<212> DNA
<213> Homo sapiens
<400> 6
cctgtgtcag catgtgggac ctggttctct ccatcgcctt gtctgtgggg tgcactggtg 60
ccgtgcccct catccagtct cggattgtgg gaggctggga gtgtgagaag cattcccaac 120
cctggcaggt ggctgtgtac agtcatggat gggcacactg tgggggtgtc ctggtgcacc 180
cccagtgggt gctcacagct gcccattgcc taaagaagaa tagccaggtc tggctgggtc 240
ggcacaacct gtttgagcct gaagacacag gccagagggt ccctgtcagc cacagcttcc 300
cacagccgct ctacaatatg agccttctga agcatcaaag ccttagacca gatgaagact 360
ccagcatcga accagaggag tgagtcttca gtgtgtgagc ctccatctc 409
<210> 7
<211> 595
<212> DNA
<213> Homo sapiens
<400> 7
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
4
gcatgtggga cctggttctc tccatcgcct tgtctgtggg gtgcactggt gccgtgcccc 60
tcatccagtc tcggattgtg ggaggctggg agtgtgagaa gcattcccaa ccctggcagg 120
tggctgtgta cagtcatgga tgggcacact gtgggggtgt cctggtgcac ccccagtggg 180
tgctcacagc tgcccattgc ctaaagaaga atagccaggt ctggctgggt cggcacaacc 240
tgtttgagcctgaagacacaggccagagggtccctgtcagccacagcttcccacacccgc300
tctacaatatgagccttctgaagcatcaaagccttagaccagatgaagactccagccatg360
acctcatgctgctccgcctgtcagagcctgccaagatcacagatgttgtgaaggtcctgg420
gcctgcccacccaggagccagcactggggaccacctgctacgcctcaggctggggcagca480
tcgaaccagaggagtggtaaagacacttgtggggtgagtcatccctactcccaacatctg540
gaggggaaaggtgagtgaagaccctaattctgggctgcaatctgaaagctaacca 595
<210> 8
<211> 350
<212> DNA
<213> Homo sapiens
<400>
8
ggttctctccatcgccttgtctgtggggtgcactggtgagattggggggataaaggaagg60
ggggcgggttctgactcttatgctgaagcccttttcgtcccacccagtgccccagcctcg120
tcccttcagcccacagttcagcccagacaatgtgcccctgactcttccccccgaggctat180
ctggcctgagacaacaaatgctgcctcccaccccgagtctggcactgggactttcagaac240
tcctcctttcctgactctttgccccagacccgtcattcaatggctagctttttccatggg300
aaaaacacga gcacccccaa ccacaacggc cagttctctg attccctaaa 350
<210> 9
<211> 118
<212> DNA
<213> Homo sapiens
<400> 9
ggttctctcc atcgccttgt ctgtggggtg cactgacccg tcattcaatg gctagctttt 60
tccatgggaa aaacacgagc acccccaacc acaacggcca gttctctgat tccctaaa 118
<210> 10
<211> 159
<212> DNA
<213> Homo sapiens
<400> 10
ggttctctcc atcgccttgt ctgtggggtg cactggtgag attgggggga taaaggaagg 60
ggggcgggtt ctgactctta tgctgaagcc cttttcctcc cacccagtgc cccagcctcg 120
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
tccccccaac cacaacggcc agttctctga ttccctaaa 159
<210> 11
<211> 293
<212> DNA
<213> Homo sapiens
<400> 11
cagtcatgga tgggcacact gtgggggtgc tttggagccc ccccagcggg gccgtacaca 60
ccatgcaccc tggccgtacc gaaactagtg gaaccctgct atctgccgag ctagaggcat 120
agctcgacta ttctatagtg gcaactatac ccccaggttt ttttctcagg aatagccagg 180
tctggctggg tcggcacaac ctgtttgagc ctgaagacac aggccagagg gtccctgtca 240
gccacagctt cccacacccg ctctacaata tgagccttct gaagcatcaa agc 293
<210> 12
<211> 313
<212> DNA
<213> Homo sapiens
<400> 12
cagtcatgga tgggcacact cctgttttct aactcactgt ctgtatttca ccacgactat 60
atctccccga cccctgtgct tttctcactg tttctttttc ttccctttgg agtctccctt 120
atcctcccct gccccatcta cctttcccca ttttctctct cctcatgcat ccaccccctt 180
cctccccagg aatagccagg tctggctggg tcggcacaac ctgtttgagc ctgaagacac 240
aggccagagg gtccctgtca gccacagcct cccacacccg ctctacaata tgagccttct 300
gaagcatcaa agc 313
<210> 13
<211> 211
<212> DNA
<213> Homo sapiens
<400> 13
agctcaatgt gtgtgcatgt gagggggtgc ctgtcattgc acagcactct ctgcaggaca 60
tCCCtCCdCC C'tCatgCCtC tCCtCCtCCC CCtgCtaCtC CdC3CtCCt C agatgCCCCC 120
gtggcctccc tcctttttct ctcccacact gtatcacccc tggcttcctc tctgctgttt 180
ctccttctct ctctgacttc ccgcatcctt t 211
<210> 14
<211> 216
<2l2> DNA
<213> Homo sapiens
<400> 14
ggagtgacga tgaggatgac ctgggggtgg ctccaggcat tgtccccacc tgggccctca 60
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
6
cccagcctcc ctcacaggct cctggccctc agtctctccc ctccactcca ttctccacct 120
acccacagtg ggtcattctg atcactgaac tgaccatgcc agccctgccg atggcccact 180
tgtctgtaat ggggtgcttc aaggtatcac atcatg 216
<210> 15
<211> 159
<212> DNA
<213> Homo sapiens
<400> 15
ccactacaga gccctcactc cagccccagc tgcaggaata gccaggtctg gctgggtcgg 60
cacaacctgt ttgagcctga agacacaggc cagagggtcc ctgtcagcca cagcttccca 120
cacccgctct acaatatgag ccttctgaag catcaaagc 159
<210> 16
<2l1> 455
<212> DNA
<213> Homo sapiens
<400>
16
ggagagctgtgtcaccatgtgggtcccggttgtcttcctcaccctgtccgtgacgtggat60
tggtgagaggggccatggtgggggggatgcaggagagggagccatccctgactgtcaagc120
tgaggctctttgccccccaacccagcaccccagctcccagctgctttactaaaggggaag180
ttcctgggcatctccgtgtttctctttgtggggctcaaaacctccaaggacctctctcaa240
tgccattggttccttggaccgtatcactggtccatctcctgagcccctcaatcctatcac300
agtctactgacttttcccattcagctgtgagtgtccaaccctatcccagagaccttgatg360
cttggcctcccaatcttgccctaggatacccagatgccaaccagacacctccttcttcct420
agccaggctatctggcctgagacaacaaatgggtc 455
<210> 17
<211> 444
<212> DNA
<213> Homo sapiens
<400>
17
ggagagctgtgtcaccatgtgggtcccggttgtcttcctcaccctgtccgtgacgtggat60
tggtgagaggggccatggttggggggatgcaggagagggagccagccctgactgtcaagc120
tgaggctctttcccccccaacccagcaccccagcccagacagggagctgggctcttttct180
gtctctcccagccccactccaagcccatacccccagcccctcccacttaccccagaactt240
tctccccattgcccagccagctccctgctcccagctgctttactaaaggggaagttcctg300
ggcatctccgtgtttctctttgtggggctcaaaacctccaaggacctctctcaatgccat360
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
7
tggttccttg gaccgtatca ctggtccatc tcctgagccc ctcaatccta tcacagtcta 420
ctgacttttc ccattcagct gtga 444
<210> 18
<211> 193
<212> DNA
<213> Homo sapiens
<400> 18
ggagagctgt gtcaccatgt gggtcccggt tgtcttcctc accctgtccg tgacgtggat 60
tgctgtgagt gtccaaccct atcccagaga ccttgatgct tggcctccca atcttgccct 120
aggataccca gatgccaacc agacacctcc ttcttcctag ccaggctatc tggcctgaga 180
caacaaatgg gtc 193
<210> 19
<21l> 256
<212> DNA
<213> Homo sapiens
<400> 19
ggagagctgt gtcaccatgt gggtcccggt tgtcttcctc accctgtccg tgacgtggat 60
tggtgagagg ggccatggtt ggggggatgc aggagaggga gccagccctg actgtcaagc 120
tgaggctctt tccccccaac cctatcccag agaccttgat gcttggcctc ccaatcttgc 180
cctaggatac ccagatgcca accagacacc tccttcttcc tagccaggct atctggcctg 240
agacaacaaa tgggtc 256
<210> 20
<211> 159
<212> DNA
<213> Homo sapiens
<400> 20
ggagagctgt gtcaccatgt gggtcccggt tgtcttcccc aaccctatcc cagagacctt 60
gatgcttggc ctcccaatct tgccctagga tacccagatg ccaaccagac acctccttct 120
tcctagccag gctatctggc ctgagacaac aaatgggtc 159
<210> 21
<211> 133
<212> DNA
<213> Homo sapiens
<400> 21
ggagagctgt gtcaccatgt tggtcccggt tgtcttcctc accctgtccg tgacgtggat 60
tggataccca gatgccaacc agacacctcc ttcttcctag ccaggctatc tggcctgaga 120
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
ô
caacaaatgg gtc 133
<210> 22
<211> 767
<212> DNA
<213> Homo sapiens
<400>
22
ggagagctgtgtcaccatgtgggtcccggttgtcttcctcaccctgtccgtgacgtggat 60
tggtgctgcacccctcatcctgtctcggattgtgggaggctgggagtgcgagaagcattc 120
ccaaccctggcaggtgcttgtggcctctcgtggcagggcagtctgcggcggtgttctggt l80
gcacccccagtgggtcctcacagctgcccactgcatcaggaagtgagtaggggcctgggg 240
tctggggagcaggtgtctgtgtcccagagaaataacagctgggcattttccccaggataa 300
cctctaaggccagccttgggactgggggagagagggaaagttctggttcaggtcacatgg 360
ggaggcagggttggggctggaccaccctccccatggctgcctgggtctccatctgtgtcc 420
ctctatgtctctttgtgtcgctttcattatgtctcttggtaactggcttcggttgtgtct 480
tttcgtgggactattatgttgttttttttcctctcttctctgtcttcagtatccatatct 540
ccgcctcttattccattctttcttcaggacatccctcctctcttgggaggcgcagagaag 600
gagtggttcc agctggagct gggaggggca attgagggag gaggaaggag aagggggaag 660
gaaaacaggg tatgggggaa aggaccctgg ggagggaagt ggaggatgca tccttgggcc 720
tgcaggccag gctgcctacc cacttggaaa cccacgccaa agccgca 767
<210> 23
<211> 394
<212> DNA
<213> Homo sapiens
<400>
23
ggagatgtgtcaccatgtgggtcccggttgtcttcctcaccctgtccgtgacgtggattg 60
gtgctgcacccctcatcctgtctcggattgtgggaggctgggagtgcgagaagcattccc 120
aaccctggcaggtgcttgtggcctctcgtggcagggcagtctgcggcggtgttctggtgc 180
acccccagtgggtcctcacagctgcccactgcatcaggaatctccatatccccccctctc 240
tctgtccttctctagtccctctctagccagtgtgtctcaccctgtatctctctgccaggc 300
tctgtctctcggtctctgtctcacctgtgccttctccctactgagcacacgcatgggatg 360
ggcctggggg gaccctgaga aaaggaaggg cttt 394
<210> 24
<211> 328
<212> DNA
<213> Homo sapiens
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
9
<400> 24
ggagagctgt gtcaccatgt gggtcccggt tgtcttcctc accctgtccg tgacgtggat 60
tgcaccccct ctgcaggcgc tgcgcccctc atcctgtctc ggattgtggg aggctgggag 120
tgcgagaagc attcccaacc ctggcaggtg cttgtggcct ctcgtggcag ggcagtctgc 180
ggcggtgttc tggtgcaccc ccagtgggtc ctcacagctg cccactgcat caggaagtga 240
gtaggggcct ggggtctggg gagcaggtgt ctgtgtccca gaggaataac agctgggcat 300
tttccccagg ataacctcta aggccagc 328
<210> 25
<211> 226
<212> DNA
<213> Homo sapiens
<400> 25
ggagagctgt gtcaccatgt gggtcccggt tgtcttcctc accctgagct tgtggcctct 60
cgtggcaggg cagtctgcgg cggtgttctg gtgcaccccc agtgggtccc cacagctgcc 120
cactgcatca ggaagtgagt aggggcctgg ggtctgggga gcaggtgtct gtgtcccaga 180
ggaataacag ctgggcattt tccccaggat aacctctaag gccagc 226
<210> 26
<211> 220
<212> DNA
<213> Homo sapiens
<400> 26
ggagagctgt gtcaccatgt gggtcccggt tgtcttcctc accctgtccg tgacgtggat 60
tgggcagtct gcggcggtgt tctggtgcac ccccagtggg tcctcacagc tgcccactgc 120
atcaggaagt gagtaggggc ctggggtctg gggagcaggt gtctgtgtcc cagaggaata 180
acagctgggc attttcccca ggataacctc taaggccagc 220
<2l0> 27
<211> 805
<212> DNA
<213> Homo sapiens
<400>
27
ggagagctgtgtcaccatgtgggtcccggttgtcttcctcaccctgtccgtgacgtggat60
tggagctgcgcccctcatcctgtctcggattgtgggaggctgggagtgcgagaagcattc120
ccaaccctggcaggtgcttgtggcctctcgtggcagggcagtctgcggcggtgttctggt180
gcacccccagtgggtcctcacagctgcccactgcatcaggaagtgagtaggggcctgggg240
tctggggagcaggtgtctgtgtcccagaggaataacagctgggcattttccccaggataa300
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
cctctaaggccagccttgggactgggggagagagggaaagttctggttcaggtcacatgg360
ggaggcagggttggggctggaccaccctccccatggctgcctgggtctccatctgtgtcc420
ctctatgtctctttgtgtcgctttcattatgtctcttggtaactggcttcggttgtgtct480
ctccgtgtgactattttgttctctctctccctctcttctctgtcttcagtctccatatct540
ccccctctctctgtccttctctggtccctctctagccagtgtgtctcaccctgtatctct600
ctgccaggctctgtctctcggtctctgtctcacctgtgccttctcccttctgaacacacg660
cacgggatgggcctggggggaccctgagaaaagaaaggaccctggggagcgaagtggagg720
atacaaccttgggcctgcaggccaggctacctacccacttggaaacccacgccaaagccg780
catctacagctgagccactctgagg 805
<210> 28
<211> 684
<212> DNA
<213> Homo sapiens
<400>
28
ggagagctgtgtcaccatgtgggtcccggttgtcttcctcaccctgtccgtgacgtggat60
tggcgctgcgcccctcatcctgtctcggattgtgggaggctgggagtgcgagaagcattc120
ccaaccctggcaggtgcttgtggcctctcgtggcagggcagtctgcggcggtgttctggt180
gcacccccagtgggtcctcacagctgcccactgcatcaggaacaaaagcgtgatcttgct240
gggtcggcacagtctgtttcatcctgaagacacaggccaggtatttcaggtcagcctgcc300
gagatcacggatgctgtgaaggtcatggacctgcccacccaggagccagcactggggacc360
acctgctacgcctcaggctggggcagcattgaaccagaggagttcttgatgccaaagaaa420
cttcagcgtgtggatctccatgttatttgcaatgacgtgtgtgcgcaagttcagcctcag480
aaggtgatcaagttcatacggtgtgctggacgctggacagcgggcaaaagcacctgctgg540
ggtgattttgggggagcgcttgtctgtaatggtgtggttcaaggtatcatgtcatggtgc600
agtgatccatctatcctagccgaaaggagttgcctgtccaccaaggtggtgcattaccgg660
aagtggatcaaggacaggcatcgt 684
<210> 29
<2l1> 560
<212> DNA
<213> Homo sapiens
<400> 29
ggagagctgt gtcaccatgt gggtcccggt tgtcttcctc accctgtccg tgacgtggat 60
tggtgctgca cccctcatcc tgtctcggat tgtgggaggc tgggagtgcg agaagcattc 120
ccaaccctgg caggtgcttg tggcctctcg tggcagggca gtctgcggcg gtgttctggt 180
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
11
gcacccccagtgggtcctcacagctgcccactgcatcaggaagccaggtgatgactccag 240
cattgaaccagaggagttcttgaccccaaagaaacttcagtgtgtggacctccatgttat 300
ttccaatgacgtgtgtgcgcaagttcaccctcagaaggtgaccaagttcatgctgtgtgc 360
tggacgctggacagggggcaaaagcacctgctcgggtgattttgggggcccacttgtctg 420
taatggtgtgcttcaaggtatcacgtcatggggcagtgaaccatctatcctgcccgaaag 480
gccttccctgtacaccaaggtggtgcattaccggaagtggatcaaggacaccatcgtggc 540
caacccctga gcacccatat 560
<210>
30
<211>
541
<212>
DNA
<2l3>
Homo
sapiens
<400>
30
ggagagctgtgtcaccatgtgggtcccggttgtcttcctcaccctgtccgtgacgtggat 60
tggtgctgcacccctcatcctgtctcggattgtgggaggctgggagtgcgagaagcattc 120
ccaaccctggcaggtgcttgtggcctctcgtggcagggcagtctgcggcggtgttctggt 180
gcacccccagtgggtcctcacagctgcccactgcatcaggaacaaaagcgtgatcttgct 240
gggtcggcacagcctgtttcatcctgaagacacaggccaggtatttcaggtcagccacag 300
cttcccacacccgctctacgatatgagcctcctgaagaatcgattcctcaggccaggtga 360
tgactccagcattgaaccagaggagttcttgaccccaaagaaacttcagtgtgtggacct 420
ccatgttatttccaatgacgtgtgtgcgcaagttcaccctcagaaggtgaccaagttcat 480
gctgtgtgctggacgctggacagggggcaaaagcacctgctcgggtgattctgggggccc 540
a 541
<210> 31
<211> 525
<212> DNA
<213> Homo sapiens
<400>
31
tcctgtgtcagcatgtgggtcccggttgtcttcctcaccctgtccgtgacgtggattggc60
gctgcgcccctcatcctgtctcggattgtctctcgtggcagggcagtcggcggcggtgtt120
ctggtgcacccccagtgggtcctcacagctgcccactgcatcaggaagccaggtgatgac180
tccagccacgacctcatgctgctccgcctgtcagagcctgccgagatcacggatgctgtg240
aaggtcatggacctgcccacccaggagccagcactggggaccacctgctacgcctcaggc300
tggggcagcattgaaccagaggagtgtacgcctgggccagatgtcttgaccccaaagaaa360
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
12
cttcagtgtg tggacctcca tgttatttcc aatgacgtgt gtgcgcaagt tcaccctcag 420
aaggtgacca agttcatgct gtgtgctgga cgctggacag ggggcaaaag cacctgctcg 480
ggtgattctg ggggcccact tgtctgtaat ggggtgcttc aaggt 525
<210> 32
<211> 524
<212> DNA
<213> Homo sapiens
<400>
32
ggagagctgtgtcaccatgtgggtcccggttgtcttcctcaccctgtccgtgacgtggat60
tggcgctgcgcccctcatcctgtctcggattgtgggaggctgggagtgcgagaagcattc120
ccaaccctggcaggtgcttgtggcctctcgtggcagggcagtctgcggcggtgttctggtl80
gcacccccagtgggtcctcacagctgcccactgcatcaggagcttgaccccaaagaaact240
tcagtgtgtggacctccatgttatttccaatgacgtgtgtgcgcaagttcaccctcagaa300
ggtgaccaagttcatgctgtgtgctggacgctggacagggggcaaaagcacctgctgggg360
tgattctgggggcccacttgtctgtaatggtgtgcttcaaggtatcacgtcatggggcag420
tgaaccatgtgccctgcccgaaaggccttcgctgtacaccaaggtggtgcattaccggaa480
gtggatcaaggacaccatcgtggccaacccctgagcacccctat 524
<210> 33
<211> 430
<212> DNA
<2l3> Homo sapiens
<400> 33
ggagagctgtgtcaccatgtgggtcccggttgtcttcctcaccctgtccgtgacgtggat60
tggcgctgcgcccctcatcctgtctcggattgtgggaggctgggagtgcgagaagcattc120
ccaaccctggcaggtgcttgtggcctctcgtggcagggcagtctgcggcggtgttctggt180
gcacccccagtgggtcctcacagctgcccactgcatcaggaacaaaagcgtgatcttgct240
gggtcagcattgaaccagaggagttcttgaccccaaagaaacttcagtgtgtggacctcc300
atgttatttccaatgacgtgtgtgcgcaagttcaccctcagaaggtgaccaagttcatgc360
tgtgtgctggacgctggacagggggcaaaagcacctgctggggtgattctgggggcccac420
ctgtctgtaa 430
<210> 34
<211> 290
<212> DNA
<213> Homo sapiens
<400> 34
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
13
ggagagctgt gtcaccatgt gggtcccggt tgtcttcctc accctgtccg tgacgtggat 60
tggtgctgca cccctcatcc tgtctcggat tgtgggaggc tgggagtgcg agaagcattc 120
ccaaccctgg caggtgcttg tggcctctcg tggcagggca gtctgcggcg gtgttctggt 180
gcacccccag tgggtcctca cagctgccca ctgcatcagg aacaaaagcg tgatcttgct 240
gggtcgggtg attctggggg cccacttgtc tgtaatggtg tgcttcaagg 290
<210> 35
<211> 414
<212> DNA
<213> Homo sapiens
<400> 35
cgtgacgtggattggtgagaggggccatggttggggggatgcaggagagggagccagccc 60
tgactgtcaagctgaggctctttccccccaaacccagcaccccagctccctgctcccagc 120
tgctttactaaaggggaagttcctgggcatctccgtgtttctctttgtggggctcaaaac 180
ctccaaggacctctctcaatgccattggttccttggaccgtatcactggtccacctcctg 240
aggccctcaatcctatcacagtctactgacttttcccattcagctgtgagtgcccaaccc 300
tatcccagagaccttgatgcttggcctcccaatcttgccctaggatacccagatgccaac 360
cagacacctccttcttcctagccaggctatctggcctgagacaacaaatgggtc 414
<210> 36
<211> 201
<212> DNA
<213> Homo sapiens
<400> 36
tggcaggtgc ttgtggcctc tcgtggcagg gcagtctgcg gcggtgttct ggtgcacccc 60
cagtgggtcc tcacagctgc ccactgcacc tgctacgcct caggctgggg cagcattgaa 120
ccagaggagt tcttgacccc aaagaaactt cagtgtgtgg acctccatgt tatttccaat 180
gacgtgtgtg cgcaagttca c 201
<210> 37
<211> 475
<212> DNA
<213> Homo sapiens
<400> 37
tggcaggtgc ttgtggcctc tctcgtggca gggcagtctg cggcggtgtt ctggtgcgcc 60
cccagtgggt cctcacagct gcccactgca tcaggaagtg agtaggggcc tggggtctgg 120
ggagcaggtg tctgtgtccc agaggaataa cagctgggca ttttccccag gataacctct 180
aaggccagcc ttgggactgg gggagagagg gaaagttctg gttcaggtca catggggagg 240
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
14
cagggttggg gctggaccac cctccccatg gctgcctggg tctccatctg tgtccctcta 300
tgtctctttg tgtcgctttc attatgtctc ttggtaactg gcttcggttg tgtctctccg 360
tgtgactatt ttgttctctc tctccctctc ttctctgtct cacctgtgcc ttctctcttc 420
tgaacacacg cacgggatgg gcctgggggg accctgagaa aaggaagggc tttgg 475
<210> 38
<211> 444
<212> DNA
<213> Homo sapiens
<400>
38
gtccagcccacaacagtgtttttgcctggcccgtagtcttgaccccaaagaaacttcagt60
gtgtggacctccatgttacttccaatgacgtgtgtgcgcaagttcaccctcagaaggtga120
ccaagttcatgctgtgtgctggacgctggacagggggcaaaagcacctgctcggagctgg180
accctgaagtcccttccccaccggccaggactggagcccctacccctctgttggaatccc240
tgcccaccttcttctgggagtcggctctggagacatttctctcttcttccaaagctggga300
actgctatctgttatctgcctgtccaggtctgaaagataggattgcccaggcagaaactg360
ggactgacctatctcactctctccctgcttttacccttagggtgattctgggggcccact420
tgtctgtaatggtgtgcttcaagg 444
<210>
39
<211>
659
<212>
DNA
<213> sapiens
Homo
<400>
39
gtccagcccacaacagtgtttttgcctggcccgtagtcttgaccccaaagaaacttcagt60
gtgtggacctccatgttatttccaatgacgtgtgtgcgcaagttcaccctcagaaggtga120
ccaagttcatgctgtgtgctggacgctggacagggggcaaaagcacctgctcgtgggtca180
ttctgatcaccgtactgaccatgccagccctgccgatggtcctccatggctccctagtgc240
cctggagaggaggtgtctagtcaaagagtactcctggaaggtggcctctgtgaggagcca300
cggggacagcatcctgcagatggtcctggcccttgtcccaccgacctgtctacaaggact360
gtcctcgtggaccctcccctctgcacaggagctggaccctgaagtcccttccctaccggc420
caggactggagcccctacccctctgttggaatccctgcccaccttcttctggaagtcggc480
tctggagacatttctctcttcttccaaagctgggaactgctatctgttatctgcctgtcc540
aggtctgaaagataggattgcccaggcagaaactgggactgacctatctcactctctccc600
tgcttttacccttagggtgattctgggggcccacttgtctgtaatggtgtgcttcaagg 659
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
<210> 40
<211> 409
<212> DNA
<213> Homo sapiens
<400>
40
gtccagcccacaacagtgtttttgcctggcccgtagtcttgaccccaaagaaacttcagt60
gtgtggacctccatgttatttccaatgacgtgtgtgcgcaagttcaccctcagaaggtga120
ccaagttcatgctgtgtgctggacgctggacagggggcaaaagcacctgctcggtgagtc180
atccctacccctctgttggaatccctgcccaccttcttctggaagtcggctctggagaca240
tttctctcttcttccaaagctgggaactgctatctgttatctgcctgtccaggtctgaaa300
gataggattgcccaggcagaaactgggactgacctatctcactctctccctgcttttacc360
cttagggtgattctgggggcccacttgtctgtaatggtgtgcttcaagg 409
<210> 41
<211> 378
<212> DNA
<213> Homo sapiens
<400> 41
gtccagccca caacagtgtt tttgcctggc ccgtagtctt gaccccaaag aaacttcagt 60
gtgtggacct ccatgttact tccaatgacg tgtgtgcgca agttcaccct cagaaggtga 120
ccaagttcag cacacggtta ctaggcacct gctatgcacc cagcactgcc ctagagcctg 180
ggacatagca gtgaacagac agagagcagc ccctcccttc tgtagccccc aagccagtga 240
ggggcacagg caggaacagg gaccacaaca cagaaaagct ggagggtgtc aggaggtgat 300
caggctctcg gggagggaga aggggtgggg agtgtgactg ggaggagaca tcctgcagaa 360
ggtgggagtg agcaaaca 378
<210> 42
<211> 76
<212> DNA
<213> Homo sapiens
<400> 42
tcccagagac cttgatgctt ggcctcccaa tcttgcccta ggatacccag atgccaccag 60
ccaccaacct gcaaac 76
<210> 43
<211> 247
<212> DNA
<213> Homo sapiens
<400> 43
tcccagagac cttgatgctt ggcctcccaa tcttgcccta ggatacccag atgccaacca 60
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
16
gacacctcct tcttcctagc caggctatct ggcctgagac aacaaatggg tccctcagtc 120
tggcaatggg actctgagaa ctcctcattc cctgactctt agccccagac tcttcattca 180
gtggcccaca ttttccttag gaaaaacatg aagcctctcc accagcacca gccaccaacc 240
tgcaaac 247
<210> 44
<211> 73
<212> DNA
<213> Homo sapiens
<400> 44
gtcccggttg tcttcctcac cctgtccgtg acgtggattg ccaggctatc tggcctgaga 60
caacaaatgg gtc 73
<210> 45
<211> 255
<212> DNA
<213> Homo sapiens
<400> 45
ctgaacacacgcacgggatgggcctggggggaccctgagaaaaggaaggg ctttggctgg~0
gcgcggtggctcacacctgtaatcccagcactttgggaggccaaggcagg tagatcacct120
gaggtcaggagttcgagaccagcctggccaactggtgaaaccccatctct actaaaaata180
caaaaaattagccaggctacctacccacttggaaacccacgccaaagccg catctacagc240
tgagccactctgagg 255
<210> 46
<2l1> 70
<212> DNA
<213> Homo sapiens
<400> 46
gaacacacgc acgggatggg cctgggggga ccctgagaaa agccgcatct acagctgagc 60
cactctgagg 70
<210> 47
<211> 286
<212> DNA
<213> Homo sapiens
<400> 47
tgactccctc aaggcaatag gttattctta cagcagagag gaggttgcct ggccggagtg 60
aaggatcggg gcagggtgcg agagggaaga aaagacccct cctgcagggc ctcacctggg 120
ccacaggagg acactgcttt tcctctgagg agtcaggaac tgtggatggt gctggacaga 180
agcaggacag ggcctggctc tgccgctgcc agaggctgcc gctggcctcc ctatgggatc 240
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
17
agactgcagg gagggagggc agcagggatg tggagggagt gatgat 286
<210> 48
<211> 273
<212> DNA
<213> Homo sapiens
<400> 48
tgactccctc aaggcaatag gttattctta cagcacaact catctgttcc tgcgttcagc 60
acacggttac taggcacctg ctatgcaccc agcactgccc tagagcctgg gacatagcag 120
tgaacagaca gagagcggcc cctcccttct gtagccccca agccagtgag gggcacaggc 180
aggaacaggg accacaacac agaaaagctg gagggtgtca ggaggtgatc aggctctcag 240
ggagggcagc agggatgtgg agggagtgat gat 273
<210> 49
<211> 77
<212> DNA
<213> Homo sapiens
<400> 49
tgctggacag aagcaggaca gggcctggct cagggtgatt ctgggggtcc acttgtctgt 60
aatggtgtgc ttcaagg 77
<210> 50
<211> 69
<212> PRT
<213> Homo sapiens
<400> 50
Met Trp Asp Leu Val Leu Ser Ile Ala Leu Ser Val Gly Cys Thr Gly
1 5 10 15
Ala Val Pro Leu Ile Gln Ser Arg Ile Val Gly Gly Trp Glu Cys Glu
20 25 30
Lys His Ser Gln Pro Trp Gln Val Ala Val Tyr Ser His Gly Trp Ala
35 40 45
His Cys Gly Gly Val Leu Val His Pro Gln Trp Val Leu Thr Ala Ala
50 55 60
His Cys Leu Lys Lys
<210> 51
<211> 54
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
18
<212> PRT
<213> Homo sapiens
<400> 51
Gly Ala Val Pro Leu Ile Gln Ser Arg Ile Val Gly Gly Trp Glu Cys
1 5 10 15
G1u Lys His Ser Gln Pro Trp Gln Val Ala Val Tyr Ser His Gly Trp
20 25 30
Ala His Cys Gly Gly Val Leu Val His Pro Gln Trp Val Leu Thr Ala
35 40 45
A1a His Cys Leu Lys Lys
<210> 52
<211> 49
<212> PRT
<213> Homo sapiens
<400> 52
Met Trp Asp Leu Val Leu Ser Ile Ala Leu Ser Val Gly Cys Thr G1y
1 5 10 15
Ile Ala Arg Ser Gly Trp Val Gly Thr Thr Cys Leu 5er Leu Lys Thr
20 25 30
Gln Ala Arg Gly Ser Leu Ser Ala Thr Ala Ser His Thr Arg Ser Thr
35 40 45
Ile
<210> 53
<211> 34
<212> PRT
<213> Homo sapiens
<400> 53
Gly Ile Ala Arg Ser Gly Trp Val Gly Thr Thr Cys Leu Ser Leu Lys
1 5 10 15
Thr Gln Ala Arg Gly Ser Leu Ser Ala Thr Ala Ser His Thr Arg Ser
20 25 30
Thr Ile
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
19
<210> 54
<2ll> 85
<212> PRT
<213> Homo sapiens
<400> 54
Met Trp Asp Leu Val Leu Ser Ile Ala Leu Ser Val Gly Cys Thr Gly
1 5 10 15
Ala Val Pro Leu Ile Gln Ser Arg Ile Val Gly Gly Trp Glu Cys Glu
20 25 30
Lys His Ser Gln Pro Trp Gln Val Ala Val Tyr Ser His Gly Trp Ala
35 40 45
His Cys Gly Gly Val Leu Val His Pro Gln Trp Val Leu Thr Ala A1a
50 55 60
His Cys Leu Lys Asn Leu Ala Pro Gln Glu Ser Ser Val Cys Glu Pro
65 70 75 80
Pro Ser Pro Val Gln
<210> 55
<211> 70
<212> PRT
<213> Homo sapiens
<400> 55
G1y Ala Val Pro Leu Ile Gln Ser Arg Tle Val Gly Gly Trp Glu Cys
1 5 10 15
Glu Lys His Ser Gln Pro Trp Gln Val Ala Val Tyr Ser His Gly Trp
20 25 30
Ala His Cys Gly G1y Val Leu Val His Pro Gln Trp Val Leu Thr Ala
35 40 45
A1a His Cys Leu Lys Asn Leu Ala Pro Gln G1u Ser Ser Val Cys Glu
50 55 60
Pro Pro Ser Pro Val Gln
65 70
<210> 56
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
<211> 16
<212> PRT
<213> Homo sapiens
<400> 56
Leu Ala Pro Gln Glu Ser Ser Val Cys Glu Pro Pro Ser Pro Val Gln
1 5 10 15
<210> 57
<211> 149
<212> PRT
<213> Homo sapiens
<400> 57
Met Trp Asp Leu Val Leu Ser Ile Ala Leu Ser Val G1y Cys Thr Gly
1 5 10 15
Ala Val Pro Leu Ile Gln Ser Arg Ile Val Gly Gly Trp Glu Cys Glu
20 25 30
Lys His Ser Gln Pro Trp Gln Val Ala Val Tyr Ser His Gly Trp Ala
35 40 45
His Cys Gly Gly Val Leu Va1 His Pro Gln Trp Val Leu Thr Ala Ala
50 55 60
His Cys Leu Lys Lys Asn Ser Gln Val Trp Leu Gly Arg His Asn Leu
65 70 75 80
Phe Glu Pro Glu Asp Thr Gly Gln Arg Va1 Pro Val Ser His Ser Phe
85 90 95
Pro His Pro Leu Tyr Asn Met Ser Leu Leu Lys His Gln Ser Leu Arg
100 105 110
Pro Asp Glu Asp Ser Ser His Asp Leu Met Leu Leu Arg Leu Ser Glu
115 120 125
Pro Ala Lys Ile Thr Asp Val Val Lys Glu Ser Ser Val Cys Glu Pro
130 135 140
Pro Ser Pro Val Gln
145
<210> 58
<211> 134
<212> PRT
<213> Homo sapiens
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
21
<400> 58
Gly Ala Val Pro Leu Ile Gln Ser Arg Ile Val Gly Gly Trp Glu Cys
1 5 10 15
Glu Lys His Ser Gln Pro Trp Gln Val Ala Val Tyr Ser His Gly Trp
20 25 30
Ala His Cys Gly Gly Val Leu Val His Pro Gln Trp Val Leu Thr Ala
35 40 45
Ala His Cys Leu Lys Lys Asn Ser Gln Val Trp Leu Gly Arg His Asn
50 55 60
Leu Phe Glu Pro Glu Asp Thr Gly Gln Arg Val Pro Val Ser His Ser
65 70 75 80
Phe Pro His Pro Leu Tyr Asn Met Ser Leu Leu Lys His Gln Ser Leu
85 90 95
Arg Pro Asp Glu Asp Ser Ser His Asp Leu Met Leu Leu Arg Leu Ser
100 105 110
Glu Pro Ala Lys Ile Thr Asp Val Val Lys Glu Ser Ser Val Cys Glu
115 120 125
Pro Pro Ser Pro Val Gln
130
<210> 59
<211> 12
<212> PRT
<213> Homo sapiens
<400> 59
Glu Ser Ser Val Cys G1u Pro Pro Ser Pro Val Gln
1 5 10
<210> 60
<211> 224
<212> PRT
<213> Homo sapiens
<400> 60
Met Trp Asp Leu Val Leu Ser Tle Ala Leu Ser Val Gly Cys Thr Gly
1 5 10 15
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
22
Ala Val Pro Leu Ile Gln Ser Arg Ile Val Gly Gly Trp Glu Cys Glu
20 25 30
Lys His Ser Gln Pro Trp Gln Val A1a Va1 Tyr Ser His Gly Trp Ala
35 40 45
His Cys Gly Gly Val Leu Val His Pro Gln Trp Val Leu Thr Ala Ala
50 55 60
His Cys Leu Lys Lys Asn Ser Gln Val Trp Leu Gly Arg His Asn Leu
65 70 75 80
Phe Glu Pro Glu Asp Thr Gly Gln Arg Va1 Pro Val Ser His Ser Phe
85 90 95
Pro His Pro Leu Tyr Asn Met Ser Leu Leu Lys His Gln Ser Leu Arg
100 105 110
Pro Asp Glu Asp Ser Ser His Asp Leu Met Leu Leu Arg Leu Ser Glu
115 120 125
Pro Ala Lys Ile Thr Asp Val Val Lys Val Leu Gly Leu Pro Thr Gln
130 135 140
Glu Pro Ala Leu Gly Thr Thr Cys Tyr Ala Ser Gly Trp Gly Ser Ile
l45 150 155 160
Glu Pro Glu Glu Phe Leu Arg Pro Arg Ser Leu Gln Cys Val Ser Leu
165 170 175
His Leu Leu Ser Asn Asp Met Cys Ala Arg Ala Tyr Ser Glu Lys Val
180 l85 190
Thr G1u Phe Met Leu Cys Ala Gly Leu Trp Thr Gly Gly Lys Asp Thr
195 200 205
Cys Gly Val Ser His Pro Tyr Ser Gln His Leu Glu Gly Lys Gly Glu
210 215 220
<210> 61
<211> 209
<212> PRT
<213> Homo sapiens
<400> 61
Gly Ala Val Pro Leu Ile Gln Ser Arg Ile Val Gly Gly Trp Glu Cys
1 5 10 15
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
23
Glu Lys His Ser Gln Pro Trp Gln Val Ala Val Tyr Ser His Gly Trp
20 25 30
Ala His Cys Gly Gly Val Leu Val His Pro Gln Trp Val Leu Thr Ala
35 40 45
Ala His Cys Leu Lys Lys Asn Ser Gln Val Trp Leu Gly Arg His Asn
50 55 60
Leu Phe Glu Pro Glu Asp Thr Gly Gln Arg Val Pro Val Ser His Ser
65 70 75 80
Phe Pro His Pro Leu Tyr Asn Met Ser Leu Leu Lys His Gln Ser Leu
85 90 95
Arg Pro Asp Glu Asp Ser Ser His Asp Leu Met Leu Leu Arg Leu Ser
100 105 l10
Glu Pro Ala Lys Ile Thr Asp Val Val Lys Val Leu Gly Leu Pro Thr
115 120 125
Gln Glu Pro Ala Leu Gly Thr Thr Cys Tyr A1a Ser Gly Trp G1y Ser
130 135 140
Ile Glu Pro Glu Glu Phe Leu Arg Pro Arg Ser Leu Gln Cys Val Ser
145 150 155 160
Leu His Leu Leu Ser Asn Asp Met Cys Ala Arg Ala Tyr Ser Glu Lys
165 l70 175
Val Thr Glu Phe Met Leu Cys Ala Gly Leu Trp Thr Gly Gly Lys Asp
180 185 190
Thr Cys Gly Val Ser His Pro Tyr Ser Gln His Leu Glu Gly Lys Gly
195 200 205
Glu
<210> 62
<211> 14
<212> PRT
<213> Homo sapiens
<400> 62
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
24
Val Ser His Pro Tyr Ser G1n His Leu Glu Gly Lys Gly Glu
1 5 10
<210> 63
<211> 123
<212> PRT
<213> Homo sapïens
<400> 63
Met Trp Asp Leu Val Leu Ser Tle Ala Leu Ser Va1 Gly Cys Thr Gly
1 5 10 15
Ala Val Pro Leu Ile Gln Ser Arg Ile Val Gly Gly Trp Glu Cys Glu
20 25 30
Lys His Ser Gln Pro Trp Gln Val Ala Val Tyr Ser Hïs Gly Trp Ala
35 40 45
His Cys Gly Gly Val Leu Val His Pro Gln Trp Val Leu Thr Ala Ala
50 55 60
His Cys Leu Lys Lys Asn Ser Gln Val Trp Leu Gly Arg His Asn Leu
65 70 75 80
Phe Glu Pro Glu Asp Thr Gly G1n Arg Val Pro Val Ser His Ser Phe
85 90 95
Pro G1n Pro Leu Tyr Asn Met Ser Leu Leu Lys His Gln Ser Leu Arg
100 105 110
Pro Asp Glu Asp Ser Ser Ile Glu Pro Glu Glu
115 120
<210> 64
<2l1> 108
<212> PRT
<213> Homo sapiens
<400> 64
Gly Ala Val Pro Leu Ile Gln Ser Arg Ile Val Gly Gly Trp Glu Cys
1 5 10 15
Glu Lys His Ser Gln Pro Trp Gln Val Ala Val Tyr Ser His Gly Trp
20 25 30
Ala His Cys Gly Gly Val Leu Val His Pro Gln Trp Val Leu Thr Ala
35 40 45
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
Ala His Cys Leu Lys Lys Asn Ser Gln Val Trp Leu Gly Arg His Asn
50 55 60
Leu Phe Glu Pro Glu Asp Thr Gly Gln Arg Val Pro Val Ser His Ser
65 70 75 80
Phe Pro Gln Pro Leu Tyr Asn Met Ser Leu Leu Lys His Gln Ser Leu
85 90 95
Arg Pro Asp Glu Asp Ser Ser Ile Glu Pro Glu Glu
100 105
<210> 65
<211> 5
<212> PRT
<213> Homo sapiens
<400> 65
Ile Glu Pro Glu Glu
1 5
<210> 66
<211> 165
<212> PRT
<213> Homo sapiens
<400> 66
Met Trp Asp Leu Va1 Leu Ser Ile Ala Leu Ser Val G1y Cys Thr Gly
1 5 10 15
Ala Val Pro Leu Ile Gln Ser Arg Ile Val Gly Gly Trp Glu Cys Glu
20 25 30
Lys His Ser Gln Pro Trp Gln Val Ala Val Tyr Ser His Gly Trp Ala
40 45
His Cys Gly Gly Val Leu Va1 His Pro Gln Trp Val Leu Thr Ala Ala
50 55 60
His Cys Leu Lys Lys Asn Ser Gln Val Trp Leu Gly Arg His Asn Leu
65 70 75 80
Phe Glu Pro Glu Asp Thr Gly Gln Arg Val Pro Val Ser His Ser Phe
85 90 95
Pro His Pro Leu Tyr Asn Met Ser Leu Leu Lys His Gln Ser Leu Arg
100 105 110
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
26
Pro Asp Glu Asp Ser Ser His Asp Leu Met Leu Leu Arg Leu Ser Glu
115 120 125
Pro Ala Lys Ile Thr Asp Val Val Lys Val Leu Gly Leu Pro Thr Gln
130 135 140
Glu Pro Ala Leu Gly Thr Thr Cys Tyr Ala Ser Gly Trp Gly Ser Ile
145 150 155 160
Glu Pro Glu Glu Trp
165
<210> 67
<211> 150
<212> PRT
<213> Homo sapiens
<400> 67
Gly Ala Val Pro Leu Ile Gln Ser Arg Ile Val Gly Gly Trp Glu Cys
1 5 10 15
Glu Lys His Ser Gln Pro Trp Gln Val Ala Val Tyr Ser His Gly Trp
20 25 30
Ala His Cys Gly Gly Val Leu Val His Pro Gln Trp Val Leu Thr A1a
35 40 45
A1a His Cys Leu Lys Lys Asn Ser Gln Val Trp Leu Gly Arg His Asn
50 55 60
Leu Phe Glu Pro Glu Asp Thr Gly Gln Arg Val Pro Val Ser His Ser
65 70 75 80
Phe Pro His Pro Leu Tyr Asn Met Ser Leu Leu Lys His Gln Ser Leu
85 90 95
Arg Pro Asp Glu Asp Ser Ser His Asp Leu Met Leu Leu Arg Leu Ser
100 105 110
Glu Pro Ala Lys Ile Thr Asp Val Val Lys Val Leu Gly Leu Pro Thr
115 120 125
Gln Glu Pro Ala Leu Gly Thr Thr Cys Tyr Ala Ser Gly Trp Gly Ser
130 135 140
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
27
Ile Glu Pro Glu Glu Trp
145 150
<210> 68
<211> 119
<212> PRT
<213> Homo sapiens
<400> 68
Met Trp Asp Leu Val Leu Ser Ile Ala Leu Ser Val Gly Cys Thr Gly
1 5 10 15
Glu Ile Gly Gly Ile Lys Glu Gly Gly Arg Val Leu Thr Leu Met Leu
20 25 30
Lys Pro Phe Ser Ser His Pro Val Pro Gln Pro Arg Pro Phe Ser Pro
35 40 45
G1n Phe Ser Pro Asp Asn Val Pro Leu Thr Leu Pro Pro Glu Ala Ile
50 55 60
Trp Gln Lys Thr Thr Asn Ala Ala Ser His Pro Glu Ser Gly Thr Gly
65 70 75 80
Thr Phe Arg Thr Pro Pro Phe Leu Thr Leu Cys Pro Arg Pro Val Ile
85 90 95
Gln Trp Leu Ala Phe Ser Met Gly Lys Thr Arg Ala Pro Pro Thr Thr
100 105 1l0
Thr Ala Ser Ser Leu Ile Pro
115
<210> 69
<211> 104
<212> PRT
<2l3> Homo sapiens
<400> 69
Gly Glu Ile Gly Gly Ile Lys Glu Gly Gly Arg Val Leu Thr Leu Met
1 5 10 15
Leu Lys Pro Phe Ser Ser His Pro Val Pro Gln Pro Arg Pro Phe Ser
20 25 30
Pro Gln Phe Ser Pro Asp Asn Val Pro Leu Thr Leu Pro Pro Glu Ala
35 40 45
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
28
Ile Trp Gln Lys Thr Thr Asn Ala Ala Ser His Pro G1u Ser Gly Thr
50 55 60
Gly Thr Phe Arg Thr Pro Pro Phe Leu Thr Leu Cys Pro Arg Pro Va1
65 70 75 80
Ile Gln Trp Leu Ala Phe Ser Met Gly Lys Thr Arg Ala Pro Pro Thr
85 90 95
Thr Thr Ala Ser Ser Leu Ile Pro
100
<210> 70
<211> 59
<212> PRT
<213> Homo sapiens
<400> 70
Pro Glu Ala Ile Trp Gln Lys Thr Thr Asn Ala Ala Ser His Pro Glu
1 5 10 15
Ser Gly Thr Gly Thr Phe Arg Thr Pro Pro Phe Leu Thr Leu Cys Pro
20 25 30
Arg Pro Val Ile Gln Trp Leu Ala Phe Ser Met Gly Lys Thr Arg Ala
35 40 45
Pro Pro Thr Thr Thr A1a Ser Ser Leu Ile Pro
50 55
<2l0> 71
<211> 17
<212> PRT
<213> Homo sapiens
<400> 71
Arg Arg Thr Thr Ser Thr Pro Asn His Asn Gly Gln Phe Ser Asp Ser
1 5 10 15
Leu
<210> 72
<2l1> 21
<212> PRT
<213> Homo sapiens
<400> 72
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
29
Met Trp Asp Leu Val Leu Ser Ile Ala Leu Ser Val Gly Cys Thr Asp
1 5 10 15
Pro Ser Phe Asn Gly
<210> 73
<211> 6
<212> PRT
<213> Homo sapiens
<400> 73
Asp Pro Ser Phe Asn Gly
l 5
<210> 74
<211> 56
<212> PRT
<213> Homo sapiens
<400> 74
Met Trp Asp Leu Val Leu Ser Ile Ala Leu Ser Val Gly Cys Thr Gly
1 5 10 15
Glu Ile Gly Gly Ile Lys Glu Gly Gly Arg Val Leu Thr Leu Met Leu
20 25 30
Lys Pro Phe Ser Ser His Pro Val Pro Gln Pro Arg Pro Pro Asn His
35 40 45
Asn Gly Gln Phe Ser Asp Ser Leu
50 55
<210> 75
<211> 41
<212> PRT
<213> Homo sapiens
<400> 75
Gly Glu I1e Gly Gly Ile Lys Glu Gly Gly Arg Val Leu Thr Leu Met
1 5 10 15
Leu Lys Pro Phe Ser Ser His Pro Val Pro Gln Pro Arg Pro Pro Asn
20 25 30
His Asn Gly Gln Phe Ser Asp Ser Leu
35 40
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
<210> 76
<21l> 11
<212> PRT
<213> Homo sapiens
<400> 76
Pro Asn His Asn Gly Gln Phe Ser Asp Ser Leu
1 5 10
<2l0> 77
<211> 83
<212> PRT
<213> Homo sapiens
<400> 77
Gly Glu Ile Gly Gly Ile Lys Glu G1y Gly Arg Val Leu Thr Leu Met
1 5 10 15
Leu Lys Pro Phe Ser Ser His Pro Val Pro Gln Pro Arg Pro Pro Asn
20 25 30
His Asn Gly Gln Phe Ser Asp Ser Leu Asn Pro His Pro Phe Gln Asn
40 45
Leu Lys Asn Lys Thr Lys Gln Asn Lys Ala Arg Asn Asn Ser Gly Lys
50 55 60
Thr Cys Cys Leu Thr Leu Asp Met Val Asn His Pro Lys Pro Ser Ser
65 70 75 80
Pro Ser Asn
<210> 78
<211> 71
<212> PRT
<213> Homo sapïens
<400> 78
Met Trp Asp Leu Val Leu Ser Ile Ala Leu Ser Val Gly Cys Thr Gly
1 5 10 15
Ala Val Pro Leu Ile Gln Ser Arg Ile Val Gly Gly Trp Glu Cys Glu
20 25 30
Lys His Ser Gln Pro Trp Gln Val Ala Val Tyr Ser His Gly Trp Ala
35 40 45
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
31
His Cys Gly Gly Ala Leu Glu Pro Pro Gln Arg Gly Arg Thr His His
50 55 60
Ala Pro Trp Pro Tyr Arg Asn
65 70
<210> 79
<211> 19
<212> PRT
<213> Homo sapiens
<400> 79
Ala Leu Glu Pro Pro Gln Arg Gly Arg Thr His His Ala Pro Trp Pro
1 5 10 15
Tyr Arg Asn
<210> 80
<211> 298
<212> PRT
<213> Homo sapiens
<400> 80
Met Trp Asp Leu Val Leu Ser Ile Ala Leu Ser Val Gly Cys Thr Gly
1 5 10 15
Ala Val Pro Leu Ile Gln Ser Arg I1e Val Gly Gly Trp Glu Cys Glu
20 25 30
Lys His Ser Gln Pro Trp Gln Val Ala Val Tyr Ser His Gly Trp Ala
35 40 45
His Ser Cys Phe Leu Thr His Cys Leu Tyr Phe Thr Thr Thr Ile Ser
50 55 60
Pro Arg Pro Leu Cys Phe Ser His Cys Phe Phe Phe Phe Pro Leu Glu
65 70 75 80
Ser Pro Leu Ser Ser Pro Ala Pro Ser Thr Phe Pro His Phe Leu Ser
85 90 95
Pro His Ala Ser Thr Pro Phe Leu Pro Arg Asn Ser Gln Val Trp Leu
100 105 110
Gly Arg His Asn Leu Phe G1u Pro Glu Asp Thr Gly Gln Arg Val Pro
115 120 125
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
32
Val Ser His Ser Phe Pro His Pro Leu Tyr Asn Met Ser Leu Leu Lys
130 135 140
His Gln Ser Leu Arg Pro Asp Glu Asp Ser Ser His Asp Leu Met Leu
145 150 155 160
Leu Arg Leu Ser Glu Pro Ala Lys Ile Thr Asp Val Val Lys Val Leu
165 170 175
Gly Leu Pro Thr Gln Glu Pro Ala Leu Gly Thr Thr Cys Tyr Ala Ser
180 185 190
Gly Trp Gly Ser Ile Glu Pro Glu Glu Phe Leu Arg Pro Arg Ser Leu
195 200 205
Gln Cys Val Ser Leu His Leu Leu Ser Asn Asp Met Cys Ala Arg Ala
210 215 220
Tyr Ser Glu Lys Val Thr G1u Phe Met Leu Cys Ala Gly Leu Trp Thr
225 230 235 240
Gly Gly Lys Asp Thr Cys Gly Gly Asp Ser Gly Gly Pro Leu Val Cys
245 250 255
Asn Gly Val Leu Gln Gly Ile Thr Ser Trp Gly Pro Glu Pro Cys Ala
260 265 270
Leu Pro Glu Lys Pro Ala Va1 Tyr Thr Lys Val Val His Tyr Arg Lys
275 280 285
Trp Ile Lys Asp Thr Ile Ala Ala Asn Pro
290 295
<210> 81
<21l> 57
<212> PRT
<213> Homo sapiens
<400> 81
Ser Cys Phe Leu Thr His Cys Leu Tyr Phe Thr Thr Thr Ile Ser Pro
1 5 10 15
Arg Pro Leu Cys Phe Ser His Cys Phe Phe Phe Phe Pro Leu Glu Ser
20 25 30
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
33
Pro Leu Ser Ser Pro Ala Pro Ser Thr Phe Pro His Phe Leu Ser Pro
35 40 45
His A1a Ser Thr Pro Phe Leu Pro Arg
50 55
<210> 82
<211> 70
<212> PRT
<213> Homo sapiens
<400> 82
Ser Ser Met Cys Val His Val Arg Gly Cys Leu Ser Leu His Ser Thr
1 5 10 15
Leu Cys Arg Thr Ser Leu His Pro His Ala Ser Pro Pro Pro Pro Ala
20 25 30
Thr Pro His Ser Ser Asp Ala Pro Val Ala Ser Leu Leu Phe Leu Ser
35 40 45
His Thr Val Ser Pro Leu Ala Ser Ser Leu Leu Phe Leu Leu Leu Ser
50 55 60
Leu Thr Ser Arg Ile Leu
65 70
<210> 83
<211> 69
<212> PRT
<213> Homo sapiens
<400> 83
Leu Asn Val Cys Ala Cys Glu Gly Val Pro Val Ile Ala Gln His Ser
1 5 10 15
Leu Gln Asp 21e Pro Pro Pro Ser Cys Leu Ser Ser Ser Pro Cys Tyr
20 25 30
Ser Thr Leu Leu Arg Cys Pro Arg Gly Leu Pro Pro Phe Ser Leu Pro
35 40 45
His Cys Ile Thr Pro Gly Phe Leu Ser Ala Val Ser Pro Ser Leu Ser
50 55 60
Asp Phe Pro His Pro
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
34
<210> 84
<211> 46
<212> PRT
<213> Homo sapiens
<400> 84
Gly Val Thr Met Arg Met Thr Trp G1y Trp Leu Gln Ala Leu Ser Pro
1 5 10 15
Pro Gly Pro Ser Pro Ser Leu Pro His Arg Leu Leu Ala Leu Ser Leu
20 25 30
Ser Pro Pro Leu His Ser Pro Pro Thr His Ser Gly Ser Phe
35 40 45
<210> 85
<211> 71
<212> PRT
<213> Homo sapiens
<400> 85 '
Ser Asp Asp Glu Asp Asp Leu Gly Val Ala Pro Gly Ile Val Pro Thr
1 5 10 15
Trp Ala Leu Thr Gln Pro Pro Ser Gln Ala Pro G1y Pro Gln Ser Leu
20 25 30
Pro Ser Thr Pro Phe Ser Thr Tyr Pro Gln Trp Val Ile Leu Ile Thr
35 40 45
Glu Leu Thr Met Pro Ala Leu Pro Met Ala His Leu Ser Val Met Gly
50 55 60
Cys Phe Lys Val Ser His His
65 70
<210> 86
<211> 53
<212> PRT
<213> Homo sapiens
<400> 86
Pro Leu Gln Ser Pro His Ser Ser Pro Ser Cys Arg Asn Ser Gln Val
1 5 10 15
Trp Leu Gly Arg His Asn Leu Phe Glu Pro G1u Asp Thr Gly Gln Arg
20 25 30
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
Val Pro Val Ser His Ser Phe Pro His Pro Leu Tyr Asn Met Ser Leu
35 40 45
Leu Lys His Gln Ser
<210> 87
<211> 45
<212> PRT
<213> Homo sapiens
<400> 87
His Tyr Arg Ala Leu Thr Pro Ala Pro Ala Ala Gly Ile Ala Arg Ser
1 5 10 15
Gly Trp Val Gly Thr Thr Cys Leu Ser Leu Lys Thr Gln Ala Arg Guy
20 25 30
Ser Leu Ser Ala Thr Ala Ser Hïs Thr Arg Ser Thr Ile
35 40 45
<210> 88
<211> 12
<212> PRT
<213> Homo sapïens
<400> 88
Thr Thr Glu Pro Ser Leu G1n Pro Gln Leu Gln Glu
1 5 10
<210> 89
<211> 51
<212> PRT
<213> Homo sapiens
<400> 89
Met Trp Val Pro Val Val Phe Leu Thr Leu Ser Val Thr Trp Ile Gly
1 5 10 15
Glu Arg Gly His Gly Gly Gly Asp Ala Gly Glu Gly Ala Ile Pro Asp
20 25 30
Cys Gln Ala Glu Ala Leu Cys Pro Pro Thr Gln His Pro Ser Ser Gln
35 40 45
Leu Leu Tyr
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
36
<210> 90
<211> 36
<212> PRT
<213> Homo sapiens
<400> 90
Gly Glu Arg Gly His Gly Gly Gly Asp Ala G1y Glu Gly Ala Ile Pro
1 5 10 15
Asp Cys Gln A1a Glu Ala Leu Cys Pro Pro Thr G1n His Pro Ser Ser
20 25 30
Gln Leu Leu Tyr
<210> 91
<211> 89
<212> PRT
<213> Homo sapiens
<400> 91
Met Trp Val Pro Val Val Phe Leu Thr Leu Ser Val Thr Trp Ile Gly
1 5 10 15
Glu Arg Gly His Gly Trp Gly Asp Ala Gly Glu G1y Ala Ser Pro Asp
20 25 30
Cys Gln Ala Glu Ala Leu Ser Pro Pro Thr Gln His Pro Ser Pro Asp
35 40 45
Arg Glu Leu Gly Ser Phe Leu Ser Leu Pro Ala Pro Leu Gln Ala His
50 55 60
Thr Pro Ser Pro Ser His Leu Pro Gln Asn Phe Leu Pro Ile Ala Gln
65 70 75 80
Pro Ala Pro Cys Ser Gln Leu Leu Tyr
<210> 92
<211> 74
<212> PRT
<213> Homo sapiens
<400> 92
Gly Glu Arg Gly His Gly Trp Gly Asp Ala Gly Glu Gly Ala Ser Pro
1 5 10 15
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
37
Asp Cys Gln Ala Glu Ala Leu Ser Pro Pro Thr Gln His Pro Ser Pro
20 25 30
Asp Arg Glu Leu Gly Ser Phe Leu Ser Leu Pro Ala Pro Leu Gln Ala
35 40 45
His Thr Pro Ser Pro Ser His Leu Pro Gln Asn Phe Leu Pro Ile Ala
50 55 60
Gln Pro Ala Pro Cys Ser Gln Leu Leu Tyr
65 70
<210> 93
<21l> 20
<212> PRT
<213> Homo sapiens
<400> 93
His Leu Pro Gln Asn Phe Leu Pro Ile Ala Gln Pro Ala Pro Cys Ser
1 5 10 15
Gln Leu Leu Tyr
<210> 94
<211> 47
<212> PRT
<213> homo sapiens
<400> 94
Met Trp Val Pro Val Val Phe Leu Thr Leu Ser Val Thr Trp Ile Ala
1 5 10 15
Val Ser Va1 Gln Pro Tyr Pro Arg Asp Leu Asp Ala Trp Pro Pro Asn
20 25 30
Leu Ala Leu Gly Tyr Pro Asp Ala Asn Gln Thr Pro Pro Ser Ser
35 40 45
<210> 95
<211> 32
<212> PRT
<213> Homo sapiens
<400> 95
Ala Val Ser Val Gln Pro Tyr Pro Arg Asp Leu Asp Ala Trp Pro Pro
l 5 l0 15
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
38
Asn Leu Ala Leu Gly Tyr Pro Asp Ala Asn Gln Thr Pro Pro Ser Ser
20 25 30
<210> 96
<21l> 68
<212> PRT
<213> Homo sapiens
<400> 96
Met Trp Va1 Pro Val Val Phe Leu Thr Leu Ser Val Thr Trp Ile Gly
1 5 10 15
Glu Arg Gly His Gly Trp Gly Asp Ala Gly Glu Gly Ala Ser Pro Asp
20 25 30
Cys Gln Ala Glu Ala Leu Ser Pro Gln Pro Tyr Pro Arg Asp Leu Asp
35 40 45
Ala Trp Pro Pro Asn Leu Ala Leu Gly Tyr Pro Asp Ala Asn Gln Thr
50 55 60
Pro Pro Ser Ser
<210> 97
<211> 53
<2l2> PRT
<213> Homo sapiens
<400> 97
Gly Glu Arg Gly His Gly Trp Gly Asp Ala Gly Glu Gly A1a Ser Pro
1 5 10 15
Asp Cys Gln Ala Glu Ala Leu Ser Pro Gln Pro Tyr Pro Arg Asp Leu
20 25 30
Asp Ala Trp Pro Pro Asn Leu Ala Leu Gly Tyr Pro Asp Ala Asn Gln
35 40 45
Thr Pro Pro Ser Ser
<210> 98
<211> 28
<212> PRT
<213> Homo sapiens
<400> 98
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
39
Gln Pro Tyr Pro Arg Asp Leu Asp Ala Trp Pro Pro Asn Leu Ala Leu
1 5 10 15
Gly Tyr Pro Asp Ala Asn G1n Thr Pro Pro Ser Ser
20 25
<210> 99
<211> 23
<212> PRT
<213> Homo sapiens
<400> 99
Met Trp Val Pro Val Val Phe Pro Asn Pro Ile Pro Glu Thr Leu Met
1 5 10 15
Leu Gly Leu Pro Ile Leu Pro
<210> 100
<211> 27
<212> PRT
<213> Homo sapiens
<400> 100
Met Leu Val Pro Val Val Phe Leu Thr Leu Ser Va1 Thr Trp Ile Gly
1 5 10 15
Tyr Pro Asp Ala Asn Gln Thr Pro Pro Ser Ser
20 25
<210> 101
<211> 12
<212> PRT
<213> Homo sapiens
<400> 101
Gly Tyr Pro Asp Ala Asn Gln Thr Pro Pro Ser Ser
1 5 10
<210> 102
<211> 69
<212> PRT
<213> Homo sapiens
<400> 102
Met Trp Val Pro Val Val Phe Leu Thr Leu Ser Val Thr Trp Ile Gly
1 5 10 15
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
Ala Ala Pro Leu I1e Leu Ser Arg Ile Val Gly Gly Trp Glu Cys Glu
20 25 30
Lys His Ser Gln Pro Trp G1n Val Leu Val Ala Ser Arg Gly Arg Ala
35 40 45
Val Cys Gly G1y Val Leu Val His Pro Gln Trp Val Leu Thr A1a Ala
55 60
His Cys Ile Arg Lys
<210> 103
<211> 54
<212> PRT
<213> Homo sapiens
<400> 103
Gly Ala Ala Pro Leu Ile Leu Ser Arg Ile Val Gly Gly Trp G1u Cys
1 5 10 15
Glu Lys His Ser Gln Pro Trp Gln Val Leu Val Ala Ser Arg Gly Arg
20 25 30
Ala Val Cys Gly Gly Val Leu Val His Pro Gln Trp Val Leu Thr Ala
35 40 45
Ala His Cys Ile Arg Lys
<2l0> 104
<211> 83
<212> PRT
<213> Homo sapiens
<400> 104
Met Trp Val Pro Val Val Phe Leu Thr Leu Ser Val Thr Trp Tle Gly
1 5 10 15
Ala Ala Pro Leu Ile Leu Ser Arg Ile Val G1y Gly Trp Glu Cys Glu
20 25 30
Lys His Ser Gln Pro Trp Gln Val Leu Val Ala Ser Arg Gly Arg Ala
35 40 45
Val Cys Gly G1y Val Leu Va1 His Pro Gln Trp Val Leu Thr Ala Ala
50 55 60
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
41
His Cys Ile Arg Asn Leu His Ile Pro Pro Ser Leu Cys Pro Ser Leu
65 70 75 80
Val Pro Leu
<210> l05
<211> 68
<212> PRT
<213> Homo sapiens
<400> 105
Gly Ala Ala Pro Leu Ile Leu Ser Arg Ile Val Gly Gly Trp Glu Cys
1 5 10 15
Glu Lys His Ser Gln Pro Trp Gln Val Leu Val Ala Ser Arg Gly Arg
20 25 30
Ala Val Cys Gly Gly Val Leu Val His Pro Gln Trp Val Leu Thr Ala
35 40 45
Ala His Cys Ile Arg Asn Leu His Ile Pro Pro Ser Leu Cys Pro Ser
50 55 60
Leu Val Pro Leu
<210> 106
<211> 14
<212> PRT
<213> Homo sapiens
<400> 106
Leu His Ile Pro Pro Ser Leu Cys Pro Ser Leu Val Pro Leu
1 5 10
<210> 107
<211> 75
<212> PRT
<213> Homo sapiens
<400> 107
Met Trp Val Pro Val Val Phe Leu Thr Leu Ser Va1 Thr Trp Ile Ala
1 5 10 15
Pro Pro Leu Gln A1a Leu Arg Pro Ser Ser Cys Leu Gly Leu Trp Glu
20 25 30
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
42
Ala Gly Ser Ala Arg Ser Ile Pro Asn Pro Gly Arg Cys Leu Trp Pro
35 40 45
Leu Val Ala Gly G1n Ser Ala Ala Val Phe Trp Cys Thr Pro Ser Gly
50 55 60
Ser Ser Gln Leu Pro Thr Ala Ser Gly Ser Glu
65 70 75
<210> 108
<211> 60
<212> PRT
<213> Homo sapiens
<400> 108
Ala Pro Pro Leu Gln Ala Leu Arg Pro Ser Ser Cys Leu Gly Leu Trp
1 5 10 15
Glu Ala Gly Ser Ala Arg Ser Ile Pro Asn Pro Gly Arg Cys Leu Trp
20 25 30
Pro Leu Val Ala Gly Gln Ser Ala Ala Val Phe Trp Cys Thr Pro Ser
35 40 45
Gly Ser Ser Gln Leu Pro Thr Ala Ser Gly Ser Glu
50 55 60
<210> 109
<211> 41
<212> PRT
<213> Homo sapiens
<400> 109
Met Trp Val Pro Val Val Phe Leu Thr Leu Ser Leu Trp Pro Leu Val
1 5 10 15
Ala Gly Gln Ser Ala Ala Val Phe Trp Cys Thr Pro Ser Gly Ser Pro
2p 25 30
Gln Leu Pro Thr Ala Ser Gly Ser Glu
35 40
<210> 110
<211> 30
<212> PRT
<213> Homo sapiens
<400> 110
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
43
Leu Trp Pro Leu Val A1a Gly Gln Ser Ala Ala Val Phe Trp Cys Thr
1 5 10 15
Pro Ser Gly Ser Pro Gln Leu Pro Thr Ala Ser Gly Ser Glu
20 25 30
<210> l11
<211> 39
<212> PRT
<213> Homo sapiens
<400> 111
Met Trp Val Pro Val Val Phe Leu Thr Leu Ser Val Thr Trp Ile Gly
1 5 10 15
Gln Ser Ala Ala Val Phe Trp Cys Thr Pro Ser Gly Ser Ser Gln Leu
20 25 30
Pro Thr Ala Ser Gly Ser Glu
<210> 112
<211> 24
<212> PRT
<213> Homo sapiens
<400> 112
Gly Gln Ser Ala Ala Val Phe Trp Cys Thr Pro Ser Gly Ser Ser Gln
1 5 10 15
Leu Pro Thr Ala Ser Gly Ser Glu
<2l0> 113
<211> 69
<212> PRT
<213> Homo sapiens
<400> 113
Met Trp Val Pro Val Val Phe Leu Thr Leu Ser Val Thr Trp I1e Gly
l 5 10 l5
Ala Ala Pro Leu Ile Leu Ser Arg Ile Val Gly Gly Trp Glu Cys Glu
20 25 30
Lys His Ser Gln Pro Trp Gln Val Leu Val Ala Ser Arg Gly Arg Ala
35 40 45
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
44
Val Cys Gly Gly Val Leu Val His Pro Gln Trp Val Leu Thr Ala Ala
50 55 60
His Cys Ile Arg Lys
<210> 114
<211> 54
<212> PRT
<213> Homo sapiens
<400> 114
Gly Ala Ala Pro Leu Ile Leu Ser Arg Ile Va1 Gly Gly Trp Glu Cys
1 5 10 15
Glu Lys His Ser Gln Pro Trp Gln Val Leu Val Ala Ser Arg Gly Arg
20 25 30
Ala Val Cys Gly Gly Val Leu Val His Pro Gln Trp Val Leu Thr Ala
35 40 45
Ala His Cys Ile Arg Lys
<210> 115
<211> 100
<212> PRT
<213> Homo sapiens
<400> 115
Met Trp Val Pro Val Val Phe Leu Thr Leu Ser Val Thr Trp Ile Gly
1 5 10 15
Ala Ala Pro Leu Ile Leu Ser Arg Ile Val Gly Gly Trp Glu Cys Glu
20 25 30
Lys His Ser Gln Pro Trp Gln Val Leu Val Ala Ser Arg Gly Arg Ala
35 40 45
Val Cys Gly Gly Val Leu Val His Pro Gln Trp Val Leu Thr Ala A1a
50 55 60
His Cys Ile Arg Asn Lys Ser Val Ile Leu Leu Gly Arg His Ser Leu
65 70 75 80
Phe His Pro G1u Asp Thr Gly Gln Val Phe Gln Val Ser Leu Pro Arg
85 90 95
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
Ser Arg Met Leu
100
<210> 116
<211> 85
<212> PRT
<213> Homo sapiens
<400> 116
Gly Ala Ala Pro Leu Ile Leu Ser Arg I1e Val Gly Gly Trp Glu Cys
1 5 10 15
Glu Lys His Ser G1n Pro Trp Gln Val Leu Val Ala Ser Arg Gly Arg
20 25 30
A1a Val Cys Gly Gly Val Leu Val His Pro Gln Trp Val Leu Thr Ala
35 40 45
Ala His Cys Ile Arg Asn Lys Ser Val Ile Leu Leu Gly Arg His Ser
55 60
Leu Phe His Pro Glu Asp Thr Gly Gln Val Phe Gln Val Ser Leu Pro
65 70 75 80
Arg Ser Arg Met Leu
<210> 117
<211> 6
<212> PRT
<2l3> Homo sapiens
<400> 117
Pro Arg Ser Arg Met Leu
1 5
<210> 118
<211> 177
<212> PRT
<213> Homo sapiens
<400> 118
Met Trp Val Pro Val Val Phe Leu Thr Leu Ser Val Thr Trp Ile Gly
1 5 10 15
Ala Ala Pro Leu Ile Leu Ser Arg Ile Val Gly Gly Trp Glu Cys Glu
20 25 30
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
46
Lys His Ser Gln Pro Trp Gln Val Leu Val Ala Ser Arg G1y Arg Ala
35 40 45
Val Cys Gly Gly Val Leu Val His Pro Gln Trp Val Leu Thr Ala Ala
50 55 60
His Cys Ile Arg Lys Pro Gly Asp Asp Ser Ser Ile Glu Pro Glu Glu
65 70 75 80
Phe Leu Thr Pro Lys Lys Leu Gln Cys Val Asp Leu His Val Ile Ser
85 90 95
Asn Asp Val Cys Ala Gln Val His Pro Gln Lys Val Thr Lys Phe Met
100 105 110
Leu Cys Ala Gly Arg Trp Thr Gly Gly Lys Ser Thr Cys Ser Gly Asp
l15 120 125
Ser Gly Gly Pro Leu Val Cys Asn Gly Val Leu G1n Gly Ile Thr Ser
130 135 140
Trp Gly Ser Glu Pro Ser Ile Leu Pro Glu Arg Pro Ser Leu Tyr Thr
145 150 155 160
Lys Val Va1 His Tyr Arg Lys Trp Ile Lys Asp Thr Ile Val Ala Asn
165 170 175
Pro
<210> 119
<211> 162
<212> PRT
<213> Homo sapiens
<400> 119
Gly Ala Ala Pro Leu Ile Leu Ser Arg Ile Val Gly Gly Trp Glu Cys
1 5 10 15
Glu Lys His Ser Gln Pro Trp Gln Va1 Leu Val Ala Ser Arg Gly Arg
3p 25 30
Ala Val Cys Gly Gly Val Leu Val His Pro Gln Trp Val Leu Thr Ala
35 40 45
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
47
Ala His Cys Ile Arg Lys Pro Gly Asp Asp Ser Ser Ile Glu Pro Glu
50 55 60
Glu Phe Leu Thr Pro Lys Lys Leu Gln Cys Val Asp Leu His Val Ile
65 70 75 80
Ser Asn Asp Val Cys Ala Gln Val His Pro Gln Lys Val Thr Lys Phe
85 90 95
Met Leu Cys Ala Gly Arg Trp Thr Gly Gly Lys Ser Thr Cys Ser Gly
100 105 110
Asp Ser Gly Gly Pro Leu Val Cys Asn Gly Val Leu Gln Gly Ile Thr
115 120 125
Ser Trp Gly Ser Glu Pro Ser Ile Leu Pro Glu Arg Pro Ser Leu Tyr
130 135 140
Thr Lys Val Val His Tyr Arg Lys Trp Ile Lys Asp Thr Ile Val Ala
145 150 155 160
Asn Pro
<210> 120
<211> 220
<212> PRT
<213> Homo sapiens
<400> 120
Met Trp Val Pro Val Val Phe Leu Thr Leu Ser Val Thr Trp Ile Gly
1 5 l0 15
Ala Ala Pro Leu Ile Leu Ser Arg Ile Val Gly G1y Trp Glu Cys Glu
20 25 30
Lys His Ser Gln Pro Trp Gln Val Leu Val Ala Ser Arg Gly Arg Ala
35 40 45
Val Cys Gly Gly Val Leu Val His Pro Gln Trp Val Leu Thr Ala Ala
50 55 60
His Cys Ile Arg Asn Lys Ser Val Ile Leu Leu Gly Arg His Ser Leu
65 70 75 80
Phe His Pro Glu Asp Thr Gly Gln Val Phe Gln Val Ser His Ser Phe
85 90 95
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
48
Pro His Pro Leu Tyr Asp Met Ser Leu Leu Lys Asn Arg Phe Leu Arg
100 105 110
Pro Gly Asp Asp Ser Ser Ile Glu Pro Glu Glu Phe Leu Thr Pro Lys
115 120 125
Lys Leu Gln Cys Val Asp Leu His Val Ile Ser Asn Asp Val Cys Ala
130 135 140
Gln Val His Pro Gln Lys Val Thr Lys Phe Met Leu Cys Ala Gly Arg
145 150 155 160
Trp Thr Gly Gly Lys Ser Thr Cys Ser Gly Asp Ser Gly Gly Pro Leu
165 170 175
Val Cys Asn Gly Val Leu Gln Gly Ile Thr Ser Trp Gly Ser Glu Pro
180 185 190
Ser Ile Leu Pro Glu Arg Pro Ser Leu Tyr Thr Lys Va1 Val His Tyr
195 200 205
Arg Lys Trp Ile Lys Asp Thr Ile Val Ala Asn Pro
210 215 220
<210> 121
<211> 205
<212> PRT
<213> Homo sapiens
<400> 121
Gly Ala Ala Pro Leu Ile Leu Ser Arg Ile Val Gly Gly Trp Glu Cys
1 5 10 15
Glu Lys His Ser Gln Pro Trp Gln Val Leu Val Ala Ser Arg G1y Arg
20 25 30
Ala Val Cys Gly Gly Val Leu Val His Pro Gln Trp Val Leu Thr Ala
35 40 45
Ala His Cys I1e Arg Asn Lys Ser Val Ile Leu Leu Gly Arg His Ser
50 55 60
Leu Phe His Pro Glu Asp Thr Gly Gln Val Phe G1n Val Ser His Ser
65 70 75 g0
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
49
Phe Pro His Pro Leu Tyr Asp Met Ser Leu Leu Lys Asn Arg Phe Leu
85 90 95
Arg Pro Gly Asp Asp Ser Ser Ile Glu Pro Glu Glu Phe Leu Thr Pro
100 105 110
Lys Lys Leu Gln Cys Val Asp Leu His Val Ile Ser Asn Asp Va1 Cys
115 120 125
Ala Gln Val His Pro Gln Lys Val Thr Lys Phe Met Leu Cys Ala Gly
130 135 140
Arg Trp Thr Gly Gly Lys Ser Thr Cys Ser Gly Asp Ser Gly Gly Pro
145 150 155 160
Leu Val Cys Asn Gly Val Leu Gln Gly Ile Thr Ser Trp Gly Ser Glu
165 170 175
Pro Ser Ile Leu Pro Glu Arg Pro Ser Leu Tyr Thr Lys Val Val His
180 185 190
Tyr Arg Lys Trp Ile Lys Asp Thr Ile Val Ala Asn Pro
195 200 205
<210> 122
<211> 207
<212> PRT
<213> Homo sapiens
<400> 122
Met Trp Val Pro Val Val Phe Leu Thr Leu Ser Val Thr Trp Ile G1y
1 5 10 15
Ala Ala Pro Leu Ile Leu Ser Arg Ile Val Ser Arg Gly Arg Ala Val
20 25 30
Gly Gly Gly Val Leu Val His Pro Gln Trp Val Leu Thr Ala Ala His
35 40 45
Cys I1e Arg Lys Pro Gly Asp Asp Ser Ser His Asp Leu Met Leu Leu
50 55 60
Arg Leu Ser Glu Pro Ala Glu Ile Thr Asp Ala Val Lys Val Met Asp
65 70 75 80
Leu Pro Thr Gln Glu Pro Ala Leu Gly Thr Thr Cys Tyr Ala Ser Gly
85 90 95
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
Trp Gly Ser Ile Glu Pro Glu Glu Cys Thr Pro Gly Pro Asp Val Leu
100 105 110
Thr Pro Lys Lys Leu Gln Cys Val Asp Leu His Val Ile Ser Asn Asp
115 120 125
Val Cys Ala Gln Val His Pro Gln Lys Va1 Thr Lys Phe Met Leu Cys
130 135 140
Ala Gly Arg Trp Thr Gly Gly Lys Ser Thr Cys Ser Gly Asp Ser Gly
145 150 155 160
Gly Pro Leu Val Cys Asn Gly Val Leu Gln Gly Ile Thr Ser Trp Gly
165 170 175
Ser Glu Pro Ser Ile Leu Pro Glu Arg Pro Ser Leu Tyr Thr Lys Val
180 185 190
Va1 His Tyr Arg Lys Trp Ile Lys Asp Thr Ile Val Ala Asn Pro
195 200 205
<210> 123
<211> 192
<212> PRT
<213> Homo sapiens
<400> 123
Gly Ala Ala Pro Leu Ile Leu Ser Arg Ile Val Ser Arg Gly Arg Ala
1 5 10 15
Val Gly Gly Gly Val Leu Val His Pro Gln Trp Val Leu Thr Ala Ala
20 25 30
His Cys Ile Arg Lys Pro Gly Asp Asp Ser Ser His Asp Leu Met Leu
35 40 45
Leu Arg Leu Ser Glu Pro Ala Glu Ile Thr Asp Ala Val Lys Val Met
50 55 60
Asp Leu Pro Thr Gln G1u Pro Ala Leu Gly Thr Thr Cys Tyr Ala Ser
65 70 75 80
Gly Trp Gly Ser Ile Glu Pro Glu Glu Cys Thr Pro Gly Pro Asp Val
85 90 95
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
51
Leu Thr Pro Lys Lys Leu Gln Cys Val Asp Leu His Val Ile Ser Asn
100 105 110
Asp Val Cys Ala Gln Val His Pro Gln Lys Val Thr Lys Phe Met Leu
115 120 125
Cys Ala Gly Arg Trp Thr Gly Gly Lys Ser Thr Cys Ser Gly Asp Ser
130 135 140
Gly Gly Pro Leu Val Cys Asn Gly Val Leu Gln Gly Ile Thr Ser Trp
145 150 155 160
Gly Ser G1u Pro Ser Ile Leu Pro Glu Arg Pro Ser Leu Tyr Thr Lys
165 170 175
Val Val His Tyr Arg Lys Trp Ile Lys Asp Thr Ile Val Ala Asn Pro
180 185 190
<210> 124
<211> 85
<212> PRT
<213> Homo sapiens
<400> 124
Met Trp Val Pro Val Val Phe Leu Thr Leu Ser Val Thr Trp Ile Gly
1 5 , 10 15
Ala Ala Pro Leu Ile Leu Ser Arg Ile Val Gly Gly Trp Glu Cys Glu
20 25 30
Lys His Ser Gln Pro Trp Gln Val Leu Val Ala Ser Arg Gly Arg Ala
35 40 45
Val Cys G1y Gly Val Leu Val His Pro Gln Trp Val Leu Thr Ala Ala
50 55 60
His Cys Ile Arg Asn Leu Asp Pro Lys Glu Thr Ser Val Cys G1y Pro
65 70 75 80
Pro Cys Tyr Phe Gln
<210> 125
<211> 70
<212> PRT
<213> Homo sapiens
<400> 125
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
52
Gly Ala Ala Pro Leu Ile Leu Ser Arg Ile Val Gly Gly Trp Glu Cys
1 5 10 15
Glu Lys His Ser Gln Pro Trp Gln Val Leu Val Ala Ser Arg Gly Arg
20 25 30
Ala Va1 Cys Gly Gly Val Leu Val His Pro Gln Trp Val Leu Thr Ala
35 40 45
Ala His Cys Ile Arg Asn Leu Asp Pro Lys Glu Thr Ser Val Cys Gly
50 55 60
Pro Pro Cys Tyr Phe Gln
65 70
<210> 126
<211> 16
<212> PRT
<213> Homo sapiens
<400> 126
Leu Asp Pro Lys G1u Thr Ser Val Cys Gly Pro Pro Cys Tyr Phe Gln
1 5 10 15
<210> 127
<211> 78
<212> PRT
<213> Homo sapiens
<400> 127
Met Trp Val Pro Val Va1 Phe Leu Thr Leu Ser Val Thr Trp Ile Gly
1 5 10 15
Ala Ala Pro Leu 21e Leu Ser Arg Ile Val Gly Gly Trp Glu Cys Glu
20 25 30
Lys His Ser G1n Pro Trp Gln Val Leu Val Ala Ser Arg Gly Arg Ala
35 40 45
Val Cys Gly Gly Va1 Leu Val His Pro Gln Trp Val Leu Thr Ala Ala
50 55 60
His Cys Ile Arg Asn Lys Ser Val Ile Leu Leu Gly Gln His
65 70 75
<210> 128
<211> 63
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
53
<212> PRT
<213> Homo sapiens
<400> 128
Gly Ala Ala Pro Leu Ile Leu Ser Arg Ile Val Gly Gly Trp Glu Cys
1 5 10 15
Glu Lys His Ser Gln Pro Trp Gln Val Leu Va1 Ala Ser Arg Gly Arg
20 25 30
A1a Val Cys Gly Gly Val Leu Val His Pro Gln Trp Val Leu Thr Ala
35 40 45
Ala His Cys Ile Arg Asn Lys Ser Val Ile Leu Leu Gly Gln His
50 55 60
<210> 129
<211> 144
<212> PRT
<213> Homo sapiens
<400> 129
Met Trp Val Pro Val Val Phe Leu Thr Leu Ser Val Thr Trp Ile Gly
1 5 10 15
Ala Ala Pro Leu Ile Leu Ser Arg Ile Val Gly Gly Trp Glu Cys Glu
20 25 30
Lys His Ser Gln Pro Trp Gln Val Leu Val Ala Ser Arg Gly Arg Ala
35 40 45
Val Cys Gly Gly Val Leu Val His Pro Gln Trp Val Leu Thr Ala Ala
50 55 60
His Cys Ile Arg Asn Lys Ser Val Ile Leu Leu Gly Arg Val Ile Leu
65 70 75 80
Gly Ala His Leu Ser Val Met Val Cys Phe Lys Val Ser Arg His Gly
85 90 95
Ala Val Asn His Va1 Pro Cys Pro Lys Gly Leu Pro Cys Thr Pro Arg
100 105 110
Cys Cys Ile Thr Gly Ser Gly Ser Arg Thr Pro Ser Trp Pro Thr Pro
115 120 125
Glu His Pro Tyr Gln Leu Pro Ile Val Val Asn Leu Glu Pro Trp Lys
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
54
130 135 140
<210> 130
<211> 129
<212> PRT
<213> Homo sapiens
<400> 130
Gly Ala Ala Pro Leu Ile Leu Ser Arg Ile Val Gly Gly Trp Glu Cys
1 5 10 15
Glu Lys His Ser Gln Pro Trp Gln Val Leu Val Ala Ser Arg Gly Arg
20 25 30
Ala Val Cys Gly Gly Val Leu Val His Pro Gln Trp Val Leu Thr Ala
35 40 45
A1a His Cys Ile Arg Asn Lys Ser Val Ile Leu Leu Gly Arg Val Ile
50 55 60
Leu Gly Ala His Leu Ser Val Met Val Cys Phe Lys Val Ser Arg His
65 70 75 80
Gly Ala Val Asn His Val Pro Cys Pro Lys Gly Leu Pro Cys Thr Pro
85 90 95
Arg Cys Cys Ile Thr Gly Ser Gly Ser Arg Thr Pro Ser Trp Pro Thr
100 105 1l0
Pro Glu His Pro Tyr G1n Leu Pro Ile Val Val Asn Leu Glu Pro Trp
l15 120 l25
Lys
<210> 131
<211> 67
<212> PRT
<213> Homo sapiens
<400> 131
Val Ile Leu Gly Ala His Leu Ser Val Met Val Cys Phe Lys Val Ser
1 5 10 15
Arg His Gly Ala Val Asn His Val Pro Cys Pro Lys Gly Leu Pro Cys
20 25 30
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
Thr Pro Arg Cys Cys Ile Thr Gly Ser Gly Ser Arg Thr Pro Ser Trp
35 40 45
Pro Thr Pro Glu His Pro Tyr Gln Leu Pro Ile Val Val Asn Leu Glu
50 55 60
Pro Trp Lys
<210> 132
<211> 90
<212> PRT
<213> Homo sapiens
<400> 132
Met Trp Val Pro Val Val Phe Leu Thr Leu Ser Va1 Thr Trp Ile Gly
1 5 10 15
Glû Arg Gly His Gly Trp Gly Asp Ala Gly Glu Gly Ala Ser Pro Asp
20 25 30
Cys Gln Ala Glu Ala Leu Ser Pro Gln Thr Gln His Pro Ser Ser Leu
35 40 45
Leu Pro Ala Ala Leu Leu Lys Gly Lys Phe Leu Gly Ile Ser Val Phe
50 55 60
Leu Phe Val Gly Leu Lys Thr Ser Lys Asp Leu Ser Gln Cys His Trp
65 70 75 80
Phe Leu Gly Pro Tyr His Trp Ser Thr Ser
85 90
<210> 133
<211> 75
<212> PRT
<213> Homo sapiens
<400> 133
Gly Glu Arg Gly His Gly Trp Gly Asp Ala Gly Glu Gly Ala Ser Pro
1 5 10 15
Asp Cys Gln Ala Glu Ala Leu Ser Pro Gln Thr Gln His Pro Ser Ser
20 25 30
Leu Leu Pro A1a Ala Leu Leu Lys Gly Lys Phe Leu Gly Ile Ser Val
35 40 45
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
56
Phe Leu Phe Val Gly Leu Lys Thr Ser Lys Asp Leu Ser Gln Cys His
50 55 60
Trp Phe Leu Gly Pro Tyr His Trp Ser Thr Ser
65 70 75
<210> 134
<211> 44
<212> PRT
<213> Homo sapiens
<400> 134
Ser Leu Leu Pro Ala A1a Leu Leu Lys Gly Lys Phe Leu Gly Ile Ser
1 5 10 15
Val Phe Leu Phe Val Gly Leu Lys Thr Ser Lys Asp Leu Ser Gln Cys
20 25 30
His Trp Phe Leu Gly Pro Tyr His Trp Ser Thr Ser
35 40
<210> 135
<211> 177
<212> PRT
<213> Homo sapiens
<400> 135
Met Trp Val Pro Val Va1 Phe Leu Thr Leu Ser Val Thr Trp Ile Gly
1 5 10 15
Ala Ala Pro Leu Ile Leu Ser Arg Ile Va1 Gly Gly Trp Glu Cys Glu
20 25 30
Lys His Ser Gln Pro Trp Gln Val Leu Val Ala Ser Arg Gly Arg Ala
35 40 45
Val Cys Gly Gly Val Leu Val His Pro Gln Trp Val Leu Thr Ala Ala
50 55 60
His Cys Thr Cys Tyr Ala Ser Gly Trp Gly Ser Ile G1u Pro Glu Glu
65 70 75 80
Phe Leu Thr Pro Lys Lys Leu Gln Cys Val Asp Leu His Val Ile Ser
85 90 95
Asn Asp Val Cys Ala Gln Val His Pro Gln Lys Val Thr Lys Phe Met
100 105 110
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
57
Leu Cys Ala Gly Arg Trp Thr Gly Gly Lys Ser Thr Cys Ser Gly Asp
115 120 125
Ser Gly Gly Pro Leu Val Cys Asn Gly Val Leu Gln Gly Ile Thr Ser
130 135 140
Trp Gly Ser Glu Pro Cys Ala Leu Pro Glu Arg Pro Ser Leu Tyr Thr
145 150 155 160
Lys Val Val His Tyr Arg Lys Trp Ile Lys Asp Thr Ile Val Ala Asn
165 170 175
Pro
<210> 136
<211> 69
<212> PRT
<213> Homo sapiens
<400> 136
Met Trp Val Pro Val Val Phe Leu Thr Leu Ser Val Thr Trp Ile Gly
1 5 10 15
A1a Ala Pro Leu Ile Leu Ser Arg Ile Val Gly Gly Trp Glu Cys Glu
20 25 30
Lys Hïs Ser Gln Pro Trp Gln Val Leu Val Ala Ser Arg Gly Arg Ala
35 40 45
Val Cys Gly Gly Val Leu Val His Pro Gln Trp Val Leu Thr A1a Ala
50 55 60
His Cys Ile Arg Lys
<210> 137
<211> 61
<212> PRT
<213> Homo sapiens
<400> 137
Ser Ser Pro Gln G1n Cys Phe Cys Leu Ala Arg Ser Leu Asp Pro Lys
1 5 10 15
Glu Thr Ser Val Cys Gly Pro Pro Cys Tyr Phe Gln Ser Arg Val Cys
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
58
20 25 30
Ala Ser Ser Pro Ser Glu Gly Asp Gln Val His Ala Val Cys Cys Thr
35 40 45
Leu Asp Arg Gly Gln Lys His Leu Leu Gly Ala Gly Pro
50 55 60
<210> 138
<211> 147
<212> PRT
<213> Homo sapiens
<400> 138
Pro Ala His Asn Ser Val Phe Ala Trp Pro Val Va1 Leu Thr Pro Lys
1 5 10 15
Lys Leu Gln Cys Val Asp Leu His Val Thr Ser Asn His Val Cys Ala
20 25 30
Gln Val His Pro Gln Lys Val Thr Lys Phe Met Leu Cys Ala Ala Arg
35 40 45
Trp Thr Gly Gly Lys Ser Thr Cys Ser G1u Leu Asp Pro Glu Val Pro
50 55 60
Ser Pro Pro Ala Arg Thr Gly Ala Pro Thr Pro Leu Leu Glu Ser Leu
65 70 75 80
Pro Thr Phe Phe Trp Glu Ser Ala Leu Glu Thr Phe Leu Ser Ser Ser
85 90 95
Lys Ala Gly Asn Cys Tyr Leu Leu Ser A1a Cys Pro Gly Leu Lys Asp
100 105 110
Arg Ile Ala Gln Ala G1u Thr Gly Thr Asp Leu Ser His Ser Leu Pro
115 120 125
Ala Phe Thr Leu Arg Val Ile Leu Gly Ala His Leu Ser Val Met Val
130 135 140
Cys Phe Lys
145
<210> 139
<211> 11
<212> PRT
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
59
<213> Homo sapiens
<400> 139
Val Gln Pro Thr Thr Val Phe Leu Pro Gly Pro
1 5 10
<210> 140
<211> 28
<212> PRT
<213> Homo sapiens
<400> 140
Ser Ser Pro Gln Gln Cys Phe Cys Leu Ala Arg Ser Leu Asp Pro Lys
1 5 10 15
Glu Thr Ser Val Cys Gly Pro Pro Cys Tyr Phe Gln
20 25
<210> 141
<211> 85
<212> PRT
<213> Homo sapiens
<400> 141
Pro Ala His Asn Ser Val Phe Ala Trp Pro Val Val Leu Thr Pro Lys
1 5 10 15
Lys Leu Gln Cys Val Asp Leu His Val Ile Ser Asn Asp Val Cys Ala
20 25 30
Gln Val His Pro Gln Lys Val Thr Lys Phe Met Leu Cys Ala Gly Arg
35 40 45
Trp Thr Gly Gly Lys Ser Thr Cys Ser Trp Val Ile Leu Ile Thr Glu
50 55 60
Leu Thr Met Pro Ala Leu Pro Met Val Leu His Gly Ser Leu Val Pro
65 70 75 80
Trp Arg Gly Gly Val
<210> 142
<211> 11
<212> PRT
<213> Homo sapiens
<400> 142
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
Val Gln Pro Thr Thr Val Phe Leu Pro Gly Pro
1 5 10
<210> 143
<211> 28
<212> PRT
<213> Homo sapiens
<400> 143
Ser Ser Pro G1n Gln Cys Phe Cys Leu Ala Arg Ser Leu Asp Pro Lys
1 5 10 15
Glu Thr Ser Val Cys Gly Pro Pro Cys Tyr Phe Gln
20 25
<210> 144
<211> 100
<212> PRT
<213> Homo sapiens
<400> 144
Pro Ala His Asn Ser Val Phe Ala Trp Pro Val Val Leu Thr Pro Lys
1 5 10 15
Lys Leu Gln Cys Val Asp Leu His Val Ile Ser Asn Asp Val Cys Ala
20 25 30
Gln Val His Pro Gln Lys Val Thr Lys Phe Met Leu Cys Ala Gly Arg
35 40 45
Trp Thr Gly Gly Lys Ser Thr Cys Ser Val Ser His Pro Tyr Pro Ser
50 55 60
Val Gly Ile Pro Ala His Leu Leu Leu Glu Val Gly Ser Gly Asp Ile
70 75 80
Ser Leu Phe Phe Gln Ser Trp Glu Leu Leu Ser Val Ile Cys Leu Ser
85 90 95
Arg Ser Glu Arg
100
<210> 145
<211> 11
<212> PRT
<213> Homo sapiens
<400> 145
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
61
Val Gln Pro Thr Thr Val Phe Leu Pro Gly Pro
1 5 10
<210> 146
<211> 28
<212> PRT
<213> Homo sapiens
<400> 146
Ser Ser Pro Gln Gln Cys Phe Cys Leu Ala Arg Ser Leu Asp Pro Lys
1 5 10 15
Glu Thr Ser Val Cys Gly Pro Pro Cys Tyr Phe Gln
20 25
<210> 147
<211> 61
<212> PRT
<213> Homo sapiens
<400> 147
Pro Ala His Asn Ser Val Phe Ala Trp Pro Val Val Leu Thr Pro Lys
1 5 10 15
Lys Leu Gln Cys Val Asp Leu His Val Thr Ser Asn Asp Val Cys Ala
20 25 30
Gln Val His Pro Gln Lys Val Thr Lys Phe Ser Thr Arg Leu Leu Gly
35 40 45
Thr Cys Tyr Ala Pro Ser Thr Ala Leu Glu Pro Gly Thr
50 55 60
<2l0> 148
<211> 4
<212> PRT
<213> Homo sapiens
<400> 148
Ser Gln Arg Pro
1
<210> 149
<211> 25
<212> PRT
<213> Homo sapiens
<400> 149
Pro Arg Asp Leu Asp Ala Trp Pro Pro Asn Leu Ala Leu Gly Tyr Pro
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
62
1 5 10 15
Asp Ala Thr Ser His Gln Pro Ala Asn
20 25
<210> 150
<211> 12
<212> PRT
<213> Homo sapiens
<400> 150
Pro Glu Thr Leu Met Leu Gly Leu Pro Ile Leu Pro
1 5 10
<210> 151
<211> 4
<212> PRT
<213> Homo sapiens
<400> 151
Ser Gln Arg Pro
1
<210> 152
<211> 25
<212> PRT
<213> Homo sapiens
<400> 152
Pro Arg Asp Leu Asp Ala Trp Pro Pro Asn Leu Ala Leu Gly Tyr Pro
1 5 10 15
Asp Ala Asn Gln Thr Pro Pro Ser Ser
20 25
<210> l53
<211> 12
<2l2> PRT
<213> Homo sapiens
<400> 153
Pro Glu Thr Leu Met Leu Gly Leu Pro Ile Leu Pro
1 5 10
<210> 154
<211> 26
<212> PRT
<213> Homo sapiens
<400> 154
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
63
Met Trp Val Pro Val Val Phe Leu Thr Leu Ser Va1 Thr Trp Ile Ala
1 5 10 15
Arg Leu Ser Gly Leu Arg Gln Gln Met Gly
20 25
<210> 155
<211> 11
<212> PRT
<213> Homo sapiens
<400> 155
Ala Arg Leu Ser Gly Leu Arg Gln Gln Met Gly
1 5 ZO
<210> 156
<21l> 80
<212> PRT
<213> Homo sapiens
<400> 156
Leu Asn Thr Arg Thr Gly Trp Ala Trp Gly Asp Pro Glu Lys Arg Lys
1 5 l0 15
Gly Phe Gly Trp Ala Arg Trp Leu Thr Pro Va1 Ile Pro Ala Leu Trp
20 25 30
Glu Ala Lys Ala Gly Arg Ser Pro Glu Val Arg Ser Ser Arg Pro Ala
35 40 45
Trp Pro Thr Gly Glu Thr Pro Ser Leu Leu Lys Ile Gln Lys Ile Ser
50 55 60
Gln Ala Thr Tyr Pro Leu Gly Asn Pro Arg Gln Ser Arg Ile Tyr Ser
65 70 75 80
<210> 157
<211> 11
<212> PRT
<213> Homo sapiens
<400> 157
Glu His Thr His Gly Met Gly Leu Gly Gly Pro
1 5 10
<210> l58
<211> 11
<212> PRT
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
64
<213> Homo sapiens
<400> 158
Glu His Thr His Gly Met G1y Leu Gly G1y Pro
1 5 10
<210> 159
<211> 18
<212> PRT
<213> Homo sapiens
<400> 159
Asn Thr Arg Thr Gly Trp Ala Trp Gly Asp Pro Glu Lys Ser Arg Ile
1 5 10 15
Tyr Ser
<210> 160
<211> 21
<212> PRT
<213> Homo sapiens
<400> 160
Thr His Ala Arg Asp Gly Pro G1y Gly Thr Leu Arg Lys Ala Ala Ser
1 5 10 15
Thr Ala Glu Pro Leu
<210> 161
<211> 19
<212> PRT
<213> Homo sapiens
<400> 161
Asp Ser Leu Lys Ala Ile Gly Tyr Ser Tyr Ser Arg Glu Glu Val Ala
1 5 10 15
Trp Pro Glu
<210> l62
<211> 5
<212> PRT
<213> Homo sapiens
<400> 162
Thr Pro Ser Arg Gln
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
1 5
<210> 163
<211> 23
<212> PRT
<213> Homo sapïens
<400> 163
Asp Ser Leu Lys Ala Ile Gly Tyr Ser Tyr Ser Thr Thr His Leu Phe
1 5 10 15
Leu Arg Ser Ala His Gly Tyr
<210> 164
<211> 5
<212> PRT
<213> Homo sapiens
<400> 164
Thr Pro Ser Arg Gln
1 5
<210> 165
<211> 25
<212> PRT
<213> Homo sapiens
<400> 165
Cys Trp Thr Glu Ala Gly Gln Gly Leu Ala Gln Gly Asp Ser Gly Gly
1 5 10 15
Pro Leu Val Cys Asn Gly Val Leu Gln
20 25
<210> 166
<211> 25
<212> PRT
<213> Homo sapiens
<400> 166
Ala Gly Gln Lys Gln Asp Arg Ala Trp Leu Arg Va1 Ile Leu Gly Val
1 5 10 15
His Leu Ser Val Met Val Cys Phe Lys
20 25
<210> 167
<211> 11
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
66
<212> PRT
<213> Homo sapiens
<400> 167
Leu Asp Arg Ser Arg Thr Gly Pro G1y Ser Gly
1 5 10
<210> 168
<211> 24
<212> DNA
<213> Homo sapiens
<400> 168
ccagcacccc agctcccagc tgct 24
<210> 169
<211> 24
<212> DNA
<213> Homo sapiens
<400> 169
ccataccccc agcccctccc actt 24
<210> 170
<211> 24
<212> DNA
<213> Homo sapiens
<400> 170
gtgacgtgga ttgctgtgag tgtc 24
<210> 171
<211> 24
<212> DNA
<213> Homo sapiens
<400> 171
aggctctttc cccccaaccc tatc 24
<210> 172
<211> 24
<212> DNA
<213> Homo sapiens
<400> 172
gtgacgtgga ttggataccc agat 24
<210> 173
<211> 24
<212> DNA
<213> Homo sapiens
<400> 173
tcegcctctt attccattct ttct 24
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
67
<210> 174
<211> 24
<212> DNA
<213> Homo sapiens
<400> 174
gcatcaggaa tctccatatc cccc 24
<210> 175
<211> 24
<212> DNA
<213> Homo sapiens
<400> 175
tgacgtggat tgcaccccct ctgc 24
<2l0> 176
<211> 24
<212> DNA
<213> Homo sapiens
<400> 176
gtcttcctca ccctgagctt gtgg 24
<210> 177
<211> 24
<212> DNA
<213> Homo sapiens
<400> 177
cttcctcacc ctgagcttgt ggcc 24
<2l0> 178
<211> 24
<212> DNA
<213> Homo sapiens
<400> 178
tgacgtggat tgggcagtct gcgg 24
<210> 179
<211> 24
<212> DNA
<213> Homo sapiens
<400> 179
gagaaaagaa aggaccctgg ggag 24
<210> 180
<211> 24
<212> DNA
<213> Homo sapiens
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
68
<400> 180
atttcaggtc agcctgccga gatc 24
<210> 181
<211> 24
<212> DNA
<213> Homo sapiens
<400> 181
ctgcatcagg aagccaggtg atga 24
<210> 182
<211> 24
<212> DNA
<213> Homo sapiens
<400> 182
gtgatgactc cagcattgaa ccag 24
<210> 183
<211> 24
<212> DNA
<213> Homo sapiens
<400> 183
gtctcggatt gtctctcgtg gcag 24
<210> 184
<211> 24
<212> DNA
<213> Homo sapiens
<400> 184
ctgggccaga tgtcttgacc ccaa 24
<210> 185
<211> 25
<2l2> DNA
<213> Homo sapiens
<400> 185
tgcatcagga atcttgaccc caaag 25
<210> 186
<211> 24
<212> DNA
<213> Homo sapiens
<400> 186
ttgctgggtc agcattgaac caga 24
<210> 187
<211> 24
<212> DNA
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
69
<213> Homo sapiens
<400> 187
atcttgctgg gtcgggtgat tctg 24
<210> 188
<211> 24
<212> DNA
<213> Homo sapiens
<400> 188
cttgctgggt cgggtgattc tggg 24
<210> 189
<211> 24
<212> DNA
<213> Homo sapiens
<400> 189
ctgcccactg cacctgctac gcct 24
<210> 190
<211> 25
<212> DNA
<2l3> Homo sapiens
<400> 190
ccctctcttc tctgtctcac ctgtg 25
<210> 191
<211> 24
<212> DNA
<213> Homo sapiens
<400> 191
agcacctgct cggagctgga ccct 24
<210> 192
<211> 24
<212> DNA
<213> Homo sapiens
<400> 192
aagcacctgc tcgtgggtca ttct 24
<210> 193
<211> 24
<212> DNA
<213> Homo sapiens
<400> 193
cacctgctcg gtgagtcatc ccta 24
<210> 194
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
7~
<211> 24
<212> DNA
<213> Homo sapiens
<400> 194
agaaggtgac caagttcagc acac 24
<210> 195
<211> 24
<212> DNA
<213> Homo sapiens
<400> 195
acccagatgc caccagccac caac 24
<210> 196
<21l> 24
<212> DNA
<213> Homo sapiens
<400> 196
ccttaggaaa aacatgaagc ctct 24
<210> 197
<211> 24
<212> DNA
<213> Homo sapiens
<400> 197
gtgacgtgga ttgccaggct atct 24
<210> 198
<211> 24
<2l2> DNA
<213> Homo sapiens
<400> 198
aaaattagcc aggctaccta ccca 24
<210> 199
<2l1> 24
<212> DNA
<213> Homo sapiens
<400> 199
ccctgagaaa agccgcatct acag 24
<210> 200
<211> 25
<212> DNA
<213> Homo sapiens
<400> 200
ggttattctt acagcagaga ggagg 25
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
71
<210> 201
<211> 24
<2l2> DNA
<213> Homo sapiens
<400> 201
gctctcaggg agggcagcag ggat 24
<210> 202
<211> 24
<212> DNA
<213> Homo sapiens
<400> 202
ggcctggctc agggtgattc tggg 24
<210> 203
<211> 24
<212> DNA
<213> Homo sapiens
<400> 203
tcacttctca ggaatagcca ggtc 24
<210> 204
<211> 24
<212> DNA
<213> Homo sapiens
<400> 204
gatgttgtga aggagtcttc agtg 24
<210> 205
<211> 24
<212> DNA
<213> Homo sapiens
<400> 205
tggggtgcac tggaatagcc aggt 24
<210> 206
<211> 24
<212> DNA
<213> Homo sapiens
<400> 206
ctggagggga aagggtgatt ctgg 24
<210> 207
<211> 24
<212> DNA
<213> Homo sapiens
<400> 207
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
72
ttgcctaaag aatcttgcgc ccca 24
<210> 208 '
<211> 24
<212> DNA
<213> Homo sapiens
<400> 208
gaaccagagg agtgagtctt cagt 24
<210> 209
<211> 24
<212> DNA
<213> Homo sapiens
<400> 209
tgaagactcc agcatcgaac calta 24
<210> 210
<211> 24
<212> DNA
<213> Homo sapiens
<400> 210
aaccagagga gtggtaaaga cact 24
<210> 211
<211> 25
<212> DNA
<213> Homo sapiens
<400> 211
ctgactcttc cccccgaggc tatct 25
<210> 212
<211> 24
<212> DNA
<213> Homo sapiens
<400> 212
tggggtgcac tgacccgtca ttca 24
<210> 213
<211> 24
<212> DNA
<2l3> Homo sapiens
<400> 213
cagcctcgtc cccccaacca caac 24
<210> 214
<211> 24
<212> DNA
<213> Homo sapiens
CA 02478572 2004-09-09
WO 03/076610 PCT/FR03/00833
73
<400> 214
tagtggaacc ctgctatctg ccga 24
<210> 215
<211> 24
<212> DNA
<213> Homo sapiens
<400> 215
ttttctcagg aatagccagg tctg 24
<210> 216
<211> 24
<212> DNA
<213> Homo sapiens
<400> 216
gatgggcaca ctcctgtttt ctaa 24
<210> 217
<211> 24
<212> DNA
<213> Homo sapiens
<400> 217
acatccctcc accctcatgc ctct 24
<210> 218
<211> 24
<212> DNA
<213> Homo sapiens
<400> 218
agtctctccc ctccactcca ttct 24
<210> 219
<211> 24
<212> DNA
<213> Homo sapiens
<400> 219
cctgccgatg gcccacttgt ctgt 24
<210> 220
<211> 24
<212> DNA
<213> Homo sapiens
<400> 220
ccccagctgc aggaatagcc aggt 24