Note : Les descriptions sont présentées dans la langue officielle dans laquelle elles ont été soumises.
CA 03113884 2021-03-23
WO 2020/064855
PCT/EP2019/075897
1
PROCEDE D'ANALYSE AUTOMATISEE DES CONTRACTIONS CELLULAIRES D'UN
ENSEMBLE DE CELLULES BIOLOGIQUES.
DOMAINE DE L'INVENTION
La présente invention concerne le domaine de l'analyse des contractions
cellulaires d'un
ensemble de cellules biologiques contractiles, qu'elles soient in-vitro ou in-
vivo.
Une telle analyse des contractions permet par exemple de déduire certaines
propriétés, par
exemple physiques ou physiologiques, des cellules biologiques.
Si une telle analyse peut être réalisée par des individus manuellement, il
existe un besoin
d'automatisation, ce qui permet d'une part de pouvoir traiter de gros volumes
de données et
d'autre part d'obtenir des résultats plus objectifs et plus robustes.
Il est proposé ici une solution permettant l'analyse des contractions
cellulaires d'un ensemble
de cellules biologiques contractiles, basée sur un traitement numérique
d'images dudit ensemble
de cellules.
Par concision, on entend indistinctement cellules , cellules biologiques
et cellules
biologiques contractiles .
Par exemple, les cellules sont des cellules souches pluripotentes induites, ou
IPS pour
Induced Pluripotent Stem cells en Anglais, qui ont le potentiel de se
différencier en n'importe
quelle cellule du corps humain, en particulier des cellules contractiles
composant le muscle
cardiaque ou cardiomyocytes .
Les cardiomyocytes différenciés à partir d'IPS sont particulièrement
intéressantes car le
coeur est un organe qui se régénère peu, il est difficile d'avoir accès aux
cardiomyocytes d'un
patient. Or les maladies cardiaques sont souvent héréditaires, le patrimoine
génétique est donc
important et l'intérêt de la présente invention à ce sujet sera discuté
ultérieurement.
Par concision, seules ces cardiomyocytes seront décrits ci-après. Bien
entendu, l'invention
n'est pas limitée à ce type de cellules et concerne tout type de cellule
biologique contractile :
cellules cardiaques, cellules musculaires.
Les cellules souches pluripotentes induites, dérivées à partir d'un patient
malade ou d'une
personne saine, différenciées en cardiomyocytes, présentent une contraction
spontanée, rythmée,
CA 03113884 2021-03-23
WO 2020/064855
PCT/EP2019/075897
2
qui peut être obtenue en quelques semaines, avec l'avantage particulier de
conserver le
patrimoine génétique du patient. Elles peuvent donc être utilisées
avantageusement pour tester de
nouveaux médicaments et étudier les pathologies associées à ces médicaments, y
compris les
effets secondaires.
Toutefois, les cellules souches pluripotentes induites, différenciées en
cardiomyocytes, ne
servent pas uniquement à tester des médicaments sur des cellules malades mais
peuvent
également être utilisées pour tester la toxicité de substances sur des
cellules saines.
Auparavant, il était quasiment impossible de travailler directement sur des
cellules
cardiaques humaines car les prélèvements cardiaques sont très compliqués.
Maintenant, grâce
aux cellules souches pluripotentes (issues de simples échantillons de peau, de
sang ou d'urine), il
est facile d'obtenir des cardiomyocytes humains, sains ou malades.
Bien qu'il existe des techniques disponibles pour caractériser les
cardiomyocytes telles que
par exemple l'électrophysiologie ou la microscopie AFM pour Atomic Force
Microscope en
Anglais, de telles techniques sont coûteuses, souvent difficiles à appliquer à
grandes échelles et à
mettre en place, et elles exigent souvent des consommables spéciaux
nécessitant de repiquer les
cellules, ce qui n'est pas toujours possible ni désirable.
Il est proposé ici une solution plus complète, plus flexible, plus simple et
moins chère. Elle
est facilement accessible et permet la caractérisation de paramètres des
contractions des cellules.
En outre, la présente invention présente l'avantage de ne pas nécessiter de
fluorescence.
RESUME DE L'INVENTION
Selon un premier de ses objets, l'invention concerne un procédé d'analyse
automatisée des
contractions cellulaires d'un ensemble de cellules biologiques, comprenant :
- Déterminer la position d'un ensemble de points remarquables (11) sur la
première image
(10) d'une séquence temporelle d'images dudit ensemble de cellules
biologiques, ladite séquence
comprenant une première image (10) et un ensemble d'au moins une deuxième
image (20).
Il est essentiellement caractérisé en ce qu'il comprend en outre des étapes
consistant à:
- Déterminer la position dudit ensemble de points remarquables (11) sur la
deuxième image
(20),
Et pour au moins un point remarquable (11) :
CA 03113884 2021-03-23
WO 2020/064855
PCT/EP2019/075897
3
- Déterminer le déplacement dudit point remarquable (11) entre la première
image (10) et la
deuxième image (20), par comparaison de la position dudit premier point
remarquable (11) de la
première image (10) et la position dudit deuxième point remarquable (11)
correspondant de la
deuxième image (20).
On peut prévoir en outre :
- Définir une première zone d'intérêt (12) autour dudit point remarquable
(11) de la
première image (10),
- Calculer l'intensité de ladite première zone d'intérêt (12) de la
première image (10),
- Définir une deuxième zone d'intérêt (22) sur la deuxième image (20), de
préférence
centrée sur un point sur la deuxième image (20) ayant les mêmes coordonnées
que ledit point
remarquable (11) de la première image (10), ladite deuxième zone d'intérêt
(22) étant de même
forme et de mêmes dimensions que la première zone d'intérêt (12),
- Déplacer la deuxième zone d'intérêt (22) sur un ensemble de positions
dans une fenêtre
d'arrivée (23) de la deuxième image (20), et à chaque position :
o calculer l'intensité de la deuxième zone d'intérêt (22), et
o calculer une corrélation entre l'intensité de la deuxième zone d'intérêt
(22) de la deuxième
image (20) et l'intensité de première zone d'intérêt (12) de la première image
(10).
On peut prévoir que l'étape consistant à:
- Déterminer le déplacement dudit point remarquable (11) entre la première
image (10) et la
deuxième image (20),
Comprend des étapes préalables consistant à :
- réduire la résolution de la première image (10) et la résolution de la
deuxième image (20)
selon une pyramide d'images ;
- rechercher la première zone d'intérêt (12) de la première image (10) dans
l'une des
images de la pyramide d'images.
De préférence, pour une plus grande robustesse, le calcul de corrélation est
répété
séquentiellement sur la paire de première et deuxième image, en utilisant les
mêmes images à
plus faibles résolutions pour commencer.
On peut prévoir des étapes consistant à, pour chaque fenêtre d'arrivée (23) :
- calculer une matrice de corrélation correspondant à l'ensemble de
positions de la
deuxième zone d'intérêt (22) dans ladite fenêtre,
- détecter la position du pic de corrélation dans ladite matrice de
corrélation, et
CA 03113884 2021-03-23
WO 2020/064855
PCT/EP2019/075897
4
- assigner comme position du point remarquable (11) sur la deuxième image
(20) ladite
position correspondant au pic de corrélation.
On peut prévoir des étapes consistant à :
- Afficher sur un écran d'affichage au moins l'une parmi la première image
(10) et la
deuxième image (20), et
- Sur au moins une image affichée, représenter graphiquement un vecteur
entre ledit point
remarquable (11) de la première image (10) et ledit point remarquable (11) de
la deuxième image
(20).
Ledit vecteur représente graphiquement le déplacement du point remarquable
entre la
première et la deuxième image.
On peut prévoir des étapes consistant à :
- Déterminer une onde de contraction cellulaire de l'ensemble de cellules
biologiques par
calcul du déplacement dudit point remarquable entre la première image (10) et
l'ensemble d'au
moins une deuxième image (20) en fonction du temps (t), et
- Calculer au moins l'une des valeurs parmi :
o l'amplitude (A) de l'onde de contraction cellulaire de l'ensemble de
cellules biologiques,
o la vitesse de propagation de l'onde de contraction cellulaire de l'ensemble
de cellules
biologiques,
o au moins un gradient de vitesse de propagation de l'onde de contraction
cellulaire de
l'ensemble de cellules biologiques,
o la période (T) de l'onde de contraction cellulaire de l'ensemble de
cellules biologiques,
o la fréquence (V) de l'onde de contraction cellulaire de l'ensemble de
cellules biologiques,
o l'intervalle de temps entre :
= la première image (10) et
= la première deuxième image (20) de la séquence pour laquelle le
déplacement du point remarquable (11) entre la première image (10) et la
deuxième image (20) est maximal ;
o l'intervalle de temps entre :
= la deuxième image (20) pour laquelle le déplacement du point
remarquable (11) entre la première image (10) et la deuxième image (20)
est maximale, et
= la première deuxième image (20) de la séquence pour laquelle le
déplacement du point remarquable (11) entre la première image (10) et la
deuxième image (20) est nulle.
CA 03113884 2021-03-23
WO 2020/064855
PCT/EP2019/075897
On peut prévoir une étape consistant à détecter pendant une période (T) au
moins de l'onde
de contraction cellulaire si l'amplitude de ladite onde de contraction
cellulaire :
- Passe par un nombre de maximum (El, E3) prédéterminé et dont la valeur
est supérieure
5 à une valeur seuil prédéterminée, ou
- passe par un nombre de minimum (E2) prédéterminé et dont la valeur est
inférieure à une
valeur seuil prédéterminée.
On peut prévoir en outre une étape de criblage pharmacologique.
On peut prévoir en outre une étape préalable de microscopie à contraste de
phase dudit
ensemble de cellules biologiques pour obtenir ladite séquence temporelle
d'images.
Selon un autre de ses objets, l'invention concerne un programme d'ordinateur
comprenant
des instructions de code de programme pour l'exécution des étapes du procédé
selon l'invention,
lorsque ledit programme est exécuté sur un ordinateur.
D'autres caractéristiques et avantages de la présente invention apparaîtront
plus clairement
à la lecture de la description suivante donnée à titre d'exemple illustratif
et non limitatif et faite en
référence aux figures annexées.
DESCRIPTIF DES DESSINS
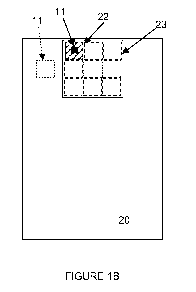
- la figure 1A illustre une première image comprenant un point remarquable
et une
première zone d'intérêt selon l'invention, et
- la figure 1B illustre une deuxième image comprenant ce même point
remarquable mais
déplacé et une deuxième zone d'intérêt selon l'invention, correspondant au
déplacement
du point remarquable après une contraction des cellules imagées sur la figure
1A, et le
déplacement de la deuxième zone d'intérêt dans une fenêtre d'arrivée,
- la figure 2A illustre une onde de contraction de cellules saines,
calculée selon l'invention,
- la figure 2B illustre une onde de contraction de cellules affectées,
calculée selon
l'invention,
- la figure 3A illustre la fréquence de l'onde de contraction cardiaque des
cellules de 3
individus,
- la figure 3B illustre la fréquence moyenne de contraction cardiaque des
cellules des 3
individus de la figure 3A, après une phase de maturation,
CA 03113884 2021-03-23
WO 2020/064855
PCT/EP2019/075897
6
- la figure 30 illustre l'amplitude de l'onde de contraction cardiaque des
cellules des 3
individus de la figure 3A,
- la figure 3D illustre l'amplitude moyenne de contraction cardiaque des
cellules des 3
individus de la figure 3A, après une phase de maturation.
DESCRIPTION DETAILLEE
La présente invention est basée sur un traitement d'images. On prévoit donc
d'enregistrer
préalablement une séquence temporelle d'images d'un ensemble de cellules. Le
traitement
d'image peut être réalisé en temps réel sur une séquence temporelle d'images,
ou en différé, à
partir d'une séquence temporelle d'images enregistrée préalablement.
La séquence d'images comprend une première image 10 (par exemple au temps t=1)
et un
ensemble d'au moins une deuxième image 20, en l'espèce consécutive à la
première image 10,
c'est à dire une deuxième image 20 (par exemple au temps t=1+dt), une
troisième image (par
exemple au temps t=1+2dt), une quatrième image (par exemple au temps t=1+3dt),
etc. avec dt un
intervalle de temps prédéterminé.
Par exemple, la séquence d'images peut être enregistrée sous forme de vidéo.
On peut prévoir que la séquence d'images est obtenue grâce à une étape
préalable de
microscopie à contraste de phase dudit ensemble de cellules biologiques, en
particulier sous
forme monocouche.
Pour simplifier, on considère que la première image 10 est la première image
10 de la
séquence et que la deuxième image 20 est la deuxième image 20 de la séquence.
On prévoit de déterminer la position d'un ensemble de points remarquables sur
la première
image 10. Puisque les cellules sont contractiles, il est fortement probable
que la position de ces
points remarquables varie d'une image à l'autre. On prévoit donc de déterminer
la position de cet
ensemble de points remarquables sur la deuxième image 20, comme décrit plus en
détails ci-
après.
De préférence, on prévoit de déterminer la position de l'ensemble de points
remarquables
sur chaque image de la séquence.
CA 03113884 2021-03-23
WO 2020/064855
PCT/EP2019/075897
7
Par concision, on entend indistinctement par deuxième image : la deuxième
image de la
séquence, l'une des images de la séquence, plusieurs images de la séquence ou
toutes les
images de la séquence, autre que la première image 10.
Identification d'un point remarquable 11 sur la 1è' image.
On prévoit d'identifier un ensemble d'au moins un point remarquable 11 dans la
première
image 10. Par concision, on entend indistinctement un ensemble d'au moins un
point
remarquable 11 et un point remarquable 11 .
Par point remarquable , on entend un pixel ou un ensemble de pixels
adjacents deux à
deux qui présente une luminosité ou une intensité (où par intensité on entend
l'intensité lumineuse
mesurée par un capteur, c'est à dire le nombre de photons par unité de temps
et de surface dudit
capteur) supérieure à une valeur seuil ; ou pour lequel le gradient de
contraste ou d'intensité, selon
.. une direction et une distance prédéfinies, est supérieur à une valeur seuil
prédéfinie.
Typiquement, les points remarquables sont les points les plus lumineux de la
première
image 10, c'est à dire des maxima locaux d'intensité, ou les plus contrastés.
On peut par exemple
utiliser une fonction LOCALMAX du logiciel Matlab (marque déposée) ou d'autres
fonctions
équivalentes de logiciels équivalents appliqués à la première image 10.
Une fois les points remarquables identifiés, les coordonnées des points
remarquables sont
connues.
Si la cellule a subi une contraction entre la première image 10 et la deuxième
image 20,
alors au moins un point remarquable 11 de la première image 10 a une position
différente sur la
deuxième image 20, qu'il convient de déterminer comme décrit ultérieurement.
Une fois la position d'un point remarquable 11 sur la première image 10 et la
position dudit
point remarquable 11 sur la deuxième image 20 connus, on peut alors déterminer
le déplacement
dudit point remarquable 11 entre la première image 10 et la deuxième image 20.
En effet, par comparaison de la position (des coordonnées) dudit point
remarquable 11 de la
première image 10 et la position dudit point remarquable 11 de la deuxième
image 20, et
connaissant l'intervalle de temps séparant la première et la deuxième image
20, on peut calculer le
déplacement dudit point remarquable 11, c'est à dire au moins l'un parmi :
CA 03113884 2021-03-23
WO 2020/064855
PCT/EP2019/075897
8
- la vitesse de déplacement du point remarquable 11 entre sa position sur
la première
image 10 et sa position sur la deuxième image 20, et
- la distance séparant la position du point remarquable 11 sur la première
image 10 et sa
position sur la deuxième image 20.
Identification de la position du point remarquable 11 sur la 2ème image.
La position du point remarquable sur la deuxième image 20 est déterminée comme
suit.
On prévoit tout d'abord de définir une première zone d'intérêt 12 autour dudit
point
remarquable 11 de la première image 10.
Une zone d'intérêt est l'ensemble des pixels compris dans une sous-partie
d'une image et
qui présente une forme prédéterminée, en l'espèce un rectangle dont le
barycentre est le point
remarquable 11. Les coordonnées de la première zone d'intérêt sont donc
connues.
On peut alors calculer l'intensité de ladite première zone d'intérêt 12 de la
première image
10.
Ensuite, de manière similaire, on prévoit de définir une deuxième zone
d'intérêt 22, sur la
deuxième image 20.
La deuxième zone d'intérêt 22 (sur la deuxième image 20) présente la même
forme et les
mêmes dimensions que la première zone d'intérêt 12 (sur la première image 10),
et de préférence
présente initialement la même position.
Il s'agit alors de déterminer la position de la deuxième zone d'intérêt 22 sur
la deuxième
image 20 telle que ladite position correspond au déplacement du point
remarquable 11 entre la
première image 10 et la deuxième image 20.
A cet effet, on prévoit de déplacer la deuxième zone d'intérêt 22 sur un
ensemble de
positions prédéterminées dans une fenêtre d'arrivée 23 de la deuxième image
20, la fenêtre
d'arrivée 23 ayant elle-même une forme, une dimension et une position
prédéterminées.
A chaque position de la deuxième zone d'intérêt 22 dans la fenêtre d'arrivée
23 de la
deuxième image 20, on prévoit de calculer l'intensité (ou niveaux de gris) de
la deuxième zone
d'intérêt 22, et de calculer une corrélation entre l'intensité de la deuxième
zone d'intérêt 22 de la
CA 03113884 2021-03-23
WO 2020/064855
PCT/EP2019/075897
9
deuxième image 20 pour cette position et l'intensité de première zone
d'intérêt 12 de la première
image 10.
Par exemple sur la figure 1 B, on a représenté en pointillés fins la position
de la première
zone d'intérêt 12 est en grands pointillés un ensemble de positions
prédéterminées de deuxième
zone d'intérêt 22. En l'espèce, à titre purement illustratif, on a représenté
neuf positions adjacentes
pour la deuxième zone d'intérêt 22.
Chaque valeur de corrélation calculée pour une position donnée de la deuxième
zone
d'intérêt 22 dans la fenêtre d'arrivée 23 est enregistrée comme coefficient
d'une matrice de
corrélation, qui comprend autant de colonnes et de lignes que de pixels de
déplacement de la
deuxième zone d'intérêt 22.
On peut alors détecter, c'est à dire calculer, la position du pic de
corrélation dans ladite
matrice de corrélation.
On peut alors sélectionner la position de la deuxième zone d'intérêt 22 qui
présente le
maximum de corrélation (pic de corrélation), c'est à dire la position pour
laquelle plus l'intensité de
la deuxième zone d'intérêt 22 est proche de l'intensité de la première zone
d'intérêt 12, pour
définir, c'est à dire assigner, la position du point remarquable 11 de la
deuxième image 20, ce qui
assure la détermination de la position du point remarquable 11 sur la deuxième
image 20.
De préférence on calcule autant de matrices de corrélation que de points
remarquables, par
image de l'ensemble d'au moins une deuxième image 20. A titre purement
illustratif, pour une
séquence d'images de 101 images, c'est à dire une première image 10 et un
ensemble de 100
deuxième image 20, et un ensemble de 25 points remarquables par deuxième image
20, on
calcule ainsi 251 00=2500 matrices de corrélation. De préférence, on prévoit
donc d'effectuer les
calculs de matrices de corrélation en parallèle, en l'espèce sur carte
graphique.
La taille de la fenêtre d'arrivée 23 de la deuxième image 20 peut être
proportionnelle à la
taille de la zone d'intérêt de la première image 10.
Par exemple, la taille de la fenêtre d'arrivée 23 de la deuxième image 20 est
égale à la taille
de la zone d'intérêt de la première image 10. Par exemple, la deuxième zone
d'intérêt 22 est
déplacée de un pixel pour chaque position dans la fenêtre d'arrivée 23.
CA 03113884 2021-03-23
WO 2020/064855
PCT/EP2019/075897
On peut aussi prévoir une étape consistant à estimer préalablement le
déplacement moyen
des points d'intérêts et dimensionner la taille de la fenêtre d'arrivée 23 en
fonction dudit
déplacement moyen des points d'intérêts.
5 Si un point remarquable 11 se déplace sur une position qui n'est pas
comprise dans l'une
des positions de la deuxième zone d'intérêt 22 de la fenêtre d'arrivée 23,
alors il n'est pas visible
sur la deuxième image 20.
Pour limiter ce risque et optimiser les calculs, on peut prévoir une étape de
création de
10 pyramides d'images qui consiste à obtenir une représentation multi-
résolution de la première
image 10 ou de la deuxième image 20, depuis l'image initiale jusqu'à une image
très grossière de
celle-ci.
Ainsi, on peut prévoir de réduire la résolution de la deuxième image 20 selon
une pyramide
d'images, en l'espèce une pyramide Gaussienne, et d'enregistrer l'ensemble
d'images obtenues,
chaque image ayant une résolution et une taille correspondantes.
On peut alors rechercher la première zone d'intérêt 12 de la première image 10
dans l'une
au moins des images de la pyramide d'images, et de préférence en boucle sur
l'ensemble des
images de la pyramide, en commençant par celle de plus petite taille.
Comme la taille des images de la pyramide est inférieure à celle de l'image
d'origine mais
que la taille de la zone d'intérêt reste la même, le ratio entre les deux
diffère à chaque image de la
pyramide, ce qui permet d'une part d'obtenir une solution robuste et d'autre
part de pouvoir estimer
le déplacement du point remarquable 11 entre la première et la deuxième image
20.
Grâce à la pyramide d'images, l'estimation du déplacement est réalisée au
moins
grossièrement sur les images de la pyramide de petite taille et de plus en
plus précis à chaque
image de taille supérieure.
Typiquement, on peut utiliser à cet effet un algorithme de suivi de mouvement
ou de suivi de
points, en l'espèce l'algorithme KLT (pour Kanade¨Lucas¨Tomasi), connu
notamment pour
l'asservissement des caméras embarquées, appliqué à chaque image de la
pyramide d'images.
D'un point de vue graphique, on peut prévoir d'afficher sur un écran
d'affichage au moins
l'une parmi la première image 10 et la deuxième image 20 et, sur au moins une
image affichée, on
CA 03113884 2021-03-23
WO 2020/064855
PCT/EP2019/075897
11
peut prévoir de représenter graphiquement un vecteur entre ledit point
remarquable 11 de la
première image 10 et ledit point remarquable 11 de la deuxième image 20.
Par exemple, on peut prévoir de tracer un vecteur sur la première image 10 ou
la deuxième
image 20 dont l'origine est la position du point remarquable 11 dans la
première image 10 et dont
l'extrémité est la position du point remarquable 11 dans la deuxième image 20,
ce qui permet
d'illustrer graphiquement le déplacement du point remarquable 11 entre deux
images successives
de la séquence.
D'une image à l'autre on peut obtenir des vecteurs de champ de déplacement,
qui
contiennent une information spatiale et temporelle du déplacement de points
remarquables, donc
de la contraction.
On peut calculer la norme de l'ensemble des vecteurs d'une image et
l'intensité du champ de
vecteurs par moyenne de tous les vecteurs.
Le traitement d'images ainsi réalisé permet de calculer le déplacement d'un
point
remarquable 11 entre la première image 10 et l'ensemble de deuxièmes images.
Le temps
séparant la première image 10 de l'ensemble des deuxièmes images de la
séquence est connu.
La distance séparant la position du point remarquable 11 sur la première image
10 et la position du
point remarquable 11 sur l'ensemble de deuxièmes images est calculée, en se
basant sur la taille
des pixels, connue.
Ainsi, on peut déterminer la variation du déplacement d'un point remarquable
11 en fonction
du temps t, cette variation temporelle étant une onde de contraction
cellulaire, comme illustré sur
la figure 2A.
En moyennant les déplacements de l'ensemble des points remarquables, on peut
déterminer
une onde de contraction cellulaire de l'ensemble de cellules biologiques.
On peut prévoir d'afficher une courbe représentative de l'onde de contraction
cellulaire sur
un écran d'affichage.
On peut prévoir une étape d'interpolation de la courbe représentative de
l'onde de
contraction cellulaire.
CA 03113884 2021-03-23
WO 2020/064855
PCT/EP2019/075897
12
Grâce aux valeurs de la courbe, et le cas échéant par transformée de Fourier,
on peut
prévoir de calculer au moins l'une des valeurs parmi :
o l'amplitude A de l'onde de contraction cellulaire de l'ensemble de
cellules
biologiques, et notamment l'amplitude maximale Amax, c'est à dire la distance
maximale entre la position dudit point remarquable 11 de la première image 10
et
l'ensemble des positions correspondantes du point remarquable 11 parmi
l'ensemble d'au moins une deuxième image 20,
o la vitesse de propagation de l'onde de contraction cellulaire de
l'ensemble de
cellules biologiques, c'est à dire la vitesse de propagation dudit point
remarquable
11, qui est un marqueur du couplage entre cellules adjacentes,
o les gradients de vitesse de propagation de l'onde de contraction
cellulaire de
l'ensemble de cellules biologiques,
o la période T de l'onde de contraction cellulaire de l'ensemble de
cellules
biologiques,
o la fréquence 1/T de l'onde de contraction cellulaire de l'ensemble de
cellules
biologiques,
o le temps de montée Tm, c'est à dire l'intervalle de temps entre :
= la première image 10 et
= la première deuxième image 20 de la séquence pour laquelle la distance
entre le point remarquable 11 de la deuxième image 20 et le point
remarquable 11 de la première image 10 est maximale ;
o le temps de descente Td, c'est à dire l'intervalle de temps entre :
= la deuxième image 20 pour laquelle la distance entre le point remarquable
11 de la deuxième image 20 et le point remarquable 11 de la première
image 10 est maximale, et
= la première deuxième image 20 de la séquence pour laquelle la distance
entre le point remarquable 11 de la deuxième image 20 et le point
remarquable 11 de la première image 10 est nulle.
Ainsi, il est possible de déterminer la vitesse et la durée de contraction, la
vitesse et la durée
de relaxation, la fréquence et l'amplitude de contraction.
On peut aussi prévoir, de détecter pendant une période au moins de l'onde de
contraction
cellulaire si l'amplitude de ladite onde de contraction cellulaire passe par:
- au moins deux extrema dont la valeur est supérieure à une valeur seuil
prédéterminée,
OU
- comprend un nombre d'extrema supérieur ou inférieur à une valeur
seuil prédéterminée.
CA 03113884 2021-03-23
WO 2020/064855
PCT/EP2019/075897
13
Comme illustré sur la figure 2B l'amplitude de l'onde de contraction passe par
un premier
maximum El, un minimum E2 et un deuxième maximum E3. Cependant, la valeur du
minimum E2
de la figure 2B est très inférieure à la valeur du minimum E2 de la figure 2A,
ce qui peut être
détecté par comparaison à une valeur seuil.
Il est ainsi possible de détecter des variations dans le rythme des
contractions cellulaires, et
de détecter des événements contractiles aberrants.
Il est ainsi possible de tester la sécurité, l'efficacité ou la toxicité d'une
molécule particulière
sur des cellules biologiques, en particulier humaines, préalablement à tout
test sur un modèle
animal. On peut donc prévoir une étape de criblage pharmacologique.
Par exemple, on enregistre l'onde de contraction cellulaire pour un individu
sain (figure 2A),
on soumet ces cellules à une molécule particulière et on enregistre l'onde de
contraction cellulaire
dans ce contexte (figure 2B). On peut ainsi détecter les effets physiologiques
de la molécule in-
vitro mais aussi directement sur des cellules ayant un patrimoine génétique
désiré.
La présente invention trouve donc un intérêt particulier pour le test de
nouvelles molécules,
de nouveaux médicaments, mais également dans un contexte de pharmacovigilance,
ou encore
pour étudier des pathologies associées, y compris les effets secondaires,
comme décrit ci-
dessous.
Analyse vidéo
Chaque point de la figure 3A, 3B, 3C et 3D correspond au résultat ou
traitement des cellules
d'un individu, correspondant par exemple à l'une des courbes illustrées sur la
figure 2A ou la figure
2B.
Sur les figures 3A, 3B, 3C et 3D:
- H représente un individu sain ;
- M1 représente un individu présentant une première anomalie connue, par
exemple une
mutation génétique ;
- M2 représente un individu présentant une deuxième anomalie connue, par
exemple une
autre mutation génétique, ou un second clone de M1 .
CA 03113884 2021-03-23
WO 2020/064855
PCT/EP2019/075897
14
La figure 3A représente la fréquence de l'onde de contraction cardiaque pour 3
individus.
Clairement la fréquence moyenne de l'individu H est supérieure à celle de
l'individu Ml, elle-même
supérieure à celle de l'individu M2.
La figure 3B illustre la réponse des cellules, c'est à dire la fréquence
moyenne de
contraction, des individus H, M1 et M2 après une phase de maturation des
cellules.
La figure 3C représente l'amplitude moyenne de l'onde de contraction cardiaque
pour les 3
mêmes individus.
La figure 3D illustre la réponse des cellules, c'est à dire l'amplitude
moyenne de contraction,
des individus H, M1 et M2 après une phase de maturation des cellules.
Il est clair que les effets d'une molécule donnée sur la contraction des
cellules peuvent ainsi
.. être mesurés. Ce qui peut permettre non seulement de tester directement par
exemple l'effet d'une
molécule, en particulier thérapeutique et par exemple destinée typiquement au
traitement d'une
pathologie cardiaque, mais aussi de tester les effets secondaires sur des
cardiomyocytes humains
ou d'animaux d'une molécule thérapeutique destinée typiquement au traitement
d'une pathologie
d'un autre organe, par exemple du foie.
Au-delà de l'imagerie de contractions cellulaires l'invention peut être mise
en oeuvre dans
d'autres domaines que la biologie, par exemple en chimie ou en physique, pour
l'étude par
imagerie de tout phénomène ondulatoire, résonnant ou vibratoire, de préférence
périodique.
Nomenclature
El Premier maximum
E2 Premier minimum
E3 Deuxième maximum
10 Première image
11 Point remarquable
12 Première zone d'intérêt
20 Deuxième image
22 Deuxième zone d'intérêt
23 Fenêtre d'arrivée