Note : Les descriptions sont présentées dans la langue officielle dans laquelle elles ont été soumises.
CA 02766704 2011-12-23
WO 2011/000933 PCT/EP201(1/(159413
Variants améliorés d'une phytase
La présente invention relève du domaine de l'amélioration des protéines et en
particulier
celui des protéines améliorées par évolution moléculaire. Elle porte sur un
variant d'une
phytase, dit amélioré, en ce qu'il présente une thcrmostabilité et/ou une
activité supérieures
à la phytase d'origine. L'invention porte également sur un acide nucléique
codant pour ce
variant, une cassette ou un vecteur d'expression contenant ce variant, une
cellule hôte
exprimant ce variant, une composition comprenant ce variant ainsi que les
utilisations de
ceux-ci, principalement dans la préparation d'additifs alimentaires et
l'alimentation
animale.
Le phytate est le principal composé de stockage du phosphore chez la plante.
Cette
molécule, aussi nommée acide phytique ou inositol-hexa-phosphate (InsP6 ou
.myo-inositol
hexakisphosphate), consiste en un cyclo-hexane sur lequel sont branchés six
groupements
phosphates. Le phytate représente environ 70% du phosphate végétal, les 30%
restants
étant présents sous forme libre. Par ailleurs, les résidus phosphate du
phytate chélatent les
cations divalents et trivalents tels que les ions métalliques calcium, fer,
zinc, magnésium,
cuivre, manganèse et molybdène essentiels à la nutrition.
Les phytases sont des enzymes qui hydrolysent le phytate: ces enzymes libèrent
naturellement un ou deux phosphates, rarement trois, alors adsorbables dans le
système
digestif ; les autres produits de la réaction étant rarement de l'inositol-tri-
phosphate,
principalement de l'inositol tétra- et penta-phosphate (Figure 1). Les
phytases constituent
une famille d'enzymes largement représentées tiens la nature : de très
nombreux
organismes, de la bactérie à la plante en passant par les champignons et
certains animaux,
en expriment une 011 plusieurs. Les mammifères, cependant, n'en expriment
aucune ; les
ruminants ou les animaux polygastriques tels que vaches et chevaux possèdent
des
microorganismes endogènes propres à leur tractus gastrointestinal qui peuvent
dégrader le
phytate, ce qui n'est pas le cas des animaux monogastriques tels que porcs et
volailles
principalement, pour qui le phytate provenant des plantes ingérées ne
représente donc pas
une source utilisable de phosphore. Il est donc nécessaire d'ajouter des
phosphates libres à
leur alimentation. Cependant, une partie de ces phosphates libres ajoutés, et
l'essentiel du
phytate ingéré par les animaux sont rejetés dans l'environnement. Le phytate
est alors
CA 02766704 2011-12-23
WO 2011/000933 PCT/EP2010/059413
dégradé par les bactéries du sol et se retrouve, au final, dans les nappes
phréatiques et les
rivières sous forme de phosphate libre. Dans les régions à forte concentration
d'élevage,
ces rejets agro-industriels de phosphates, sous forme libre ou sous forme de
phytate
représentent une pollution importante, qui se traduit notamment par la
prolifération
d'algues vertes au niveau des rivières et des fleuves. Outre l'aspect
esthétique, ces algues
ont un fort impact écologique car elles entrent en compétition avec les
espèces végétales
locales notamment au niveau de la consommation de l'oxygène dissous.
Une manière pour permettre que le phosphate stocké sous forme de phytate soit
utilisable
comme source de phosphate par les mammifères est l'introduction de phytase
exogène
dans les aliments. L'ajout de cette enzyme représente ainsi une alternative à
l'utilisation de
phosphate inorganique comme complément alimentaire. Par ailleurs, en rendant
le
phosphate du phytate accessible, elles permettent également une meilleure
accessibilité aux
ions métalliques chélates sur le phosphate du phytate, ainsi qu'aux protéines
liées au
phytate, les rendant nutritionnellement disponibles. Des phytases sont
aujourd'hui utilisées
de façon relativement systématique dans l'alimentation animale. Elles
permettent de
remplacer partiellement les phosphates et rendent également plus accessibles
les protéines,
les acides aminés, et le calcium.
H existe cependant plusieurs limitations à l'utilisation des phytases dans
l'alimentation
animale :
- L'efficacité insuffisante des phytases à libérer les phosphates constitue
une
première limitation. Des phosphates sont toujours ajoutés à l'alimentation des
animaux même en présence de phytases exogènes. Pourtant, si tout le phytate
était
converti en phosphates libres, aucune supplémentation ne serait plus
nécessaire ; il
existe donc un réel besoin à proposer des phytases plus actives.
- La plupart des enzymes actuellement utilisées ne peuvent pas être ajoutées
directement aux aliments, parce qu'elle ne résistent pas au procédé de
granulation,
qui implique le chauffage à 95 C des aliments pendant 90 secondes. Les enzymes
sont donc sprayées sur les aliments après l'étape de granulation, ce qui
représente
un coût supplémentaire et constitue une seconde limitation ; il existe donc
une forte
demande à posséder des phytases plus thermostables.
L'objectif de la présente invention est de dépasser les limites actuelles en
générant une
phytase suffisamment active pour impacter sensiblement le besoin de
supplémentation en
CA 02766704 2011-12-23
WO 2011/000933 PCT/EP201(1/(159413
3
phosphates, et suffisamment thermostable pour pouvoir être ajoutée directement
à
l'alimentation des animaux, sans avoir à utiliser de technique de sprayage.
Nombreuses sont les phytases qui ont fait l'objet de publications et de
demandes de
brevets, voire qui ont déjà été utilisées dans des applications agro-
industrielles. Aucune n'a
cependant permis de s'affranchir du besoin de supplémentation en phosphates
dans
l'alimentation d'animaux d'élevage et n'est suffisamment thermostable pour
ètre ajoutée
directement à l'alimentation des animaux, sans avoir à utiliser une technique
de sprayage.
La plupart des publications se rapportent aux phytases d'Aspergillus niger et
d' Escherichia
cou. D'autres phytases d'origine microbienne ou provenant de plantes ont
également été
étudiées avec comme problématique d'obtenir des phytases thermostables
possédant une
activité spécifique supérieure à celles d'Aspergillus figer (250 U.mg -1). Les
phytases
décrites dans la littérature et les demandes de brevet proviennent de
champignons
(basidiomycètes et ascomycètes), de levures et de bactéries.
Les phytases isolées de champignons sont nombreuses et proviennent
rnajoritairement de
la famille Aspergillus mais également d'Absidia, Acrophialophora, Agrocybe,
Calcarisporiella, Cheatotnium, Corynascus, Mucor, Mycelia, Myriococcum,
Penicillium,
Peniophora, Rhizottzucor, Rhizopus ou encore Trametes, Sporotrichum,
Neurospora,
Trichodertna, Cladosporium, Mycellophthora, Taleromyces, Thielavia, Humicola,
Mafflus, Thermoascus.
De nombreuses inlbrmations sont disponibles sur les activités spécifiques et
Kiri de ces
enzymes telles que dans Wyss et al 1999 (Appl Environ Microbiol 65 (2) 367-
373).
Globalement, les phytases d'Aspergillus se caractérisent par des Km élevés
variant de 5 à
20 iM. une température optimale de 57 C et un pH optimal de 5,5 excepté pour
Aspergillus fumigatus (pH opt. = 6) mais des activités spécifiques très
faibles allant de 100
à 250 U.mg-1.
Certaines phytases provenant d'autres champignons ont montré des propriétés
intéressantes
notamment des activités spécifiques élevées telles que la phytase de Trametes
pubescens
ou la phytase de Peniophora lycii (respectivement 1200 U. mg-1 et 1080 Llang-1
dans WO
98/28408). Cladosporium sp. montre une phytase intéressante possédant une
activité
spécifique de 900 U.mg-1 et un Km de 15,2 1.iM mais une température optimale
basse à
C. Egalement, la 6-phytase de C'eriporia .sp. présente une activité spécifique
de 11040
CA 02766704 2011-12-23
WO 2011/000933 PCT/EP201(1/(159413
4
U.mg-1. Plusieurs données complémentaires ont été publiées par Lassen et al.
2001 (Appl
Environ Microbiol 67 (10) 4701-4707 comparant la thermostabilité de phytases
de
basidiomycètes notamment Peniophora lycii, Ceriporia sp., Trametes pubeseens
et
Aspergillus nager; il ressort que ces enzymes présentent des Tm (température à
laquelle
l'enzyme est active à 50%) assez proches allant de 55 C (Trametes pubescens) à
60 C
(Peniophora lyeii) mais des activités résiduelles (pourcentages d'activité
résultant d'une
préincubation de 60 minutes à 80 C dans du sodium acétate [0,1M}, pH 5,5) très
variables
allant de 15% (Trametes puheseens) à 62% (Peniophora Lycii).
De nombreuses sociétés ont déposé des demandes de brevet sur ces enzymes
notamment
Danisco/Genencor (W02001012792, Penicillium subtilis ; W02003038035,
Trichoderma
reesei ; W02003038111, Peniciiium. Mumicola, Emericella.
Fusarium).
ABEnzynies/ROAL (EP0659215, phytases d'Aspergillus produites par Trichoderma
reesei), DSM/Roche (ÉP684313, Apergillus terreus, Aspergillus lirmigatus,
Aspergillus
nidulans, Talaromaces therrnophilus), BASF (W02003102174, Aspergillus),
Adisseo
(W02003054199, Penicillium), Choongang Biotech Ltd. (W02005056835, Penicillium
oxalicum).
Parmi les phytases bactériennes, plusieurs ont été décrites provenant de
Bacillus subtilis
(Paver and Jagannathan, 1982, Journal of Bacteriology 151:1102-1108),
Pseudomonas
(Cosgrove, 1970, Australian Journal of Biological Sciences 23:1207-1220), et
Klehsiella.
Plusieurs phytases provenant d'E. cou l ont été rapportées dans la
littérature. Greiner et al,
dans Arch. Biochem. Biophys., 303, 107-113, 1993, ont purifie et caractérisé
une nouvelle
phytase d'E. cou; d'autres ont été rapportées par Uni et al., 2000, Nat.
Struct. Biol. 7:
108-113, Oshima et al.. 1996, DNA Research. 3:137-155, Touati and Danchin,
1987,
Biochimie, 69:215-221, Rodriguez et al., 2000, Arch. Biochern. Biophys,
382:105-112,
Kretz, US patent 5,876,997 ('rom E. cou i 13, and appA by Dassa et al., 1990,
J. Bacteriol.
172:5497-5500.
Des mutants de phytase d'E. col! ont été obtenus par génie génétique résultant
en des
thermostabilités et des activités spécifiques améliorées (Rodriguez et al.,
2000, Arch.
13iochem. Biophys., 382:105-112, Lanahan et al.. 2003. US patent application
20030157646). Cependant. aucune de ces mutations n'a permis une production
suffisante
de ces enzymes d'origine procaryote dans des organismes de production
eucaryotes.
CA 02766704 2011-12-23
WO 2011/000933 PCT/EP201(1/(159413
L'objectif de la présente invention est de fournir une enzyme recombinante qui
soit
adaptable aux procédés industriels permettant son utilisation comme additif
alimentaire,
principalement dans l'alimentation animale.
5 Dans la demande W02002048332, Diversa a identifié, à partir d'un BLAST de
génomes
bactériens disponibles à la date de l'invention, en utilisant le gène appA
d'E. cou, une
nouvelle protéine de Yersinia pestis présentant une activité phytase. Cette
protéine présente
une particularité remarquable en ce qu'elle possède une activité spécifique de
4400 U/mg.
Aucune autre caractéristique biochimique concernant cette protéine n'est
précisé dans cette
demande. Cette demande semblait montrer l'activité élevée potentielle de
phytase
provenant des bactéries de la famille Yersinia.
Le 20 octobre 2005, la séquence protéique ayant pour référence ZP_00832361, a
été
soumise à la base de données NCBI. Cette séquence est celle d'une protéine
hypothétique
de Yersinia intermedia ATCC 29909 dont la séquence nucléotidique
correspondante est
présentée sous la référence NZ AALF01000052 région : 1889...3214. Cette
séquence
protéique a été obtenue par la traduction de la séquence nucléotidique
correspondante
provenant du séquençage complet du génome de la souche Yersinia intermedia
ATCC
29909. Bien que le domaine PRKI 0172 semble prédire une activité phytase,
aucun élément
expérimental n'accompagne cette prédiction.
Celle-ci semble avoir été cependant confirmée par l'isolement d'une phytase
très proche,
d'une nouvelle souche de Yersinia intermedia provenant d'un prélèvement de sol
d'un
glacier. nommée H-27 et présentée dans la demande W02007128160. La phytase
issue de
la souche H-27 est référencée dans la base de données NCBI sous le numéro
d'entrée
AB195370.1 pour la séquence nucléotidique et DQ986462 pour la séquence
protéique; elle
présente 98 % d'identité en acides aminés et 97% d'identité avec la séquence
nucléotidique
codante de la phytase hypothétique de Yersinia intermedia ATCC 29909.
La phytase de la demande W02007128160 présente une activité spécifique élevée
supérieure à 3000 U/mg, du même ordre que l'activité spécifique enregistrée
pour Yersinia
pestis dans W02002048332. Dans cette demande W02007128160, les
caractéristiques
biochimiques intrinsèques de la protéine sont revendiquées à savoir un poids
moléculaire
de 45,5 kDa, un pH optimal de 4,0-5,0, une température optimale de 50-60 C, un
pl
théorique de 7.7. une activité spécifique supérieure à 3000 U/mg et une
résistance élevée à
la pepsine et à la trypsine.
CA 02766704 2011-12-23
WO 2011/000933 PCT/EP201(1/(159413
6
La présente invention permet de dépasser les limites actuelles en proposant un
variant
amélioré par évolution moléculaire de la phytase de Yersinia intermedia. Par
variant
amélioré on entend un variant présentant une thermostabilité et / ou une
activité
supérieures à la phytase d'origine de Yersinia intermedia. lui permettant
d'être utilisé dans
des procédés industriels et en particulier comme additif alimentaire.
Comme il sera décrit dans la suite du texte et à l'aide des figures associées,
la présente
demande décrit un variant amélioré d'une phytase dont la séquence est SEQ ID N
1 ou un
dérivé fonctionnel de celui-ci, caractérisé en ce qu'il comprend au moins une
substitution
sur un des acides aminés du groupe consistant en P3, V4, A5, P8, T9, (31 0,
V16. V17, L19,
S20, R21, 1122, 023, V24, R25, S26, P27,128, K29, Q30, T31, Q32, L33, M34,
D36, P39,
K41, W45, A49, 050, Y51, L52, T53, G56, A57, V60, Y67, 075, A78. C8I, D92,
V93,
D94, Q95, R96, T97, R98, L99, T100, 0101, A103, VI 16, V125, D126, F129,
11130,
P131. V132, D133, DI40, T142, Q143 + 11145, A147, LI52, P155, L156, 158, 158
+
S160, F167, A177, C182, G189, D193, N196, F197, K201, K206, P207, T209, K210,
V211, S212, L213, L217, A218, L219, S220, S221, T222, L223, 0224, E225, 1226,
F227,
L228, L229, Q230, N231, Q233, A234, P236, R.242,1250, S251, L252, L253, L255,
11256,
N257, Q259, F260, 0261, M263, A264, Y268, 1(273, G274, P276, L277, Q292, 0293,
P297, P300, Q301, 0308, 0309, H310, 1)311, T312, N313, 1314, A315, N316, G322,
A323, Q326, P331, D332, N333, T334, P335, P336, 0337, 0338, 0339, V341, 343,
13349, Q352, R353, Y354, 1355, A370, E371, 1(376, P379, A380, 0381, 1)388,
E391,
S393, G394. et P4I4, les positions étant indiquées dans SEQ ID N 1. De
préférence, le
variant amélioré de la phytase dont la séquence est SEQ ID N 1 ou un dérivé
fonctionnel
de celui-ci comprend au moins une substitution sélectionnée parmi le groupe
consistant en
P3L, P3V, V4G, A5P, P8N, P8V, T91, T9Q, T9S, T9Y, GIOA, GIOP, V16M, V17W,
L I9G, S20C, R21F, H22A, H22S, H22Y, 023S, V24C, R25C, S26C, P27F, T28N, T28S,
T28V, K29N, Q30C, Q30D, Q30R, Q32R, L33R, M34C, D36N, P39N, K41G, W45C,
G500, G50E, Y51G, Y51N, Y51Q, Y51W. L52C, L52G, T53C, G56C, A57C, V60I,
Y67F, Y67W, 075R, A78P, C8IN, D92R, V93G, D94G, D94S, Q95N, Q95V, R96A,
T97N, R98N, R98T, L99C, GIOIC, A103C, V1 16C, V125N, D126Q, F129W, 11130N,
1-1130Q, 11130R, 11130W. HI30Y, P131S, V132W, 01330, DI33P, D133R, 0133V,
0133W, D140E, 0140F, 0140N, T142N, Q143N + 11145T. A147C, 1,152N, LI52P,
P155N, PI55T, LI56N, E158N, E158N + S160T, FI67N. A177N, A177S, A177T,
C 1 82N, G189N, D193C, N196C, F197V, K.2.01N, K206A, P207N, P207S, P207T,
T209C,
CA 02766704 2011-12-23
WO 2011/000933 PCT/EP201(059413
7
K.210C, IC210E, K21ON, K210S, K210T, K210V, K210Y, V2I1C, V2110, S212N,
L217N, A218N, L219V, S220N, L223S, E225D, F227S, L229H, Q230N, Q230T, N231K,
Q233S, Q233T, A234K, P236N, R242N, 1250S. 1250T, S25IN, L252M, L255T, H256A,
H256E, H256P, N257I, Q259K, Q259S, Q259T, Q259Y, D261F, M263L, A264N. A264P.
Y268C, Y268E, Y268N, K273N, G274C, G274S, P276L, Q292P, G293N, P297N, P297S,
P300N, Q301L, G308S, H310N. H310R, D3I 1A, D311E, D311G, T312D, T312N, T312P,
T312V, N313F, N313R, 1314E, 1314M, N316C, 0322C, A323E, Q326S, Q326T, P331R,
D332A, D332L, D332N, D332Q, N333V, P335C, P3356, P335R, P335S, P335T, 0339C,
V341A, V341E. E343A, E3430, D349N, D349S, D349T, Q352S, Q352T, R353C,
Y354N, I355W, A370D, A370T, E37IS, E371T, K376S, P379L, P379S, P379T, A380S,
A380T. 0381S, D388C, E391N, S393G, G394S, 0394T, et P414Q, les positions étant
indiquées dans SEQ ID N I. De préférence, le variant amélioré de la phytase
dont la
séquence est SEQ ID NI ou un dérivé fonctionnel de celui-ci comprend au moins
une
combinaison de substitutions sélectionnées parmi les substitutions du groupe
précédent.
SEQ ID N 1 est la séquence figurant dans la base de données NCBI déposée le
20 octobre
2005 sous le numéro d'entrée ZP 00832361, ne comprenant cependant pas la
séquence
signal de 23 acides aminés à l'extrémité 5' de la protéine. SEQ ID N 1
correspond ainsi
aux résidus 24-441 figurant dans le ntunéro d'entrée précédemment cité. SEQ ID
N 1
contient également la séquence nucléique codant pour la séquence protéique
précédente,
référencée sous le numéro NZ__AALF01000052 dans la base NCB1. Un acide
nucléique
codant pour un variant selon la présente invention peut facilement être
préparé sur la base
de cette séquence par les techniques bien connues de l'homme du métier, par
exemple par
mutagenèse dirigée du codon à modifier, pour obtenir la substitution d'acide
aminé désirée.
Ainsi, la séquence du variant amélioré de la phytase selon la présente
invention correspond
à SEQ ID N' I incluant la ou les substitutions sélectionnées.
SEQ ID N 2 reprend uniquement la séquence protéique de SEQ ID N I.
Dans un mode de réalisation particulier, le variant amélioré de la présente
invention
comprend une unique substitution.
Dans un mode de réalisation préféré, le variant amélioré de la présente
invention ou un
dérivé fonctionnel de celui-ci comprend au moins une substitution sur un des
acides
aminés du groupe consistant en K29, Q30, Y51, L52, 075, C81, V93, Q95, R98,
L99.
CA 02766704 2011-12-23
WO 2011/000933 PCT/EP2010/o59413
F129, 1-1130, D140, T142, P155, F167, A177, 0189, K201, K210, L219, 1250,
S251, L252,
L255, M263, Y268, 0274, Q292, G293, P297, 0308, N316, Q326, D349, et E391, les
positions étant indiquées dans SEQ ID N 1. Dans un autre mode de réalisation
préféré, le
variant amélioré de la présente invention ou un dérivé fonctionnel de celui-ci
comprend
des substitutions sur un des acides aminés du groupe consistant en K29, Q30,
Y51, L52,
075, C81, V93, Q95, R98, L99, F129, H130, D140, T142, P155, F167, A177, 0189,
K201, K210, L219, 1250, S251, L252, L255, M263, Y268, 0274, Q292, 0293, P297.
0308, N316, Q326, D349, et E391, les positions étant indiquées dans SEQ ID
N*1. De
préférence, les substitutions sur les acides aminés K29, Q30, Y51, L52, 075,
C81, V93,
Q95, R98, L99, F129, 11130, D140, T142, P155, F167, A177, 0189, K201, K210,
L219,
1250, S251, L252, L255, M263, Y268, 0274, Q292, G293, P297, 0308, N316, Q326,
D349, et E391, sont sélectionnées parmi le groupe consistant en K29N, Q30D,
Y510,
Y51N, Y51Q, Y51W, L520, G75R, C81N, V93G, Q95N, R98T, L99C, F129W, HI30Y,
D140F, DI4ON, T142N, P155N, P155T, F167N, A177N, A177S, A177T, G189N, K201N,
K210N, K210S, L219V, I250S, 1250T, S251N, L252M, L255T, M263L, Y268N, G274C,
Q292P, 029314, P29714, G308S, N316C, Q326S, Q326T, D349S, D349T, et E391N, les
positions étant indiquées dans SEQ ID N 1.
Dans un mode de réalisation encore plus préféré, le variant amélioré de la
présente
invention ou un dérivé fonctionnel de celui-ci comprend des substitutions sur
un des acides
aminés du groupe consistant en K29, Q30, Y51, L52, 075, V93, R98, L99, F129,
11130,
D140, T142, P155, F167, A177, K201, K210, L219, S251, L252, L255, M263, Y268,
0274, Q292, 0293, 0308, N316, Q326 et E391, les positions étant indiquées dans
SEQ ID
N'l. De préférence, les substitutions sur les acides aminés précédents sont
sélectionnées
parmi le groupe consistant en K29N, Q30D, Y510, Y5 1Q, Y5 IW, L52G, G75R,
V93G,
R98T, L99C, F129W, H130Y, D140F, T142N, P155T, F167N, A177N, A177S, A177T,
IC201N, K210S, L219V, S251N, L252M, L255T, M263L, Y268N, G274C, Q292P,
G293N, 0308S, N316C, Q326S, Q326T, et E391N, les positions étant indiquées
dans SEQ
ID N 1.
Dans un autre mode de réalisation particulier, le variant amélioré de la
présente invention
ou un dérivé fonctionnel de celui-ci comprend une combinaison de substitutions
sélectionnée parmi le groupe consistant en G274C + N316C, T142N + A177T +
Q326T,
K210S + Y268E + Q292P, DI4OF + Y268E + Q292P, F167N + Y268E + Q292P, T142N
CA 02766704 2011-12-23
WO 2011/000933 PCT/EP201(1/(159413
9
+ A177T + 1C210S + Q326T, T142N + A177T + K2.10S + Y268E + Q292P + Q326T,
T142N + A177T + K210S + Y268E + Q292P + Q326T + G274C + N316C, L52C + L99C
+ T142N + A177T + K210S + Y268E + Q292P + Q326T, les positions étant indiquées
dans SEQ ID N 1. Dans un mode préféré de ce mode de réalisation particulier,
le variant
amélioré de la présente invention ou un dérivé fonctionnel de celui-ci
comprend une
combinaison de substitutions consistant en T142N + A177T + K210S + Y268E +
Q292P +
Q326T, les positions étant indiquées dans SEQ ID le 1. Dans un autre mode
préféré de ce
mode de réalisation particulier le variant amélioré de la présente invention
ou un dérivé
fonctionnel de celui-ci comprend une combinaison de substitutions consistant
en T142N +
A177T + K210S + Y268E + G274C + Q292P + N316C + Q326T, les positions étant
indiquées dans SEQ ID /4 I. Dans un autre mode préféré de ce mode de
réalisation
particulier le variant amélioré de la présente invention ou un dérivé
fonctionnel de celui-ci
comprend une combinaison de substitutions consistant en T142N +- A177T + K210S
+
Q326T, les positions étant indiquées dans SEQ ID N 1. Dans un autre mode
préféré de ce
mode de réalisation particulier le variant amélioré de la présente invention
ou un dérivé
fonctionnel de celui-ci comprend une combinaison de substitutions consistant
en G274C +
N316C, les positions étant indiquées dans SEQ ID NI' 1. Dans un autre mode
préféré de ce
mode de réalisation particulier le variant amélioré de la présente invention
ou un dérivé
fonctionnel de celui-ci comprend une combinaison de substitutions consistant
en T142N +
A177T + Q326T, les positions étant indiquées dans SEQ ID N 1. Dans un autre
mode
préféré de ce mode de réalisation particulier le variant amélioré de la
présente invention ou
un dérivé fonctionnel de celui-ci comprend une combinaison de substitutions
consistant en
K210S + Y268E + Q292P, les positions étant indiquées dans SEQ ID N I. Dans un
autre
mode préféré de ce mode de réalisation particulier, le variant amélioré de la
présente
invention ou un dérivé fonctionnel de celui-ci comprend une combinaison de
substitutions
consistant en DI4OF + Y268E + Q292P. Dans un autre mode préféré de ce mode de
réalisation particulier, le variant amélioré de la présente invention ou un
dérivé fonctionnel
de celui-ci comprend une combinaison de substitutions consistant en F167N +
Y268E +
Q292P.
La présente invention concerne un variant amélioré d'une phytase dont la
séquence est
SEQ ID N 1 ou un dérivé fonctionnel de celle-ci comprenant la ou les
substitutions
sélectionnées.
CA 02766704 2011-12-23
WO 2011/000933 PCT/EP201(1/(159 4
13
La présente invention concerne également un acide nucléique codant pour un
variant
amélioré de la phytase selon la présente invention ou un dérivé fonctionnel de
celui-ci, une
cassette d'expression comprenant un acide nucléique selon la présente
invention, et un
vecteur comprenant un acide nucléique ou une cassette d'expression selon la
présente
5 invention. Le vecteur peut être sélectionné de préférence parmi un
plasmide, un phage, un
phagemide et un vecteur viral.
La présente invention concerne également une composition comprenant au moins
un
variant amélioré de la phytase dont la séquence est SEQ Ill N 1 ou un dérivé
fonctionnel
10 de celui-ci avec la ou tes substitutions sélectionnées selon la présente
invention. Elle
concerne également tout mélange solide, liquide ou gazeux comprenant un
certain
pourcentage d'au moins un variant amélioré de la phytase selon la présente
invention. Elle
concerne également les mélanges contenant préférentiellement un, deux, trois,
quatre, cinq
ou dix variants améliorés de la phytase selon la présente invention ou des
dérivés
fonctionnels de ceux-ci. La présente invention concerne également des
préparations ou
compositions de phytase contenant un certain pourcentage d'au moins un variant
amélioré
de la phytase selon la présente invention ou un dérivé fonctionnel de celui-ci
et une ou
plusieurs autres enzymes ayant des propriétés avantageuses.
La présente invention concerne l'utilisation d'un variant amélioré de la
phytase selon la
présente invention ou un dérivé fonctionnel de celui-ci, pour la préparation
d'additif
alimentaire. L'utilisation d'un variant amélioré de la phytase selon la
présente invention ou
un dérivé fonctionnel de celui-ci concerne les procédés industriels qui
permettent la
libération des minéraux et en particulier du phosphate des plantes, soit in
vitro dans le cas
de traitement des aliments avant leur ingestion par le variant amélioré de la
phytase selon
la présente invention, soit in vivo par administration dudit variant
directement aux animaux
avant ou avec leur alimentation.
La présente invention concerne l'utilisation d'un acide nucléique, d'une
cassette
d'expression ou d'un vecteur codant et / ou contenant au moins un variant
amélioré de la
phytase dont la séquence est SEQ ID N 1 ou un dérivé fonctionnel de celui-ci
avec la ou
les substitutions sélectionnées selon la présente invention, pour transformer
ou transfecter
une cellule hôte. Elle concerne en outre une cellule hôte comprenant un acide
nucléique,
une cassette d'expression ou un vecteur codant un variant amélioré de la
phytase selon la
CA 02766704 2011-12-23
WO 2011/000933 PCT/EP201(1/(159413
11
présente invention ou un dérivé fonctionnel de celui-ci. La présente invention
concerne
également l'utilisation d'un tel acide nucléique, d'une telle cassette
d'expression, d'un tel
vecteur ou d'une telle cellule hôte pour produire un variant amélioré de la
phytase selon la
présente invention ou un dérivé fonctionnel de celui-ci. Elle concerne
également une
méthode de production d'un variant amélioré de la phytase selon la présente
invention
comprenant la transformation ou transfection d'une cellule hôte par un acide
nucléique,
une cassette d'expression ou un vecteur selon la présente invention, la mise
en culture de la
cellule hôte transformée ou transfectée et la récolte du variant amélioré de
la phytase ou un
dérivé fonctionnel de celui-ci, produit par la cellule hôte. La cellule hôte
peut-être
procaryote ou eucaryote. Ainsi, la cellule hôte peut être un microorganisme.
de préférence
une bactérie, une levure ou un champignon. La cellule hôte peut également être
une cellule
de mammifère telle qu'une cellule COS7 ou CHO.
Par dérivé fonctionnel est entendu toute enzyme dérivée du variant de la
phytase de la
présente invention comprenant des modifications structurales tout en
préservant une
activité phytase. Ces modifications peuvent être, par exemple, un prolongement
de
l'enzyme par addition de nouveaux domaines, des substitutions partielles ou
entières de
domaines telles que des remplacements de stretehes d'acides aminés par des
acides
aminés d'autres enzymes pouvant conférer d'autres fonctions/propriétés. Dans
dérivé
fonctionnel est également compris une forme dimérisée du variant de l'enzyme
de la
présente invention, homo- ou hétérodirnérique, ou encore polymérique,
présentant des
propriétés améliorées, tel k que la thermostabilité par exemple, de par la
multiplication des
domaines. Dans dérivé fonctionnel est également compris une forme
chimérique du
variant de la phytase de la présente invention, fusionné avec une autre
protéine/enzyme
d'intérêt ou avec un/des domaines de cet enzyme d'intérêt. Par dérivé
fonctionnel est
également entendu un fragment fonctionnel du variant de la phytase de la
présente
invention préservant l'activité de la phytase. Cette activité peut être
mesurée selon l'un des
protocoles décrits dans les exemples 4 et 5. Le fragment peut comprendre 250,
275, 300,
325. 350, 375, 380, 385, 390, 395, 400, 405, 410 ou 415 acides aminés
consécutifs de la
phytase selon la présente invention. Ce fragment fonctionnel peut également
être dimérisé
ou polymérisé et/ou fusionné avec une autre protéine/enzyme d'intérêt ou avec
un/des
domaines de celle-ci.
CA 02766704 2011-12-23
WO 2011/000933 PCT/EP201(1/(159413
12
Par variant ou mutant est entendu une séquence nucléotidique présentant
des
mutations par rapport à une séquence nucléotidique de référence. Ces mutations
peuvent
être silencieuses dues à la dégénérescence du code génétique; dans ce cas la
protéine
codée par le variant est identique à la protéine codée par la séquence
nucléotidique de
référence. Ces mutations peuvent également engendrer des substitutions
d'acides aminés
dans la protéine codée par le variant par rapport à la protéine codée par la
séquence
nucléotidique de référence. Dans variant sont incluses les séquences
contenant des
mutations obtenues par mutagénèse dirigée. L'expression variant est
attribuée aux
séquences nucléotidiques ainsi qu'aux séquences protéiques codées par lesdites
séquences
nucléotidiques, présentant lesdites mutations.
Le variant amélioré de la phytase selon la présente invention ou un dérivé
fonctionnel de
celui-ci peut comprendre d'autres substitutions sur un des acides aminés du
groupe
consistant en P3, V4, A5, P8. T9, 010, V16, V17, 119, S20, R21, 1122, 023,
V24, R25,
S26, P27, T28, K29, Q30, T31, Q32, L33, M34, 036, P39, 1(41, W45, A49, 050,
Y51,
152, T53, G56. A57, V60, Y67õ 075, A78, C81, 092, V93, D94, Q95, R96, T97,
R98,
L99, TI00, 0101, A103. V116, V125, 0126, F129, H130, P131, V132, 0133, 0140,
T142. Q143 + 11145, A147, L152, P155, 1156, E158, E158 + S160, F167, A177,
C182,
0189, 0193, N196, F197, K201, K206, P207, T209, K210, V211, S212, L213, L217,
A218, L219, S220, S221, T222, L223, 0224, E225, 1226, F227, L228, L229, Q230,
N231,
Q233, A234. P236, R242, 1250, S251, L252, L253, L255, 11256, N257, Q259, F260,
0261,
M263, A264, Y268, K273, 0274, P276, L277, Q292, 0293, P297, P300, Q301, 0308,
0309, 11310, 0311, T312, N313, 1314, A315, N316, 0322, A323, Q326, P33I. D332,
N333, T334, P335, P336, 0337, 0338, 0339, V341, E343, 0349, Q352, R353, Y354,
1355, A370, E371, 1(376, P379, A380, 0381, 0388, E391, S393, 0394, et P414 non
décrite dans le groupe P3L, P3V, V4G, A5P, P8N, P8V, T91, T9Q, T9S, T9Y, GlOA,
GlOP, V16M, V 17W, Ll9G, S20C, R21F, 1122A, 1122S, F122Y, G23S, V24C, R25C,
S26C. P27F, T28N, T28S, T28V, K29N, Q30C, Q300, Q30R, Q32R, L33R, M34C,
036N, P39N, K410, W45C, 050D, 050E, Y5IG, Y51N, Y5I Q, Y51W, L52C, L52G,
T53C, 056C, A57C, V60I, Y67F, Y67W, 075R, A78P, C81N, 092R. V930, 0940,
D94S, Q95N, Q95V, R96A, T97N, R98N, R98T, L99C, 0101C, A103C, V1 16C, V125N,
0126Q, 17129W, H130N, H130Q, H130R, 11130W. 11130Y. P131S, V132W, 0133G.
0133P, D133R, 0133V, 0133W, 0140E, 0140F, 01401'!, T142N, Q143N + I-1145T,
A147C, L152N, 1,152P, P155N, P155T, 11561'!, E158N, E158N -F S160T, F167N.
A177N,
CA 02766704 2011-12-23
WO 2011/000933 PCT/EP201(1/(159413
13
A177S, A177T, C182N, G189N, DI93C, N196C, F197V, K20IN. K206A, P207N. P207S,
P207T, T209C, K210C, K210E, K2 ION, K210S, K210T, K210V, K210Y, V2I1C,
V211G, S212N. L217N, A218N, L219V, S220N, L223S, E225D, F227S, L229H, Q230N,
Q230T, N231K, Q233S, Q233T, A234K, P236N, R242N, I250S, 1250T, S251N. L252M,
1.255T, H256A, H256E, H256P, N257I, Q259K, Q259S, Q259T, Q259Y, D261 F, M263L,
A264N, A26413. Y268C, Y268E, Y268N, K273N, G274C, G274S, P276L, Q292P,
G293N, P297N, P297S, P300N, Q301L, G308S, 11310N, H310R, D31I A, D311E, D311G,
T312D, T312N, T312P, T312V, N313F, N313R, 1314E, 1314M, N316C, G322C, A323E,
Q326S, Q326T, P331R, D332A, D332L, D332N, D332Q, N333V, P335C, P3350, P335R,
P335S, P335T, 0339C, V341A, V341E, E343A, E3430, D349N, .D349S, D349T. Q352S,
Q352T, .R353C, Y354N, 1355W, A370D, A370T, E371S, E37IT, K376S, P379L, P379S,
P379T, A380S, A380T, 0381S, D388C, E391N, S393G, G394S, G394T, et P414Q, les
positions étant indiquées dans SEQ >I 1 ou des combinaisons de substitutions
issues de
ce groupe, comme mentionné précédemment. Par exemple, ces substitutions
peuvent être
des substitutions dites conservatives , c'est-à-dire des substitutions à
l'intérieur d'un
groupe d'acides aminés présentant des caractéristiques similaires ou
équivalentes, tels que
les acides aminés ayant un faible encombrement stérique. les acides aminés
acides,
basiques, polaires, hydrophobes et aromatiques selon le tableau ci-dessous :
Faible encombrement
Ala (A) Gly (0) Sér (S) Thr (T)
stérique
Acides Asp (D) Glu (E)
Basiques Arg (R) His (H) Lys (K)
Polaires Asn (N) Gin (Q)
Hydrophobes Ile (1) Leu (L) Met (M) Val (V)
Aromatiques Phe (F) Tyr (Y) Trp (W)
Ainsi, par exemple. le variant amélioré de la présente invention ou un dérivé
fonctionnel de
celui-ci peut comprendre des substitutions équivalentes à la substitution
P276L décrite
dans le groupe précédent, telles que les substitutions P276I, P276M ou P276V
selon la
classification du tableau précédent. L'interprétation précédente s'entend
également pour
/5 les combinaisons de substitutions. Par ailleurs, le variant amélioré de
la phytase selon la
présente invention ou un dérivé fonctionnel de celui-ci peut comprendre
d'autres mutations
non décrites dans ce groupe, de préférence des substitutions, notamment
certaines connues
dans le domaine. Dans un mode de réalisation particulier, le variant amélioré
de la phytase
CA 02766704 2011-12-23
WO 2011/000933 PCT/EP201(1/(159413
14
ou un dérivé fonctionnel de celui-ci comprend au maximum 40, 35, 30, 25, 20,
15, 14, 13,
12, 11, 10, 9, 8, 7, 6, 5, 4, 3, 2 substitutions ou 1 substitution par rapport
à la phytase
sauvage, notamment par rapport à SEQ ID W1.
Par variant amélioré, on entend un variant présentant des propriétés
améliorées et en
particulier, une thermostabilité et/ou une activité spécifique et/ou une
expression
augmentée par rapport à la phytase parente. Egalement. le variant amélioré de
la présente
invention peut présenter une plus grande résistance à la protéolyse, par les
protéases ou
autre. L'augmentation d'une/des propriétés du variant amélioré de la phytase
selon la
présente invention est d'au moins 5 %, de préférence au moins 10, 20, 30, 40,
50, 60, 70,
80, 90 ou 100 ou d'un facteur 2, 5, 10 ou 100 par rapport aux propriétés de la
phytase
parente, mesurées dans les mêmes conditions expérimentales. Dans un mode de
réalisation
préféré, ces augmentations sont d'au moins 20 %. La thermostabilité de la
phytase peut
être mesurée en suivant les procédures détaillées dans l'exemple 5. L'activité
spécifique de
la phytase peut être mesurée en suivant les procédures détaillées dans
l'exemple 4.
L'expression de la phytase peut être mesurée en suivant les procédures
détaillées dans les
exemples 2 et 3.
La visualisation de structures modèles en 3D utilisant des logiciels tels que
swiss-model
(http://www.expasy.ch/) et spdbv v4.01 (GlaxoStnithKline) permet souvent de
construire
des hypothèses d'explications en rapport avec des modifications structurales
d'enzymes
entrainant des changements d'activité et/ou de propriétés, particulièrement en
ce qui
concerne les liens possibles entre des acides aminés adjacents. De telles
visualisations
permetttent également. dans une approche prédictive, de cibler certains
résidus pour des
expériences de mutagenèse. Par exemple, lorsqu'une amélioration de la
thermostabilité est
recherchée, les résidus ciblés peuvent être ceux qui permettent une
rigidification de la
structure secondaire. Une telle rigidification peut être obtenue de
différentes manières ; par
exemple, les résidus ciblés peuvent être substitués par un résidu proline qui,
classiquement,
génère peu de rotamères et par conséquent rigidifie la structure secondaire à
laquelle il
appartient. Egalement, une rigidification des structures secondaires peut être
obtenue en
générant de nouvelles liaisons hydrogène et de nouveaux ponts salins ; la
visualisation de
structures modèles en 3D permet de cibler les résidus permettant
l'établissement de telles
liaisons avec les résidus structuralement proches. Une autre approche que la
visualisation
de modèles en 3D permet, lorsqu'une amélioration de la thermostabilité et / ou
de l'activité
CA 02766704 2011-12-23
WO 2011/000933 PCT/EP201(1/(159413
est recherchée, est la modification des charges portées par un résidu et les
contraintes
stériques qui en découlent. 11 est connu que dans certains cas le substrat /
produit d'une
enzyme participe à la stabilisation de la conformation 313 de l'enzyme et peut
conférer une
thermostabilité accrue. Il est évident que la visualisation de telles
structures modèles en 3D
5 permet de cibler des résidus pour augmenter d'autres paramètres de
l'enzyme présentant un
intérêt industriel tels que l'activité, l'expression, ou la résistance à la
protéolyse par
exemple. Il est également évident que cette approche par visualisation de
structures
modèles en 3D est un outil prédictif permettant de construire des stratégies
de mutagénèse
sans aucunement garantir une quelconque amélioration de propriétés
enzymatiques.
Par vecteur d'expression, il est entendu que le vecteur d'expression peut être
n'importe
quel type de vecteur (notamment, plasmide, virus, etc...) recombinant,
permettant
d'exprimer la séquence nucléotidique du variant amélioré de la présente
invention. Le
choix de ce vecteur d'expression dépend de sa compatibilité avec l'hôte
d'expression ciblé,
dans lequel il est transformé ou transfeeté. Ce vecteur peut être linéaire ou
circulaire fermé.
Il peut se répliquer de façon autonome, c'est-à-dire être une entité
extrachromosomique
dont la réplication est indépendante du chromosome de l'hôte qui le contient,
un plasmide,
un élément extrachromosornal. un minichromosome ou un chromosome artificiel. A
l'opposé, le vecteur peut, quand il est introduit dans la cellule hôte,
s'intégrer dans le
génome de l'hôte pour se répliquer en même temps que lui. Egalement, plusieurs
vecteurs
peuvent être nécessaires à l'expression du variant amélioré de la présente
invention et
peuvent être utilisés simultanément, ainsi qu'un transposon.
Les vecteurs permettant l'expression du variant amélioré de la présente
invention, peuvent
contenir un ou plusieurs marqueurs qui permettent une sélection facile des
cellules hôtes
transformées ou transfectées. Ces marqueurs de sélection sont typiquement des
gènes dont
le produit confère un avantage à leur hôte et permet, par exemple, une
résistance
bactérienne à un antibiotique. une prototrophie pour les auxotrophes, une
résistance à des
métaux lourds etc.. .Des exemples de marqueurs de sélection bactériens sont
les gènes qui
confèrent une résistance à des antibiotiques tels que l'ampicilline, la
kanamycine, la
tétracycline et le chloramphénicol notamment. Les marqueurs appropriés à la
sélection
dans les levures sont par exemple les gènes ADE2, 1-11S3, LEU2, LYS2, MET3,
TRP1 et
URA3 notamment. Des exemples de marqueurs utilisés dans les champignons
filamenteux
sont amdS (acétamidase), argB (ornithine carbamoyltransférase), bar
(phosphinothricine
acétyltransférase). hph (hygromycine phosphotransférase), niaD (nitrate
réductase), pyrG
CA 02766704 2011-12-23
WO 2011/000933 PCT/EP201(1/(159413
16
(orotidine-5'-phosphate décarboxylase), sC (sulfate adényltransférase) et trpC
(anthranilate
synthase), notamment. Les vecteurs permettant l'expression du variant amélioré
de la
présente invention, peuvent ne pas contenir de marqueurs de sélection.
Le vecteur, dans le cas d'une réplication autonome, doit contenir une origine
de réplication
adaptée à la cellule hôte. Des exemples d'origine de réplication bactérienne
sont,
notamment, celtes des plasmides pBR322, pUC19, pACYC I 77 et pACYC184
permettant
une réplication dans Escherichia col!, et pUB110, pE194, pTA1060 et pAM[beta]
permettant une réplication dans Bacillus. Des exemples d'origine de
réplication dans les
levures sont, sans que cette liste soit exhaustive, les origines de
réplication 2 microns,
ARS I, ARS4, la combinaison de ARS1 et CEN3 et la combinaison de ARS4 et CEN6.
L'origine de réplication peut également contenir une mutation qui lui permet
d'être
sensible à la température. Des exemples d'origine de réplication utiles dans
les
champignons filamenteux sont AMA1 et ANSI d'Aspergillus nidulans (Gems et al.,
1991,
Gene 98 :61-67; Cullen et al., 1987. Nucleic Acids Research 15: 9163-75; WO
00/24883).
Le vecteur, dans le cas d'une intégration dans le génome de la cellule hôte,
doit permettre
son intégration grâce à la séquence codante du variant amélioré de la présente
invention ou
de n'importe quelle autre séquence adéquate dans le vecteur, via une
recombinaison
homologue ou non homologue. 11 peut également contenir des séquences d'acides
nucléiques supplémentaires pour diriger son intégration dans le génome de la
cellule hôte.
Pour maximiser les chances d'intégration dans le génome de l'hôte, les
séquences
d'intégration doivent être d'une longueur suffisante, telle que 100 à 10000
paires de bases,
préférentiellement 400 à 10000, de façon encore plus préférentielle 800 à
10000 paires de
bases. Les séquences d'intégration peuvent être codantes ou non codantes.
La séquence nueléoticlique signal de 23 codons présente à l'extrémité 5'
du gène de la
phytase de Yersinia intermedia (telle que référencée dans NCBI sous les
numéros
NZ_AALF01000052 et ZP_00832361) et clivée dans la forme mature, contribue à la
sécrétion de l'enzyme dans son hôte d'origine. La présence d'une telle
séquence est
classique et bien connue de l'homme du métier. Le changement de cette
séquence, et son
remplacement par une séquence adéquate, est également un paramètre bien connu
de
l'homme du métier lorsqu'une expression du gène considéré est souhaitée dans
un autre
organisme ou dans un autre compartiment cellulaire via un vecteur plasmidique
ou autre
choisi en adéquation avec l'organisme d'expression souhaité. Ainsi, cette
séquence peut
CA 02766704 2011-12-23
WO 2011/000933 PCT/EP201(1/(159413
17
être remplacée par la séquence signal d'autre gènes telles que celle de
Pe1.13, PhoA, OmpA
ou fl-lactamase notamment, pour son expression dans un hôte procaryote. Lors
de
l'expression dans une levure telle que Pichia pastoris les séquences signal
présentes à
l'extrémité 5' du gène de la phytase de Yersinia intermedia peuvent être les
séquences
signal PI-101 et afactor respectivement des gènes d'une phosphatase et de
l'afactor de cet
organisme. Lors de l'expression dans Saccharomyces cerevisiae, la même
séquence signal
afactor peut être utilisée. Lors de l'expression dans Yarrowia lypolytica, la
séquence signal
présente à l'extrémité 5' du gène de la phytase de Yersinia intermedia peut
être la séquence
signal XPR2 du même gêne XPR2 d'une protéase de cet organisme.
Par cellule hôte, il est entendu que la cellule peut être procaryote ou
eucaryote ; il peut
s'agir d'une bactérie gram positive telle que Bacillus alkalophilus, Bacillus
aznyloliquefaciens, Bacillus hrevis, Bacillus circulons, Bacillus clous fi,
Bacillus coagulons,
Bacillus lautus, Racillus tennis. Bacillus licheniformis, Bacillus megaterium,
Bacillus
stearothermophilus, Bacillus subtilis, Bacillus thuringiensis, Streptomyces
lividans ou
Streptomyces murinusque, notamment ou une bactérie gram négative telle
qu'Escherichia
coli ou Pseudomonas sp. par exemple, sans que cette liste ne soit limitative.
La présente
invention concerne également une méthode de production d'une phytase ou d'un
variant de
celle-ci soluble et active dans une bactérie, de préférence Escherichia coli,
comprenant
l'expression d'un acide nucléique codant la phytase ou le variant de celle-ci
dans une
bactérie. de préférence Escherichia colt et, facultativement la récupération
de la phytase
ainsi exprimée.
La cellule hôte peut être une levure du genre Candida, Hansenula,
Kluyveromyces, Pichia,
Saccharomyces, Schizosaccharomyces, ou Yarrowia. notamment. Préférentiellement
la
cellule hôte peut être Saccharomyces carlshergensis, Saccharomyces cerevisiae,
Saccharomyces diastaticus, Saccharomyces douglasii, Saccharomyces kluyveri,
Saccharomyces norbensis, Saccharomyces ()vermis, Kluyveromyces lactis, Pichia
posions ou Yarrowia lipolytica.
La cellule hôte peut être un champignon filamenteux du genre Acremonium,
Aspergillus,
Aureobasidium, Bjerk,andera, Ceriporiopsis, C'oprinus, con lus. Cryptococcus,
Filibasidium, Fusarium, Humicola, Magnaporthe, Mucor. Myceliophz hora,
Neocallimastix, Neurospora, Paecilomyces. Penicilliton, Phanerochaete,
Phlehia,
Piromyces, Pleurotus, Schizophyllzim, Talaromyces, Thermoascus, Thielavia,
CA 02766704 2011-12-23
WO 2011/000933 PCT/EP201(1/(159413
18
Tolypoclaclium. Trametes, ou Trichoderma. Préférentiellement la cellule hôte
peut être
Aspergillus awamori, Aspergillus fumigatus, Aspergillus foeticlus, Aspergillus
japonicus,
Aspergillus nidulans, A.spergillu.s figer, Aspergillus oryzae, Caldariomyces
fumago,
Fusarium bactridivides. Fusarium cerealis. Fusarium crookwellense, Fusarium
culmorum,
Fusarium grarninearum, Fusarium heterosporum, Fusarium negundi. Fusarium
oxysporum, .Fusarium reticulatum, Fusarium roseum, Fusarium sambucinum,
Fusarium
sarcochroum, Fusarium sporotrichioicles, Fusariurn sulphureum, Fusarium
torulosum.
Fusarium trichothecioldes, Fusarium venenatum, Wel-lu:radera achasta,
Ceriporiopsis
aneirina, Ceriporiopsis aneirincr, Ceriporiopsis caregiea, Ceriporiopsis
gilvescens,
Ceriporiopsis pannoçinta, C'eriporiopsis rivulosa, Ceriporiopsis subraffa,
Ceriporiopsis
subvermispora, Coprinus cinereus, Coriolus birmanes, Humicola insoleras,
Humicola
lanuginosa, Mucor miehei, Myceliophthora thermophila, Neurospora crassa,
Penicillium
purpurogenum, Phanerochaete chrysosporiurn, Phlebia radiata, Pleurotus
eryngii,
Thielavia terrestris, 7'rametes villosa, Trametes vers icolor, .Trichoderma
harzianum,
Trichoderma koningii, Trichoderma longibrachiaturn, Trichoderma reesei, ou.
Trichoderma viride.
La cellule hôte peut être une cellule de mammifère telle que COS7 ou CHO (US
4,889,803 ; US 5,047,335).
Par acide nucléique on entend de l'ADN (ADNc ou ADNg), de PARN ou un mélange
des
deux. L'acide nucléique peut comprendre des nucléotides modifiés, comprenant
par
exemple une liaison modifiée, une base purique ou pyrimidique modifiée, ou un
sucre
modifié. Il peut être préparé par toutes méthodes connues de l'homme du
métier, dont la
synthèse chimique, la recombinaison, la mutagenèse, etc._
L'acide nucléique codant un variant amélioré de la phytase selon la présente
invention peut
être optimisé en termes de codons le constituant pour maximiser son expression
dans un
hôte particulier différent de son organisme d'origine. Le code génétique
universel étant
dégénéré, il existe plusieurs codons (codon = triplet de nucléotides) qui
codent pour un
acide aminé donné. Ces codons homonymes ne sont pas employés au hasard parce
que les
ARNt (ARN de transfert) correspondants n'existent pas dans toutes les cellules
aux mêmes
concentrations. De sorte que certains codons auront moins de chances de
s'exprimer dans
les tissus où l'ARNt correspondant est rare. Cette donnée, bien connue de
l'homme du
métier, est un paramètre important à considérer lorsqu'on effectue
l'expression dans un
hôte donné différent de l'organisme d'origine d'un transgène particulier. Les
tables de
CA 02766704 2011-12-23
WO 2011/000933 PCT/EP201(1/(159413
19
fréquence d'utilisation des codons dans un organisme particulier sont
publiques et bien
connues de l'homme du métier. Ainsi, les acides nucléiques codant pour les
variants
améliorés de la phytase selon la présente invention peuvent nécessiter une
optimisation
pour favoriser leur expression dans l'hôte de production choisi.
Par pourcentage d'identité)> ou identité entre deux séquences d'acide
nucléique ou
d'acides aminés au sens de la présente invention, on entend désigner un
pourcentage de
nucléotides ou de résidus d'acides aminés identiques entre les deux séquences
à comparer,
obtenu après le meilleur alignement, ce pourcentage étant purement statistique
et les
différences entre les deux séquences étant réparties au hasard et sur toute
leur longueur. Le
meilleur alignement ou alignement optimal est l'alignement pour lequel le
pourcentage
d'identité entre les deux séquences à comparer, comme calculé ci- après, est
le plus élevé.
Les comparaisons de séquences entre deux séquences d'acide nucléique ou
d'acides aminés
sont traditionnellement réalisées en comparant ces séquences après les avoir
alignées de
manière optimale, ladite comparaison étant réalisée par segment ou par fenêtre
de
comparaison pour identifier et comparer les régions locales de similarité de
séquence.
L'alignement optimal des séquences pour la comparaison peut être réalisé,
outre
manuellement, au moyen de l'algorithme d'homologie locale de Smith et Waterman
(1981)
(Ad. App. Math. 2 : 482), au moyen de l'algorithme d'homologie locale de
Neddleman et
Wunsch (1970) J. Mol. Biol. 48 : 443), au moyen de la méthode de recherche de
similarité
de Pearson et Lipman (1988) (Proc. Natl. Acad. Sci. USA 85 : 2444), au moyen
de
logiciels informatiques utilisant ces algorithmes (GAP, BESTF1T, FASTA et
TFASTA
dans le Wisconsin Geneties Software Package, Genetics Computer Group, 575
Science
Dr., Madison, WI). Le pourcentage d'identité entre deux séquences d'acide
nucléique ou
d'acides aminés est déterminé en comparant ces deux séquences alignées de
manière
optimale par fenêtre de comparaison dans laquelle la région de la séquence
d'acide
nucléique ou d'acides aminés à comparer peut comprendre des additions ou des
délétions
par rapport à la séquence de référence pour un alignement optimal entre ces
deux
séquences. Le pourcentage d'identité est calculé en déterminant le nombre de
positions
identiques pour lesquelles le nucléotide ou le résidu d'acide aminé est
identique entre les
deux séquences, en divisant ce nombre de positions identiques par le nombre
total de
positions dans la fenêtre de comparaison et en multipliant le résultat obtenu
par 100 pour
obtenir le pourcentage d'identité entre ces deux séquences. Ainsi, des acides
aminés
conservés ou équivalents à ceux présents dans le variant amélioré de la
présente invention
CA 02766704 2011-12-23
WO 2011/000933 PCT/EP201(1/(159413
peuvent être retrouvés dans des phytases d'autres organismes. Ainsi,
l'amélioration
conférée par la ou les substitutions sélectionnées dans le variant amélioré de
la présente
invention peut se retrouver en effectuant une substitution équivalente dans
une phytase
d'un autre organisme que celui dont est originaire la phytase de la présente
invention. Il est
5 entendu que ces substitutions équivalentes sont comprises dans le
périmètre de la présente
invention.
Le variant amélioré de la phytase selon la présente invention peut être
utilisé dans les
réactions et procédés mentionnés précédemment sous une forme purifiée ou
partiellement
10 purifiée. La purification du variant amélioré de la phytase selon la
présente invention peut
être basique et réalisée notamment par lyse et filtration du contenu des
flasques ou
conteneurs de production et/ou par des étapes de centrifugation, et/ou par
précipitations
successives et sélectives à l'ammonium sulfate et/ou par évaporation. Ces
procédures
basiques permettent d'obtenir des fractions du variant amélioré de la phytase
selon
15 l'invention présentant une forte augmentation d'activité spécifique. La
purification du
variant amélioré selon l'invention peut être complète et nécessiter
différentes étapes bien
connues de l'homme du métier notamment des étapes de chromatographie
(échangeuse
d'ions, d'affinité, hydrophobe, par exclusion de taille), d'électrophorèse
(préparative par
concentration isoélectrique) [Protein Purification, J.C. Janson and Lars
Ryden, V01
20 Publishers, New York. 19891.
La fraction purifiée ou partiellement purifiée du variant amélioré de la
phytase selon la
présente invention peut être utilisée dans les réactions et procédés
mentionnés
précédemment sous une forme immobilisée ou non. Les procédés pour immobiliser
le
variant amélioré de ta présente invention sur des supports organiques ou
inorganiques sont
bien connus de l'homme du métier. Ces derniers peuvent être notamment des
polyacrylamides, des agaroses, des celluloses, des sephadex ou dextrans, des
billes de verre
poreuses, des hydroxydes d'aluminium ou de titanium.
Par composition on entend généralement une composition comprenant au moins
un
variant amélioré de la phytase dont la séquence est SEQ ID N 1 avec la ou les
substitutions
sélectionnées selon la présente invention. Elle concerne également tout
mélange solide,
liquide ou gazeux comprenant un certain pourcentage d'au moins un variant
amélioré de la
phytase selon la présente invention. Elle concerne également les mélanges
contenant un,
deux, trois. quatre, cinq ou dix variants améliorés de la phytase selon la
présente invention.
CA 02766704 2011-12-23
WO 2011/000933 PCT/EP201(1/(159413
21
Généralement, les compositions contenant des phytases sont des compositions
liquides ou
dites sèches.
Les compositions liquides ne nécessitent pas de contenir autre chose que la
phytase
hautement purifiée. Cependant des stabilisateurs tels que glycérol. sorbitol
ou
monopropylène glycol peuvent être ajoutés. Ladite composition peut également
contenir
d'autres additifs tels que sels, sucres, conservateurs, agents tamponnant vis
à vis du pH,
des protéines et du phytate. Des compositions liquides typiques sont des
compositions
aqueuses ou des suspensions à base d'huile. Les compositions liquides peuvent
être
ajoutées à l'alimentation avant ou après une étape optionnelle de granulation.
Les compositions dites sèches peuvent être des compositions sèches par
congélation. par
pulvérisation ou des compositions sèches extrudées ; dans ces cas ladite
composition ne
nécessite pas de contenir autre chose que l'enzyme sous sa forme sèche. Les
compositions
sèches peuvent également être des granulés qui peuvent être mixés ou prêts à
l'être avec un
composant alimentaire, ou former un composant d'un pré-mélange. La taille des
granulés
d'enzyme est, de manière préférée, compatible avec celle des autres composants
du
mélange. Ceci représente un moyen sécurisé et pratique pour incorporer une/des
enzymes
dans des aliments.
Par exemple, une formulation stable d'enzyme peut être préparée par
pulvérisation d'une
mixture liquide de phytase sur un composant tel que le tourteau de soja puis
séchage de
l'ensemble. La diminution du pourcentage d'humidité et les interactions de
liaison de la
phytase avec le composant protègent l'enzyme des facteurs environnementaux
extérieurs
telles que les températures extrêmes utilisées lors de la fabrication du
composant
alimentaire. Egalernent, la présentation sous forme sèche de la préparation de
phytase peut
augmenter sa stabilité en diminuant l'activité de potentielles enzymes
protéolytiques
pouvant être présentes à l'état de traces à l'issue des étapes de fermentation
liquides lors du
procédé de production. La préparation sèche de phytase peut, par exemple, être
utilisée
comme un supplément alimentaire dans l'industrie de production de volailles et
de porcs.
A partir d'une préparation sèche d'enzyme, des granulés sont préparés en
utilisant des
techniques d'agglomération bien connues de l'homme du métier, dans un mixeur à
l'intérieur duquel un matériel de remplissage et l'enzyme sont co-agglomérés
pour former
des granulés. Les granulés sont préparés à partir de matrices sur lesquelles
l'enzyme peut
venir s'absorber ou sur lesquelles une couche d'enzymes peut être appliquée.
Des
CA 02766704 2011-12-23
WO 2011/000933 PCT/EP201(1/(159413
22
matériaux typiques pouvant servir de matrice sont des sels tels que du sulfate
disodique.
D'autres matrices potentielles peuvent être, ou à base de, tale, argile,
silicate de
magnésium, silicate d'aluminium ou fibres de cellulose. Optionnellement, des
agents
liants tels que les dextrines peuvent être inclus dans les granulés.
Des agents entraîneurs peuvent être indus sous une forme quelconque des
composants ci-
après : amidon, manioc, pomme de terre, riz, blé, mas...Des sels peuvent
également être
ajoutés.
Optionnellement, les granulés peuvent être recouverts par des mélanges dédiés
spécifiquement, et en particulier des mélanges hydrophobes, à base d'huile de
palme, de
suif de b uf, et si besoin, d'autres additifs tels que carbonate de calcium et
argile.
Par ailleurs, la préparation de phytase peut contenir d'autres agents tels que
des agents
colorants, des composés aromatiques, des stabilisants, des vitamines, des
minéraux ainsi
que d'autres enzymes ou mélanges d'enzymes ayant des propriétés avantageuses.
Ceci est
particulièrement vrai pour les pré-mélanges.
Par additif alimentaire, on entend un composant pratiquement pur ou une
composition
contenant plusieurs composants destinés à être ajoutés à un aliment. En
particulier, cet
additif est destiné à devenir un composant à part entière dudit aliment et a,
pour objectif
d'affecter, de modifier, d'augmenter une/des propriétés dudit aliment. Ainsi,
une
préparation de phytase utilisée comme additif alimentaire signifie une phytase
qui n'est pas
un composant naturel de l'aliment dans lequel elle est ajoutée, ou qui n'est
pas présente
dans cet aliment à sa concentration naturelle, ou qui est ajoutée séparément
des autres
composants de l'aliment, seule ou en association avec d'autres additifs
alimentaires.
Typiquement, un additif alimentaire contient plusieurs composants tels que
vitamines,
minéraux, agents entraîneurs, excipients, autres enzymes ou mélanges d'enzymes
ayant des
propriétés avantageuses.
Par préparation de phytase comme additif alimentaire prêt à l'emploi ou
phytase comme
additif alimentaire prêt à l'emploi, on entend un additif alimentaire qui
n'est pas produit in
situ dans la nourriture ou l'alimentation animale. Une telle phytase ou
préparation peut être
donnée directement comme aliment aux êtres humains ou aux animaux et de
manière
préférée, directement après mélange avec d'autres constituants dudit aliment.
Par exemple,
un additif alimentaire selon cet aspect de la présente invention est combiné
avec d'autres
CA 02766704 2011-12-23
WO 2011/000933 PCT/EP201(1/(159413
23
composés pour produire un aliment. Il est inclus dans ces autres composés une
ou plusieurs
autres enzymes, de préférence thermostables, des additifs alimentaires
vitaminés, des
additifs alimentaires minéraux, des acides aminés comme additifs alimentaires.
Le résultat
de ce mélange ou cette combinaison de composés peut être mélangé dans une
proportion
appropriée avec d'autres composés tels que des suppléments protéiques ou
céréaliers pour
former l'aliment final. Les procédés de fabrication de ces aliments peuvent
être réalisés en
utilisant tout appareil bien connu par l'homme du métier, telle qu'une machine
à double
granulation, une granuleuse à vapeur. un expandeur ou un extrudeur.
Par préparation de phytase ou composition de phytase de la présente invention,
on entend
des préparations ou compositions qui contiennent une quantité significative
d'au moins un
variant amélioré de la phytase selon la présente invention et une ou plusieurs
autres
enzymes ayant des propriétés avantageuses pour la préparation d'aliments. De
telles
enzymes peuvent appartenir à la liste suivante sans que celle-ci soit
exhaustive : alpha-
galactosidases, beta-galactosidases, en particulier les lactases, d'autres
phytases, beta-
eiticanases, en particulier des endo-beta-1,4-glucanases et des endo-beta-
1,3(4)-glucanases,
cellulases, xylosidases, galactanases, en particulier des arabinogalactan-endo-
1,4-beta-
galactosidases et des arabinogalactan-endo-1,3-beta-galactosidases,
endoglucanases. en
particulier des endo-1,2-beta-glucanase, endo-1,3-alpha-glucanase, et endo-1,3-
beta-
glucanase, des enzymes déradant les pectines, en particulier des pectinases.
pectinestcrases, des pectine lyases, polygalacturonases,
arabinanases,
rhamnogalacturonases, rhamnonalacturonan-acetyl-esterases, rhamnogalacturonan-
alpha-
rhamnosidase, pectate lyases. des alpha-galacturonisidases, mannanases, beta-
mannosidases, mannan-acetyl-esterases, xylan-acetyl-estemses, proteases,
xylanases,
arabinoxylanases et enzymes lipolytiques telles que lipases, phospholipases et
cutinases.
La supplémentation de l'additif alimentaire aux animaux, selon la présente
invention, peut
être faite avant ou de manière simultanée par rapport au repas. De manière
préférée, la
supplémentation se fait en même temps que le repas.
(inc quantité efficace de phytase pouvant être ajoutée dans les aliments est
d'environ 10 à
20000 PPU/kg d'aliment; de manière préférée entre 10 et 15000 PF'U/kg; de
manière
encore préférée entre 10 et 10000 PPU/kg. En particulier de 100 à 5000 PPI
'kg,
particulièrement de 100 à 2000 PPU/kg d'aliment.
CA 02766704 2011-12-23
WO 2011/000933 PCT/EP201(1/(159413
24
Dans le périmètre de la présente invention est également comprise
l'utilisation d'un variant
amélioré de la phytase selon la présente invention dans la fabrication
d'aliments destinés à
la consommation humaine ou à l'alimentation animale. Des grains ou des farines
destinés à
l'alimentation humaine peuvent être traités avec de la phytase pour réduire
leur teneur en
phytate, permettant un accroissement de la valeur nutritionnelle de ces
produits en
augmentant la disponibilité de minéraux essentiels tels que fer, calcium et
zinc par
exemple. Au delà de la valeur nutritionnelle, un tel traitement par la phytase
peut
augmenter l'efficacité de la production de cet aliment. Par exemple,
l'addition de phytase à
des flocons de soja blancs durant le procédé d'extraction de protéines du soja
peut
augmenter le rendement et la qualité des protéines extraites. La phytase est
active durant la
fabrication des aliments mais pas dans le produit final. Ceci est
particulièrement vrai dans
la fabrication et la cuisson des pâtes pour boulangerie. De façon similaire,
dans la
production d'alimentation animale, des grains de soja ou de colza peuvent être
prétraités
par la phytase avant leur fabrication chou conditionnement final. Un tel
prétraitement
permet de dégrader des éléments anti-nutritionnels tels que le phytate et
augmenter la
qualité et la valeur nutritionnelle des aliments en question. Dans ce cas, la
phytase peut ou
ne pas être toujours active dans le tractus digestif des animaux après leur
ingestion des
aliments.
Est également comprise dans le périmètre de l'invention, l'utilisation d'un
variant amélioré
de la phytase selon la présente invention comme agent facilitateur de la
transformation des
aliments. En particulier, la phytase selon la présente invention peut être
utilisée comme un
supplément dans l'alimentation humaine pour faciliter la digestion. Par
exemple, un/des
cachets contenant une quantité adéquate de phytase peuvent être ingérés par
une personne
avant la consommation de nourriture afin de fournir une enzyme active à son
tractus
digestif. Le bénéfice de cette ingestion de phytase est particulièrement
remarquable dans le
cas de consommation d'une nourriture ne pouvant être traitée par la phytase
durant sa
fabrication.
La phytase selon la présente invention peut être avantageusement utilisée avec
des
animaux mono- ou polygastriques. spécialement les jeunes veaux. Les régimes
destinés
aux poissons et crustacés peuvent également être traités par la phytase afin
d'améliorer les
rendements de conversion entre la nourriture fournie et la croissance des
animaux, ainsi
CA 02766704 2011-12-23
WO 2011/000933 PCT/EP2010/059413
que réduire les quantités de phosphate excrété dans les systèmes de production
intensifs.
La nourriture traitée selon la présente invention peut-être fournie à la
volaille (dindes,
canards, oies, perdrix, poules, poulets de chair) aux pores, aux chevaux, aux
bovins, aux
caprins, aux canins, aux félins. Elle s'adresse particulièrement à la volaille
et aux porcs.
5 incluant sans que cela ne soit limitatif, les poules, poulets de chair.
dindes, canards et oies.
La phytase selon la présente invention est utilisée pour produire de nouvelles
combinaisons
d'ingrédients alimentaires ou aliments avec des qualités avantageuses. Par
exemple, elle
peut être utilisée pour produire des aliments ayant une teneur abaissée en
phosphate
10 inorganique. Cette teneur est ajustée en fonction de la quantité et de
l'activité de la phytase
ajoutée, présente dans l'aliment final, ou active dans un des ingrédients
alimentaires
rentrant dans la composition de l'aliment final. D'une manière préférée. un
tel aliment peut
contenir des ingrédients tels que micronutriments, vitamines, acides aminés et
des
quantités efficaces et optimisées de phytase et de phosphate inorganique
telles que la
15 quantité de phytase est comprise entre 50 et 20000 unités de phytase par
kilo d'aliment et
la quantité de phosphate inorganique est inférieure à 0,45%.
Préférentiellement, ces deux
quantités se situent entre 100 et 10000 unités de phytase par kilo d'aliment
et moins que
0.225% de phosphate inorganique d'une manière encore préférée entre 150 et
10000
unités de phytase par kilo d'aliment et moins de 0,15% de phosphate
inorganique : d'une
20 manière encore plus préférée entre 250 et 20000 unités de phytase par
kilo d'aliment et
aucun phosphate inorganique ajouté. Ces nouvelles combinaisons présentent des
intérêts
divers tels que la réduction des rejets de phosphate dans l'environnement et
l'optimisation
des rendements de conversion entre la nourriture fournie et la croissance des
animaux,
particulièrement recherchée dans la production intensive en élevage.
La présente invention sera décrite plus en détail dans les exemples suivants
qui n'ont en
aucun cas un caractère limitatif.: à l'aide des tableaux et figures associés
suivants :
Tableau 1 : Mutants isolés par l'approche THR1TM.
Tableau 2 : Liste des mutants présentant un site supplémentaire de
glycosylation classés en
fonction de leurs pourcentages d'accessibilité aux solvants respectifs.
Tableau 3 : Liste des couples de mutations permettant l'addition de ponts
disul titres
supplémentaires.
Tableau 4 : Coefficients d'extinction d'activité 80-20 pour différents
mutants.
CA 02766704 2011-12-23
WO 2011/000933 PCT/EP201(1/(159413
26
Tableau 4A: Coefficients d'extinction d'activité 80-20 pour les mutants K210S,
Y268E et Q292P.
Tableau 4B: Détails des données permettant le calcul du coefficient
d'extinction
d'activité 80-20 du mutant K210S.
Tableau 4C: Coefficients d'extinction d'activité 80-20 pour les mutants T142N,
Al 77T et Q326T.
Tableau 4D: Coefficient d'extinction d'activité 80-20 pour le mutant
G274C1N316C.
Tableau 5 : Listes des mutants ciblant une amélioration d'activité
Tableau 5A: Liste des positions ciblées par une distance inférieure ou égale à
10
Angstriims autour du site catalytique de l'enzyme.
Tableau 5B Liste des substitutions ciblant une amélioration d'activité
caractérisées
dans une première série d'expériences.
Figure 1: Dégradation du phytate par une phytase
Figure 2: Principe de la technologie THRTm
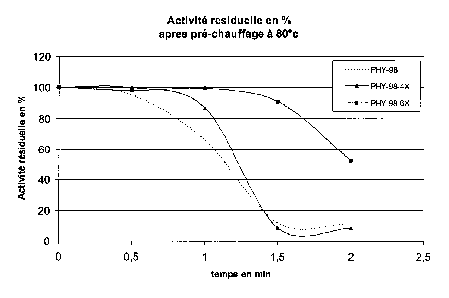
Figure 3 : Mesure des activités résiduelles des mutants PHY-98-4X et PHY-98-6X
produits
par Saccharomyces cerevisiae après un préchauffage de 0 à 2 minutes à 80 C.
Figure 4: Mesure des activités résiduelles des mutants PHY-98-4X et PHY-98-6X
produits
par Saccharomyces cerevisiae après un préchauffage 15 minutes à des
températures de
45 C à 65 C.
Figure 5 : Mesure des activités résiduelles des mutants PHY-98-4X et PHY-98-6X
produits
par Saccharomyces cerevisiae après un préchauffage de 1 minute à des
températures de
60 C à 80 C.
Figure 6A: Mesure des activités résiduelles des mutants PHY-98-6X et PHY-98-6X-
ssl 1
produits par Pichia pastoris après un préchauffage de 0 à 30 minutes à 80 C.
Figure 6B: Mesure des activités résiduelles des mutants PHY-98-6X et PHY-98-6X-
ss 1 1
produits par Pichia pastoris après un préchauffage de 0 à 5 minutes à 80 C.
Figure 7: Mesure des niveaux d'activités relatifs des mutants P1-JY-98-4X et
PHY-98-6X
comparativement à l'enzyme d'origine PHY-98 produits par Saccharomyces
cerevisiae. en
fonction du temps.
Figure 8: Gel SUS-PAGE 12% de surnageants de production pour différents
mutants
exprimés par Pichia pastoris. digérés ou non par l'endoglycosidase Hf.
Exemptes:
CA 02766704 2011-12-23
WO 2011/000933 PCT/EP201(1/(159413
17
Exemple 1: Obtention des variants améliorés de la phytase de Yersinia
intermedia
Contructions alasmidiques :
La réalisation des variants améliorés de la phytase selon la présente
invention a nécessité la
construction de différents vecteurs plasmidiques permettant la réalisation des
expériences
de mutagenèse dirigée nécessaires à l'obtention de banques de variants ou
mutants ainsi
que l'expression de ces mutants dans différents hôtes de criblage ou de
production.
Constructions dans pET25b:
Selon des techniques de biologie moléculaire bien connues de l'homme du
métier, l'ORF
(Open Reading Frame) 72_00832361 correspondant à la séquence NCBI ATec 29909
et à
la séquence nueléotidique correspondante NZ_AALF01000052 région : 1889...3214
a été
clonée dans le vecteur plasmidique pET25b. Avant clonage, l'ORF a été délétée
de sa
séquence signal d'origine (23 premiers acides aminés) remplacée par la
séquence signal de
la phytase di Escherichia colt. Cette séquence signal de la phytase
d'Escherichia cou a été
préalablement clonée dans le vecteur pET25b afin d'optimiser l'expression de
la phytase
de Yersinta intermedia dans E. cou. Le vecteur pET25b permet également
d'exprimer la
protéine d'intérêt sous la forme d'une protéine de fusion avec un tag 6His
facilitant
l'isolement et/ou la purification de la protéine d'intérêt selon des méthodes
bien connues
de l'homme du métier. Ce vecteur contenant la phytase de Yersinia intermedia
fusionnée à
1a séquence signal de la phytase d'E. cou a été transformé dans une souche
d'E. coui
BL21(DE3) délétée du gène AppA codant pour la phytase endogène d'E. coll.
Constructions dans pNCK:
L'ORF de ZP_00832361 a été clonée selon des techniques de biologie moléculaire
bien
connues de l'homme du métier dans le vecteur pNCK. Ce vecteur permet
d'exprimer dans
un microorganisme thermophile Thermus therenophilus une protéine de fusion
entre la
protéine d'intérêt et un gène de résistance thermostable à la kanamycine selon
le procédé
décrit dans la demande de brevet W02006134240 et correspondant à la technique
propriétaire de Biométhodes TI IRTm.
Constructions dans pYES2 :
L'ORF de ZP 00832361 a été clonée selon des techniques de biologie moléculaire
bien
connues de l'homme du métier dans le vecteur pYES2. Ce vecteur, contenant le
peptide
CA 02766704 2011-12-23
WO 2011/000933 PCT/EP201(1/(159413
28
signal de la phytase d'Aspergillus Pilger en position 5' de l'ORF
213_00832361, a permis
l'expression de la phytase de Yersinia intermedia par Saccharomyces
cerevisiae.
Constructions dans pP1C9
L'ORF de ZP 00832361 a été clonée selon des techniques de biologie moléculaire
bien
connues de l'homme du métier dans le vecteur pP1C9. Ce vecteur, contenant le
peptide
signal de rathctor de Pichia pastoris (invitrogen) en position 5' de l'ORF
ZP_00832361,
a permis l'expression de la phytase de Yersinia intermedia par Pichia parlons.
Construction de banques de mutants dans e4NCK, criblue et obtention de
variants
améliorés :
Plusieurs banques de mutants de la phytase de Yersiinia intertnedia ont été
créées par la
technique propriétaire de Blométhodes Massive Mutagenesis décrite dans US
7,202,086
ou dans Saboulard et al (Biotechniques, 2005 Sep 39(3) : 363-8). Brièvement,
des
olinonueléotides mutagènes ont été synthétisés pour réaliser des mutants de la
phytase sur
chacun des acides aminés constituant l'enzyme. Les banques de mutants ont été
construites
à partir du vecteur pNCK contenant le gène de la phytase, selon le protocole
de Massive
Mutagenesis décrit dans US 7,202,086 ou dans Saboulard et al (Biotechniques,
2005 Sep
39(3) : 363-8), puis transformées et amplifiées dans la souche d'Escherichia
col! D1-11013.
Les mutants de la phytase contenus dans ces banques ont ensuite été criblés
par la
technique Ti Hem décrite dans la demande de brevet W02006134240, dont le
principe est
résumé dans la figure 2. La possibilité offerte par le système de sélectionner
en une seule
étape un très grand nombre de molécules mutantes permet de travailler avec des
banques
de très haute diversité. Une telle banque, ciblant tous les résidus de la
protéine. en \ iron 10g
clones, a été construite grâce à Massive Mutagenesis .
Brièvement, les banques de mutants sont transformées dans des cultures de
Thermus
thermophilus rendues compétentes. Les transformants sont mis à pousser en
milieu liquide
à 70 C pour réaliser des précultures qui seront ensuite étalées sur un milieu
solide
contenant des concentrations stringcntes en kanamycine de l'ordre de 25
itG/ral. Après une
incubation de 48 heures à 70 C, seuls les mutants dont la structure protéique
résiste à la
température de sélection et dont le repliement est correct permettent un
repliement
fonctionnel du gène de résistance thermostable à la kanamycine et donc peuvent
pousser en
présence de kanamycine. Par cette approche, différents mutants ont été isolés,
leurs ADN
plasmidiques isolés et séquencés. 1,es différents mutants ont révélé un total
de 138
CA 02766704 2011-12-23
WO 2011/000933 PCT/EP201(1/(159413
19
substitutions sur 89 positions présentées dans le tableau 1. Les différentes
substitutions ont
été introduites individuellement dans la phytase clonée dans le vecteur
plasmidique
pET25b afin d'être exprimés dans Escherichia col! et caractérisés plus en
détail au niveau
de leur activité et de leur thermostabilité. L'exemple 4 détaille les
protocoles de mesure
d'activité utilisés et l'exemple 5 détaille les protocoles de caractérisation
de l'activité
résiduelle des mutants en fonction de la température. Une première série
d'expériences a
permis d'identifier au moins 5 mutants ayant une thermostabilité augmentée par
rapport à
l'enzyme sauvage. Parmi ceux-ci, 3 mutants contiennent respectivement des
substitutions
sur les acides aminés K210, Y268 et Q292, particulièrement les substitutions
K210S,
Y268E et Q292P. Ces mutants présentent un coefficient d'extinction d'activité
entre 80 %
et 20 % d'activité résiduelle respectivement de 1,75, 1,98 et 2,30 tels que
présentés dans le
tableau 4A. Ces indices sont calculés par le rapport entre les différences de
températures
permettant d'une part le maintien de 80% de l'activité résiduelle et d'autre
part de 20% de
cette activité résiduelle pour les différents variants comparativement à
l'enzyme sauvage.
Le détail du calcul de cet indice est montré pour le mutant K210S dans le
tableau 4B.
La visualisation de structures modèles en 3D utilisant des logiciels tels que
swiss-model
(http://www.expasy.chn et spdbv v4.01 (GlaxoSmithKline) permet d'avancer
certaines
explications concernant les gains de thermostabilité obtenus pour les 3
mutants mentionnés
ci-dessus. K210 est un résidu potentiellement accessible au solvant et
localisé dans une
structure secondaire de type feuillet , éventuellement impliqué dans la
liaison et / ou
l'interaction avec le substrat (phytate) ou les produits issus de la réaction.
K210 semble
donc être un résidu important notamment à cause de sa charge positive et de
son volume
important pouvant générer des contraintes stériques et électrostatiques vis à
vis de l'entrée
du substrat ou de la sortie des produits du site actif. La perte de cette
charge positive dans
la substitution K210S et la modification de la contrainte spatiale associée
peuvent donc
perturber globalement la réaction (entrée du substrat sortie des produits +
solvatation de
la cavité du site actif) et modifier les constantes cinétiques de celle-ci, et
plus
particulièrement les Km apparents pour les différents substrats générés au
cours de la
réaction. Il est connu, dans certains cas, que les substrat / produit peuvent
participer à la
stabilisation de la structure 3D de la protéine et conférer une
thermostabilité accrue. Il est
envisageable que cette substitution facilite, par exemple, le positionnement
du substrat en
augmentant le nombre de conformères tolérés dans le site actif et/ou puisse
également
faciliter l'évacuation des phosphates libérés au cours de la réaction en
empêchant que
ceux-ci ne soient trop présents dans la cavité du site actif, présence qui
pourrait clairement
CA 02766704 2011-12-23
WO 2011/000933 PCT/EP201(1/(159413
perturber le complexe enzyme-substrat et déstabiliser l'ensemble de la
structure. Y268 est
un résidu potentiellement accessible au solvant et localisé dans une structure
secondaire de
type "boucle". En modélisant la substitution Y268E à l'aide des logiciels
précédemment
mentionnés, on constate qu'on augmente le nombre de liaisons hydrogènes
potentielles
5 avec les résidus structuralernent proches (avec la fonction CO peptidique
du résidu Q143
par exemple), mais aussi, que cette substitution peut engendrer un pont salin
supplémentaire avec notamment K146. Globalement, on constate une
rigidification de la
région en question qui pourrait expliquer le gain en thermostabilité constaté.
Q292 est
également un résidu potentiellement accessible au solvant, localisé dans une
structure
10 secondaire de type boucle . La substitution par un résidu proline est
une approche
d'ingénierie relativement classique dans ce type de structures secondaires
faiblement
conservées. Ce type de résidu génère peu de rotamères et va, par conséquent,
rigidifier la
structure secondaire dans laquelle il se situe et ainsi conférer dans certains
cas, un gain de
thennostabilité que pourrait donc engendrer la substitution Q292P.
Construction de mutants possédant des sites supplémentaires de glycosvlation
Certaines modifications post-traductionnelle.s comme les glycosylations sont
décrites
comme stabilisant les protéines. Les signaux de N-glycosylations dans une
séquence
protéique sont NxT ou NxS. De tels sites ont été introduits dans la phytase de
Yersinia
intermedia clonée dans le vecteur pYES2, soit par la technique propriétaire de
Biométhodes Massive Mutagenesis comme mentionné ci-dessus ou par une
technique de
mutagenèse bien connue de l'homme du métier, telle qu'une PCR par
recouvrement, en
introduisant un résidu T à la position +2 par rapport à un résidu N présent
dans la phytase
de Yersinia intermedia ou en introduisant un résidu N à la position ¨2 par
rapport à un
résidu S ou T. Les positions sur lesquelles des sites de glycosylation ont été
introduits sont
classées en fonction de leur pourcentage d'accessibilité aux solvants (% ASA)
par
utilisation du logiciel disponible â: http://rnobylexpbs.univ-paris-
diderot.fr/cgi-
bin/portal.py?from=ASA (Richmond, TJ. Suivent accessible surface area and
excluded
volume in proteins. J. Mol. Biol..178. 63-89 (1984). Préférentiellement, 22
substitutions
sur des résidus ayant un pourcentage d'accessibilité aux solvants > 35 % ont
été
sélectionnées. Encore plus préférentiellement, 10 substitutions sur des
résidus ayant un
pourcentage d'accessibilité aux solvants > 70 % ont été sélectionnées. Ces
différents
variants construits permettant l'ajout de sites de glycosylations sont classés
dans le tableau
2 en fonction de leurs pourcentages d'accessibilité aux solvants respectifs.
CA 02766704 2011-12-23
WO 2011/000933 PCT/EP201(1/(159413
31
Les constructions mutantes ont été transformées dans Saccharomyces cerevisiae
et
caractérisées plus en détail au niveau de leur activité et de leur
thermostabilité. L'exemple
détaille les protocoles de caractérisation de l'activité résiduelle des
mutants en fonction
de la température. Une première série d'expériences a permis d'identifier
plusieurs mutants
5 avec un site supplémentaire de glycosylation possédant une
thermostabilité augmentée par
rapport à l'enzyme sauvage. Ces mutants contiennent des substitutions sur les
acides
aminés T142. A177 et Q326 caractérisés par un pourcentage d'accessibilité aux
solvants>
70%. Plus particulièrement, les substitutions sur ces acides aminés sont T1
42N, A177T et
Q326T. Ces mutants présentent un coefficient d'extinction d'activité entre 80
% et 20 %
d'activité résiduelle respectivement de 1,62, 1,62 et 1.52 tels que présentés
dans le tableau
4C. Ces indices sont calculés par le rapport entre les différences de
températures
permettant d'une part le maintien de 80% de l'activité résiduelle et d'autre
part de 20% de
cette activité résiduelle pour les différents variants comparativement à
l'enzyme sauvage.
Le détail du calcul de cet indice est montré pour le mutant K210S dans le
tableau 413.
Construction de mutants possédant des ponts disulfure supplémentaires :
Plusieurs couples de résidus ont été identifiés pour leurs distance et
orientation compatibles
avec la formation d'un pont disulfure et remplacés par des résidus cystéine.
L'identification de ces couples a été faite par visualisation de structures
modèles ou
homologues de phytases sous format pdb en utilisant un programme type Swisspdb
Viewer
(GlaxoSmithKline) et en tenant compte des distances optimales entre résidus et
orientation
des chaînes latérales de ceux-ci. Par cette approche. 12 paires de résidus
situés à environ 2
AngstrZims ont été sélectionnées et sont listées dans le tableau 3:
Les résidus précédents ont été introduits dans la phytase de Yersinia
interrneclia clonée
dans le vecteur pYES2, soit par la technique propriétaire de Biométhodes
Massive
Mutagenesis comme mentionné ci-dessus ou par une technique de mutagenèse bien
connue de l'homme du métier, telle qu'une PCR par recouvrement. Les
constructions
mutantes ont été transformées dans Saccharomyces cerevisiae et caractérisées
plus en
détail au niveau de leur activité et de leur thermostabilité. L'exemple 5
détaille les
protocoles de caractérisation de l'activité résiduelle des mutants en fonction
de la
température. Une première série d'expériences a permis d'identifier un mutant
possédant
un pont disulfure supplémentaire et une thermostabilité améliorée. Ce mutant
contient deux
substitutions sur les résidus G274 et N316. plus particulièrement les
substitutions G274C
et N316C. Ce mutant présente un coefficient d'extinction d'activité entre 80 %
et 20 %
CA 02766704 2011-12-23
WO 2011/000933 PCT/EP201(1/(159413
3,
d'activité résiduelle de 2,33 tel que présenté dans le tableau 4D. Cet indice
est calculé par
le rapport entre les différences de températures permettant d'une part le
maintien de 80%
de l'activité résiduelle et d'autre part de 20% de cette activité résiduelle
pour le variant
SS11 comparativement à l'enzyme sauvage. Le détail du calcul de cet indice est
montré
pour le mutant K210S dans le tableau 413.
Construction de mutants ciblant une amélioration d'activité :
Plusieurs résidus ont été ciblés pour identifier des mutants présentant une
activité
améliorée. L'identification de ces résidus a été faite par visualisation de
structures modèles
ou homologues de phytases sous format pdb en utilisant un programme type
Swisspdb
Viewer (GlaxoSmithKline). Le critère de sélection choisi est la situation des
résidus à une
distance inférieure ou égale à 10 Angstr6ms autour du site catalytique de
l'enzyme. Par
cette approche 76 positions ont été sélectionnées et sont listées dans le
tableau 5A. Une
diversité totale, par l'utilisation d'oligonucléotides NNS a été introduite au
niveau de ces
positions dans la phytase de Yersinia intermedia clonée dans le vecteur pYES2,
soit par la
technique propriétaire de Biométhodes Massive Mutagenesie comme mentionné ci-
dessus ou par une technique de mutagenèse bien connue de l'homme du métier,
telle
qu'une PCR par recouvrement. Ainsi, pour chacune des positions listées dans le
tableau
5A. les mutants comportant individuellement l'une des 19 substitutions
possibles parmi la
liste des 20 acides aminés existants ont été construits ; selon le code à une
lettre
communément adopté, ces 20 acides aminés sont : A, R, N, D, C, E, Q. G. H, I.
L, K, M, F,
P, S, T, W. Y et V. Les constructions mutantes sont transfiartnées dans
Saccharomyces
cerevisiae et caractérisées plus en détail au niveau de leur activité et de
leur thermostabilité
respectives. L'exemple 5 détaille les protocoles de caractérisation de
l'activité résiduelle
des mutants en fonction de la température. Une première série d'expériences a
permis de
caractériser 14 mutants contenant les substitutions listées dans le tableau
5B.
Combinaison des améliorations :
Les différentes approches utilisées permettent de cumuler les gains de
thermostabilité et/ou
d'activité et/ou de résistance à la protéolyse obtenues par les criblages et
caractérisations
des différents mutants dans chacune des approches THRTm, ajout de sites de
glycosylation,
ajout de ponts disulfures et ciblage de l'activité. Une première série
d'expériences a permis
de construire plusieurs mutants combinant les améliorations de thermostabilité
obtenues
par criblage des banques selon THRTm, l'ajout de nouveaux sites de
glycosylation et l'ajout
CA 02766704 2011-12-23
WO 2011/000933 PCT/EP201(1/(159413
33
de ponts disulfures. Ces mutant ont été construits par la technique
propriétaire de
Biométhodes Massive Mutagenesis comme mentionné ci-dessus. Plusieurs mutants
ont
été construits comprenant les combinaisons de substitutions G274C + N316C,
T142N +
A177T + Q326T, K210S + Y268E + Q292P, DI4OF + Y268E + Q292P, FI67N + Y268E
+ Q292P, T142N + A177T + K210S + Q326T, T142N + A177T + K210S + Y268E +
Q292P + Q326T, T142N + A177T + K210S + Y268E + Q292P + Q326T + G274C +
N316C. Les constructions mutantes ont été transformées dans Saccharomyces
cerevisiae et
caractérisées plus en détail au niveau de leur activité et de leur
thermostabilité. L'exemple
5 détaille les protocoles de caractérisation de l'activité résiduelle des
mutants en fonction
de la température. Les figures 3 à 7 présentent les résultats des
caractérisations de certains
de ces mutants en particulier les mutants PHY-98-4X, PHY-98-6X et PHY-98-6X-SS
Il
contenant respectivement les combinaisons de mutations T142N + A177T + K210S +
Q326T, T142N + A177T + K210S + Y268E + Q292P + Q326T et T142N + A177T +
K210S + Y268E + Q292P + Q326T + G274C + N316C, les positions étant indiquées
dans
SEQ ID N 1. Trois souches bactériennes contenant respectivement l'enzyme
d'origine
PHY-98 et les mutants PHY-98-6X et PH Y-98-6X-SS11 mentionnés ci-dessus,
clonés
dans le vecteur plastnidique pYES2 comme décrit par ailleurs, ont fait l'objet
d'un dépôt
de matériel biologique sous les références respectives CNCM 1-4172, CNCM 1-
4173 et
CNCM 1-4174, le 24 juin 2009. Ces différents mutants peuvent servir de base
pour ajouter
une ou des substitutions supplémentaires provenant des différentes approches
de sélection
et montrant une/des améliorations d'activité/therrnostabilité afin de créer de
nouvelles
combinaisons de mutations cumulant les gains d'améliorations ou montrant des
synergies
dans les améliorations dues à ces nouvelles combinaisons de mutations.
Exemple 2: Expression des variants améliorés de la phytase de Yersinia
imermedia
dans Saccharomyces cerevisiae
La plasmide pYES2 contenant le variant amélioré de la phytase de Yersinia
haermedia
comme décrit précédemment a été transformé par électroporation dans la souche
de levure
Saccharomyces cerevisiae APho4 et les transformants ont été sélectionnés sur
milieu solide
SD-U. Plusieurs clones ont été mis à pousser en préculture en milieu SD-U
liquide pendant
une nuit à 30 C sous agitation. Ces précultures ont permis d'ensemencer autant
de culture
dans le milieu de production YP Galactose 2%. Les productions ont été
réalisées sur une
nuit à 30 C sous agitation.
CA 02766704 2011-12-23
WO 2011/000933 PCT/EP201(1/(159413
34
Exemple 3 : Expression des variants améliorés de la phytase de Yersinia
intermedia
dans Pichia Pastoris
Le plasrnide pP1C9 contenant le variant amélioré de la phytase de Versifia
intermedia
comme décrit précédemment a été transformé dans des cellules de Pichia
pastoris rendues
compétentes. Après sélection des colonies contenant le plasmide, une colonie a
été mise à
pousser pendant 16 heures à 28 C sous agitation dans 50 ml de milieu BMG pour
former
une préculture. La production des variants est contrôlée par des temps
d'induction
différents au méthanol 0,5% selon la quantité désirée dudit variant. Avant
induction, la
Densité Optique (DO) des précultures a été mesurée à 600nrn : pour
l'induction, des DO
optimales de 2 à 6 uDO sont exigées. Les précultures ont alors été
centrifugées et
resuspendues dans un milieu BMM contenant 0,5% de méthanol à une DO de
démarrage
comprise entre 1 et 30 uDO/m1 selon les temps d'induction envisagés : luDO/m1
pour une
induction de 96 heures, 6 uDO/m1 pour une induction de 72 heures et 30 uDO/m1
pour une
induction de 48 heures. Du méthanol 100% est ajouté toutes les 24 heures à une
concentration finale de 0,5%. Ces procédures de production ont été utilisées
pour des
productions en Erlenmeyer adaptées à des volumes allant de 10 ml à 200 ml.
Pour des volumes supérieurs, les productions ont été réalisées en bioréacteur
de 5 à 50 L
adapté à des volumes de production allant de 2 à 20 L. Le type de bioréacteur
utilisé
(Applikon) permet un contrôle automatisé de la pousse des levures par mesures
régulières
de la Densité Optique à 600 nm et de la pression en Oxygène (p02) optimisant
le maintien
des conditions d'induction par méthanol. La procédure utilisée a été adaptée
du protocole
de fermentation en Pichia pastoris d'Invitrogen Pichia expression kit; a
manual of
methods for expression of recombinant proteins in Pichia pastoris ¨ Version
M du 11
janvier 2002). La figure 8 présente un Gel SDS-PAGE 12% de surnageants de
production
pour différents mutants exprimés par Pichia pastoris, digérés ou non par
l'endoglycosidase
lif (New England Biolabs P0703). Les pistes du gel notées Phy 98, Phy 98 6x et
Phy 98 6x
SS11 correspondent à 10 G de surnageants de production des molécules PHY-98,
PHY-
98-6X et PHY-98-6X-SS11 décrits dans l'exemple I. Le mutant Phy 98-6X-SS5 est
un
mutant contenant les substitutions L52C + 1.99C 4- TI 42N + Al 77T F K21OS +
Y268E +
Q292P + Q326T. Les pistes Phy98 montrent le profil de migration de la phytase
sauvage
de Yersinia intermedia, non glycosylée puisque ne possédant pas de sites
potentiels de N-
glycosylations NxS/T ceci est illustré par l'absence de différence de
migration après
CA 02766704 2011-12-23
WO 2011/000933 PCT/EP201(1/(159413
digestion par l'endoglycosidase Hf. Les pistes Phy 98 6x. Phy 98 6x SS5 et Phy
98 6x
SS11, en absence de digestion par l'endoglycosidase Hf, montrent les profils
de migration
des phytases mutantes présentant des masses moléculaires supérieures dues à la
glycosylation respective des mutants. Cette glycosylation est montrée, après
digestion par
5 l'endoglycosidase Hf (pistes notées digéré Endo Hf ), par un retour du
profil de
migration des différents mutants vers celui de la phytase sauvage, même si
dans certains
cas la digestion par l'endoglycosidase peut être incomplète.
Exemple : Mesure de l'activité des variants améliorés de la phytase de
Yersinia
10 interinedia
L'activité des variants de la phytase de Yersinia intermedia ainsi que
l'activité de la
protéine d'origine prise comme contrôle, a été mesurée à l'aide d'un test
colorimétrique
mesurant le phosphate libéré en présence d'une solution de phytate prise comme
substrat.
15 Brièvement, 40 pi de surnageant de l'échantillon à tester (dilué ou non
en tampon acétate
de sodium) ou 40 gl d'une solution étalon d'une gamme de phosphate ont été
mélangés à
Ill de phytate (20 WB. La réaction a été incubée classiquement 15 minutes à 37
C. La
réaction a été stoppée par 80 gl de NaOH à 0,5M ou TCA 20%. 60 gl du volume
total de la
réaction de 160 MI ont été transférés pour la révélation avec 60 gl d'une
solution Fe-Mo.
20 La réaction de révélation a été laissée pendant 15 minutes à l'abri de
la lumière avant
d'être lu dans un spectrophotomètre à 620 nm. Toutes les solutions de la
réaction ont été
réalisées avec de l'eau ppi sans phosphate. Le tampon réactionnel est un
tampon acétate de
sodium 0,25 M pH 4,5. Le substrat utilisé est une solution de phytate réalisée
dans le
tampon réactionnel précédent à partir d'une solution stock à 200g/1. La
solution de
25 révélation a été formée extemporanément par 4 volumes de solution de Mo
mélangés à I
volume de solution de Fe. La solution de Mo est une solution de Molybdate à
0,0I2M et la
solution de Fe est une solution de fer il à 0,38M.
Pour le calcul de l'activité une gamme étalon de phosphate a été réalisée à
partir d'une
gamme de KH2PO4 allant de 0 à 10 ttmol/ml. En utilisant les conditions
réactionnelles
30 décrites ci-dessus, l'activité phytase des échantillons à tester a été
calculée en appliquant la
formule suivante : Activité phytase en IJ/m1 = [(DO 620 mn) x (facteur de
dilution)] /
[(pente gamme étalon) x (temps de réaction en minutes)].
Le calcul des concentrations en protéines a été déterminé selon la méthode
classique de
Bradford bien connu de l'homme du métier.
CA 02766704 2011-12-23
WO 2011/000933 PCT/EP201(1/(159413
36
La figure 7 montre des niveaux d'activités relatifs en fonction du temps (0 à
15 minutes)
supérieurs pour les mutants PHY-98-4X et PHY-98-6X comparativement à l'enzyme
d'origine PHY-98 dans Saccharomyces cerevisiae.
Exemple 5 : Caractérisation de la thermostabilité des variants améliorés de la
phytase
de Yervinia intermedia
La thermostabilité des variants isolés de la phytase de Yersinia intermedia a
été déterminée
en mesurant une activité résiduelle des différents surnageants de production
des variants
soit après un préchauffage à une température constante pendant des temps
variables, soit
après un temps fixe de préchauffage à des températures variables. Dans le
premier cas, les
variants sont préchauffés à 80 C pendant des temps allant de 0 à 30 minutes.
Dans le
second cas, deux types d'activité résiduelle des variants ont été mesurés,
soit après un
préchauffage de 15 minutes à des températures allant de 45 C à 65 C, soit
après un
préchauffage de 1 minute à des températures allant de 60 C à 80 C. Les
activités
résiduelles ont été déterminées dans le premier cas en % de l'activité du même
échantillon
sans préchauffage ou dans le second cas en % de l'activité du même échantillon
préchauffé à la première température mesurée, les deux activités références
étant
considérées comme les points 100%. L'activité résiduelle est montrée en
valeurs brutes de
Densité Optique à 600 nm après un préchauffage de 15 minutes dans la figure 4.
Les figures 3, 4 et 5 présentent des résultats obtenus pour les varianis PlY-
98-4X et PHY-
98-6X exprimés par Saccharomyces cerevisiae par rapport à l'enzyme d'origine
PUY-98.
La figure 3 montre les activités résiduelles supérieures des deux mutants PHY-
98-4X et
PHY-98-6X après un préchauffage de 0 à 2 minutes à 80 C par rapport à l'enzyme
d'origine PHY-98. La figure 4 montre les activités résiduelles supérieures des
deux
mutants PHY-98-4X et PHY-98-6X après 15 minutes de préchauffage à des
températures
variables allant de 45 C à 65 C. La figure 5 montre les activités résiduelles
supérieures
des deux mutants PH.Y-98-4X et PUY-98-6X au delà de 60 C en utilisant 1 minute
de
préchauffage.
Les figures 6a) et 6b) montrent les activités résiduelles supérieures des
mutants PHY-98-
6X et PHY-98-6X-SS11, exprimés par Pichia pastoris, après des temps de
préchauffage à
80 C de respectivement 0 à 30 minutes et 0 à 5 minutes.
CA 02766704 2011-12-23
WO 2011/000933 PCT/EP201(1/(159413
37
Exemple 6: llermostabilité des mutants dans des essais de granulation
Des tests de granulation peuvent être menés pour déterminer la thermostabilité
des
différents mutants par rapport à l'enzyme sauvage et des enzymes existantes
et/ou
commerciales. Les différentes phytases peuvent être incorporées dans les
procédés de
formation et de formulation de granulés destinés à être ajoutés à
l'alimentation animale,
par exemple.
Ces granulés peuvent être formés en mixant/pétrissant dans les mêmes
conditions des
surnageants de production des mutants et enzymes référentes devant être
comparées, avec,
par exemple, une matrice composée d'amidon de maïs et d'eau. La matrice de
granulation
peut contenir des pourcentages relatifs phytase/maïs/eau différents.
Classiquement, après
pétrissage, la matrice peut être extrudée avec une extrudeuse semblable au
type NICATM E-
220 et directement sphéronisée à partir d'un sphéroniseur NICATm ou de type
Fuji
Paudatrm QJ-400G. Les particules obtenues sont ensuite séchées sur un sécheur
à lit
fluidisé du type Glatt GPCG 1.1. L'activité phytase dans les granulés est
généralement
comprise entre 2500 et 3000 FTU/g.
Les granulés formés peuvent être mélangés à la nourriture. Selon l'importance
en volume
des tests, la quantité de nourriture formée peut être variable. Par exemple,
250 g de
granulés peuvent être mélangés avec 25 kg de nourriture pour former un
prémélange. Ce
prémélange peut être incorporé juste avant le test à 225 kg de nourriture, par
exemple, de la
même composition. De façon non limitative, une nourriture pour volaille
typique peut être
composée en pourcentage de 45 à 50% de maïs, 0 à 5% de pois, 0 à 4,5% de
farine de
colza, 0 à 4,5% de farine de graines de tournesol, 0 à 2,5% de gluten de fleur
de maïs, 6 à
10% de fèves de soja entières, environ 25% de tourteau de soja, environ 4% de
tapioca, 1
à 3,5% d'huile de soja, 0 à 4% de graisse animale, 0,5 à 1% d'un cocktail de
vitamines
(Mervit 100), environ 1% de calcaire en poudre, 0,2 à 1,3% de phosphate
monocalcique.
0,1 à 0,4% de sels, 0 à 0,3% de bicarbonate de sodium (Na1.1093), 0,05 à 0,3%
de L-
Lysine, 0,15 à 0,25% de DL-Méthionine et 0 à 0,05% de L-Thréonine. Le
prémélange de
l'ordre de 25 kg est typiquement mélangeable dans un mélangeur planétaire type
Collete
MF90 pendant 10 minutes. Le mélange de l'ordre de 225 kg peut être mélangé
dans un
mélangeur du type Nauta 1200 Litres. Des échantillons de ce mélange sont
prélevés à ce
stade pour déterminer l'activité et la stabilité des mutants et des phytases
de référence
avant la formation des granulés finaux. Le mélange de l'ordre de 250 kg est
typiquement
dosé dans un mélangeur/conditionneur par une vis de dosage à une vitesse
d'environ 600
CA 02766704 2011-12-23
WO 2011/000933 PCT/EP201(1/(159413
.38
kg /h, où il est chauffé par injection de vapeur jusqu'à environ 95 C. Le
temps de résidence
total est d'environ 10 à 30 secondes après quoi le mélange chaud est dirigé
vers une presse
à granulation. Pour les tests, les tailles de granulés pouvant être fabriqués
sont du type 5/45
mm (largeur/longueur) ou 3/65 mm. La température des granulés à la sortie de
la presse est
typiquement de 82 à 83 C pour le premier type de granulés et 91 à 93 C pour le
second
type. A la suite de cette étape, les granulés sont refroidis sur un tapis de
refroidissement où
des échantillons sont récupérés pour déterminer l'activité et la stabilité des
mutants et des
phytases de référence après la formation des granulés finaux. Des rendements
de
granulation en terme d'activité peuvent ainsi être obtenus pour chaque mutant,
comparativement aux enzymes de référence, en faisant les rapports d'activité
après et avant
l'étape de granulation. Un protocole de mesure de l'activité phytase est donné
dans
l'exemple 4. Egalement, un protocole standard de mesure de l'activité phytase
adapté à ces
procédés est publié sous la référence : van.Engelen et al.. Journal of AOAC
International
1994, 77 :760-764.
Exemple 7: Tests dans des essais d'alimentation animale
Plusieurs approches peuvent être utilisées pour mesurer l'efficacité des
mutants de
l'invention à libérer le phosphate du phytate in vivo afin de contribuer à la
croissance des
animaux, comparativement à des phytases référentes.
Différents animaux tels que des porcs peuvent être intégrés dans des
protocoles bien
établis. Ceux-ci ont un accès libre à de l'eau et à un régime typique
constitué par exemple
de 674 de maïs, 28% de farine de soja. 1% de calcaire en poudre. 0,1% L-
Lysine, 1%
d'huile de maïs, 0,25% de cocktail vitaminé classiquement utilisé, 0,5% de
sels, 0,5%
d'antibiotiques. Les déchets de nourriture sont collectés quotidiennement. La
prise de
poids des animaux est mesurée hebdomadairement pour calculer le gain moyen par
jour, la
prise de nourriture moyenne quotidienne et le rapport gain/prise. Les mutants
présentant un
intérêt particulier et les meilleurs performances sont typiquement ceux qui
présentent un
rapport gain/prise accru.
Egalement des modèles in vitro existent qui permettent de simuler la digestion
dans le
tractus d'un animal monogastrique. Par exemple des échantillons de nourriture
composés
de 30% de farine de soja et 70% de farine de maïs peuvent être supplémentés
avec du
calcium de phosphate à un taux de 5 Wkg de nourriture et préincubés à 40 C, pH
3 pendant
30 minutes, suivi d'une addition de pepsine à 3000 U/g de nourriture et
différents dosages
CA 02766704 2011-12-23
WO 2011/000933 PCT/EP201(1/(159413
39
de phytase compris entre 0 (blanc témoin) et 1 U phytase/g de nourriture.
Différents
mutants de phytase peuvent être testés comparativement à des phytases
référentes. Les
différents échantillons sont incubés à 40 C, d'abord à pH 3 pendant 60 minutes
puis à pH 4
pendant 30 minutes. Les réactions sont alors stoppées et le phytate et les
inositol phosphate
sont extraits par addition d'acide chlorhydrique à une concentration finale de
0,5M, une
incubation pendant 2 heures à 40 C, suivi d'un cycle de congélation-
décongélation et
d'une heure d'incubation à 40 C.
Le phytate et les inositol phosphates sont séparés par chromatographie
échangeuse d'ions à
haute performance tel que décrit dans Chen. Q.C. and Li, B. W. (2003) et
journal of
Chromatography A 1018, 41-52 ainsi que dans Skoglund. E., Carlson. N.G. and
Sandberg,
A.S. (1997) et J. Agric. Food Chem. 45, 431-436. Le phosphate relargué est
calculé
comme la différence entre k phosphate lié aux inositol phosphates dans des
échantillons
traités avec une phytase comparativement à des échantillons non traités avec
une phytase.
Les mutants intéressants relarguent une plus grande quantité de phosphate.
Dépôt de matériel biologique :
Trois souches bactériennes contenant les constructions PHY-98, PHY-98-6X et
PHY-98-
6X-SSI 1 utilisées dans les exemples précédents ont été déposées et acceptées
par la
Collection Nationale de Cultures de Microorganismes (CNCM) de l'Institut
Pasteur, 25,
Rue du Docteur Roux, 75724 Paris Cedex France, selon les termes du Traité de
Budapest :
Références d'identification Dates de dépôt Numéros d'enregistrement des
des souches déposées matériels biologiques reçus
PHY-98 24 juin 2009 CNCM 1-4172
PHY-98-6X 24 juin 2009 CNCM 1-4173
PHY-98-6X-SS11 24 juin 2009 CNCM 1-4174
CA 02766704 2011-12-23
WO 2011/000933
PCT/EP201(1/(159413
Littérature citée :
Cosgrove Di (1970) Inositol phosphate phosphatases of microbiological origin.
Inositol
phosphate intermediates in the dephosphorylation of the hexaphosphates of myo-
inositol,
scyllo-inositol, and D-chiro-inositol by a bacterial (Pseudomonas sp.)
phytase. Australian
5 Journal of Biological Sciences 23:1207-1220
Cullen et al. (1987) Sequence and eentromere proximal location of a
transformation
enhancing fragment ansl from Aspergillus nidulans. Nucleic Acids Research 15 :
9163-75
10 Dassa J. et al. (1990) The complete nucleotide sequence of the
Escherichia cou i gene appA
reveals significant homology between pH 2.5 acid phosphatase and glucose-1 -
phosphatase.
J. Bacteriol. 1'72:5497-5500
Gerns et al. (1991) An autonomously replicating plasmid transfonns Aspergillus
nidulans
15 at high frequency. Gefle 98 :61-67
Greiner et al. Purification and characterization of two phytases from E. Coli.
Ardt.
Biochem. Biophys., 303, 107-113, 1993
20 Jansen J.C. and Ryden L (1989) Protein Purification, VCH Publishers, New
York
Karhunen T, Mântylâ A, Nevalainen KMH, Suominen PL (1993) High frequency one-
step
gene replacement inTrichoderma reesei. I. Endoglucanase I overproduction. Mol
Gen
Genet 241: 515-522
Laemmli UK (1970) Cleavage of structural protein during the assembly of the
head of
bactedophage T4. Nature 227: 680-685
Lim et al., 2000. Crystal structures of Escherichia cou i phytase and its
complex with
phytate. Nat. Struet. Biol. 7: 108-113
Needleman et Wunsch (1970) A general method applicable to the seareh for
similarities in
the amino acid sequence of two proteins. J. Mol. Biol. 48 : 443
CA 02766704 2011-12-23
WO 2011/000933 PCT/EP201(1/(159413
41
Oshima et al. (1996) A 718-kb DNA sequence of the Escherichia cou i K-12
genome
corresponding to the 12.7-28.0 min region on the linkage map. DNA Research,
3:137-155
Pearson et Lipman (1988) Improved tools for biological sequence comparison.
Proc. Natl.
Acad. Sci. USA 85 (8) : 2444-8
Penttilâ M, Nevelainen 11, RJttô M, Salminen E, Knowles J (1987) A versatile
transformation system for the celltdolytic filamentous fungus Trichoderma
reesei. Gene
61: 155-164
Powar VK and Jagannathan V (1982) Purification and properties of phytate-
specific
phosphatase from Bacillus subtilis. Journal of Bacteriology 151:1102-1108
Raeder U, Broda P (1985) Rapid preparation of DNA from iilanientous fungi.
Lett Appl
Microbiol 1: 17-20
Richmond, u (1984) Solvent accessible surface area and excluded volume in
proteins. J.
Mol. Biol.,178, 63-89
Rodriguez et al. (2000) Site-directed mutagenesis improves catalytic
efficiency and
tbermostability of Escherichia cou i pH 2.5 acid phosphatase/phytase expressed
in Pichia
pastoris. Arch. Biochem. Biophys., 382:105-112
Saboulard et al (2005) High-throughput site-directed mutagenesis using
oligonucleotides
synthesized on DNA chips. Biotechniques Sep 39(3) : 363-8)
Smith et Waterman (1981) Ad. App. Math. 2 : 482
Touati and Darichin (1987) The structure of the promoter and amino terminal
region of the
pli 2.5 acid phosphatase structural gene (appA) of E. cou: a negative control
of
transcription rnediated by cyclic AMP. Biochimie, 69:215-221
CA 02766704 2011-12-23
WO 2011/000933
PCT/EP2010/059413
42
Wyss et al (1999) Biochemical Characterization of Fungal Phytases. Appl
Environ
Microbiol 65 (2) 367-373
FEUILLE DE REMPLACEMENT (REGLE 26)
CA 02766704 2011-12-23
WO 2011/000933
PCT/EP2010/059413
43
TABLEAU 1
P3L P3V V4G A5P P8N P8V
T91 T9Q T9S T9Y G10A G1OP
V16M V17W L19G S20C R21F H22A
H22S H22Y G23S R25C P27F T28N
T28S T28V Q3OR Q32R L33R T41G
G5OD G50E V601 Y67F Y67W G75R
A78P D92R D94G D94S Q95V R96A
T97N V125N D126Q H130N H130Q H13OR
H 130W P131S V132W D133G D133P D133R
D133V D133W D140E D140F L152N L152P
L156N F167N C182N F197V K206A T209C
K210C K210E K210S K210T K210V K210Y
V211C V211G L217N L223S E225D F227S
L229H Q230T N231K A234K P236N S251N
L252M H256A H256E H256P N257I ' Q259K
Q259Y D261F A264P Y268N Y268E K273N
G274S P276L Q292P P297S Q301L H31OR
D311A D311E D311G T312D T312N T312P
T312V N313F N313R 1314E 1314M A323E
P331R D332A D332L D332Q N333V P335C
P335G P335R P335S V341A V341E E343A
E343G D349N R353C Y354N V355W A370D -
A370T K376S P379L G381S S393G P414Q
FEUILLE DE REMPLACEMENT (REGLE 26)
CA 02766704 2011-12-23
WO 2011/000933 PCT/EP201(1/(159413
44
TABLEAU 2
_
< 35% ASA < 35% AS,-. < 35% ASA 35<x<70% r''..L;:\ > 70 % ASA
,
Q7N S220N P3355 Y51N K29N -
D36N Q230N P335T C81N T142N
,
P39N Q2335 Q352S Q95N A177N _
R98N . Q233T . Q352T D140N A177S
Q143N + 1-114512 R242N E3715 - P155N A177T
E158N Q2595 E371T G189N K201N
E158N + 5160T Q259T P379S K210N G293N
-
P207N A264N P379T i2505 0326S
: 7S F,273N A380S 1250T W26T
_
P207T PT.:Wtii-1 ' A380T P297N E391N '
S212N 14310N G3995 , D349S
A218N D332N G394T D349T ,
_
CA 02766704 2011-12-23
WO 2011/000933
PCT/EP2010/059413
TABLEAU 3
SEFt20 - GLY339
VAL24 - GLY56
SER26 - TRP45
GLN30 - ME T34 _
LEU52 - LEU99
T11R53 - GLY56
ALA57 - ALA103
GLY101 - VAL116
ALA147 - TYR268
ASP193 - ASN196
GLY274 - ASN31 6
GLY322 - ASP388
FEUILLE DE REMPLACEMENT (REGLE 26)
CA 02766704 2011-12-23
WO 2011/000933
PCT/EP2010/059413
46
TABLEAU 4
Tableau 4A
Mutants K210S Y268E Q292P
Coefficients d'extinction d'activité 80-20 1,75 1,98 2,3
Tableau 4B
Sauvage K210S
T C 80% 50,77 50,51
T C 20% 53,44 55,17
Delta 80-20 2,67 4,66
R(K210S/Sauv) 1,74531835
Tableau 4C
Mutants T142N A177T Q326T
1¨Coefficients d'extinction d'activité 80-20 1,62 1,62 1,52
Tableau 4D
Mutants G274C/N316C(SS11L
[-Coefficients d'extinction d'activité 80-20 2,33
FEUILLE DE REMPLACEMENT (REGLE 26)
CA 02766704 2011-12-23
WO 2011/000933 PCT/EP2010/059413
47
TABLEAU 5
Tableau SA
L19 S20R21 H22 G23 V24
R25 . S26 - P27 T28 K29 Q30
T31 M34.., A49 G50 Y51 L52
D92 V93 094 095 R96 T97
, _
R98 T100 V125 D126 F129 H130
. ... .,
in 0133 P207 T209 K210 V211 S212 .
L213 L217, A218, L219, S220, S221,
T222 L223 G224 E225 1226 F227
L228 L229 , L253 L.255 H256 N257
Q259 F260 M263 A264 - k273 - L277 '
=
G308 G309 H310 0311 T312 _ N313 ....
1314 A315 N316 0332 N333 T334
P335 P336 G337 G338
Tableau 58
030D Y51G Y510 ,(51W L52G _____ V93G
_
R98T F129µ/V H130Y P 155T L219V L255T
M263L G 308S